基于表型性状构建中国花生地方品种骨干种质
2020-03-23闫彩霞李春娟宋秀霞孙全喜苑翠玲赵小波单世华
闫彩霞 王 娟 张 浩 李春娟 宋秀霞 孙全喜 苑翠玲 赵小波 单世华,*
基于表型性状构建中国花生地方品种骨干种质
闫彩霞1王 娟1张 浩1李春娟1宋秀霞2孙全喜1苑翠玲1赵小波1单世华1,*
1山东省花生研究所, 山东青岛 266100;2菏泽市牡丹区农业农村局, 山东菏泽 274000
中国花生地方品种遗传多样性丰富, 是花生新品种选育的重要亲本来源。本研究以种质库保存的2741份地方种质为材料, 基于种植区划和植物学类型分组, 平方根法确定取样量, 组内按13个表型数据进行UPGMA聚类分析, 类内随机取样, 构建骨干种质。利用检验、测验、卡方测验、极差、表型保留比例、表型相关性等对骨干种质代表性进行检验和评价; 并利用主成分分析和直方图对骨干种质进行确认。结果表明, 构建了包含259份种质的中国花生地方品种骨干种质, 占全部种质的9.4%, 包括多粒型14份、珍珠豆型85份、龙生型42份、普通型103份、中间型15份。在< 0.05概率条件下, 骨干种质13个性状的均值、方差、变异系数、香农指数与全部种质无显著差异, 且保留了全部种质的分布范围、表型保留比例和表型相关性; 二者的植物学类型组成和生态分布是一致的, 具有相似的遗传结构和分布频率。建立的骨干种质很好地代表了全部种质的遗传变异和群体结构, 可为花生种质创新和优异等位基因发掘奠定良好的基础。
花生; 地方品种; 表型性状; 骨干种质; 代表性评价
花生栽培种(L.)起源于南美洲, 隶属于豆科花生属(), 一年生, 异源四倍体(AABB), 推测是由二倍体野生种经过自然杂交和染色体加倍而形成的[1]。自多途径引进后, 在我国多变的气候及复杂的地理环境下形成了遗传多样性较为丰富的种质资源。目前国家中期种质库中的保有量已达7000多份, 居世界第3位[2], 其中地方品种共4638份, 因其变异广泛、适应性好、配合力高及对病害、虫害及逆境等的抗性, 是花生品种改良的重要亲本来源。追溯建国以来育成花生品种的系谱, 有25个地方品种直接或间接参与育成了85%的品种, 其中40%的亲缘来源于伏花生、徐州68-4、狮头企、粤油551、徐州402和粤选58。地方品种成为花生育种的“骨干亲本”[3], 但是, 在育种实践中用作亲本的不足百份, 特别是多粒型种质和龙生型种质有效利用甚少[4]。同时, 花生育种存在着严重的“近亲繁殖”, 导致品种遗传基础狭窄, 适应性、抗病(逆)性减退, 产量上很难取得重要突破, 因此, 拓宽现有花生品种的遗传基础已迫在眉睫。
地方品种具有丰富的多样性, 这为品种选育和遗传研究提供了广阔的遗传基础。然而, 其庞大的资源数量反而阻碍了作物的品种改良和新品种选育。构建核心种质(core collection)或骨干种质(key germplasm), 即以最小的资源份数最大限度地代表整个资源的遗传多样性[5], 无疑是解决这一矛盾的有效策略, 这为地方种质的深入评价、创新利用和基因挖掘开辟了新的途径。迄今为止, 基于表型性状和分子标记数据, 已构建了水稻、小麦、玉米、大豆、棉花、燕麦、谷子、芝麻、糜子、黍稷等[6]多种作物的核心(骨干)种质。核心种质的取样比例、取样策略以及有效性评价等理论研究也取得了进展, 为核心(骨干)种质的构建及代表性评价提供了坚实的理论依据。
作物的表型多样性是遗传多样性的外在表现, 国内外研究机构均利用表型数据构建了花生核心种质, 如美国的831份核心种质[7]和112份微核心种质[8], 国际半干旱地区热带作物研究所(ICRISAT)的1704份核心种质[9]和184份微核心种质[10], 姜慧芳等[2]构建的298份小核心种质, 并且开展了大量的性状评价与抗病(虫或逆)性鉴定[11-17]。本研究以建国以来收集保存的2741份代表性地方品种为材料, 以13个表型性状数据为基础, 构建中国花生地方品种骨干种质, 对其代表性进行评价, 为地方品种的保护与利用、花生种质的创新利用及品种的遗传改良提供理论依据。
1 材料与方法
1.1 材料
2741份有明确地理来源的中国栽培花生地方品种, 其有关数据来自山东省花生研究所编写的《中国花生品种资源目录》及中国农业科学院油料作物研究所编写的《中国花生品种资源目录(续编一、二)》。涉及生育期、百果重、百仁重、出仁率、株高、株型、开花习性、分枝型、植物学类型9个农艺性状和粗蛋白含量、粗脂肪含量、油酸含量、亚油酸含量4个品质性状。质量性状根据其表型进行赋值(表1), 数量性状数据采用0.5个标准差为间距进行标准化, 分为10级(1级≤-2, 10级 >+2, 中间每级间差0.5,为性状平均值,为标准差)。
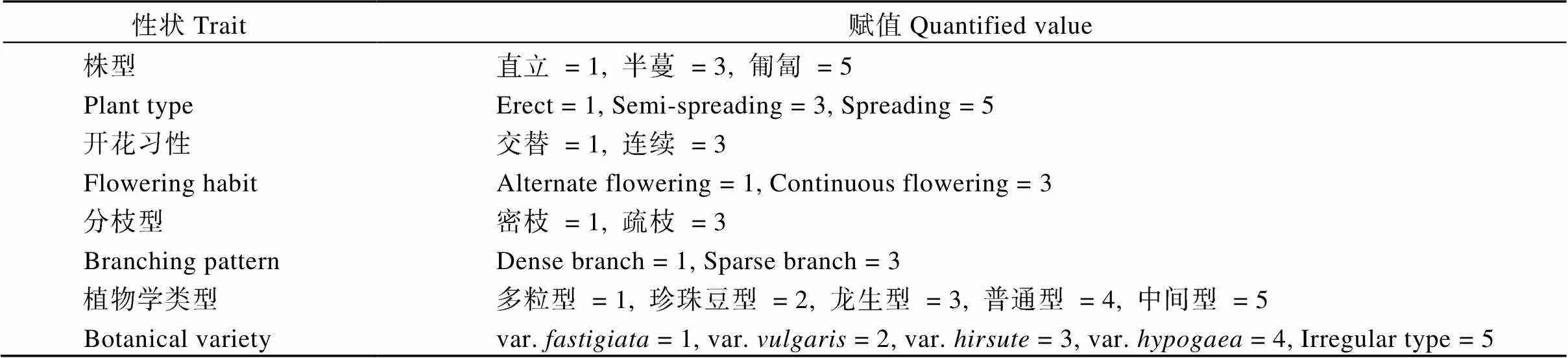
表1 花生质量性状赋值
1.2 分组及取样比例的确定
根据中国栽培花生的七大地理种植区划(黄河流域花生区、长江流域花生区、东南沿海花生区、云贵高原花生区、黄土高原花生区、东北花生区和西北花生区)和五大植物学类型(多粒型、珍珠豆型、龙生型、普通型、中间型)分组。采取常用的平方根法(square root strategy, S法)系统取样, 即分组取样量由整个组内资源份数的平方根值占各组平方根之和的比例来决定。

其中,N为第组的取样数,n为第组的品种数,n为第组的品种数,为总分组数,为总品种数。
1.3 聚类分析
采用SPSS 22.0统计分析软件, 以组为单位采用类平均数法(UPGMA)进行各性状的系统聚类分析, 类内利用Microsoft Excel提供的Randbetween函数随机取样。
1.4 骨干种质的代表性检测
利用SPSS 22.0中的测验、测验分别判断全部种质和骨干种质13个性状的均值、方差、变异系数及Shannon-Weaver多样性指数(′)是否有差异, 卡平方(c2)测验用于检验两者的植物学类型组成、生态分布及13个性状的表型分布频率是否一致。变异系数(CV) = (标准偏差SD/平均值Mean)×100%, 用于比较全部种质和骨干种质的离散程度。

其中,P表示某性状第级的分布频率,为总分级数, 用于评价2个群体的遗传多样性水平。极差分析和表型相关性分析分别用于确定全部种质的变异范围和性状相关性是否在骨干种质中得到了相应保持。表型保留比例(the ratio of phenotypic retention, RPR)用于检测骨干种质中是否保留了全部种质足够的变异。
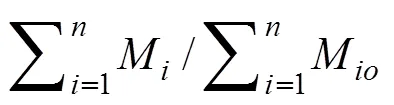
其中,M为全部种质中某性状的表型变异数,M为骨干种质中该性状的表型变异数,为性状总数)。
1.5 骨干种质的确认
利用主成分分析和直方图比较全部种质与骨干种质基于主成分的样品分布图及13个性状的分布频率, 对构建的骨干种质有效性进行确认。
2 结果与分析
2.1 骨干种质的构建、植物学组成及生态分布
全部花生种质被分成了26组, 每组种质从1到623份不等。每组的取样量为1~47份。UPGMA法聚类分析后随机取样, 得到242份种质, 初步评价后补充极值材料和特殊性状种质17份, 提取9.4%的中国花生地方品种构建骨干种质。该骨干种质包含全部种质的植物学类型, 其中多粒型14份(5.4%)、珍珠豆型85份(32.8%)、龙生型42份(16.2%)、普通型103份(39.8%)、中间型15份(5.8%)。卡方测验表明, 骨干种质代表了全部种质的植物学类型组成(c2=1.600,=0.809)(表2)。另外, 骨干种质包含了全部种质的生态分布, 其中黄河流域花生区75份(29.0%)、长江流域花生区75份(29.0%)、东南沿海花生区63份(24.3%)、云贵高原花生区16份(6.2%)、黄土高原花生区13份(5.0%)、东北花生区15份(5.8%)和西北花生区2份(0.8%), 骨干种质代表了全部种质的生态分布(c2=5.232,=0.514), 其中, 西北花生区由于种质较少, 仅有的2份种质被全部取样(表2)。
2.2 全部种质与骨干种质的平均值、方差和极差比较
测验表明, 4个形态性状和9个数量性状的平均值在全部种质与骨干种质中无显著差异, 且骨干种质中植物学类型、百果重和亚油酸含量这3个性状的平均值都大于全部种质的平均值(表3)。测验表明, 除出仁率的方差在全部种质与骨干种质中差异显著外, 其余12个性状的方差均为齐性(表3)。此外, 骨干种质大部分性状的方差高于全部种质, 表明骨干种质遗传冗余度明显减小, 变异率更高。极差分析表明, 全部种质4个形态性状的变异范围100%保留在骨干种质中; 除百仁重(保留范围64.3%)、出仁率(保留范围77.2%)外, 其余7个性状变异范围的86%~100%保留在骨干种质中(表4)。由此可见, 骨干种质对全部种质的性状的变异幅度具有良好的代表性。
2.3 变异系数和Shannon-Weaver多样性指数的比较
Shannon-Weaver多样性指数和变异系数常用来比较不同样品的表型特征、等位基因的丰富度和均匀度。从表4可以看出, 全部种质和骨干种质13个性状的′和变异系数是非常相似的。全部种质的平均′为1.631±0.153, 变异系数为29.009±5.964; 骨干种质的这2个指标分别为1.672±0.154和30.125±5.765, 骨干种质略高于全部种质。成对双样本测验表明, 骨干种质′极显著高于全部种质(=0.002), 变异系数在2个群体中差异不显著(=0.123), 说明骨干种质的样本足够大, 且有效去除了总资源中的冗余, 保留了全部种质的遗传多样性, 变异均匀度显著提高。
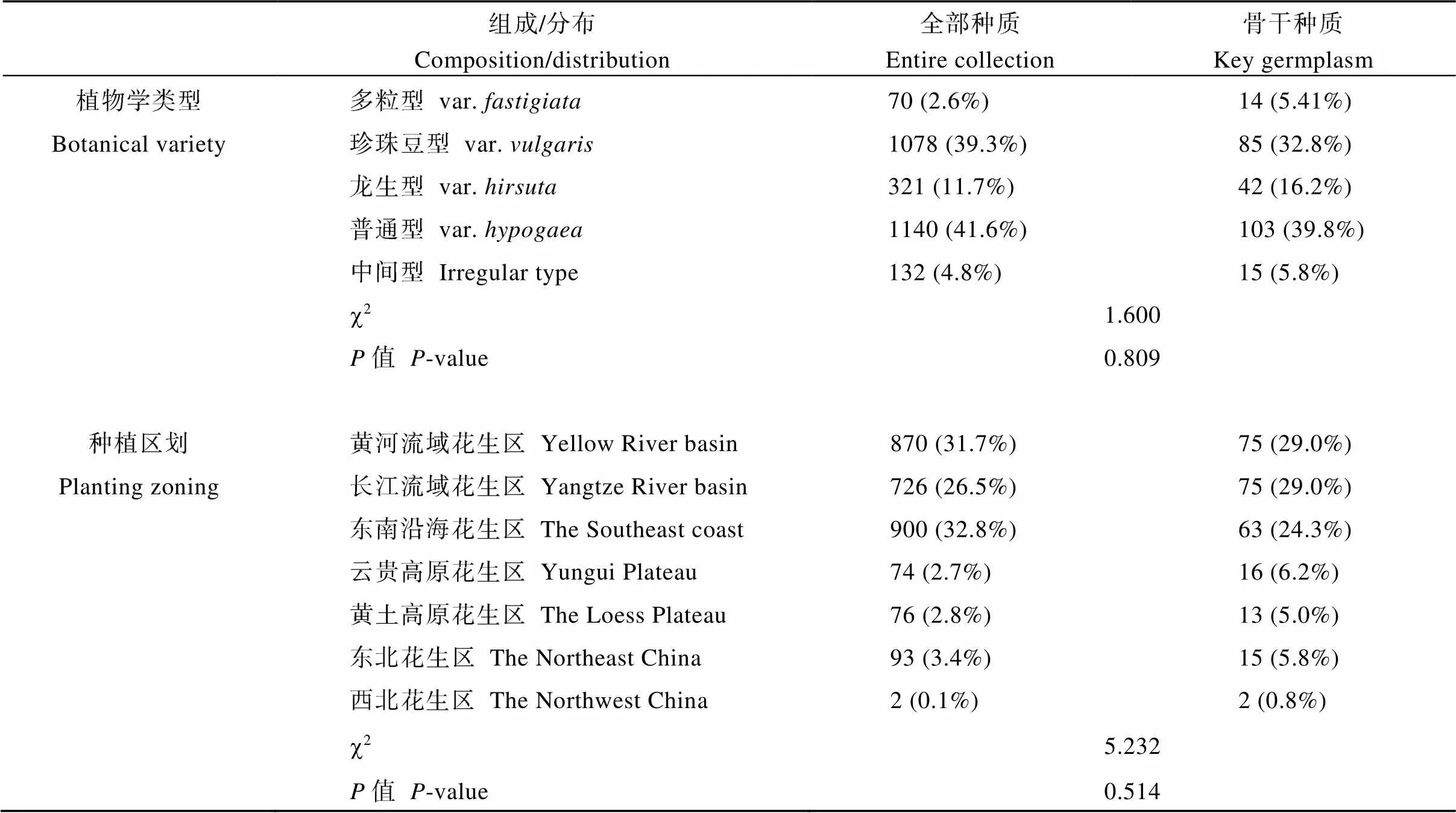
表2 骨干种质和全部种质的植物学类型组成、生态分布及其卡方测验
> 0.05表明差异不显著。> 0.05 means the difference is insignificant.
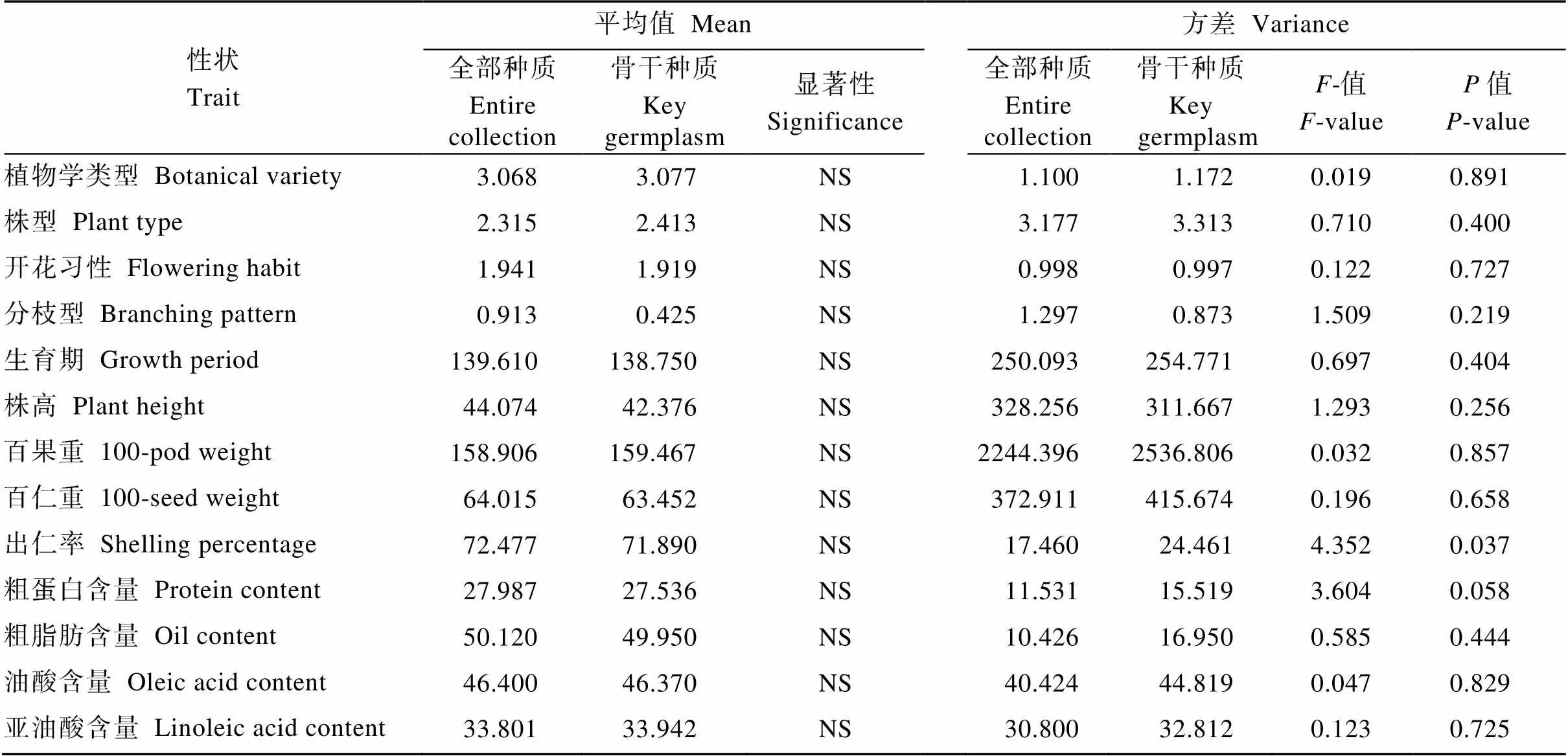
表3 全部种质与骨干种质13个性状平均值和方差的比较
> 0.05表示差异不显著,< 0.05表示差异显著, NS表示差异不显著。
> 0.05 means the difference is insignificant;< 0.05 means the difference is significant; NS: not significant.
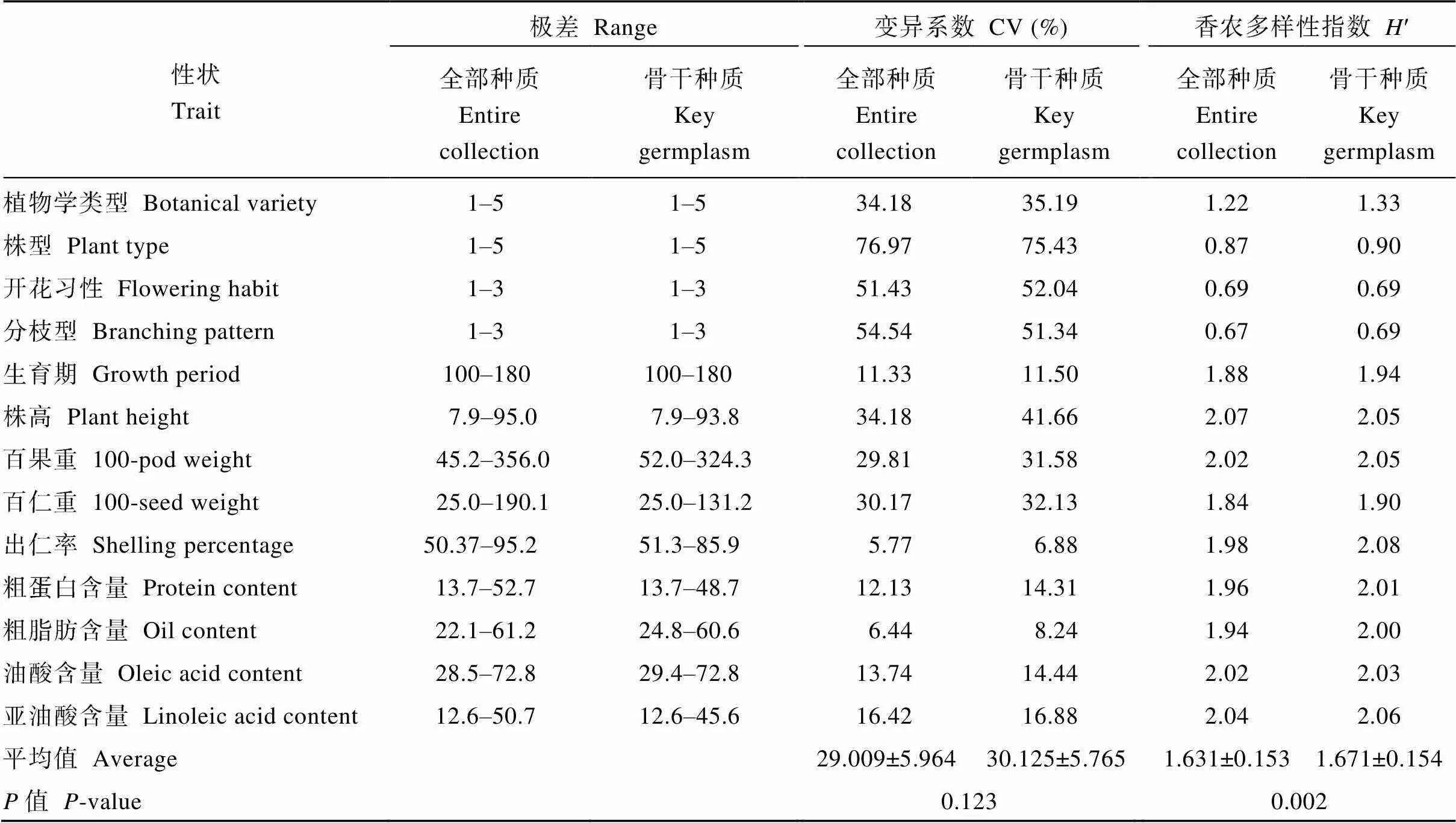
表4 全部种质与骨干种质极差、变异系数和遗传多样性指数的比较
> 0.05表明差异不显著,< 0.01表明差异极显著。
> 0.05 means the difference is insignificant;< 0.01 means the difference is positively significant.
2.4 分布频率和表型保留比例的比较
从表5可以看出, 全部种质13个性状的102个表型分级均包含在骨干种质中。对2个群体13个性状的分布频率进行Chi平方测验, 差异均不显著, 表明2个样本的性状分布是一致的, 骨干种质可代表全部种质的变异。植物学类型、百仁重和粗脂肪含量的表型保留比例较大, 是补充了一些特殊种质和极值材料所致; 其余10个性状的表型保留比例均比较合适, 表明骨干种质保留了全部种质丰富的变异, 且丰度更高。
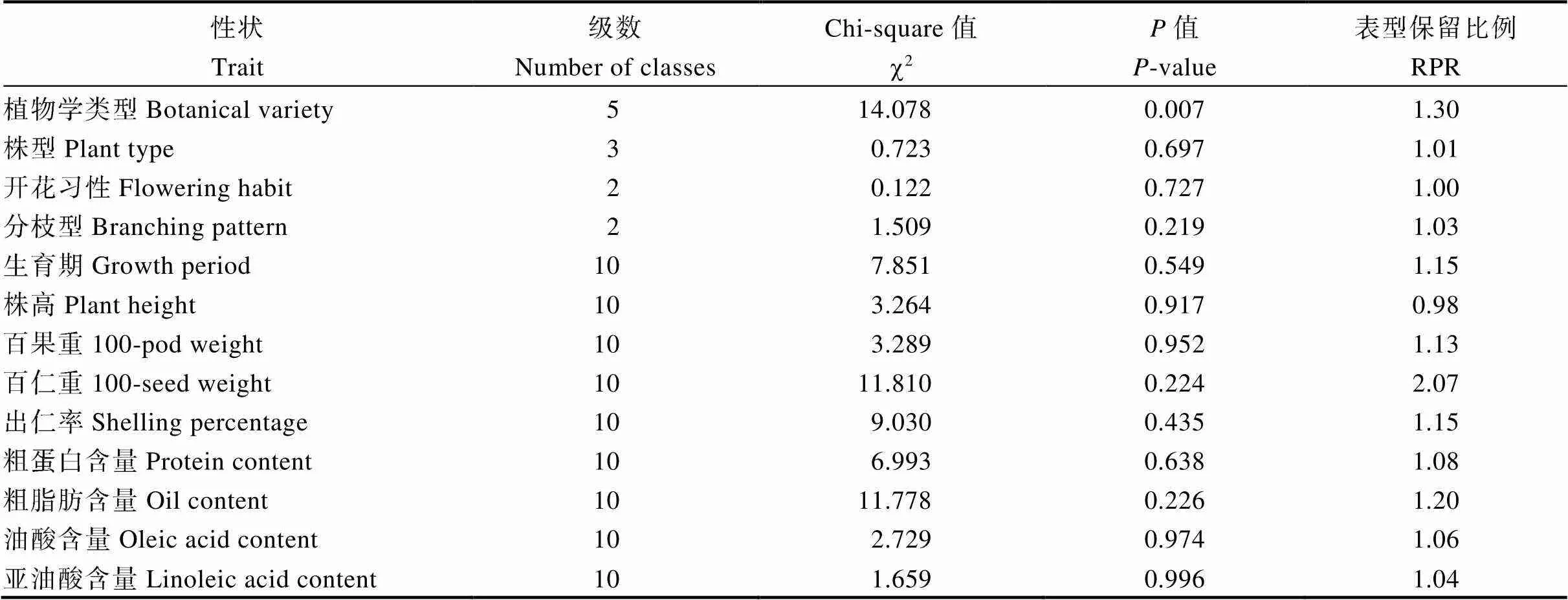
表5 全部种质与骨干种质13个性状的分布频率和表型保留比例的比较
> 0.05表明差异不显著,< 0.01表明差异极显著。RPR: 表型保留比例。
> 0.05 means the difference is insignificant;< 0.01 means the difference is positively significant. RPR: the ratio of phenotypic retention.
2.5 表型相关分析
一个具有代表性的骨干种质除应具有较小的遗传冗余外, 还应保留原群体固有的性状间的遗传关联。对13个性状的表型相关性分析表明, 64对性状在全部种质中呈极显著正相关, 其中有59对与骨干种质中的相关性是一致的, 39对在骨干种质保持了极显著正相关(表6), 因此, 骨干种质较好地保持了全部种质的表型相关性。不同性状间的极显著相关性意味着在育种实践中, 可以不直接筛选难测量的性状, 而优先筛选与其极显著相关的易测量性状, 从而较快地实现育种目的。比如, 由于分枝型和油酸含量极显著负相关(全部种质中=-0.789**, 骨干种质=-0.685**), 那么在杂交后代中要获得高油酸的单株, 就可以优先筛选分枝较多的单株, 直到形成稳定的品系后, 再测定其油酸含量。

表6 在全部种质和骨干种质中均显著相关的性状

(续表6)
**表示相关达到0.01极显著性水平,*表示相关达到0.05显著水平。
**indicates significant correlation at≤ 0.01;*indicates significant correlation at≤ 0.05.
2.6 骨干种质的确认
利用主成分分析对所构建的骨干种质进行确认, 从骨干种质和全部种质基于第1、第2主成分的样品分布图可知(图1), 全部种质中大量的样品集中在散点图的左右方并存在较重的相互重叠, 表明这些样品存在较高的遗传相似性, 群体的遗传冗余程度高。9.4%的平方根法取样后, 骨干种质样品分布的重叠程度得到了显著降低, 但仍保留了全部种质的几何形状和特征, 且较多外围的个体入选到骨干种质中, 表明建立的骨干种质既去除了全部种质的大部分遗传冗余, 又确保了骨干种质的代表性。此外, 绘制了4个植物学性状与9个数量性状表型分布的直方图, 可以看出, 植物学类型、开花习性、株型和分枝型4个植物学性状在全部种质和骨干种质中的分布非常吻合; 9个数量性状在全部种质和骨干种质中都表现出广泛的变异, 且大部分变异的分布频率较为一致, 均基本符合正态分布(图2)。因此, 骨干种质很好地保留了全部种质的遗传多样性和群体结构, 确保了骨干种质的有效性。
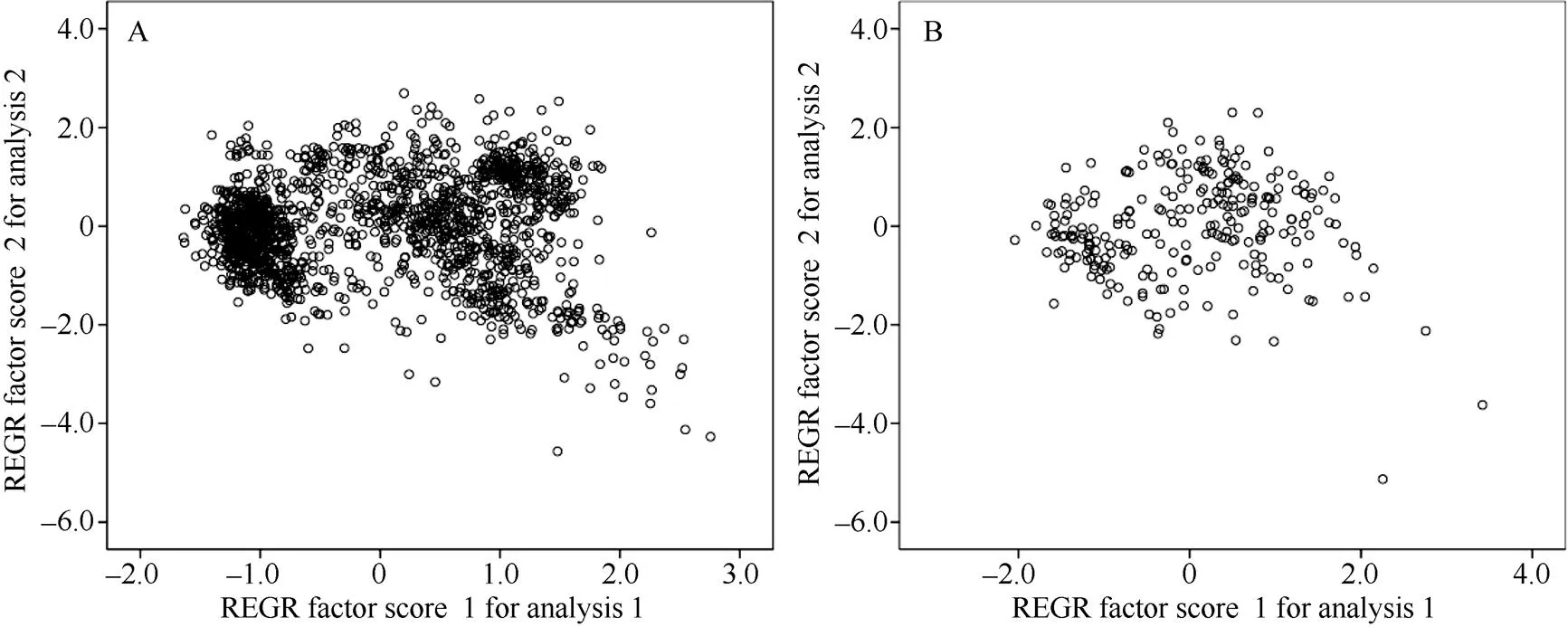
图1 全部种质与9.4%取样比例骨干种质的样品主成分分布图
A: 全部种质的样品分布图; B: 骨干种质的样品分布图。
A: scatter diagram for entire collection; B: scatter diagram for the key germplasm.
3 讨论
3.1 取样策略对骨干种质构建的影响
要建立质量高、代表性强的骨干种质, 采用的取样策略很关键。为使构建的骨干种质能更有效地代表全部种质最大的遗传多样性, 一般采用2种取样策略, 即逐步聚类法与分层取样+系统聚类法。前者多见于基础数据为分子标记数据, 少见于农艺数据。由于具有最大相似系数或最近遗传距离的种质能够聚在一起, 从而筛除相同或相近的种质。如刘娟等[18]基于ISSR分子数据, 采用UPGMA多次聚类抽样法+位点优先取样策略构建了包含31份种质的新疆野杏核心种质。张春雨等[19]基于SSR分子数据, 采用UPGMA多次聚类法+位点优先取样策略构建了包含25份种质的新疆野苹果核心种质, 并利用基因多样度、香农指数、主坐标及SRAP数据分析确认了其代表性。徐益等[20]则是将农艺性状聚类分析和SSR分子标记聚类分析相结合, 遴选出84份黄麻核心种质, 可以最大限度地代表300份黄麻种质资源的遗传多样性。刘遵春等[21]基于15个数量性状数据, 采用最短距离法逐步聚类+优先取样法构建了包含60份材料的新疆野苹果核心种质。后者常见于以农艺数据为基础数据的, 以分子数据的较少。考虑到作物遗传多样性的发生、发展层次和分布极不均匀, 一般在建立核心种质之前, 将资源归类, 实行系统分组取样, 从而更有效地达到以最小的重复代表最大的遗传多样性[22]。常见的分组方法基于植物学分类、系谱起源、地理分布、农业生态区、生长习性等。在系统分组的基础上, 基于表型或分子标记数据计算的遗传距离进行聚类分析, 将材料划分成不同的类群, 类内一般采用随机取样法或位点优先取样法。潘英华等[23]先按照地理分布将普通野生水稻分组, 再利用SSR数据进行多次UPGMA聚类, 构建了包含351份种质的广西普通野生稻核心种质。常利芳等[24]先将144份材料分成超甜玉米和普甜玉米2组, 再根据SSR数据进行UPGMA聚类分析+位点优先取样, 构建了包含33份种质的甜玉米核心种质。任丽平等[25]先按品种特点、地理来源、生长习性分组, 再结合EST-STS标记和SSR标记分析, 在500余份甘蓝型油菜种质中遴选出87份核心种质。刘艳阳等[26]根据地理来源分组, 组内比例法聚类抽样, 结合SSR标记位点优先取样策略进行UPGMA逐步聚类, 在5020份芝麻种质资源中遴选出501份核心种质。本研究按中国花生地方品种的七大地理种植区划和五大植物学类型, 将全部种质划分为26个组, 再根据13个性状对组内材料进行UPGMA聚类分析, 类内随机取样, 构建的骨干种质的均值、方差、极差、变异系数、香农指数、分布频率、表型保留比例等与全部种质基本无显著差异, 且很好地保留了全部种质的表型相关性。
3.2 组内取样量与取样比例的确定
作物遗传结构的差异, 导致在分组聚类的基础上组内取样量的确定方法也各不相同。常用的有平方根法、比例法、对数法及多样性法。刘三才等[27]针对普通小麦核心种质进行抽样方法比较表明, 平方根法能提高优良类别的频率, 有利于实现小麦核心种质在育种上的利用。此外, 合理的取样比例也是构建骨干种质的重要环节, 其大小与原始群体的数量规模、评价数据类型及物种遗传结构有关。目前, 国内外所构建各类作物核心种质的取样比例基本为5%~30%, 一般在10%左右。Brown[28]提出样品数不少于3000时, 以5%~10%的取样比例就可以代表原始群体70%的变异。Reddy等[29]构建鹰嘴豆核心种质过程中, 原始种质为16,991份, 核心种质为1956份, 取样比例约为10%。Diwan等[30]对美国一年生苜蓿资源的研究表明, 7%是适宜的筛选比例。魏兴华等[31]研究450份浙江籼稻地方种质的变异, 建立了12.5%的核心种质。Zewdie等[32]构建的高粱核心种质也采用了10%的取样比例。本研究从2741份花生地方品种中选取了242份骨干种质, 初步评价后补充极值材料和特殊性状种质17份, 取样比例为9.4%, 基本符合核心种质的规模。
3.3 骨干种质的评价与确认
骨干种质的评价就是检验其代表性和有效性。李自超等[33]认为遗传多样性指数、表型方差、表型分布频率、变异系数、表型保留比率等是衡量骨干种质的重要参数。本研究通过检验、测验、卡方测验对骨干种质与全部种质比较表明, 多数性状的均值、方差、变异系数、香农指数、分布频率等无显著差异; 除百仁重和出仁率外, 大部分性状的变异范围均保留在骨干种质中。另外, 二者在植物学组成(c2=1.600,=0.809)、生态分布(c2=5.232,=0.514)上也有很好的一致性, 表型保留比例和表型相关性都得到了稳定的保持, 可见本研究构建的骨干种质代表了全部种质的遗传多样性。出仁率的方差在2个群体间较大, 可能与出仁率的数据缺失较多有关。骨干种质保留了百仁重64.3%的变异范围, 可适当补充部分超大仁材料; 出仁率的保留范围为77.2%, 是由一份出仁率95.20%的种质比同级其他种质高出约10%所致, 差异在可接受范围内。利用主成分分析和直方图对骨干种质进一步确认, 259份种质的遗传冗余较小, 离散程度和分布特点与全部种质一致; 骨干种质的性状变异广泛, 分布频率与全部种质较符合, 证实了骨干种质的代表性和有效性。
4 结论
基于种植区划和植物学类型分组, 平方根法确定取样量, 组内按表型数据进行UPGMA聚类分析, 类内随机取样, 构建了中国花生地方品种的259份骨干种质, 占全部种质的9.4%, 其均值、方差、极差、变异系数、香农指数、表型保留比例等与全部种质无显著差异, 植物学类型组成和生态分布一致, 且保持了全部种质的表型分布频率和表型相关性。主成分分析和直方图进一步确认了骨干种质的遗传多样性和群体结构。本研究建立的骨干种质具有很好的代表性。
[1] Kochert G, Halward T, Branch W D, Simpson C E. RFLP variability in peanut () cultivars and wild species., 1991, 81: 565–570.
[2] 姜慧芳, 任小平, 廖伯寿, 黄家权, 陈本银. 中国花生核心种质的建立. 武汉植物学研究, 2007, 25: 289–293. Jiang H F, Ren X P, Liao B S, Huang J Q, Chen B Y. Establishment of peanut core collection in China., 2007, 25: 289–293 (in Chinese with English abstract).
[3] 沈一, 鄂志国, 刘永惠, 陈志德. 中国花生品种及其系谱数据库的构建. 中国油料作物学报, 2015, 37: 571–575. Shen Y, E Z G, Liu Y H, Chen Z D. Database construction of Chinese peanut varieties and their genealogy., 2015, 37: 571–575 (in Chinese with English abstract).
[4] 姜慧芳, 段乃雄. 花生种质资源在育种中的利用. 中国种业, 1998, (2): 24–25. Jiang H F, Duan N X. The application of germplasm resources in peanut breeding., 1998, (2): 24–25 (in Chinese with English abstract).
[5] Frankel O H, Brown A H D. Plant genetic resources today: a critical appraisal. In: Holden J H W, Williams J T, eds. Crop Genetic Resources: Conservation and Evaluation. London: George Allen and Unwin, 1984. pp 249–257.
[6] 贾继增, 高丽锋, 赵光耀, 周文斌, 张卫健. 作物基因组学与作物科学革命. 中国农业科学, 2015, 48: 3316–3332. Jia J Z, Gao L F, Zhao G Y, Zhou W B, Zhang W J. Crop genomics and crop science revolutions., 2015, 48: 3316–3332 (in Chinese with English abstract).
[7] Holbrook C C, Anderson W F, Pittman R N. Selection of a core collection from the U.S. germplasm collection of peanut., 1993, 33: 859–861.
[8] Holbrook C C, Dong W B. Development and evaluation of a mini core collection for the U.S. peanut germplasm collection., 2005, 45: 1540–1544.
[9] Upadhyaya H D, Bramel P J, Ortiz R, Singh S. Developing a mini core of peanut for utilization of genetic resources., 2002, 42: 2150–2156.
[10] Upadhyaya H D, Ortiz R, Bramel P J, Sube S. Development of a groundnut core collection using taxonomical, geographical and morphological descriptors., 2003, 50: 139–148.
[11] Chamberlin K D C, Hassan A M, Mark E P. Evaluation of the U.S. peanut mini core collection using a molecular marker for resistance toJagger., 2010, 172: 109–115.
[12] Mukri G, Hajisaheb L, Nadaf R S B, Gowda M V C, Upadhyaya H D, Sujay V. Phenotypic and molecular dissection of ICRISAT mini core collection of peanut (L.) for high oleic acid., 2012, 131: 418–422.
[13] Upadhyaya H D, Dwivedi S L, Vadez V, Hamidou F, Singh S, Varshney R K, Liao B. Multiple resistant and nutritionally dense germplasm identified from mini core collection in peanut., 2013, 54: 679–693.
[14] Sudini H, Upadhyaya H, Reddy S V, Mangala U N, Kumar K V. Resistance to late leaf spot and rust diseases in ICRISAT’s mini core collection of peanut (L.)., 2015, 44: 557–566.
[15] Hovav R, Badani H, Ginzberg I, Hovav R, Badani H, Ginzberg I, Chedvat I, Brand Y, Galili S. Evaluation of a peanut collection for shell-colour traits in two diverse soil types., 2012, 131: 148–154.
[16] 黄莉, 任小平, 张晓杰, 陈玉宁, 姜慧芳. ICRISAT花生微核心种质农艺性状和黄曲霉抗性关联分析. 作物学报, 2012, 38: 935–946. Huang L, Ren X P, Zhang X J, Chen Y N, Jiang H F. Association analysis of agronomic traits and resistance toin the ICRISAT peanut mini-core collection., 2012, 38: 935–946 (in Chinese with English abstract).
[17] 任小平, 廖伯寿, 张晓杰, 雷永, 黄家权, 晏立英, 陈玉中, 姜慧芳. 中国花生核心种质中高油酸材料的分布和遗传多样性. 植物遗传资源学报, 2011, 12: 513–518. Ren X P, Liao B S, Zhang X J, Lei Y, Huang J Q, Yan L Y, Chen Y Z, Jiang H F. Distributing and genetic diversity of high oleic acid germplasm in peanut (L.) core collection of China., 2011, 12: 513–518 (in Chinese with English abstract).
[18] 刘娟, 廖康, 赵世荣, 曹倩, 孙琪, 刘欢. 利用ISSR分子标记构建新疆野杏核心种质资源. 中国农业科学, 2015, 48: 2017–2028. Liu J, Liao K, Zhao S R, Cao Q, Sun Q, Liu H. The core collection construction of Xinjiang wild apricot based on ISSR molecular markers., 2015, 48: 2017–2028 (in Chinese with English abstract).
[19] 张春雨, 陈学森, 张艳敏, 苑兆和, 刘遵春, 王延龄, 林群. 采用分子标记构建新疆野苹果核心种质的方法. 中国农业科学, 2009, 42: 597–604. Zhang C Y, Chen X S, Zhang Y M, Yuan Z H, Liu Z C, Wang Y L, Lin Q. A method for constructing core collection of Malussieversii using molecular markers., 2009, 42: 597–604 (in Chinese with English abstract).
[20] 徐益, 张列梅, 郭艳春, 祁建民, 张力岚, 方平平, 张立武. 黄麻核心种质的遴选. 作物学报, 2019, 45: 1672–1681. Xu Y, Zhang L M, Guo Y C, Qi J M, Zhang L L, Fang P P, Zhang L W. Core collection screening of a germplasm population in jute (spp.)., 2019, 45: 1672–1681 (in Chinese with English abstract).
[21] 刘遵春, 张春雨, 张艳敏, 张小燕, 吴传金, 王海波, 石俊, 陈学森. 利用数量性状构建新疆野苹果核心种质的方法. 中国农业科学, 2010, 43: 358–370. Liu Z C, Zhang C Y, Zhang Y M, Zhang X Y, Wu C J, Wang H B, Shi J, Chen X S. Study on method of constructing core collection of Malussieversii based on quantitative traits., 2010, 43: 358–370 (in Chinese with English abstract).
[22] Zeuli P L S, Qualset C O. Evaluation of five strategies for obtaining a core subset from a large genetic resource collection of durum wheat., 1993, 87: 295–304.
[23] 潘英华, 徐志健, 梁云涛. 广西普通野生稻群体结构解析与核心种质构建. 植物遗传资源学报, 2018, 19: 498–509. Pan Y H, Xu Z J, Liang Y T. Genetic structure and core collection of common wild rice (Griff.) in Guangxi., 2018, 19: 498–509 (in Chinese with English abstract).
[24] 常利芳, 白建荣, 李锐, 张丛卓, 张效梅, 杨瑞娟. 基于SSR标记构建甜玉米群体的核心种质. 玉米科学, 2018, 26(3): 40–49.Chang L F, Bai J R, Li R, Zhang C Z, Zhang X M, Yang R J. Construction of a core collection of sweet corn populations based on SSR markers., 2018, 26(3): 40–49 (in Chinese with English abstract).
[25] 任丽平, 倪西源, 黄吉祥, 雷伟侠, 曹明富, 赵坚义. 甘蓝型油菜一个代表性核心种质的遴选. 中国农业科学, 2008, 41: 3521–3531. Ren L P, Ni X Y, Huang J X, Lei W X, Cao M F, Zhao J Y. Core collection of a representative germplasm population in., 2008, 41: 3521–3531 (in Chinese with English abstract).
[26] 刘艳阳, 梅鸿献, 杜振伟, 武轲, 郑永战, 崔向华, 郑磊. 基于表型和SSR分子标记构建芝麻核心种质. 中国农业科学, 2017, 50: 2433–2441. Liu Y Y, Mei H X, Du Z W, Wu K, Zheng Y Z, Cui X H, Zheng L. Construction of core collection of sesame based on phenotype and molecular markers., 2017, 50: 2433–2441 (in Chinese with English abstract).
[27] 刘三才, 曹永生, 郑殿升, 刘春华, 陈梦英. 普通小麦核心种质抽样方法的比较. 麦类作物学报, 2001, 21(2): 42–45. Liu S C, Cao Y S, Zheng D S, Liu C H, Chen M Y. Comparing of strategies for developing core collection from common wheat., 2001, 21(2): 42–45 (in Chinese with English abstract).
[28] Brown A H D. Core collections: a practical approach to genetic resources management., 1989, 31: 818–824.
[29] Reddy L J, Upadhyaya H D, Gowda C L L, Sube S. Development of core collection in pigeonpea [(L.) Millspaugh] using geographic and qualitative morphological descriptors., 2005, 52: 1049–1056.
[30] Diwan N, McIntosh M S, Bauchan G R. Methods of developing a core collection of annual Medicago species., 1995, 90: 755–761.
[31] 魏兴华, 颜启传, 应存山, 张丽华, 章林平. 建立浙江地方籼型稻种资源的核心样品的研究. 中国水稻科学, 1999, 13(2): 81–85.Wei X H, Yan Q C, Ying C S, Zhang L H, Zhang L P. A core collection of Zhejiang traditionalrice germplasm., 1999, 13(2): 81–85 (in Chinese with English abstract).
[32] Zewdie Y, Tong N, Bosland P. Establishing a core collection of Capsicum, using a cluster analysis with enlightened selection of accessions., 2004, 51: 147–151.
[33] 李自超, 张洪亮, 曹永生, 裘宗恩, 魏兴华, 汤圣祥, 余萍, 王象坤. 中国地方稻种资源初级核心种质取样策略研究. 作物学报, 2003, 29: 20–24.Li Z C, Zhang H L, Cao Y S, Qiu Z E, Wei X H, Tang S X, Yu P, Wang X K. Studies on the sampling strategy for initial core collection of Chinese ingenious rice., 2003, 29: 20–24 (in Chinese with English abstract).
Developing the key germplasm of Chinese peanut landraces based on phenotypic traits
YAN Cai-Xia1, WANG Juan1, ZHANG Hao1, LI Chun-Juan1, SONG Xiu-Xia2, SUN Quan-Xi1, YUAN Cui-Ling1, ZHAO Xiao-Bo1, and SHAN Shi-Hua1,*
1Shandong Peanut Research Institute, Qingdao 266100, Shandong, China;2Agricultural and Rural Bureau of Mudan District, Heze 274000, Shandong, China
Chinese peanut landraces are important parent resources in peanut breeding due to their abundant genetic diversity. In this study, a total of 2741 original accessions from peanut seed bank were divided into 26 groups based on their botanical variety and ecological distribution. The key accessions were established based on the analysis of 13 phenotypic traits by the square root strategy, UPGMA clustering within groups and random sampling in individual clusters, and evaluated by-test,-test, Chi-squared test, ranging, the ratio of phenotypic retention, and phenotypic correlation analyses. Finally, the principal components analysis (PCA) and the histogram analysis were used to re-confirm the key germplasm. The total of 259 as a key germplasm was selected, accounting for 9.4% of total accessions, which included 14 of var., 85 of var., 42 of var., 103 of var.and 15 of irregular type. There were no significant differences (< 0.05) in means, variance, coefficient of variation, and Shannon-weaver diversity index for 13 phenotypic traits between key germplasm and entire collection. The key germplasm preserved the distribution range, the ratio of phenotypic retention and the phenotypic correlation of primary collection, with similar composition of botanical variety and ecological distribution. PCA and the histogram confirmed the homogeneity of genetic structure and distribution frequency between two collections. Thus, this key germplasm can represent the genetic variability and population structure of entire collection, and enhance innovation of peanut genetic resources and exploitation of elite alleles.
peanut; landrace; phenotypic traits; key germplasm; representative evaluation
2019-07-17;
2019-12-26;
2020-01-17.
10.3724/SP.J.1006.2020.94101
单世华, E-mail: shansh1971@163.com, Tel: 0532-87629307
E-mail: cxyan335@sina.com, Tel: 0532-87626756
本研究由泰山学者特聘专家项目(ts201712080), 中央引导地方科技发展专项资金, 农业科研杰出人才及其创新团队培养(13190194), 青岛市民生科技计划项目(17-3-3-49-nsh), 山东省农业良种工程项目(2017LZN033, 2017LZGC003), 山东省农业产业技术体系项目(SDAIT-04-02)和山东省农业科学院农业科技创新工程项目(CXGC2016A01)资助。
This study was supported by the Taishan Scholars Project (ts201712080), the Central Guidance for Local Science and Technology, the Outstanding Talents and Innovation Team in Agricultural Research (13190194), the Qingdao Science and Technology Plan for the Public Benefit (17-3-3-49-nsh), the Fine Breeding Project of Shandong Province (2017LZN033, 2017LZGC003), the Shandong Agriculture Research System (SDAIT-04-02), and the Agricultural Science and Technological Innovation Project of Shandong Academy of Agricultural Science (CXGC2016A01).
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20200117.1338.004.html
