抗青枯病兼大果和高出仁率的花生新种质创制
2020-03-23李威涛徐志军郭建斌喻博伦陈玉宁周小静罗怀勇陈伟刚任小平姜慧芳
李威涛 徐志军 蔡 岩 郭建斌 喻博伦 黄 莉 陈玉宁 周小静 罗怀勇 刘 念 陈伟刚 任小平 姜慧芳
抗青枯病兼大果和高出仁率的花生新种质创制
李威涛 徐志军 蔡 岩 郭建斌 喻博伦 黄 莉 陈玉宁 周小静 罗怀勇 刘 念 陈伟刚 任小平 姜慧芳*
中国农业科学院油料作物研究所 / 农业部油料作物生物学与遗传育种重点实验室, 湖北武汉 430062
青枯病是影响花生产量和品质的重要土传性细菌病害, 百果重和出仁率是与花生产量相关的重要性状。本研究利用远杂9102和徐州68-4杂交构建的RIL群体, 在B02染色体上定位到青枯病抗性主效QTL。结合前期对百果重和出仁率QTL的定位结果发现, 所涉及的3个性状的主效QTL分布在不同的染色体上。以RIL群体基因型数据和多个环境的青枯病抗性、百果重和出仁率表型数据为基础, 利用与主效QTL紧密连锁分子标记筛选出6份聚合抗青枯病、荚果大、出仁率高3种优良性状的新种质, 可以作为育种中间材料或亲本培育高产抗病新品种。本研究利用分子标记辅助选择和表型鉴定相结合有效筛选抗病高产种质, 为未来花生育种提供了新思路。
花生新种质; 青枯病抗性; 百果重; 出仁率
花生(L.)是我国重要的油料作物, 在2016—2017年, 我国花生总产量为1729万吨[1], 约占世界总产量4398万吨[2]的40%。提高单位面积产量和改进产品质量是我国花生产业发展的关键[3]。但是由青枯雷尔氏菌()引发的花生青枯病(bacterial wilt diseases, BW)严重威胁着花生生产。花生青枯病是细菌性土传病害, 全生育期都可发病。我国花生青枯病发生面积占花生播种面积的10%以上, 危害程度居世界首位[4]。花生一旦感染青枯病, 轻者减产10%, 重者减产30%, 甚至更多[5]。青枯病还可以增加黄曲霉毒素污染的程度, 进而影响食用安全质量[4,6]。百果重(hundred pod weight, HPW)能够直接影响花生的产量, 是花生的重要产量性状[7-8]。出仁率(shelling percentage, SP)是花生籽仁重量占荚果重量的比例, 成熟花生的出仁率不仅与花生的产量相关, 还与果壳厚度及脱壳难易程度相关。因此, 出仁率也是花生遗传改良的重要性状。
我国自20世纪70年代中期鉴定出“协抗青”、“台山三粒肉”等抗青枯病种质以来, 通过杂交育种已培育出中花2号、中花6号、粤油256、粤油202、粤油200、粤油79等抗性品种, 并应用于生产, 极大地缓解了青枯病的病情[9]。但是, 我国青枯病区应用的抗病花生品种的抗源几乎都来源于少数几个疏枝亚种种质, 其抗性与高产优质存在负相关[10], 导致抗病品种的荚果普遍偏小, 产量比具相同生态适应性的非抗病品种低20%左右[9]。因此, 生产上迫切需要抗青枯病的高产大果花生品种。通过选配具有不同性状的杂交亲本, 应用分子标记辅助选择育种(marker assisted selection, MAS)[11]可加速兼具抗青枯病、大果、高出仁率品种的育种进程。
近年来, 国内外对花生青枯病抗性[12-18]、荚果大小[19-23]和出仁率[22-27]的QTL定位已取得一定进展。Wang等[15]和Luo等[16]通过SNP技术鉴定出与花生青枯病抗性相关的主效QTL, 但是, 由于SNP标记必须转换成其他标记如KASP标记才能在育种选择中应用, 因此, 存在费用较高问题。Luo等[20]、李振动等[21]、蔡岩等[24]通过SSR技术鉴定出与荚果大小和出仁率相关的主效QTL, 但它们与青枯病抗性QTL的关系如何, 以及能否用其创制抗青枯病的大果高出仁率品种未见分析。本研究拟在这些研究的基础之上, 通过简单易操作的SSR技术定位青枯病抗性QTL, 结合前期鉴定的百果重[20]和出仁率主效QTL[26-27], 筛选抗青枯病大果高出仁率种质, 为高产抗青枯病花生品种的培育提供理论依据和材料基础。
1 材料与方法
1.1 试验材料
以远杂9102(♀)×徐州68-4(♂)构建RIL群体。远杂9102的百果重、出仁率和青枯病抗性多年多点鉴定的均值分别为185.98 g、81.65%、77.82%, 是中小果、高出仁率和抗青枯病品种, 徐州68-4的相应值分别为252.91 g、75.81%、16.87%, 是大果、低出仁率和感青枯病品种。青枯病抗性分子标记检测所用花生材料均由中国农科院油料作物研究所提供。
1.2 青枯病抗性、百果重和出仁率的测定
2013年, 将RIL群体F5及其亲本种植于湖北红安病圃(2013HA), 2014年和2015年, 将F6:7及其亲本种于四川南充病圃(2014NC, 2015NC), 于开花下针期调查种植材料萎蔫和死亡的情况, 辨别枯萎症状, 剔除其他病害造成的死苗, 以存活率作为抗性判断标准。于2013年至2014年在武汉中国农业科学院油料作物研究所试验田连续种植RIL群体的F5~F6代及亲本材料, 2015年在武汉和襄阳种植F7代及亲本材料, 收获晒干后, 每个家系100个成熟饱满干荚果的重量即为百果重。选取每个家系成熟饱满干荚果500 g, 剥壳后的仁重占荚果重量的百分率即为出仁率。采用Microsoft Excel 软件统计与计算数据。
1.3 QTL定位
利用软件QTLNetwork 2.0[28]进行QTL定位和效应估计, 默认参数设置, QTL的显著水平为0.05, 得到QTL相关信息。以“q+性状的英文名称首字母+所在连锁群”命名QTL, 若同一个连锁群上有不同环境下控制相同性状QTL, 则在连锁群后加“.”和数字区分。
1.4 目的基因型家系的筛选及表型鉴定
本实验室前期已经完成了RIL群体F5830个SSR分子标记的基因型鉴定[27]。SSR标记分为显性标记和共显性标记, 对于共显性标记, 母本带型记为“a”, 父本带型记为“b”, 杂合带型记为“h”。对于显性标记, 若母本等位基因为显性, 则父本带型记为“b”, 和母本带型相同的带型都记为“d”; 若父本等位基因为显性, 则母本带型记为“a”, 和父本带型相同的带型记为“c”。以此为基础, 根据目标QTL两侧标记, 选择具有青枯病抗性、大果和高出仁率标记的家系。对筛选获得的家系进行青枯病抗性、百果重和出仁率表型分析, 基因型和目标表型一致的即目的家系。
2 结果与分析
2.1 青枯病抗性相关QTL定位
以前期实验室构建的遗传连锁图谱[26]和青枯病抗性表型数据[15-16]为基础, 利用QTLNetwork 2.0[28]软件在3年单一环境下进行QTL定位分析, 共扫描到6个QTL, 分布在A01、B02、B10染色体上(表1和图1)。在2013HA环境下, 检测到3个QTL, 分布于A01、B02和B10染色体上, 贡献率在8.36%~ 21.45%之间; 在2014NC环境下, 检测到1个QTL, 位于B02染色体上, 贡献率为27.76%; 在2015NC环境下, 检测到2个QTL, 分别位于B02和B10染色体上, 贡献率分别为45.21%和6.49%。对比QTL在连锁群上的位置发现, 2013HA环境下检测到的QTL与2014NC环境下检测到的QTL和2015NC环境下检测到的QTL置信区间相同, 都在91.5~92.5 cM (AH TE0775~AHGS2344)之间, 是重复检测到的QTL, 贡献率在21.45%~45.21%之间, 为主效QTL。
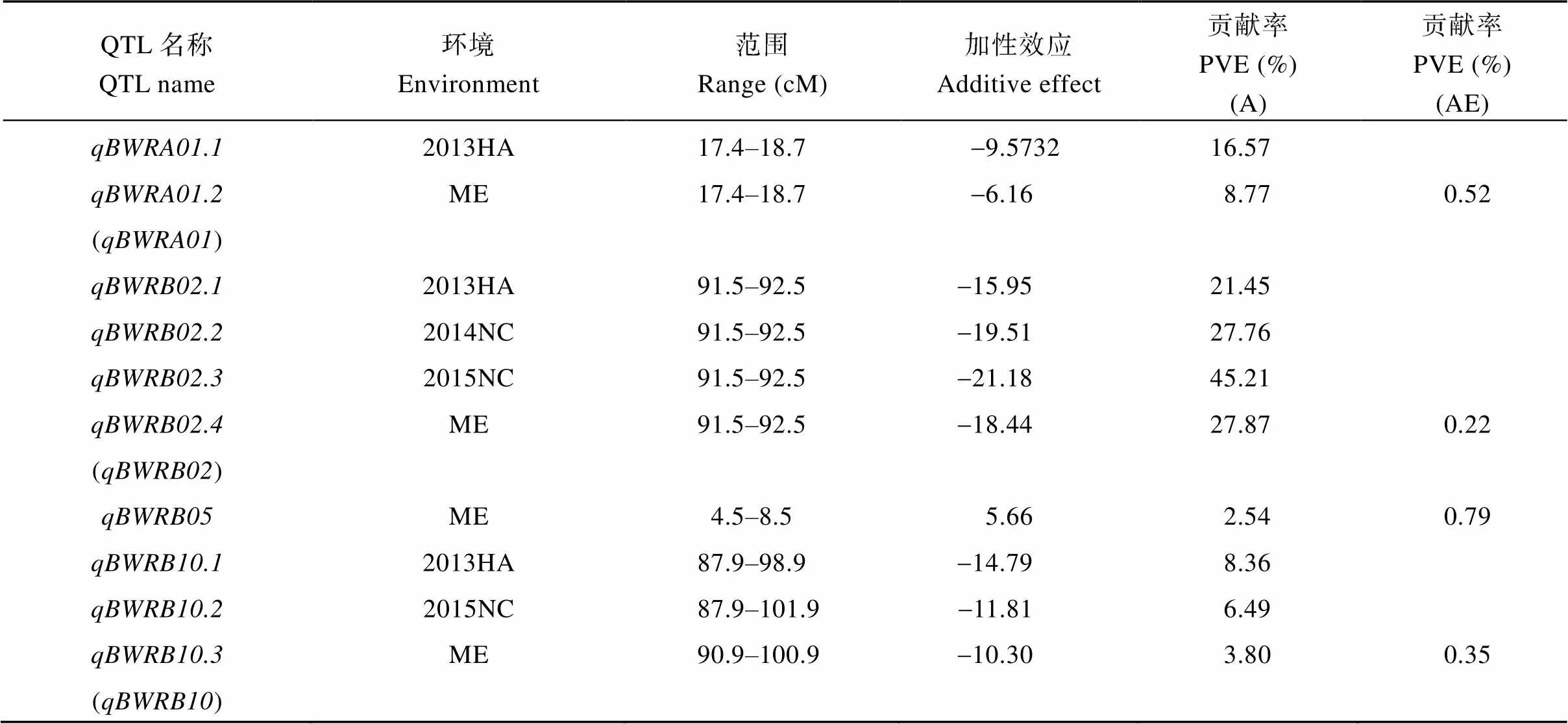
表1 利用QTLNetwork 2.0检测到的青枯病抗性QTL及参数
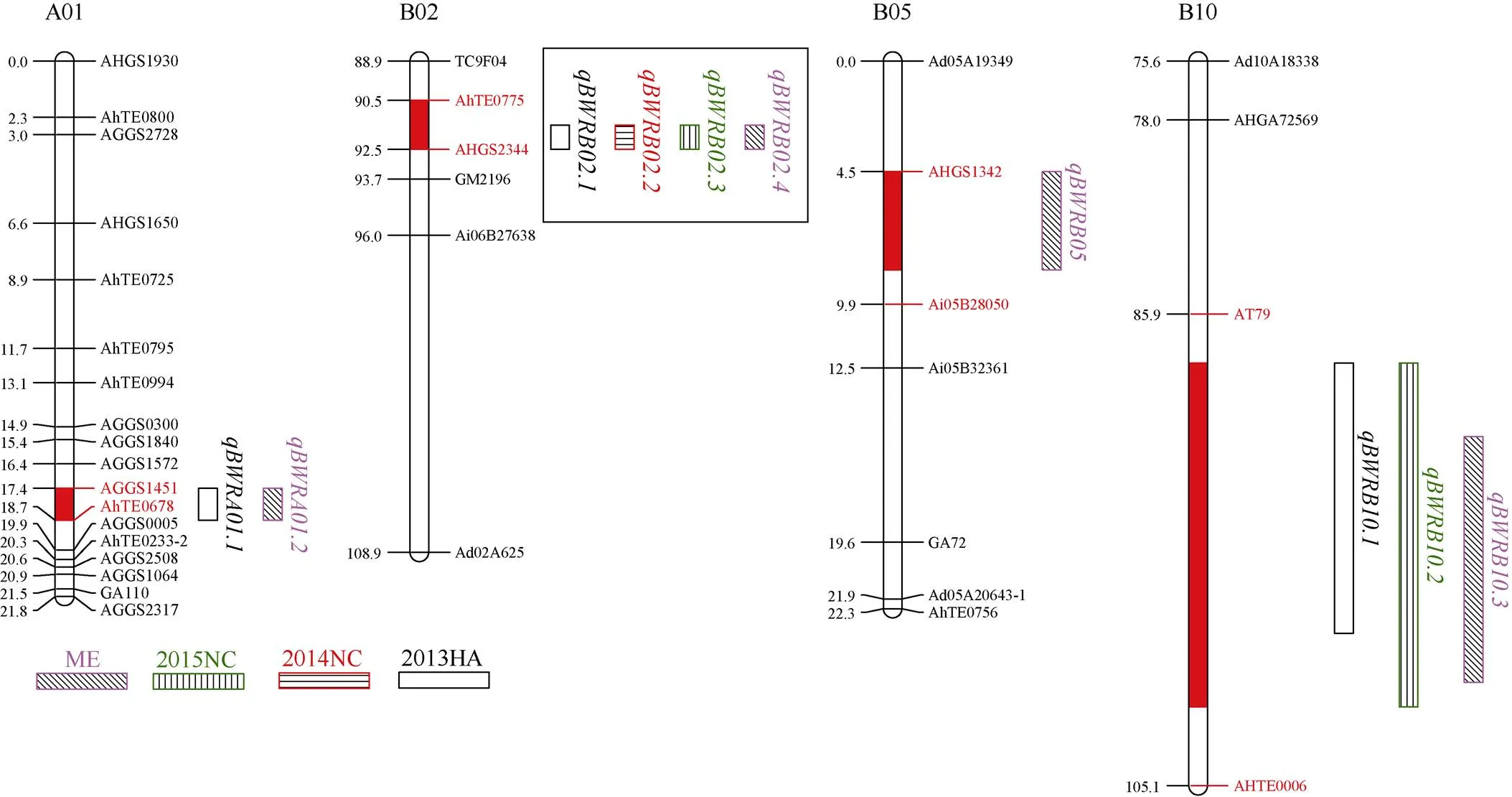
图1 抗青枯病QTL在遗传图谱上的位置
方框内为主效QTL。QTL surrounded by rectangle represents major QTL.
为了进一步分析QTL与环境的互作, 利用QTLNetwork 2.0[28]软件对3个环境(ME环境)进行联合QTL分析, 共扫描到4个QTL, 分布在A01、B02、B05和B10染色体上(表1和图1), 贡献率为2.54%~ 27.87%。其中与2013HA单环境检测的为染色体上的同一个位点, 贡献率为8.77%;与单环境的、和为同一个位点, 贡献率为27.87%, 是主效QTL;是在ME环境下检测到的QTL, 贡献率为2.54%;与单环境的、为同一个位点, 贡献率为3.8%。在ME环境下进行QTL 与环境的互作效应分析, 所有QTL均与环境间存在互作, 互作贡献率在0.22%~0.79%之间(表1), 远小于加性效应贡献率, 表明花生对青枯病的抗性主要受遗传因素影响。
2.2 抗青枯病种质筛选
利用青枯病主效QTL区间的标记AhTE0775和AHGS2344筛选RIL群体, 得到目标QTL区段来自抗性亲本远杂9102的家系57个。结合这些材料多年来的青枯病抗性鉴定结果, 在2013年的抗性鉴定中, 21份材料的抗性与远杂9102相当或高于远杂9102; 在2014年的抗性鉴定中, 38份材料的抗性与远杂9102相当或高于远杂9102; 在2015年的抗性鉴定中, 43份材料的抗性与远杂9102相当或高于远杂9102。综合标记鉴定结果和3年的抗性鉴定结果, 有35个家系青枯病抗性与远杂9102相当或高于远杂9102。
2.3 抗青枯病分子标记AHGS2344有效性及实用性验证
利用QTLNetwork 2.0[28]定位的青枯病抗性QTL位置在92.5 cM, 在AhTE0775和AHGS2344之间, 而AHGS2344与QTL连锁更紧密, 因此选择AHGS2344探究是否可以筛选其他材料。选择本实验室已经多年多点鉴定的抗病材料和感病材料进行PCR检测, 其抗病材料包括远杂9102及与远杂9102有亲本关系的材料中花21、J8、J11、J64、J91、J92, 中花6号及与中花6号有亲本关系的材料QT1315、QT1319; 感病材料包括徐花13, 徐州68-4, 与远杂9102有亲本关系的材料J12, 与中花6号有亲本关系的材料QT1322、QT1299、QT1306、QT1314, 结果表明, AHGS2344在所有抗病材料中的带型为A, 在感病材料中的带型为B, 抗病材料和感病材料带型明显分化(表2)。因此, AHGS2344在鉴定青枯病材料中是有效及实用的。
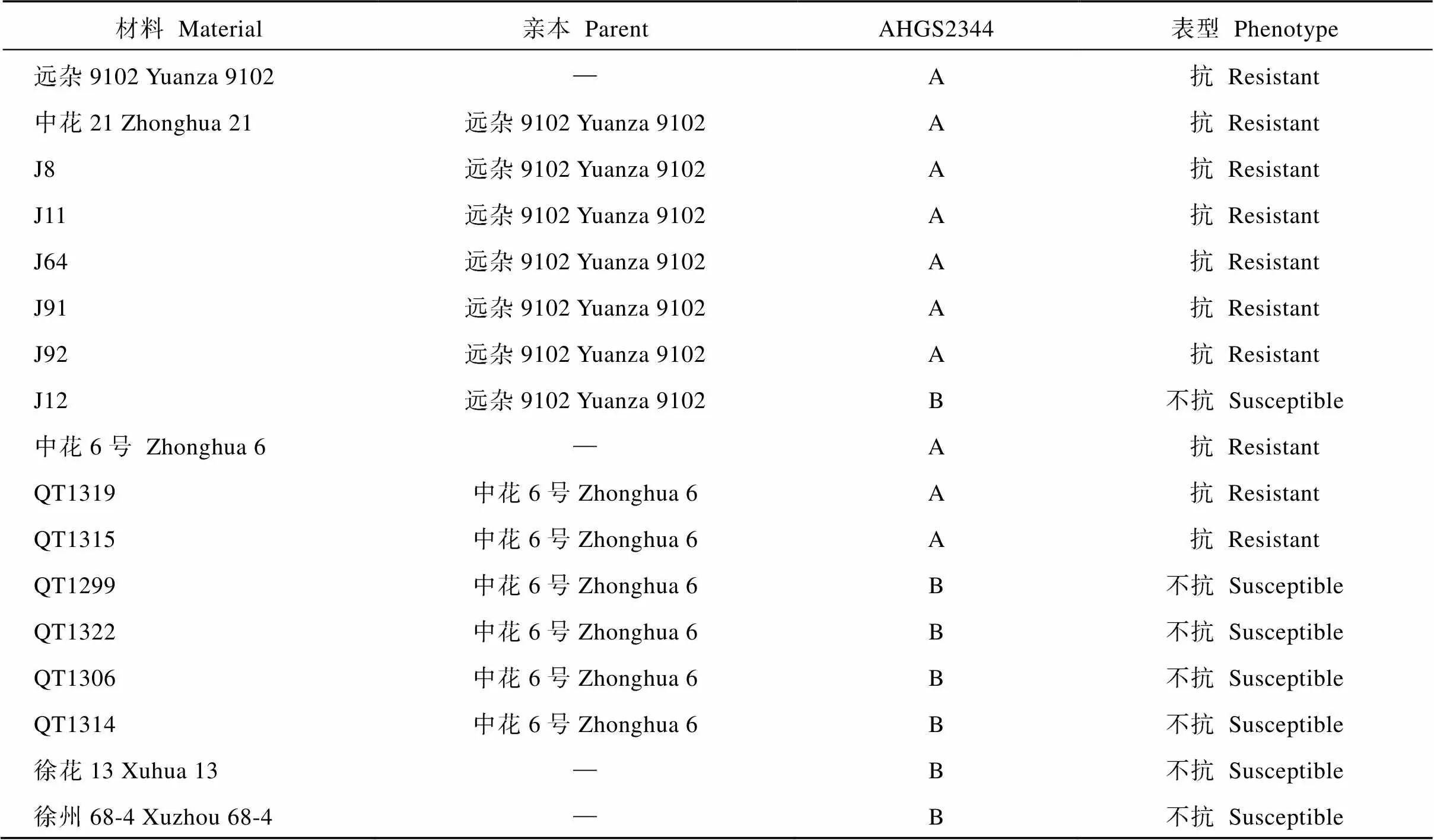
表2 不同青枯病抗性的品系和品种中分子标记AHGS2344的基因型
2.4 抗青枯病兼具大果和高出仁率种质筛选
利用前期通过该群体分析获得的百果重主效QTL侧翼分子标记Ad05A20499和Ad05A20262, 对上述35个抗青枯病材料进行筛选, 得到目标QTL区域片段来自大果亲本徐州68-4的家系13个。结合2013—2016年连续4年的百果重鉴定结果, 获得抗青枯病兼具大果材料12份。
进一步利用该群体出仁率主效QTL侧翼分子标记AGGS1606和AHGS2130对上述12个抗青枯病兼具大果材料进行筛选, 得到目标QTL区域片段来自高出仁率亲本远杂9102的家系6个。结合2013—2016年连续4年的出仁率鉴定结果, 获得抗青枯病兼具大果高出仁率材料6份(表3)。

表3 通过与QTL紧密连锁的分子标记创制的兼具3种优良性状的新种质
3 讨论
花生青枯病是影响花生产量和品质的重要因素之一, 培育和种植抗病品种是防治该病最经济有效的途径, 鉴定青枯病抗性QTL, 开发分子标记并应用于生产, 可大大缩短育种年限, 降低成本, 提高育种效率。本研究利用SSR图谱[26]及3年青枯病抗性表型数据, 定位了抗青枯病QTL, 其主效QTL位于B02染色体上。最近本实验室利用SNP数据对青枯病抗性QTL进行了定位, 其结果显示主效QTL同样位于B02染色体上[15-16], 将本研究主效QTL侧翼标记AhTE0775和AHGS2344映射至花生基因组物理图谱上, 发现其所在物理区间是一致的, 一方面说明利用SSR标记构建的遗传连锁图谱定位QTL的准确性, 另一方面更加明确了B02染色体上存在控制青枯病抗性的主效基因。本研究利用与抗青枯病主效QTL连锁更为紧密的SSR分子标记AHGS2344检测非本研究所用RIL群体的其他抗病和感病材料, 以进行有效性和实用性的验证, 结果表明, AHGS2344可以很明显地区分抗病材料和感病材料, 在实际育种中是有效和实用的。本团队前期利用SNP数据转换成KASP分子标记应用于分子育种, 而本研究开发的SSR标记AHGS2344相比KASP标记, 具有方便快捷和低费用的特点, 可直接应用于分子标记辅助选择(molecular marker assisted selection, MAS)育种。
青枯病、出仁率[26-27]和百果重[20]的主效QTL定位结果表明, 3种性状由不同的基因位点控制, 理论上是可以利用染色体片段重组获得聚合3种优良性状的材料。本研究利用与青枯病主效QTL紧密连锁的分子标记AhTE0775和AHGS2344, 对RIL群体进行基因型筛选, 结合3年青枯病抗性鉴定结果, 得到了青枯病抗性与远杂9102相当或高于远杂9102的家系35个; 再利用百果重主效QTL侧翼分子标记Ad05A20499、Ad05A20262和出仁率主效QTL侧翼分子标记AGGS1606、AHGS2130筛选35个抗青枯病家系, 结合百果重及出仁率的多年表型鉴定结果, 得到了抗青枯病兼大果高出仁率的家系6个。因此, 本研究结果印证了在花生中利用QTL聚合多种优质性状的可行性。
近年来, 花生青枯病的遗传改良方面取得了很大的进展。但是育种中选用抗病亲本的遗传基础狭窄, 抗源种质的荚果小, 产量潜力低, 导致目前生产上应用的抗病品种产量水平普遍较低[29]。因此, 迫切需要培育抗青枯病且高产的花生品种应用于生产。通过分子标记辅助筛选和表型筛选的办法(MAPS)来聚合基因或QTL并培育优良品种可以大大减少传统育种方法中表型筛选的工作量, 由此显著提高育种效率。利用分子标记选择目标基因, 其准确性主要取决于标记与目标基因的连锁程度, 连锁得越紧密, 可靠性就越高。本研究应用的分子标记与目标基因间距离还很大, 侧翼分子标记基因型结果显示存在目标QTL, 但表型还是有很大比例是不符合的, 其符合率对于青枯病抗性为61.4%、百果重为68.75%、出仁率为35.6%, 聚合3种QTL优质性状的符合率仅为27.3%。因此, 缩小QTL区间, 精细定位基因, 得到更为紧密连锁分子标记或基因对未来花生分子标记辅助育种意义重大。本研究通过构建RIL群体结合MAS育种方法, 筛选出抗青枯病兼具大果高出仁率种质6份, 这些种质在多年多点的表型鉴定一致, 对于提高抗青枯病品种的产量水平具有重要意义。另外, 通过QTL聚合创制优质材料, 为未来高效的花生育种提供了新的思路。
4 结论
获得与青枯病抗性相关主效QTL (), 利用分子标记辅助选择得到抗青枯病家系35个, 抗青枯病兼大果高出仁率的家系6个。
[1] 中国统计年鉴2017. http://www.stats.gov.cn/tjsj/ndsj/2017/indexch.htm. China Statistical Yearbook 2017. http://www.stats.gov.cn/tjsj/ ndsj/2017/indexch.htm (in Chinese).
[2] Luo H, Pandey M K, Khan A W, Guo J, Wu B, Cai Y, Huang L, Zhou X, Chen Y, Chen W, Liu N, Lei Y, Liao B, Varshney R K, Jiang H. Discovery of genomic regions and candidate genes controlling shelling percentage using QTL-seq approach in cultivated peanut (L.)., 2018. doi: 10.1111/pbi.13050.
[3] 王耀波, 张艺兵, 张鹏, 门爱军. 入世后中国花生产业发展前景及促进出口的对策. 花生学报, 2003, 32(S1): 24–29. Wang Y B, Zhang Y B, Zhang P, Men A J. Development prospect of peanut industry in China and measures to promote exports after entering WTO., 2003, 32(S1): 24–29 (in Chinese).
[4] 雷永, 王圣玉, 李栋, 姜慧芳, 廖伯寿. 花生抗青枯病种质对黄曲霉菌产毒的抗性反应. 中国油料作物学报, 2004, 26: 69–71. Lei Y, Wang S Y, Li D, Jiang H F, Liao B S. Evaluation of resistance to aflatoxin production among peanut germplasm with resistance to bacterial wilt., 2004, 26: 69–71 (in Chinese with English abstract).
[5] 姜慧芳, 任小平, 雷永, 廖伯寿, Mace E, Crouch J H. 花生青枯病抗性分子标记的初步研究. 花生学报, 2003, 32(S1): 319–323. Jiang H F, Ren X P, Lei Y, Liao B S, Mace E, Crouch J H. Preliminary study on molecular markers of resistance to peanut bacterial wilt., 2003, 32(S1): 319–323 (in Chinese).
[6] 肖达人, 王圣玉, 瞿桢, 张洪玲. 花生抗黄曲霉毒素污染研究进展. 花生科技, 1999, (S1): 124–129. Xiao D R, Wang S Y, Qu Z, Zhang H L. Recent advances of resistance to aflatoxin contamination in peanut., 1999, (S1): 124–129 (in Chinese).
[7] Gomes R L F, Lopes  C D A. Correlations and path analysis in peanut., 2005, 5: 105–112.
[8] Selvaraj M G, Narayana M, Schubert A M, Ayers J L, Baring M R, Burow M D. Identification of QTLs for pod and kernel traits in cultivated peanut by bulked segregant analysis., 2009, 12: 1–10.
[9] 陈本银, 姜慧芳, 廖伯寿, 任小平. 中国花生青枯病抗性遗传改良研究进展. 中国农学通报, 2007, 23(8): 369–372. Chen B Y, Jiang H F, Liao B S, Ren X P. Progress on groundnut genetic enhancement for bacterial wilt resistance.2007, 23(8): 369–372 (in Chinese with English abstract).
[10] 姜慧芳, 任小平, 廖伯寿, 傅廷栋. 抗青枯病花生资源的种子大小及主要品质性状的遗传分化. 中国油料作物学报, 2006, 28: 144–150. Jiang H F, Ren X P, Liao B S, Fu T D. Genetic diversity of peanut genotypes with resistance to bacterial wilt based on seed characters., 2006, 28: 144–150 (in Chinese with English abstract).
[11] 王亚琦, 孙子淇, 郑峥, 黄冰艳, 董文召, 汤丰收, 张新友. 作物分子标记辅助选择育种的现状与展望. 江苏农业科学, 2018, 46(5): 6–12. Wang Y Q, Sun Z Q, Zheng Z, Huang B Y, Dong W Z, Tang F S, Zhang X Y. Current situation and prospect of crop molecular marker-assisted selection breeding., 2018, 46(5): 6–12 (in Chinese).
[12] 姜慧芳, 陈本银, 任小平, 廖伯寿, 雷永, 傅廷栋, 马朝芝, Mace E, Crouch J H. 利用重组近交系群体检测花生青枯病抗性SSR标记. 中国油料作物学报, 2007, 29: 26–30. Jiang H F, Chen B Y, Ren X P, Liao B S, Lei Y, Fu T D, Ma C Z, Mace E, Crouch J H. Identification of SSR markers linked to bacterial wilt resistance of peanut with RILs., 2007, 29: 26–30 (in Chinese with English abstract).
[13] 彭文舫, 姜慧芳, 任小平, 吕建伟, 赵新燕, 黄莉. 花生AFLP遗传图谱构建及青枯病抗性QTL分析. 华北农学报, 2010, 25(6): 81–86. Peng W F, Jiang H F, Ren X P, Lyu J W, Zhao X Y, Huang L. Construction of AFLP genetic linkage map and detection of QTLs for bacterial wilt resistance in peanut (L.)., 2010, 25(6): 81–86 (in Chinese with English abstract).
[14] 任小平. 花生青枯病抗性的分子标记研究. 中国农业科学院硕士学位论文, 北京, 2005. Ren X P. A Study on Molecular Marker for Resistance to Bacterial Wilt in Peanut (L.). MS Thesis of Chinese Academy of Agricultural Sciences, Beijing, China, 2005 (in Chinese with English abstract).
[15] Wang L, Zhou X, Ren X, Huang L, Luo H, Chen Y, Chen W, Liu N, Liao B, Lei Y, Yan L, Shen J, Jiang H. A major and stable QTL for bacterial wilt resistance on chromosome B02 identified using a high-density SNP-based genetic linkage map in cultivated peanut Yuanza 9102 derived population., 2018, 9: 652.
[16] Luo H, Pandey M K, Khan A W, Wu B, Guo J, Ren X, Zhou X, Chen Y, Chen W, Huang L, Liu N, Lei Y, Liao B, Varshney R K, Jiang H. Next-generation sequencing identified genomic region and diagnostic markers for resistance to bacterial wilt on chromosome B02 in peanut (L.)., 2019, 17: 2356–2369
[17] Zhang C, Chen H, Cai T, Deng Y, Zhuang R, Zhang N, Zeng Y, Zheng Y, Tang R, Pan R, Zhuang W. Overexpression of a novel peanut NBS-LRR geneenhances disease resistance toin tobacco., 2016, 15: 39–55.
[18] Zhang C, Chen H, Zhuang R, Chen Y, Deng Y, Cai T, Wang S, Liu Q, Tang R, Shan S, Pan R, Chen L, Zhuang W. Overexpression of the peanut CLAVATA1-like leucine-rich repeat receptor-like kinase AhRLK1, confers increased resistance to bacterial wilt in tobacco., 2019, 70: 5407–5421.
[19] Fonceka D, Tossim H A, Rivallan R, Vignes H, Faye I, Ndoye O, Moretzsohn M C, Bertioli D J, Glaszmann C, Courtois B, Rami J F. Fostered and left behind alleles in peanut: interspecific QTL mapping reveals footprints of domestication and useful natural variation for breeding., 2012, 12: 26.
[20] Luo H, Ren X, Li Z, Xu Z, Li X, Huang L, Zhou X, Chen Y, Chen W, Lei Y, Liao B, Pandey M K, Varshney R K, Guo B, Jiang X, Liu F, Jiang H. Co-localization of major quantitative trait loci for pod size and weight to a 3.7 cM interval on chromosome A05 in cultivated peanut (L.)., 2017, 18: 58.
[21] 李振动, 李新平, 黄莉, 任小平, 陈玉宁, 周小静, 廖伯寿, 姜慧芳. 栽培种花生荚果大小相关性状QTL定位. 作物学报, 2015, 41: 1313–1323. Li Z D, Li X P, Huang L, Ren X P, Chen Y N, Zhou X J, Liao B S, Jiang H F. Mapping of QTLs for pod size related traits in cultivated peanut (L.)., 2015, 41: 1313–1323 (in Chinese with English abstract).
[22] Huang L, He H, Chen W, Ren X, Chen Y, Zhou X, Xia Y, Wang X, Jiang X, Liao B, Jiang H. Quantitative trait locus analysis of agronomic and quality-related traits in cultivated peanut (L.)., 2015, 128: 1103–1115.
[23] Chen Y, Ren X, Zheng Y, Zhou X, Huang L, Yan L, Jiao Y, Chen W, Huang S, Wan L, Lei Y, Liao B, Huai D, Wei W, Jiang H. Genetic mapping of yield traits using RIL population derived from Fuchuan Dahuasheng and ICG6375 of peanut (L.)., 2017, 37: 17.
[24] 蔡岩, 徐志军, 李振动, 李新平, 郭建斌, 任小平, 黄莉, 陈伟刚, 陈玉宁, 周小静, 罗怀勇, 姜慧芳. 花生出仁率QTL分析及其与荚果大小的相关性. 作物学报, 2017, 43: 701–707. Cai Y, Xu Z J, Li Z D, Li X P, Guo J B, Ren X P, Huang L, Chen W G, Chen Y N, Zhou X J, Luo H Y, Jiang H F. Quantitative trait locus analysis for shelling percentage and correlation between shelling percentage and pod size related traits in., 2017, 43: 701–707 (in Chinese with English abstract).
[25] 周小静, 董洋, 张芳, 任小平, 陈玉宁, 黄莉, 陈伟刚, 廖伯寿, 雷永, 晏立英, 罗怀勇, 姜慧芳. 利用SNP标记高密度遗传图谱进行花生出仁率QTL定位. 中国油料作物学报, 2016, 38: 750–756. Zhou X J, Dong Y, Zhang F, Ren X P, Chen Y N, Huang L, Chen W G, Liao B S, Lei Y, Yan L Y, Luo H Y, Jiang H F. QTL mapping of shelling percentage using SNP-based high density genetic map in cultivated peanut., 2016, 38: 750–756 (in Chinese with English abstract).
[26] Luo H, Xu Z, Li Z, Li X, Lv J, Ren X, Huang L, Zhou X, Chen Y, Yu J, Chen W, Lei Y, Liao B, Jiang H. Development of SSR markers and identification of major quantitative trait loci controlling shelling percentage in cultivated peanut (L.)., 2017, 130: 1635–1648.
[27] 陈伟刚, 郭建斌, 徐志军, 喻博伦, 邱西克, 黄莉, 宋延滨, 陈玉宁, 周小静, 罗怀勇, 刘念, 任小平, 姜慧芳. 花生出仁率和株高的QTL定位分析. 作物学报, 2018, 44: 42–51. Chen W G, Guo J B, Xu Z J, Yu B L, Qiu X K, Huang L, Song Y B, Chen Y N, Zhou X J, Luo H Y, Liu N, Ren X P, Jiang H F. QTL Mapping for shelling percentage and plant height in cultivated peanut (L.)., 2018, 44: 42–51 (in Chinese with English abstract).
[28] Yang J, Hu C, Hu H, Yu R, Xia Z, Ye X, Zhu J. QTLNetwork: mapping and visualizing genetic architecture of complex traits in experimental populations., 2008, 24: 721–723.
[29] 任小平, 姜慧芳, 廖伯寿. 花生抗青枯病分子标记研究. 植物遗传资源学报, 2008, 9: 163–167. Ren X P, Jiang H F, Liao B S. Identification of molecular markers for resistance to bacterial wilt in peanut (L.)., 2008, 9: 163–167 (in Chinese with English abstract).
Development of novel peanut genotypes with resistance to bacterial wilt disease, large pod, and high shelling percentage
LI Wei-Tao, XU Zhi-Jun, CAI Yan, GUO Jian-Bin, YU Bo-Lun, HUANG Li, CHEN Yu-Ning, ZHOU Xiao-Jing, LUO Huai-Yong, LIU Nian, CHEN Wei-Gang, REN Xiao-Ping, and JIANG Hui-Fang*
Oil Crops Research Institute, China Academy of Agricultural Sciences / Key Laboratory of Biology and Genetic Improvement of Oil Crops, Ministry of Agriculture and Rural Affairs, Wuhan 430062, Hubei, China
Bacterial wilt is an important soil-borne bacterial disease effecting yield and quality of peanut. Hundred pod weight and shelling percentage are also related to peanut yield. In this study, the major QTLrelated to bacterial wilt resistance was identified by using the RIL population derived from thecross between Yuanza 9102 and Xuzhou 68-4. Combining with the QTL mapping results for shelling percentage and hundred pod weight in previous research, it was found that the major QTL for these three traits were distributed on different chromosomes. Based on genotype data of RIL population and phenotypic data of bacterial wilt resistance, hundred pod weight and shelling percentage in multi-environment, We screened out six novel accessions with resistance to bacterial wilt disease, large pod, and high shelling percentage from RIL population by molecular markers closely linked with major QTL, which can be used as breeding intermediates or parents in peanut breeding for high yield and high disease resistance. The results of effectively screening disease-resistant and high-yield germplasm through the combination of molecular marker-assisted selection and phenotypic identification provide a new idea for peanut breeding in the future.
novel peanut genotype; bacterial wilt diseases resistance; hundred pod weight; shelling percentage
2019-08-04;
2019-12-26;
2020-01-15.
10.3724/SP.J.1006.2020.94112
姜慧芳, E-mail: peanutlab@oilcrops.cn, Tel: 027-86711550
E-mail: 965301631@qq.com
本研究由国家自然科学基金项目(31601340, 31571713, 31761143005, 31461143022)资助。
This study was supported by the National Natural Science Foundation of China (31601340, 31571713, 31761143005, 31461143022).
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20200115.1110.012.html
