盐胁迫下水稻体内miRNA表达谱分析
2020-03-15张秀妍徐妙云郑红艳邹俊杰张笑宇王磊
张秀妍,徐妙云,郑红艳,邹俊杰,张笑宇,王磊*
(1.内蒙古农业大学园艺与植物保护学院, 呼和浩特 010018;2.中国农业科学院生物技术研究所, 北京 100081)
高盐胁迫是水稻生产中常见的非生物逆境,严重影响了水稻的生长发育和最终产量[1]。目前,提高植物耐盐性的途径主要有两种:一是改良和治理盐土,其次是深入探究盐胁迫对植物的伤害机制,挖掘植物应答盐胁迫的重要基因,通过分子育种手段培育耐盐品种。
miRNA是一类广泛存在于真核生物体内,长度为21~25 nt的单链非编码小RNA分子,参与基因的转录后调控,通过与靶mRNA完全匹配或者部分匹配的方式将靶基因降解或抑制靶基因的翻译[2-6]。大量研究发现,miRNA在植物体内应对非生物胁迫过程中发挥着重要作用,这也是近年来研究的焦点[7-11]。盐胁迫下,水稻叶片中Os-miR414、Os-miR408和Os-miR164e下调表达[12],Os-miR29上调表达[13]。这些miRNA通过与靶基因作用对植物的生长发育进行调控,进而应对环境中的盐胁迫。并且同一种miRNA在不同处理条件下表达趋势不同。例如,在水稻中,Os-miR393在盐[14]、干旱[15]、低温[14]处理后均上调表达,在铝胁迫中[16]下调表达;Os-miR319a在脱落酸处理后下调表达,在赤霉素处理后上调表达[17]。
通过高通量测序可以在全基因组水平上鉴定生物体内小RNA的表达谱、挖掘新的miRNA分子、预测靶基因、鉴定样品间的差异表达。在植物遭受非生物逆境胁迫期间,必然会应激表达一些miRNA,这些miRNA的靶基因大多是转录因子,往往参与一些重要的信号途径。但是,在盐胁迫处理不同时间,植物体内应激所产生miRNA的类型及表达量变化如何尚需研究,因此,本文利用高通量测序及实时荧光定量PCR(quantitative real-time PCR,qRT-PCR)方法,研究水稻幼苗在盐胁迫处理下不同时间点差异miRNA及其靶基因的表达模式,以期为耐逆miRNA的发现和鉴定提供理论依据。
1 材料与方法
1.1 试验材料
1.1.1供试水稻材料 本研究所用水稻材料为OsPIR1(LOC_4334762)基因沉默材料,背景为日本晴,来源于滚环复制介导发卡RNA法构建的RNAi突变体库[18]。
1.1.2试剂与试剂盒 EASYspin Plus多糖多酚复杂植物RNA快速提取试剂盒购自杭州昊鑫生物科技股份有限公司,用于RNA提取;VAHTS mRNA-seq V3 Library Prep Kit for Illumina试剂盒购自NEB公司,用于sRNA文库构建;Mon ScriptTM5×RT Ⅲ All-in-One Mix试剂盒购自莫纳公司,用于将RNA反转录为cDNA第一链;SYBR GreenTMPremixExTaqTM试剂盒购自TaKaRa公司,用于荧光定量检测基因表达。
1.2 水稻材料的培养与处理
选取当年收获的饱满水稻种子置于三角瓶中,于光照培养箱37 ℃培养一周打破休眠后,先用75%乙醇消毒3 min,无菌水洗一遍,然后用2.5%的次氯酸钠消毒30~60 min,最后用无菌水浸泡1~2 h。将已消毒的水稻种子置于酸性花肥土中用小花盆土培,每盆15株。光照培养箱温度为28 ℃,光周期为16 h光照/8 h黑暗,相对湿度为60%~80%,适时补水。待苗长至二叶期时,挑选大小一致的幼苗进行盐胁迫处理。
盐处理:选取长势一致的水稻植株150株,加入终浓度为150 mmol·L-1的盐水,补水均使用终浓度为150 mmol·L-1的盐水。在处理0、24、72 h分别对水稻根系、茎干及叶片取样,样品共分9组,分别命名为0 h-R(Root)、0 h-S(Stem)、0 h-L(Leaf)、24 h-R、24 h-S、24 h-L、72 h-R、72 h-S、72 h-L。样品立刻置于液氮中,保存于-80 ℃冰箱中备用。
1.3 小RNA文库构建
分别提取9组样品的总RNA,要求所有样品浓度 ≥ 100 ng·μL-1,总量 ≥10 μg,RIN ≥6.0,A260/A280>2.1,保证使用合格的样品进行测序。样品检测合格后,以总RNA为起始样品,分离得到18~30 nt的目的片段,利用T4 RNA连接酶分别在小RNA 3′端和5′端连接上接头,反转录合成cDNA,PCR扩增后构建sRNA文库。将文库构建好后,使用Illumia Solexa测序技术进行高通量测序,测序读长为single-end(SE)50 nt,测序工作由北京贝瑞和康生物技术有限公司完成。
1.4 已知miRNA的获取
使用Illumia Solexa高通量测序技术获得小RNA的原始数据(raw reads)。原始数据包括低质量读数、污染序列等,需将其清理获得clean reads序列,使用水稻基因组(Oryza_sativa.IRGSP-1.0)作为参考基因组进行序列比对,最后将比对上参考基因组的reads序列与已知miRNA数据库miRBase(v22.1)(http://www.mirbase.org/)中的成熟miRNA序列进行比对,与数据库序列完全相同的reads被认为是已知miRNA。
1.5 差异表达miRNA分析
将0 h-R/S/L分别与24 h-R/S/L、72 h-R/S/L对应的组织比较;24 h- R/S/L分别与72 h-R/S/L对应的组织比较,判断两组间已知miRNA表达量是否存在显著差异,使用R package edgeR软件[19]进行差异分析。首先对每个样品中miRNA的read counts数采用TPM计算度量指标(transcript per million)[20]进行归一化处理,计算统计检验的P值,根据P值和log2(fold-change)的值进行筛选。选择|log2(fold-change)|>1,P值<0.05的miRNA认为是差异表达的miRNA。
1.6 靶基因预测及功能分类
利用psRNA target平台[21](http://plantgrn.noble.org/psRNA Target)以水稻基因组序列(Oryzasativa, taranscript, RAP-DB, version 1.0,https://rapdb.dna.affrc.go.jp/download/irgsp1.html)为候选靶基因库,采用默认参数对本实验中差异表达的miRNA进行靶基因在线预测。然后使用The plant GSEA平台[22](http://structuralbiology.cau.edu.cn/PlantGSEA/tools.php)对靶基因进行GO(Gene Ontology)分类,可获得靶基因在与其相对应的mRNA序列中的分类信息。
1.7 实时荧光定量PCR(qRT-PCR)
挑选24与72 h表达趋势相同的miRNA进行qRT-PCR验证,利用Trizol法提取盐胁迫72 h组和对照组水稻根部总RNA,将总RNA反转录为cDNA第一链,之后对miRNA及靶基因进行荧光定量检测,根据TaKaRa公司的SYBR GreenTMPremixExTaqTM试剂盒说明书配置20 μL反应体系:cDNA模板2 μL,上、下游引物(表1)各0.4 μL,SYBR PremixExTaq(2×)10 μL,ROX Reference Dye (50×)0.4 μL,ddH2O 6.8 μL。

表1 qRT-PCR所使用引物序列Table 1 Primer sequences used in qRT-PCR
反应程序为:预变性94 ℃ 30 s;变性94 ℃ 5 s,退火55 ℃ 15 s,延伸72 ℃ 45 s,40个循环。每个样品3个生物学重复,所用引物均为miRNA前体序列特异引物,内参基因使用水稻Osactin2(XM_015759418.2),由生工生物(上海)有限公司合成。相对表达量计算方法采用2-ΔΔCT方法[23]。
2 结果与分析
2.1 不同组织盐处理条件下的小RNA高通量测序
为了分析水稻幼苗在高盐条件处理后表型变化的差异情况,使用浓度为150 mmol·L-1的氯化钠溶液对二叶期的水稻幼苗进行处理,处理24 h时水稻幼苗出现轻微萎蔫状态,而72 h时水稻幼苗则表现出了严重的萎蔫状态(图1)。为了分析表型变化所产生的内在原因,分别对盐处理24 h和72 h水稻幼苗的根、茎、叶组织进行取样及小RNA高通量测序。将测序后所产生的原始序列整理,去除低质量序列、过滤核糖体RNA等序列,0 h-R/S/L组分别产生1.91×107、1.69×107和2.45×107条原始序列以及1.40×107、1.24×107和1.49×107条Clean reads;24 h-R/S/L组分别产生1.70×107、1.99×107和2.19×107条原始序列以及1.28×107、1.26×107和9.69×106条Clean reads;72 h-R/S/L组分别产生3.14×107、1.62×107和3.87×107条原始序列以及2.21×107、8.99×106和2.03×107条Clean reads(表2)。

表2 水稻小RNA测序数据Table 2 Summary of small RNA data produced by solexa sequencing from rice

图1 盐胁迫水稻幼苗表型差异Fig.1 Phenotype in rice seedling under salt stress
2.2 水稻中小RNA的丰度变化
经150 mmol·L-1盐处理后,对不同时间点水稻不同组织中小RNA丰度的变化趋势进行分析,结果(图2)表明,已知miRNA的表达变化主要分四类:①受到盐胁迫后,随着处理时间的延长,水稻幼苗根部、茎部和叶片24 nt小RNA的丰度均呈下降趋势;②随着处理时间的延长,根和茎中21 nt的小RNA丰度呈先上升后下降趋势,而叶片中21 nt小RNA则持续下降;③随着处理时间的延长,根和叶中30 nt小RNA丰度呈持续上升趋势,茎中则表现为先下降后上升趋势;④在0 h-R/S/L组和24 h-R/S/L组中24 nt小RNA含量最高;在72 h-R/S/L组中则21 nt小RNA含量最高。
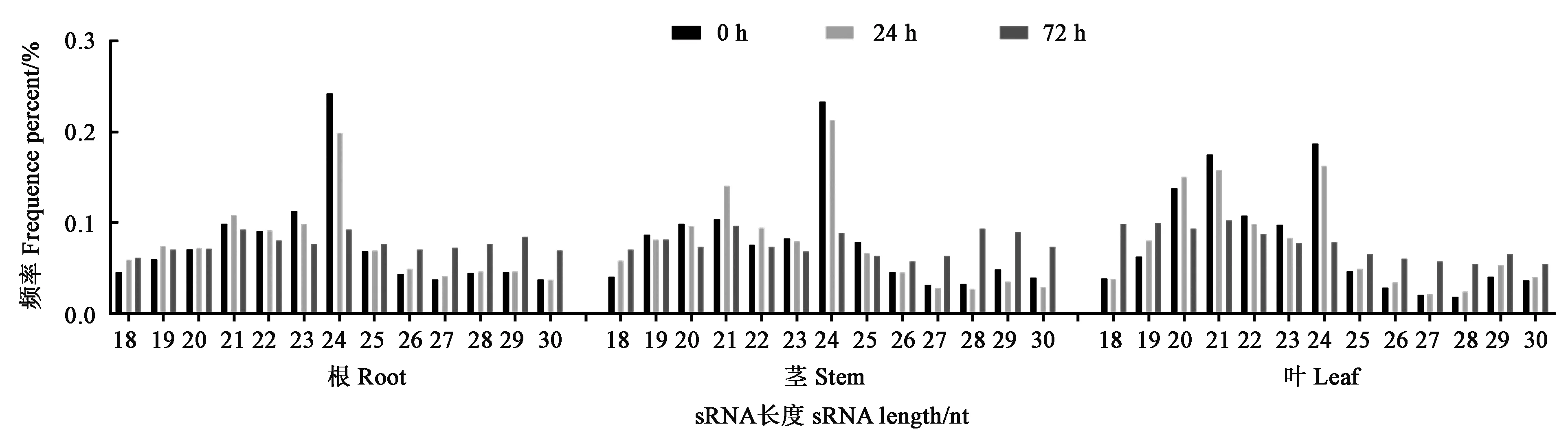
图2 不同处理时间内水稻幼苗根、茎和叶中小RNA序列长度分布Fig.2 Length distribution of the sRNAs in rice root, stem and leaf at different treatment time
一般来说,小RNA的长度区间为18~30 nt,miRNA集中在20~23 nt,siRNA(small interfering RNA)集中在20~24 nt,30 nt的小RNA在动物中主要是piRNA(piwi-interacting RNA),植物中的功能还未见相关报道。
2.3 差异表达的已知miRNA
将测序结果与miRBase数据库中(http://www.mirbase.org)水稻miRNA序列比对,0 h-R/S/L、24 h-R/S/L和72 h-R/S/L共鉴定出248个已知miRNA,来源于132个miRNA家族。其中来自于29个miRNA家族的51个miRNA在盐处理后发生显著的差异表达[|log2(fold-change)|]>1,P值<0.05)。进一步对在不同组织间差异表达miRNA的变化趋势和表达谱进行分析,为miRNA在高盐条件下的功能和作用机制的阐明奠定基础。
2.3.1根部差异表达miRNA 在150 mmol·L-1盐处理条件下,水稻根部有31个差异表达的miRNA(图3),根据差异miRNA表达量随时间的变化趋势,将其分为四类。第一类包含6个miRNA:Osa-miR444a-5p、Osa-miR530-5p、Osa-miR1878、Osa-miR5156、Osa-miR5788和Osa-miR5827,它们在24 h内表达量迅速降低,并在24至72 h不再变化;第二类包含17个miRNA:Osa-miR156k、Osa-miR169h、Osa-miR169i-5p.1、Osa-miR169j、Osa-miR169k、Osa-miR169l、Osa-miR169m、Osa-miR396c-5p、Osa-miR396e-5p、Osa-miR396f-5p、Osa-miR397a、Osa-miR397b、Osa-miR408-5p、Osa-miR1425-5p、Osa-miR2871a-5p、Osa-miR2878-5p和Osa-miR11339-5p,它们的表达量在72 h内持续降低;第三类包含5个miRNA:Osa-miR166a-5p、Osa-miR166e-5p、Osa-miR1846d-5p、Osa-miR1850.1和Osa-miR3979-5p,它们的表达量在24 h内迅速升高,在24 h至72 h内逐渐下降;第四类包含3个miRNA:Osa-miR166b-5p、Osa-miR166h-5p和Osa-miR166k-5p,它们的表达量在72 h内逐渐升高。

图3 盐胁迫下水稻根中差异表达miRNAFig.3 Differentially expressed miRNA in rice root under salt stress
差异表达的miRNA家族中,Osa-miR166家族和Osa-miR169家族成员中发生变化的成员最多,其中Osa-miR169家族的6个成员表达量下降,而Osa-miR166家族的5个成员表达量升高,推测这两个家族在根中响应盐胁迫方面起重要作用。
2.3.2茎部差异表达miRNA 茎部共有11个差异表达的miRNA(图4),分为三类。第一类包含2个miRNA:Osa-miR3979-5p和Osa-miR5795,它们在24 h内表达量迅速降低,并在24至72 h不再变化;第二类包含4个miRNA:Osa-miR166h-5p、Osa-miR171d-5p、Osa-miR171e-5p和Osa-miR528-5p,它们的表达量在24 h内迅速升高,在24 h至72 h内逐渐下降;第三类包含5个miRNA:Osa-miR5801a、Osa-miR5801b、Osa-miR814a、Osa-miR814b和Osa-miR814c,它们的表达量在72 h内持续降低。

图4 盐胁迫下水稻茎中差异表达miRNAFig.4 Differentially expressed miRNA rice instem under salt stress
对茎部差异表达的miRNA进一步分析发现,随着处理时间的增加,大部分miRNA的表达量显著下降,例如Osa-miR5801和Osa-miR814家族成员等;表达量显著升高的有Osa-miR166h和Osa-miR171家族部分成员等。结合根部分析结果可证实Osa-miR166家族对盐胁迫强烈响应。
2.3.3叶部差异表达miRNA 叶部共有9个差异表达的miRNA(图5),分为四类。第一类包含2个miRNA:Osa-miR5801b、Osa-miR812o-5p,表达量在24 h内迅速降低,在24至72 h内逐渐升高;第二类为Osa-miR2103,表达量在24 h内迅速升高,在24~72 h内逐渐下降;第三类包含5个miRNA:Osa-miR814a、Osa-miR814b、Osa-miR814c、Osa-miR5145、Osa-miR1432-5p,它们的表达量在72 h内迅速降低;第四类为Osa-miR5150-5p,在24 h内表达量迅速降低,并在24至72 h不再变化。叶部差异表达miRNA中,除Osa-miR2103显著上调表达外,其余均显著下调表达。
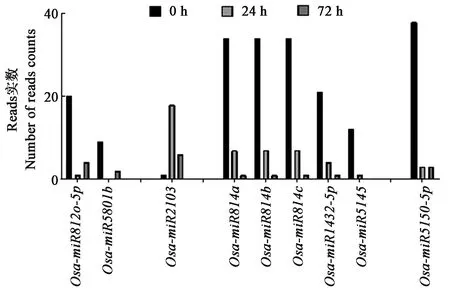
图5 盐胁迫下水稻叶中差异表达miRNAFig.5 Differentially expressed miRNA in rice leaf under salt stress
对比地上部与地下部中差异miRNA,发现有3个miRNA在根与茎中表达趋势不同:Osa-miR3979-5p在根中表达量先升高后降低,在茎中显著降低;Osa-miR166h-5p在根中表达量显著升高,在茎中升高后降低;Osa-miR5801b在茎中表达量持续降低,在叶中表现为先降低后升高。Osa-miR814家族的三个成员(Osa-miR814a、Osa-miR814b和Osa-miR814c)在茎与叶中的表达模式相同,均表现为持续降低。 说明同一miRNA在不同组织部位表达量不尽相同。
2.4 miRNA的靶基因预测与GO分类
通过psRNA target靶基因在线预测平台[21]对本实验中差异表达的miRNA进行靶基因在线预测,51个差异miRNA预测到592个符合筛选条件的靶基因。差异miRNA的靶基因主要涉及各种生理代谢进程的转录因子,如SPL、GRF、SAP、HD-ZIP、NF-YA、Laccase等。
使用The plant GSEA在线富集分析平台[22],对靶基因进行GO分类(图6),结果表明:差异表达的miRNA所对应的靶基因在细胞组分中显著富集于膜相关细胞器;在分子功能中显著富集于核糖核苷酸结合、ATP结合等过程;在生物学过程中,靶基因显著富集于含氧酸代谢、磷代谢、氮代谢等过程。
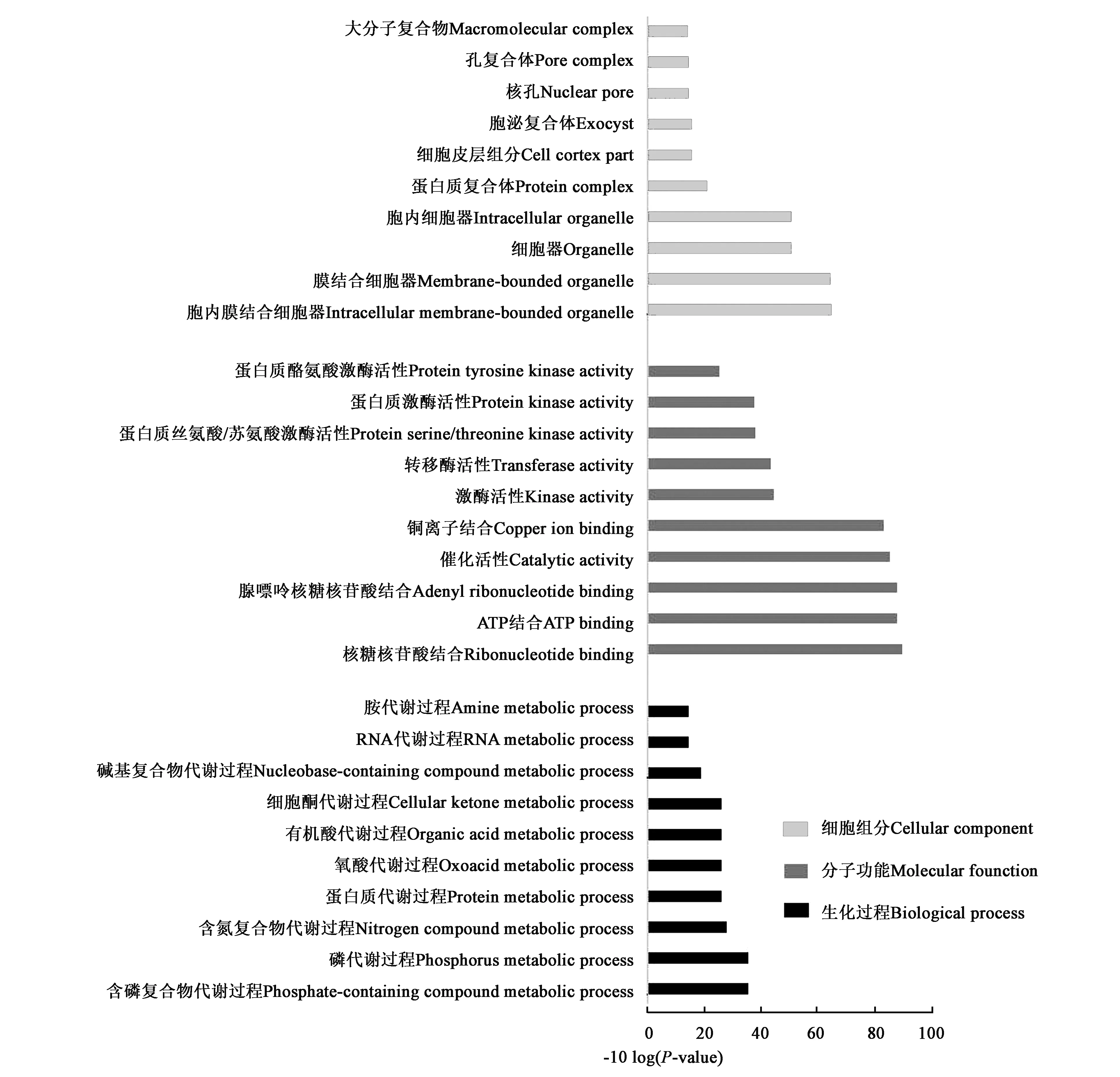
图6 miRNA靶基因GO分类Fig.6 GO classification of known-miRNA target genes
2.5 荧光定量分析差异基因
2.5.1miRNA验证 为了进一步验证逆境相关miRNA的表达,使用qRT-PCR方法分析5个在24和72 h内根部表达量趋势相同的盐胁迫响应差异miRNA基因(图7),分别来源于差异变化倍数较大的miR166家族、miR169家族和miR530家族。结果表明:盐胁迫72 h水稻幼苗根部Osa-miR166k显著上调;Osa-miR169h和Osa-miR530略下调;Osa-miR166h略上调,与高通量测序结果一致。但是Osa-miR166b表达略下调,与高通量测序结果相反。

图7 5个盐胁迫响应miRNAs相对表达量检测Fig.7 Detection of relative expression of 5 salt stress responsive miRNAs
2.5.2靶基因验证 测序结果显示24 h-R与72 h-R组中Osa-miR169家族成员变化趋势相同,且Osa-miR169家族在植物生长发育以及抵抗非生物胁迫过程中起到了重要的调节作用,在水稻中,Osa-miR169家族的靶基因NF-YA已有较为深入的研究[24]。挑选由psRNA target[21]平台在线预测到水稻Osa-miR169h的候选靶基因,对其中4个分值最高的NF-YA家族基因OsNF-YA1(Os03g0174900)、OsNF-YA2(Os03g0411100)、OsNF-YA4(Os03g0696300)和OsNF-YA10(Os12g0618600)进行qRT-PCR分析。结果表明,Osa-miR169h的候选靶基因OsNF-YA4的表达量与对照相比无显著差异,而OsNF-YA1、OsNF-YA2、OsNF-YA10在受到盐胁迫72 h水稻根部上调表达,这与Osa-miR169h的表达下调结果相一致。

图8 4个靶基因相对表达量检测Fig.8 Detection of relative expression of 4 target genes
3 讨论
miRNA在植物中几乎涉及到生长、发育、代谢调控等各个方面,包括种子萌发[25]、活性氧清除[26]、激素信号转导[27]、植物器官形态发育[28]以及逆境胁迫应答[29]等。近年来,人们相继揭示了水稻[30]、拟南芥[31]、苜蓿[32]、玉米[33]等作物盐胁迫下miRNA的差异表达模式,并探讨了相关miRNA在植物盐胁迫中的调控机制。本研究结果显示,盐胁迫下,水稻Osa-miR156k、Osa-miR169h/i/j/k/l/m、Osa-miR396c、Osa-miR396e、Osa-miR396f、Osa-miR408、Osa-miR530、Osa-miR397a、Osa-miR397b表达量下调,这与miR156在棉花[34]、玉米[35]、柳枝稷[36],miR169、miR396在互花米草[37],Osa-miR408在水稻[12]、柳枝稷[36]、miR530在毛果杨[38],miR397在柳枝稷[39]中下调结果一致;而水稻Osa-miR166a在盐胁迫后表达量下调,这与miR166在拟南芥[40]、玉米[41]中上调表达结果相反。研究还发现,Osa-miR171e、Osa-miR171d在盐处理后表达量先升高后降低,这与miR171在互花米草中表达量降低结果不完全相同[37]。这说明在不同植物中,miRNA的表达不仅具有时空特异性,其应答盐胁迫的方式也不尽相同。
本研究发现,差异表达miRNA在根中数量最多,茎中其次,叶中最少,推测其可能与水稻不同部位受到盐胁迫的接触方式有关,根系作为水稻直接感受盐分变化的器官,其对盐胁迫反应更加敏感。而茎是植物体的中轴部分,具有疏导营养物质和水分的作用,水分通过维管束中的导管到达叶片。在高盐浓度处理下的植物中的水势逐渐降低,推测地上部miRNA差异表达可能与水分胁迫有关。
本研究差异表达miRNA的靶基因的预测结果显示,Osa-miR136、Osa-miR166、Osa-miR169、Osa-miR171、Osa-miR396、Osa-miR397家族的靶基因与其他物种中已经验证的靶基因相似,其靶基因所编码的SPL、HD-ZIP、NF-YA、SCL/GRAS、GRL、Laccase作为基因调控的关键转录因子可能通过控制广泛的下游基因来响应生物和非生物胁迫。Osa-miR156k的靶基因为SPL转录因子[42],是植物特有的一类基因家族,迄今为止,已发现SPL可调节多个重要的生物过程,如叶片发育[43]、孢子形成[44]、响应GA信号[45]等,在水稻中过表达Osa-miR156导致SPL基因表达降低,继而影响了在花青素途径中的DFR基因,最终使得转基因植株的耐盐性有所提高[31]。同时,也预测到水稻Osa-miR397a、Osa-miR397b的靶基因Laccase编码的蛋白有可能与非生物胁迫直接相关。研究表明在低温条件下,拟南芥中过表达miR397a可增强转基因植株对低温胁迫的抗性[46]。Ma等[47]发现,在盐胁迫下,拟南芥中的miR171作用于靶基因GRAS,通过改变植株根系miRNA的表达情况来抵御盐胁迫。miR166的靶基因为植物特异性HD-ZIP转录因子[48],包括四个亚家族HD-ZIPⅠ~Ⅳ,其成员通过与其他蛋白互作或者参与激素介导的信号途径,调控植物的光信号传导[49]、物质的积累及运输[50]、逆境应答[51]等生物学过程。对HD-ZIP蛋白的研究表明,HD-ZIPⅠ和Ⅱ亚家族相关基因的表达受ABA、干旱以及盐胁迫的诱导[52],如水稻中HD-ZIPⅠ家族OsHOX24基因的启动子融合OsNAC基因的过表达水稻可提高水稻的耐盐性及耐旱性[53],充分说明miR166家族可响应盐胁迫。miR169的靶基因为NF-YA转录因子,前人的研究工作已经证实水稻Osa-miR169家族中Osa-miR169g和Osa-miR169o可强烈响应盐胁迫[24],并且在盐胁迫处理后Osa-miR169在水稻中存在时序性和组织特异性表达[30],而过表达miR169使得拟南芥抵御干旱和盐胁迫的能力增强[24],说明NF-YA在响应干旱及盐胁迫应答过程中具有重要作用。本研究对Osa-miR169h的靶基因NF-YA家族的四个成员进行转录水平qRT-PCR验证,结果表明,OsNF-YA1、OsNF-YA2、OsNF-YA10在受到盐胁迫72 h水稻根部上调表达,与Osa-miR169h的表达呈现相反趋势。但有关这些差异表达miRNA是如何调控其靶基因,它们的靶基因在抗盐相关通路中的作用机制仍待进一步研究。
