猪细小病毒7型Cap基因原核表达与生物信息学分析
2020-03-07王小朋刘自敏白彩霞杨侃侃蒋书东李永东
王小朋,赵 靓,刘自敏,白彩霞,杨侃侃,张 达,孙 裴,蒋书东,李永东,
王 勇1,*
(1.安徽农业大学 动物科技学院,安徽 合肥 230036;2.宁波市疾病预防控制中心,浙江 宁波 315010)
猪细小病毒(porcine parvovirus,PPV)属于细小病毒科,是一种单股线性非囊膜DNA病毒[1],主要引起初产母猪流产、畸形胎、木乃伊胎、不孕不育,及新生仔猪的死亡,但是受感染的母猪本身无明显临床症状[2-3]。感染仔猪常出现生长受阻、皮炎、非化脓性心肌炎和消瘦性综合征[4-5],该病广泛存在于世界各地,给我国乃至全世界的养猪业造成了严重的经济损失。经典的PPV(称为PPV1)于1965年从德国的一种细胞培养污染物中分离得到[6],而后发展到世界多个国家和地区,是猪群中最常见的猪细小病毒型[7]。目前,随着分子生物学诊断技术的发展,在临床上陆续发现了6种新的细小病毒基因型(PPV2-PPV7)[8-13]。其中PPV7是Palinski等[13]在2016 年利用宏基因组测序技术在美国成年猪直肠拭子中首次获得的一种新的细小病毒。此后瑞典、波兰和韩国相继报道出PPV7潜在感染的病例[14-15]。我国学者Xing等[16]首次从广东省2个猪场的64份血清中检测出PPV7呈阳性,表明我国猪群中已有PPV7的存在。
PPV7基因组大小约为4 103 bp,包括两个开放阅读框(open reading frame,ORF)、5′非编码区和3′非编码区。左侧ORF1 编码非结构蛋白(NS1),右侧ORF2编码结构蛋白(Cap)[17-19]。截至目前,PPV7的研究主要集中在分子遗传进化方面,其基因功能还未见文献报道。其中包括位于基因组保守区域的Cap基因,该基因编码PPV7的衣壳蛋白,与病毒的致病性有着密切的关系。本实验选择对PPV7Cap基因进行原核表达,同时对其进行生物信息学分析。该结果为进一步深入研究PPV7Cap基因的生物学功能提供了依据。
1 材料与方法
1.1 材料
PPV7/China/Ahmas毒株[20]由本实验室保存;pMDTM19-T载体、Solution Ⅰ及rTaqDNA聚合酶购自大连宝生物(TaKaRa)有限公司;病毒基因组DNA/RNA提取试剂盒、琼脂糖凝胶DNA回收试剂盒和DH5α感受态细胞购自天根生化科技(北京)有限公司;50×PBS缓冲液购自北京索莱宝科技有限公司;青霉素-链霉素溶液(100×)购自碧云天生物技术公司;抗GST标签的鼠源一抗和HRP标签的羊抗鼠IgG购自北京中杉金桥生物技术有限公司;DL 2 000 DNA Marker购自康为世纪生物科技有限公司;小量提取质粒试剂盒购自AXYGEN公司;其他试剂均为国产或进口分析纯。
1.2 方法
1.2.1 引物设计
根据NCBI中GenBank数据库已发表的PPV7 (登录号:KU563733)全基因序列,应用Primer Permier 5.0软件设计PPV7 Cap基因特异性引物。上游引物序列:5′-GGAATTCATGGCAGAACACATCACCCTGAGTAAC-3′(下划线处为EcoR Ⅰ酶切位点),下游引物序列:5′-GCGTCGACTTATTTTCGGCTGGTTGTTGTTGTG-3′(下划线处为和SalⅠ酶切位点),预期扩增产物长度为1 410 bp。引物由通用生物系统(安徽)有限公司合成。
1.2.2Cap基因的扩增
按照病毒基因组DNA/RNA提取试剂盒使用说明书提取DNA,并以提取的DNA为模板利用PCR技术进行扩增。PCR反应体系体积为20 μL:rTaqMix 10 μL,ddH2O 7 μL,上下游引物及模板各1 μL。反应程序:98 ℃预变性30 s;98 ℃变性10 s,60 ℃退火45 s,72 ℃延伸90 s,共30个循环;72 ℃再延伸10 min。PCR产物回收后克隆至pMDTM19-T载体,转化DH5α 感受态细胞,涂布至氨苄抗性的固体培养基,次日挑取阳性克隆菌株提取质粒,命名为pMDTM19-T-Cap,送至南京擎科生物科技有限公司测序。
1.2.3 重组质粒pGEX-6P-1-Cap的构建
将pMDTM19-T-Cap与pGEX-6P-1空载体质粒经内切酶EcoR Ⅰ和SalⅠ双酶切后电泳,目的产物胶回收后连接pGEX-6P-1载体,转化E.coliDH5α感受态细胞,涂布至氨苄抗性的固体培养基过夜培养,挑取阳性菌落提质粒,命名为pGEX-6P-1-Cap,双酶切鉴定后送到南京擎科生物科技有限公司测序。
1.2.4 重组蛋白的诱导表达及鉴定
将pGEX-6P-1-Cap与pGEX-6P-1空载体质粒分别转化E.coliRosetta感受态细胞,接种至氨苄抗性的固体培养基,37 ℃温箱培养过夜,按照1∶100比例接种Amp-LB液体培养基,37 ℃ 200 r·min-1培养,待D600值在0.6~0.8时,加入1 mmol·L-1的IPTG于37 ℃、180 r·min-1的条件下诱导表达,诱导时间分别设置为3、4、5和6 h,同时设置空载体菌诱导和未诱导对照。将诱导出的蛋白进行SDS-PAGE电泳验证。
1.2.5 Western-blot检测
取15 μL融合蛋白按照上述步骤进行SDS-PAGE电泳,凝胶半干转硝酸纤维素膜后,5%脱脂奶粉4 ℃封闭过夜,TBST洗涤3次;以鼠抗GST单克隆抗体作为一抗,37 ℃孵育1 h后TBST漂洗3次;加入1∶1 000稀释的辣根过氧化物酶(HRP)标记羊抗鼠IgG 为二抗,37 ℃孵育1 h后TBST漂洗3次。利用DAB试剂盒显色。
1.2.6 Cap蛋白的生物信息学分析
利用Protparam在线程序分析该毒株Cap蛋白的理化性质;利用SOPMA在线程序预测该蛋白的二级结构;用SWISS-MODEL在线程序预测该蛋白的三级结构;利用SingalP4.1在线程序预测该蛋白的信号肽;利用NetPhos3.1 Server在线程序预测该蛋白的磷酸化位点;利用TMHMM2.0 Server在线程序预测该蛋白的跨膜结构域;利用NetPhos3.1 Server在线程序预测该蛋白的磷酸化位点;利用DNAStar的Protean程序预测Cap蛋白的B细胞表位;利用nHLAPred的ComPred在线服务器预测Cap蛋白的CTL表位;利用ProPred在线服务器预测Cap蛋白的Th表位。
2 结果与分析
2.1 Cap基因的PCR扩增结果
以PPV7/China/Ahmas株基因组DNA为模板,经PCR扩增获得1条长度约为1 410 bp特异性条带(图1)。
2.2 重组质粒pGEX-6P-1-Cap的双酶切鉴定
将pGEX-6P-1-Cap经内切酶EcoR Ⅰ和SalⅠ双酶切后琼脂糖凝胶电泳,得到符合预期的条带(图2),测序表明重组质粒成功构建,碱基并未发生突变或缺失。
2.3 重组蛋白的SDS-PAGE分析
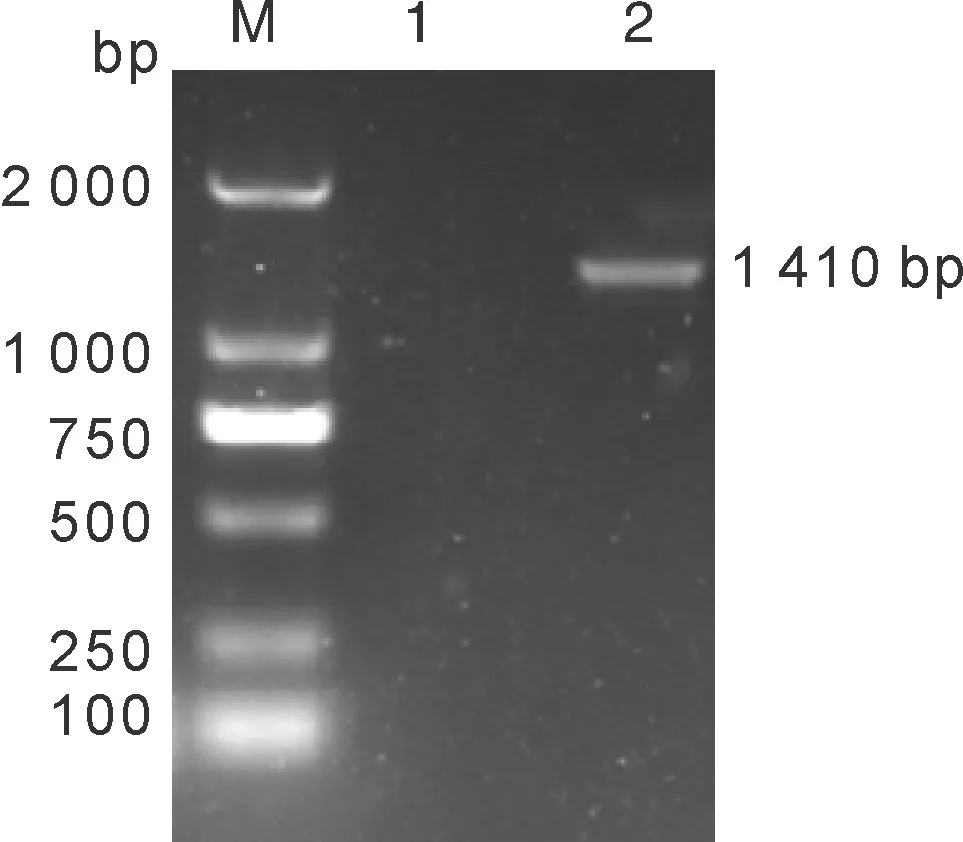
M,DL2000 DNA marker;1,阴性对照;2,Cap基因PCR产物。M, DL2000 DNA marker; 1, Negative control; 2,Cap gene PCR product.图1 Cap基因扩增产物Fig.1 PCR product of Cap gene

M,DNA marker;1,pGEX-6P-1-Cap重组质粒双酶切产物;2,pGEX-6P-1空载体阴性对照。M,DNA marker;1, pGEX-6P-1-Cap recombinant plasmid digested by double enzyme;2, pGEX-6P-1empty vector negative control.图2 重组质粒pGEX-6P-1-Cap双酶切鉴定Fig.2 Identification of recombinant plasmid pGEX-6P-1-Cap by double enzyme digestion
将鉴定正确的重组质粒pGEX-6P-1-Cap转化至E.coliRosetta感受态细胞进行SDS-PAGE 检测,可见分子质量约78 ku的特异性蛋白条带,pGEX-6P-1空载体表达对照组没有出现目的条带(图3)。
2.4 Western-blot 鉴定
利用Western-blot对蛋白进行进一步分析,结果显示,在硝酸纤维素膜上78 ku左右处有一明显条带,表明重组蛋白能被抗GST标签的一抗识别,具有良好的反应原性(图4)。
2.5 理化性质分析
利用Protparam在线程序分析该毒株Cap蛋白的理化性质,结果显示,该蛋白由469个氨基酸组成,分子式为C2473H3648N652O712S15,相对分子质量为54 385.01,理论pI(等电点)值为7.68,带负电荷的氨基酸(Asp+Glu)总数为42,带正电荷的氨基酸(Arg+Lys)总数为43,总原子数为7 500,不稳定系数为38.84,脂肪系数为56.57,总平均亲水值-0.701,为亲水性稳定蛋白。
2.6 二级结构分析
利用SOPMA在线程序预测Cap蛋白的二级结构(图5),结果显示,该蛋白二级结构包括α螺旋(h,11.09%),延伸链(e,21.96%),β转角(t,4.05%),无规则螺旋(c,62.90%)4种类型。
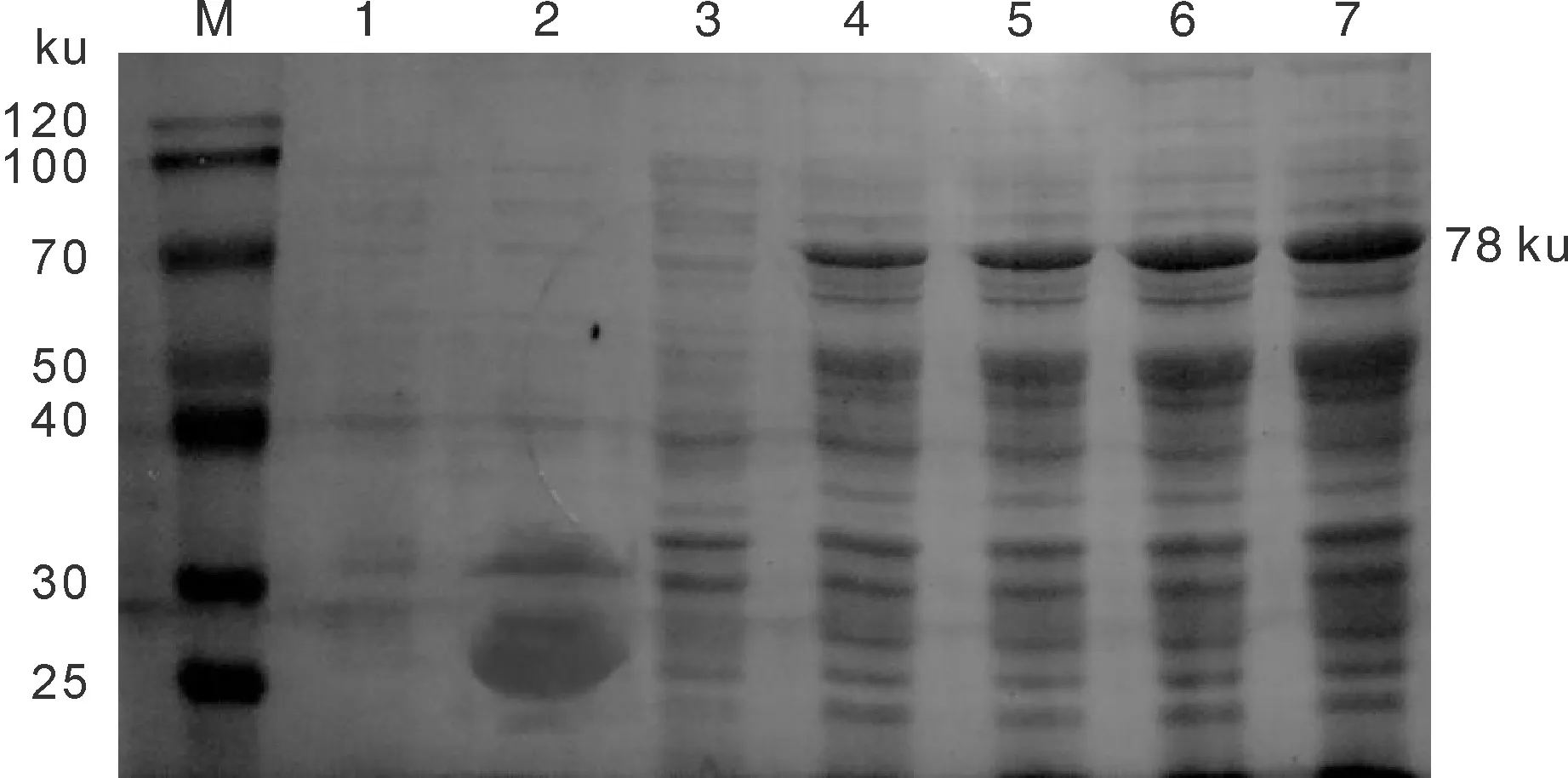
M,蛋白质分子质量标准;1,pGEX-6P-1空载体未经IPTG诱导;2,pGEX-6P-1空载体经IPTG诱导6 h;3,pGEX-6P-1-Cap未经IPTG诱导;4,pGEX-6P-1-Cap经IPTG诱导3 h;5,pGEX-6P-1-Cap经IPTG诱导4 h;6,pGEX-6P-1-Cap经IPTG诱导5 h;7,pGEX-6P-1-Cap经IPTG诱导6 h。M, Protein marker;1, pGEX-6P-1empty vector was not induced by IPTG;2, pGEX-6P-1empty vector was induced by IPTG for 6 h;3, pGEX-6P-1-Cap was not induced by IPTG;4, pGEX-6P-1-Cap was induced by IPTG for 3 h;5, pGEX-6P-1-Cap was induced by IPTG for 4 h;6, pGEX-6P-1-Cap was induced by IPTG for 5 h;7, pGEX-6P-1-Cap was induced by IPTG for 6 h.图3 pGEX-6P-1-Cap蛋白产物SDS-PAGE电泳Fig.3 SDS-PAGE electrophoresis analysis of the pGEX-6P-1-Cap protein product

M,蛋白质分子质量标准;1,pGEX-6P-1空载体经IPTG诱导表达;2,pGEX-6P-1-Cap重组质粒经IPTG诱导表达。M, Protein molecular weight marker;1, pGEX-6P-1empty vector was induced by IPTG;2, The recombinant plasmid pGEX-6P-1-Cap was induced by IPTG.图4 Western-blot鉴定Cap蛋白的体外表达Fig.4 Western-blot identification of Cap protein in vitro expression
2.7 三级结构
利用SWISS-MODEL在线程序对Cap蛋白的三级结构进行预测(图6),结果显示,所得结构有较多的无规则卷曲,与二级结构预测结果一致。
2.8 信号肽、跨膜结构域与磷酸化位点的预测分析
利用SignalP4.1在线程序和TMHMM2.0在线程序预测Cap蛋白的信号肽(图7)和跨膜结构域(图8),结果显示,Cap蛋白无信号肽和跨膜结构域。利用NetPhos3.1 Server在线程序预测Cap蛋白的磷酸化位点(图9),结果显示,该蛋白共有62个磷酸化位点,其中丝氨酸位点21个,苏氨酸位点31个,酪氨酸位点10个。
2.9 Cap蛋白B细胞表位预测
利用 DNAStar的Protean程序对Cap蛋白的B细胞潜在抗原表位进行预测(图10),结果显示,该蛋白亲水性的区域主要位于第12~41、51~64、66~72、105~125、140~155、167~200、208~242、251~297、322~352、380~393、415~469处,亲水性区段占有较大比例且整体分布比较均匀,说明蛋白亲水性较强。柔韧性的区域主要位于第17~19、23~32、53~56、70~76、80~85、89~93、105~114、121~122、132~152、168~171、177~188、192~198、209~224、232~236、253~258、263~274、283~295、326~334、337~350、359~369、384~389、394~397、418~424、429~441处,可见Cap蛋白有较多个柔性区域。抗原指数预测结果显示,蛋白中存在大量抗原指数较高的区域,表面可及性较高。结合该蛋白的二级结构、亲水性、柔韧性、抗原性和表面可及性进行综合分析(表1),该蛋白第148~149、179~182、192~196、211~213、223~224、232~234、269~270、283~285、293~294、337~340、421~423、436~437处可能存在优势抗原表位。

图5 Cap蛋白的二级结构Fig.5 Secondary structure of Cap protein

图6 Cap蛋白的三级结构Fig.6 Tertiary structure of Cap protein
2.10 T细胞表位的预测分析
2.10.1 CTL表位预测
利用nHLAPred的ComPred在线服务器,将阈值调节为0.5,分别预测HLA-A2、HLA-A*0201、HLA-A*0202、HLA-A*0203和HLA-A*0205五种不同分子的结合肽(表2),结果显示,第159~167位的YLWSAPIMA、305~313位的ILNAPIVPI可能存在Cap蛋白的CTL表位。
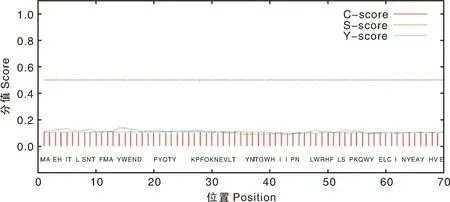
图7 PPV7 Cap蛋白的信号肽Fig.7 Signal peptide of PPV7 Cap protein
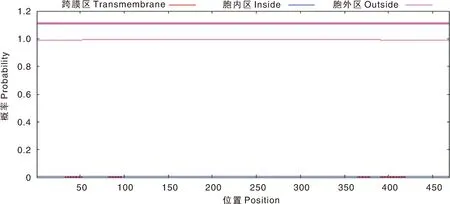
图8 PPV7 Cap蛋白的跨膜结构域Fig.8 Transmembrane domain of PPV7 Cap protein
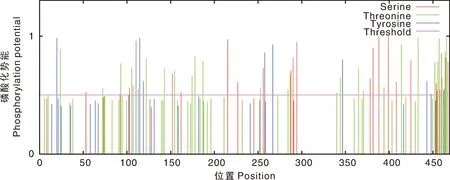
图9 PPV7 Cap蛋白的磷酸化位点Fig.9 Phosphorylation site of PPV7 Cap protein
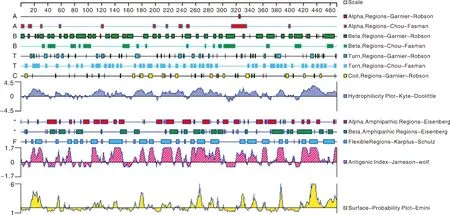
图10 PPV7 Cap蛋白的B细胞表位预测Fig.10 B cell epitope prediction of PPV7 Cap protein

表1 综合不同数据分析PPV7 Cap蛋白B细胞抗原表位的位置Table 1 Analysis of the location of the PPV7 Cap protein B cell epitopes in combination with different data
2.10.2 Th表位预测分析
利用ProPred在线服务器分别预测DRB1-0101、DRB1-0102、DRB1-0301的Th表位(表3),结果显示,第245~253位的LVKLFPYTP、309~317位的IVPIQWFWI、374~382位的FRNSIHLSC可能存在Cap蛋白的Th表位。

表2 猪细小病毒7型MAS株Cap蛋白的CTL细胞表位预测Table 2 Prediction of CTL cell epitopes of PPV7 virus MAS strain Cap protein
3 讨论
PPV7是近年来在猪细小病毒中新发现的病原,在我国广东、安徽和福建等[16,20-21]地区均报道出PPV7感染病例。但目前关于PPV7的来源尚不清楚、典型的临床症状尚不明确、分离和培养技术还未成熟、致病机制的相关研究甚少,急需安全可靠的疫苗进行免疫防控,以及诊断类生物制品进行病原诊断与抗体评价[22]。PPV7 Cap蛋白是构成该病毒衣壳的唯一结构蛋白,其携带病毒主要的抗原决定簇,可诱导机体产生特异性的免疫保护反应[23],在 PPV7的疫苗开发和诊断类生物制品研发中起到重要作用。因此,本研究在大肠埃希菌表达系统对PPV7 Cap蛋白进行表达优化,并结合生物信息学方法分析该蛋白分子特性。为下一步深入研究该蛋白的结构与功能奠定基础。
本研究以PPV7/China/Ahmas为模板扩增Cap基因,选用具有与重组蛋白融合表达率高及确保纯化蛋白纯度等优势的pGEX-6P-1大肠埃希菌表达载体[24],成功构建重组质粒pGEX-6P-1-Cap。对IPTG浓度,诱导温度、时间、转速等因素进行优化,结果显示,在37 ℃、180 r·min-1、IPTG终浓度为1 mmol·L-1条件下诱导6 h,所得目的蛋白的表达效果最佳。Western-blot结果显示,该蛋白能与抗GST标签的鼠源一抗发生特异性免疫反应,表明PPV7 Cap蛋白具有良好的反应原性,可纯化后用于制备多克隆抗体等后续试验。生物信息学分析结果显示,PPV7 Cap蛋白大小约78 ku,与SDS-PAGE结果相符,不存在跨膜结构域,有62个磷酸化位点。二级结构内无规则卷曲较多,α螺旋与β折叠较少,与三级结构结果相似。其中无规则卷曲结构突出、稳定性差,易于盘旋和扭曲,容易与抗体分子结合,因此该区域可能存在优势抗原表位[25]。结合二级结构、亲水性、柔韧性、抗原性、表面可及性等参数预测该蛋白B细胞抗原表位,有效避免了因单参数预测的局限性而导致结果误差较大[26],结果显示,在第148~149、179~182、192~196、211~213、223~224、232~234、269~270、283~285、293~294、337~340、421~423、436~437可能存在优势抗原表位。表位疫苗作为近年来新发展起来的一种新型疫苗技术,其含有能够被机体免疫细胞直接识别的MHC限制性多肽,能够产生具有细胞杀伤作用的CTL和促进中和抗体产生的Th免疫应答反应,对于清除病毒感染和提供免疫保护具有重要的作用[27-28]。因此,CTL与Th表位的筛选对表位疫苗的研究具有重要意义。利用nHLAPred的ComPred在线服务器和ProPred在线服务器分别预测CTL表位与Th表位,结果显示,在第159~167、305~313位可能存在CTL优势表位,第245~253、309~317、374~382位可能存在Th优势表位。该结果为PPV7表位疫苗的创制提供一定的方向。
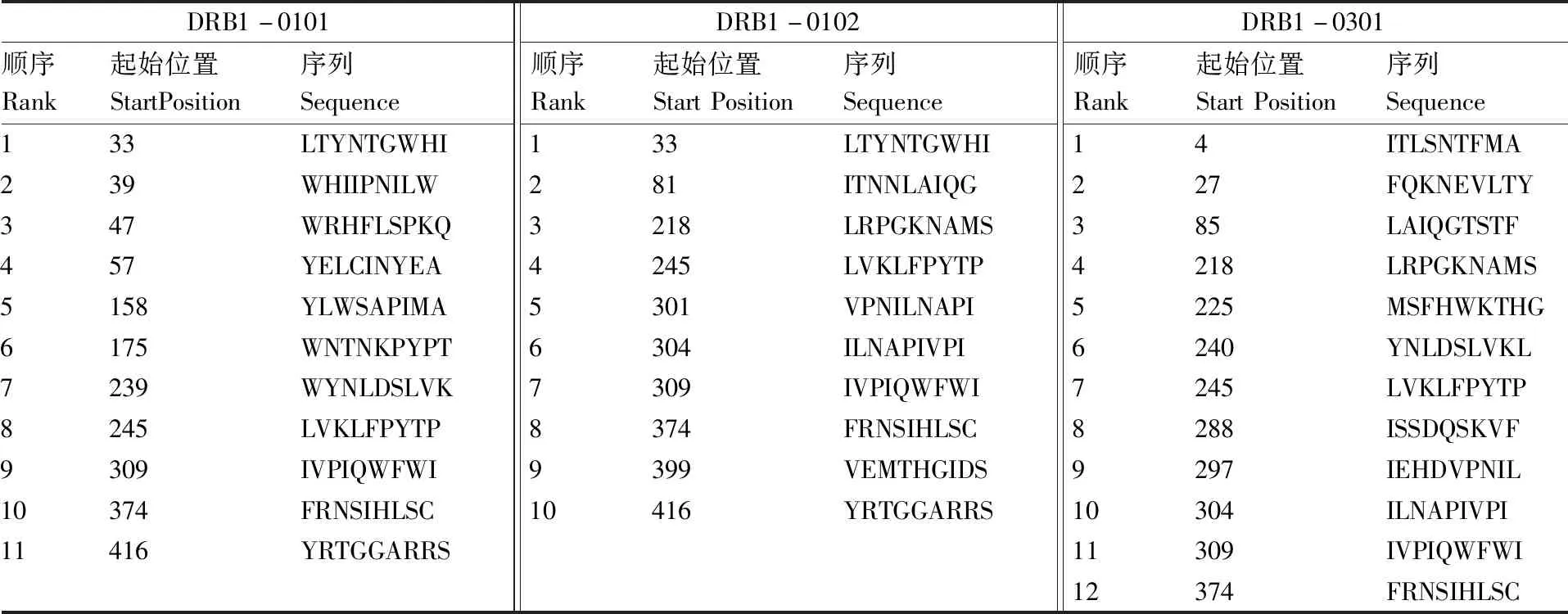
表3 猪细小病毒7型MAS株Cap蛋白的Th细胞表位预测Table 3 Prediction of Th cell epitopes of PPV7 virus MAS strain Cap protein
本研究首次通过原核表达方法对PPV7 Cap蛋白进行诱导表达,对其基本理化性质、结构与功能特性、潜在B细胞和T细胞抗原表位进行生物信息学分析。为后续开发PPV7抗体诊断试剂盒、制备Cap的单抗或多抗、研发PPV7表位疫苗等研究奠定基础。
