野生大豆阳离子质子转运体CHX19.3 与14-3-3 蛋白的相互作用研究
2020-03-04沈阳才晓溪贾博为孙明哲周伍红杨珺凯胡冰霜孙晓丽
沈阳,才晓溪,贾博为,孙明哲,周伍红,杨珺凯,胡冰霜,孙晓丽
(黑龙江八一农垦大学农学院/作物逆境分子生物学实验室,大庆 163319)
土地盐碱化严重威胁我国乃至世界农业系统的正常发展,全世界约20%的可耕种地和50%的灌溉地受盐碱化影响。中国东北松嫩平原是世界三大盐碱地之一,盐碱地面积高达370 多万hm2。因此,土地盐碱化严重制约了我国东北地区的农业生产,限制了粮食产量。随着我国人口的持续增长和可耕种土壤的退化,合理开发利用盐碱地是增加我国粮食产量的重要举措,也是实现农业可持续发展的关键因素。
作为固着的、不可移动的生命有机体,植物在长期的进化过程中,形成了一系列的应对外界胁迫的机制。如何维持胞内外离子平衡是植物应对外界逆境的关键。阳离子质子逆向转运蛋白(CPA,cation proton antiporter)作为不同单价阳离子外流耦合器[1],通过减少Na+内流,促进其外流及区隔化,保证胞质内较低的Na+含量,参与植物对外界逆境胁迫的应答[2]。根据系统发生关系,CPA 家族分为CPA1 和CPA2 两个亚家族。其中CPA1 亚家族包括研究的较为透彻的NHX 家族,而CPA2 家族包括KEA(K+-efflux antiporter)和CHX(cation/H+exchanger)[3],并且CPA2 主要存在于细菌、真菌和植物中[4]。关于植物CHX 基因家族的研究近年来才逐渐开展。迄今发现的CHX 蛋白都有约800 个氨基酸,包括N 末端约400 个氨基酸的疏水性Na+/H+交换器以及C 末端约300~400 的亲水性结构域。
植物CHXs 家族成员数目众多,拟南芥有28 个,水稻有17 个,而大豆中则有46 个[3]。目前对CHXs 的研究多集中在基因表达和功能鉴定方面。例如AtCHX13 基因与K+摄取密切相关,chx13 突变体植株对低K+敏感[5]。AtCHX14 参与调节K+平衡及K+循环再利用[6]。AtCHX17 基因表达受高盐、低K+诱导,缺K+时chx17 突变体根部K+含量减少[7-9]。AtCHX20 参与气孔保卫细胞运动[10]。
虽然目前研究已证实CHXs 在植物生长发育及逆境应答中的功能,但尚未明确其应答机制,对CHXs 互作蛋白的研究也鲜有报道。课题组前期从野生大豆中鉴定出一个耐盐碱基因CHX19.3,并利用酵母双杂交技术筛选CHX19.3 互作蛋白,发现其互作蛋白中有1 个14-3-3 蛋白。研究克隆了10 个野生大豆14-3-3 家族基因,并进一步验证GsCHX19.3与14-3-3 家族蛋白的相互作用,为深入了解其蛋白互作机制,揭示CHXs 调控耐盐碱性的分子机制提供依据。
1 材料与方法
1.1 试验材料
野生大豆G07256 由东北农业大学植物基因工程研究室提供。酵母菌(Saccharomyces cerevisiae)NMY51 菌株、大肠杆菌(Escherichia coli)DH5α 菌株、酵母双杂交表达载体pBT3-STE、pPR3-N、pOst1-NubI、pTSU2-APP 和pNubG-Fe65 为实验室保存。
1.1 GsCHX19.3 蛋白序列及拓扑结构预测
从Phytozome(http://phytozome.jgi.doe.gov/pz/portal.html)下载拟南芥同源基因的蛋白序列,利用ClustalX 程序进行蛋白序列多重比对,利用SMART在线软件(http://smart.embl -heidelberg.de/smart/set_mode.cgi?NORMAL=1)预测蛋白保守结构域,利用SOSUI 在线软件(http://harrier.nagahama-i-bio.ac.jp/sosui/)预测跨膜结构域。
1.3 GsCHX19.3 基因酵母双杂交载体构建
以pEASY-T3-GsCHX19.3 质粒为模板,采用含SfiI 酶切位点的基因特异引物(FP:ATTAACAAGGCCATTACGGCCATGATGGCGACGAGTAACAACGCG和RP:AACTGATTGGCCGAGGCGGCCCCACTCGTTGGTGTGTCT)扩增GsCHX19.3 基因CDS 区,PCR 产物回收后用SfiI 酶切,与SfiI 线性化的pBT3-STE 载体连接后转化大肠杆菌。对阳性菌落进行PCR 鉴定,并提取质粒进行酶切验证,将阳性克隆送交测序。
1.4 GsCHX19.3 诱饵蛋白毒性检测
采用LiAc 法,将pBT3-STE-GsCHX19.3 和pBT3-STE 分别转化酵母细胞NMY51,菌液涂布于SD/-Leu 筛选培养基上,30 ℃培养4 d 获得单克隆。采用pBT3-STE 载体通用引物(FP:5′- TGGCATGCATGTGCTCTG -3′ 和RP:5′ -GTAAGGTGGACTCCTTCT-3′)PCR 鉴定阳性转化子。分别挑取3 个阳性克隆,用SD/-Leu 液体培养基活化,30 ℃振荡培养过夜。将菌液浓度调整OD600=1.0,按1∶100 接种于SD/-Leu 液体培养基振荡培养,在培养4、8、12、16、20、24、48 h 测量OD600值,并绘制生长曲线。
1.5 GsCHX19.3 诱饵蛋白拓扑结构验证
采用表1 中的组合进行酵母共转化,分别涂布于SD/-Trp-Leu、SD/-Trp-Leu-His 和SD/-Trp-Leu-His-Ade 培养基上,30 ℃培养4 d 后观察菌落生长状态。统计每个反应在不同培养基上的菌落数,计算SD/-Trp-Leu-His 和SD/-Trp-Leu-His-Ade 菌落生长率即SD/-Trp-Leu-His 或SD/-Trp-Leu-His-Ade菌落数除以SD/-Trp-Leu 菌落数。其中反应3 为阳性对照,反应4 为阴性对照。若反应1 中酵母可以在SD/-Trp-Leu-His 和SD/-Trp-Leu-His-Ade 培养基上正常生长,而反应2 不能生长,则诱饵蛋白拓扑结构符合该酵母双杂交系统。
1.6 野生大豆14-3-3 基因克隆及酵母表达载体构建
采用Trizol 法提取野生大豆幼苗总RNA,经DNase 消化基因组DNA 后,反转录获得cDNA。以cDNA 为模板,采用含酶切位点的基因特异引物(表2),用高保真聚合酶PCR 扩增野生大豆GsGF14s 基因CDS 区,PCR 产物采用试剂盒回收。将PCR 产物和pPR3-N 质粒分别进行双酶切,连接后转化大肠杆菌感受态细胞。对阳性菌落进行PCR 鉴定,提取质粒进行双酶切验证,并将阳性克隆送交测序。
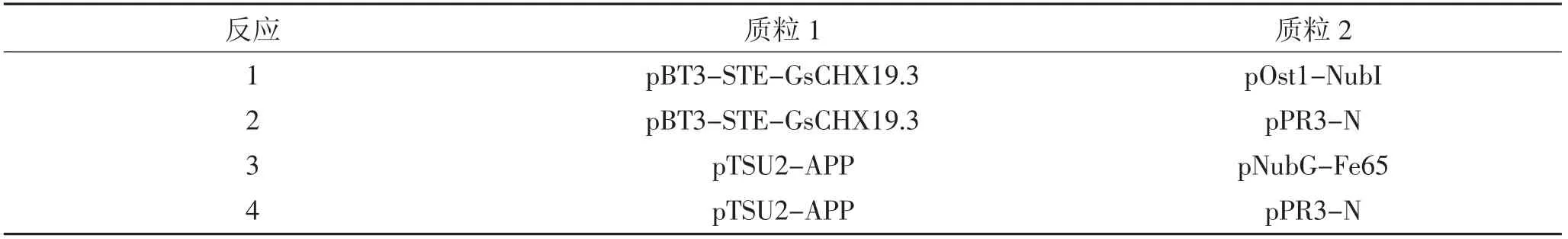
表1 GsCHX19.3 诱饵蛋白拓扑结构分析Table 1 Analysis of GsCHX19.3 topological structure

表2 野生大豆14-3-3 蛋白GsGF14s 基因特异引物Table 2 Gene specific primers of wild soybean 14-3-3 genes GsGF14s
1.7 酵母双杂交验证GsCHX19.3 与14-3-3 蛋白相互作用
将pBT3-STE-GsCHX19.3 分别与pPR3-N(阴性对照)、pOst1-NubI(阳性对照)、pPR3N-GsGF14s 共转化酵母NMY51,涂布于SD/-Trp-Leu 培养基,30 ℃培养4 d。分别采用pBT3-STE(FP:5′- TGGCATGCATGTGCTCTG-3′和RP:5′- GTAAGGTGGACTCCTTCT-3′)和pPR3-N(FP:5′- GTCGAAAATTCAAG ACAAGG-3′和RP:5′-AAGCGTGACATAACTAATTAC-3′)载体通用引物进行PCR 鉴定,获得阳性转化子。用SD/-Trp-Leu 液体培养基活化阳性克隆,将菌液浓度调整OD600=0.6,按1∶10、1∶100、1∶1 000 进行梯度稀释,取1 μL 菌液分别点点至SD/-Trp-Leu、SD/-Trp-Leu-His 和SD/-Trp-Leu-His-Ade 培养基,30 ℃培养4 d 后观察菌落生长状态。
2 结果与分析
2.1 GsCHX19.3 蛋白序列分析
前期研究克隆了野生大豆GsCHX19.3 基因全长CDS 区(2 442 bp),编码813 个氨基酸,属于Na+、K+/H+反向转运体CHX 家族。通过多重序列比对发现,GsCHX19.3 与拟南芥同源蛋白序列相似度较高(图1A)。SMART 结构域预测显示GsCHX19.3 蛋白N端含有一个典型的Na+/H+exchanger 结构域(图1B)。
2.2 GsCHX19.3 蛋白拓扑结构预测
采用SOSUI 在线软件预测GsCHX19.3 蛋白的拓扑结构,发现该蛋白N 端位于胞外,具有11 个跨膜结构域,C 端则有1 个很长的胞内结构域(图2)。因此,研究选用针对膜蛋白的基于泛素分裂的酵母双杂交系统进行蛋白互作研究。

图1 GsCHX19.3 蛋白序列及保守结构域分析Fig.1 Analysis of GsCHX19.3 protein sequences and conserved domains
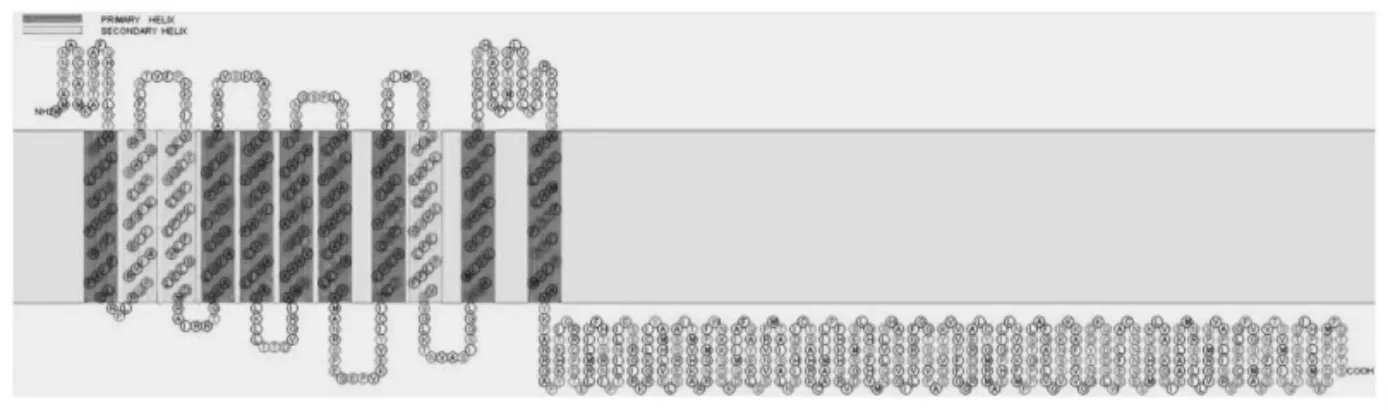
图2 GsCHX19.3 蛋白拓扑结构预测Fig.2 Prediction of GsCHX19.3 topological structure
2.3 GsCHX19.3 诱饵蛋白酵母双杂交载体构建
根据蛋白拓扑结构预测结果,研究选用pBT3-STE 诱饵载体,将GsCHX19.3 蛋白C 端与Cub-Lex-A-VP16 融合表达,使Cub-LexA-VP16 位于胞内(图3A)。采用含酶切位点的基因特异引物扩增GsCHX19.3,与线性化的载体连接后转化大肠杆菌。对阳性菌落进行PCR 鉴定(图3B),并提取质粒进行酶切验证,得到与GsCHX19.3 基因大小相符的条带(图3C),证明GsCHX19.3 诱饵蛋白酵母双杂交表达载体构建成功。
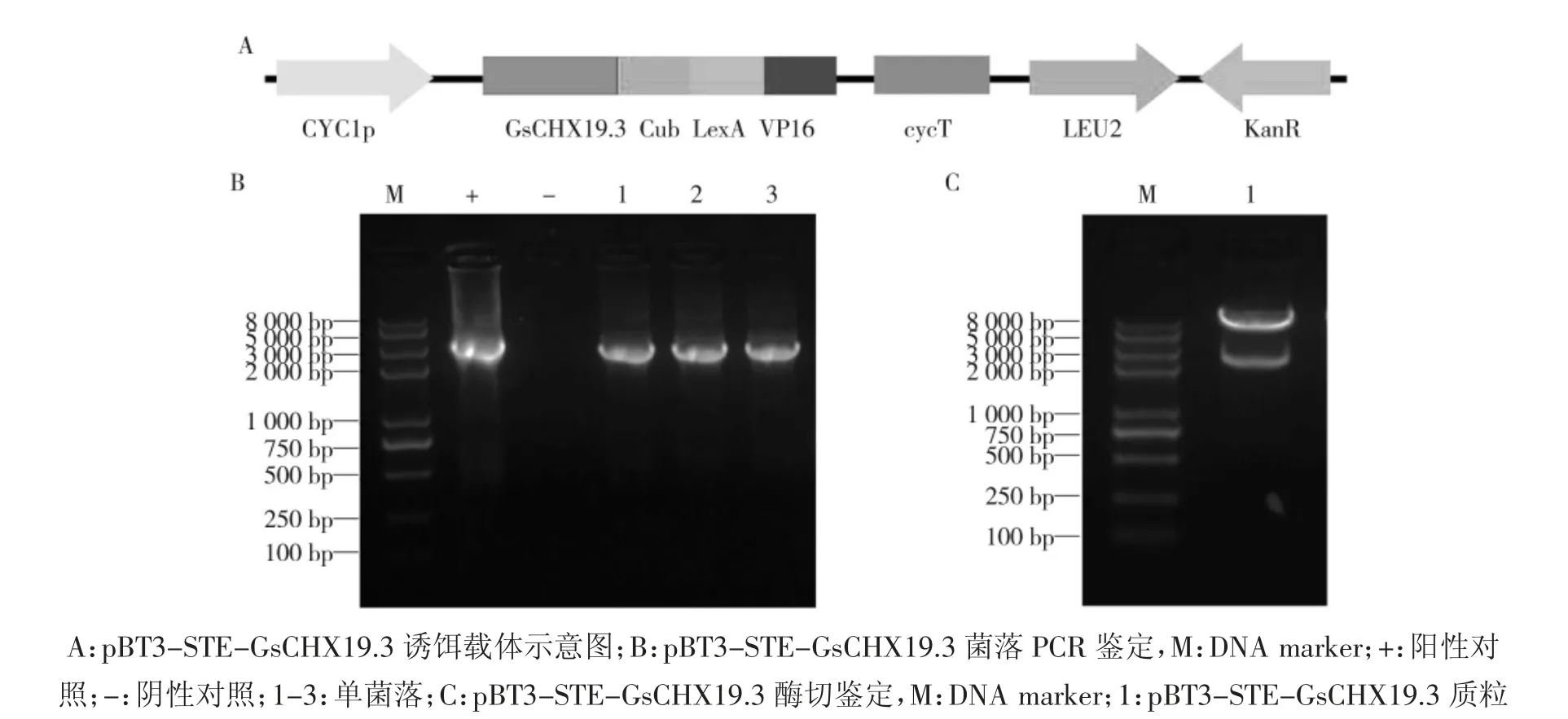
图3 GsCHX19.3 诱饵蛋白酵母双杂交载体构建Fig.3 Construction of GsCHX19.3 expression vector for yeast two hybrid assays
2.4 GsCHX19.3 诱饵蛋白毒性检测
将pBT3-STE-GsCHX19.3 和pBT3-STE 分别转化酵母细胞NMY51,测定重组酵母菌的生长曲线。如图4 所示,两种重组酵母菌在不同时间点的OD600值基本相同,生长曲线走势大致相同,说明转化pBT3-STE-GsCHX19.3 和pBT3-STE 的酵母细胞生长速度基本一致,即GsCHX19.3 蛋白的表达对酵母细胞无毒性,可进行下一步酵母双杂交实验。
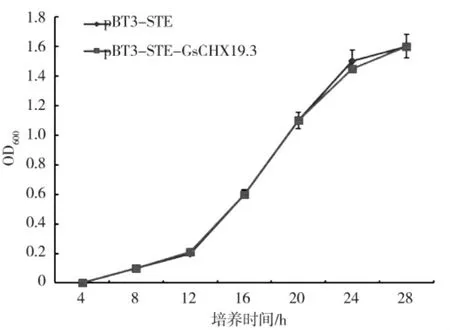
图4 GsCHX19.3 诱饵蛋白细胞毒性检测Fig.4 Cytotoxicity detection of GsCHX19.3 bait protein in yeast cells
2.5 GsCHX19.3 诱饵蛋白拓扑结构验证
采用表1 中的组合进行酵母共转化,分析重组菌在不同培养基上的生长情况,鉴定GsCHX19.3 蛋白的拓扑结构。如图5 所示,pTSU2-APP/pNubGFe65(阳性对照)重组酵母在SD/-Trp-Leu、SD/-Trp-Leu-His、SD/-Trp-Leu-His-Ade 培养基上均能正常生长(表3);而pTSU2-APP/pPR3-N(阴性对照)重组酵母在SD/-Trp-Leu 培养基上正常生长,但在SD/-Trp-Leu-His 和SD/-Trp-Leu-His-Ade 培养基上不能生长。
含有pBT3-STE-GsCHX19.3/pOst1-NubI 的酵母细胞在SD/-Trp-Leu-His 和SD/-Trp-Leu-His-Ade培养基上能正常生长,且菌落生长百分率均大于70%(表3)。这一结果表明,与拓扑结构预测结果一致,GsCHX19.3 蛋白C 端位于胞内,当GsCHX19.3-Cub-LexA-VP16 重组蛋白的Cub 与Ost1-NubI(即未突变的Nub)结合形成完整的泛素后,可释放Lex-A-VP16,使其进入细胞核激活报告基因表达。这一结果表明GsCHX19.3-Cub-LexA-VP16 融合蛋白的拓扑结构符合该酵母双杂交系统,可进行下一步实验。含pBT3-STE-GsCHX19.3/pPR3-N 的酵母细胞在SD/-Trp-Leu-His 和SD/-Trp-Leu-His-Ade 上均不能正常生长,说明GsCHX19.3-Cub-LexA-VP16 不能与突变的NubG 结合,报告基因不能表达(图5),说明SD/-Trp-Leu-His 和SD/-Trp-Leu-His-Ade 筛选培养基可作为下一步酵母双杂交实验的筛选条件。

图5 不同反应的重组酵母在不同选择培养基上的生长情况Fig.5 Growth performance of transformed yeasts on different SD medium
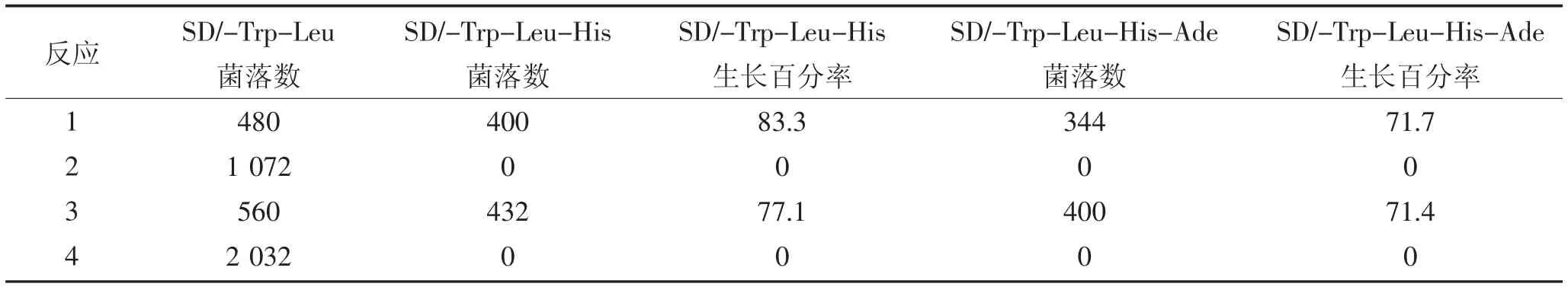
表3 GsCHX19.3 蛋白拓扑结构验证Table 3 Identification of the topological structure of GsCHX19.3 protein
2.6 野生大豆14-3-3 基因克隆及酵母表达载体构建
前期对大豆14-3-3 家族分析发现,该家族存在大量的基因复制现象,且同组内14-3-3 蛋白的序列相似度非常高[11]。为了分析GsCHX19.3 与14-3-3 家族蛋白的相互作用,研究根据家族进化关系选取了其中10 个GsGF14s 基因进行研究。
前期研究表明大豆GsGF14s 蛋白定位于细胞质或细胞核[12],且利用HMMTop 在线软件预测未发现任何跨膜结构域。因此研究选用pPR3-N 表达载体,使NubG-GsGF14s 进行融合表达(图6A)。以野生大豆cDNA 为模板,采用含酶切位点的基因特异引物扩增GsGF14s 基因,与线性化的pPR3-N 载体连接后转化大肠杆菌。对阳性菌落进行PCR 鉴定(图6B),并提取质粒进行双酶切验证,酶切得到与目标基因大小相符的条带(图6C),证明GsGF14s 基因酵母双杂交表达载体构建成功。
2.7 酵母双杂交分析GsCHX19.3 与14-3-3 蛋白相互作用
为了研究GsCHX19.3 与14-3-3 家族蛋白的相互作用,将pBT3-STE-GsCHX19.3 分别与pPR3-N(阴性对照)、pOst1-NubI(阳性对照)和pPR3N-Gs-GF14s 共转化酵母。将重组菌活化后菌液浓度调平至OD600=0.6,按1∶10、1∶100、1∶1 000 梯度稀释后,分别点点至SD/-Trp-Leu、SD/-Trp-Leu-His 和SD/-Trp-Leu-His-Ade 培养基。如图7 所示,pBT3-STEGsCHX19.3/pOst1-NubI 重组酵母在SD/-Trp-Leu、SD/-Trp-Leu-His、SD/-Trp-Leu-His-Ade 培养基上均能正常生长,而pBT3-STE-GsCHX19.3/pPR3-N 重组酵母在SD/-Trp-Leu 培养基上正常生长,但在SD/-Trp-Leu-His 和SD/-Trp-Leu-His-Ade 上不能生长。
除GsGF14f 外,其余pBT3-STE-GsCHX19.3/pPR3N-GsGF14s 重组菌均和阴性对照一样,在SD/-Trp-Leu 培养基上正常生长,但在SD/-Trp-Leu-His和SD/-Trp-Leu-His-Ade 上不能生长。而含pBT3-STE-GsCHX19.3/pPR3N-GsGF14f 的重组菌在SD/-Trp-Leu、SD/-Trp-Leu-His、SD/-Trp-Leu-His -Ade培养基上均能正常生长。上述结果表明,10 个野生大豆14-3-3 蛋白中,只有GsGF14f 和GsCHX19.3 蛋白具有相互作用,说明GsCHX19.3 和14-3-3 蛋白的相互作用具有特异性。
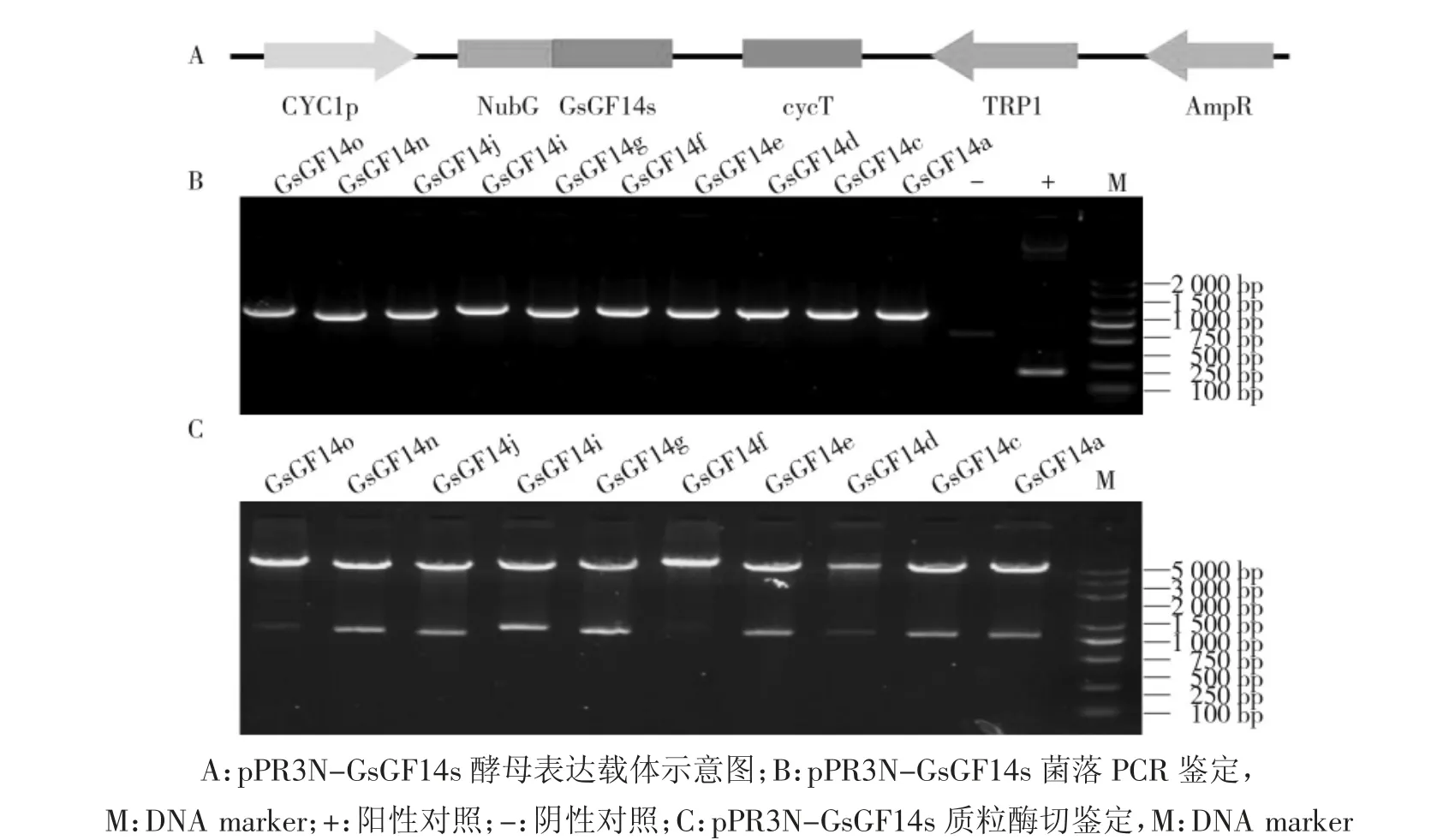
图6 GsGF14s 基因酵母双杂交表达载体构建Fig.6 Construction of GsGF14s expression vector for yeast two hybrid assays
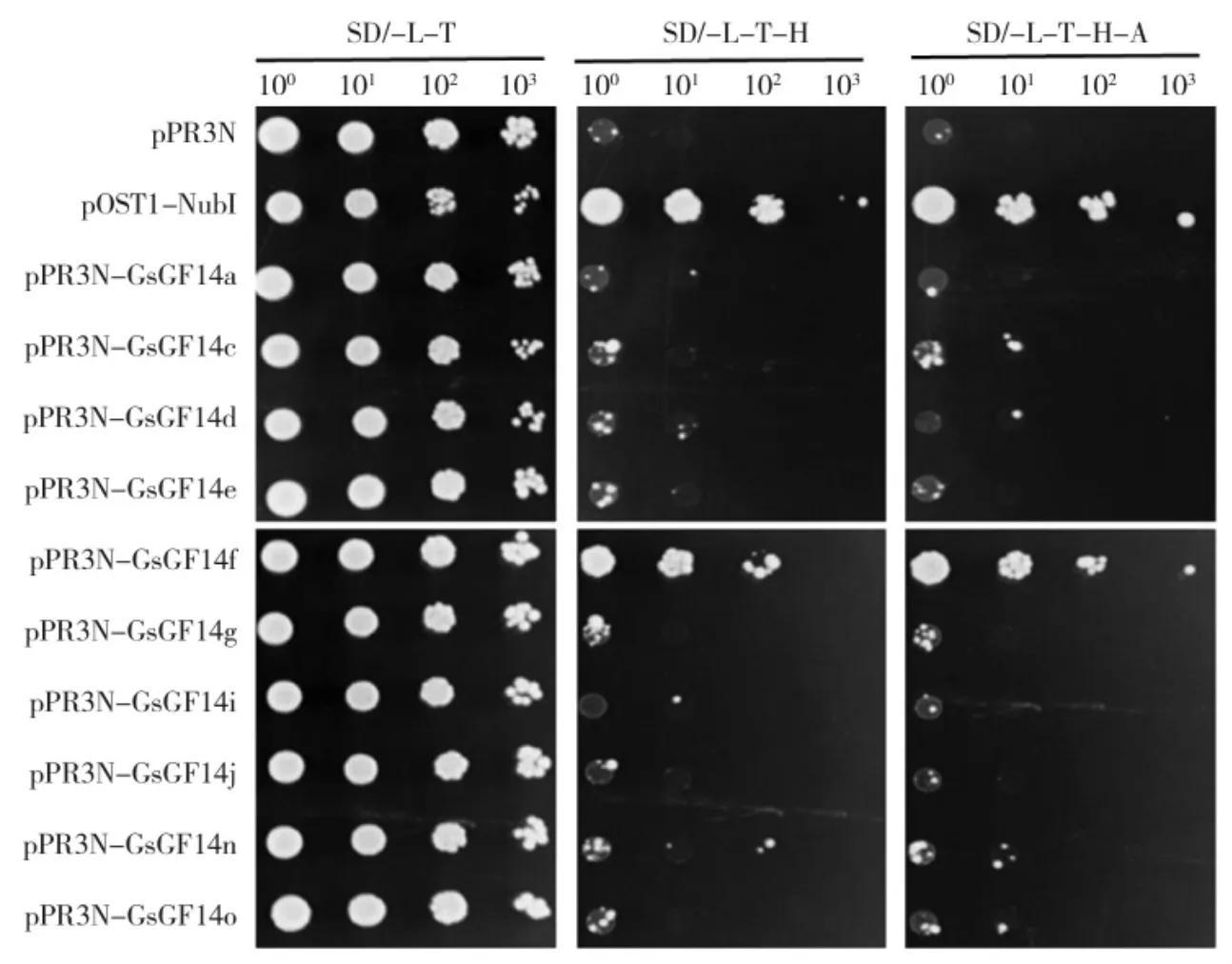
图7 酵母双杂交分析GsCHX19.3 与14-3-3 蛋白相互作用Fig.7 Y2H identification of protein interaction between GsCHX19.3 and GsGF14s
3 讨论
随着生物信息学和功能基因组学的飞速发展,科研工作者已从多种植物中鉴定了CHXs 基因家族,并揭示了其在生长发育和逆境应答中的重要角色[13]。如AtCHX16-19 参与植物生殖生长和种子发育调控过程[13]。AtCHX20 在保卫细胞中高表达,调控气孔开关[10]。AtCHX21 调控木质部Na+平衡和叶片中Na+积累[14]。然而,CHXs 基因究竟如何发挥功能?目前研究仅关注于其在离子转运和pH 平衡中的作用,尚未有研究报道CHXs 蛋白是如何被调控的。利用酵母双杂交技术筛选鉴定互作蛋白,是研究基因分子机制的一种有效途径[15-18]。与免疫共沉淀、Pull down 等其他体外分析蛋白互作的技术相比,酵母双杂交具有操作简便、快速高效等特点。
在前期研究中,课题组利用野生大豆转录组测序数据[19],筛选获得了盐碱胁迫应答的GsCHX19.3基因。该基因与拟南芥同源基因高度相似(图1A),其蛋白N 端含有一个典型的Na+/H+exchanger 结构域(图1B)。我们前期利用酵母双杂交技术筛选GsCHX19.3 互作蛋白,发现了一个14-3-3 蛋白。研究进一步鉴定了GsCHX19.3 和大豆14-3-3 家族蛋白的相互作用特异性。
由于CHXs 蛋白一般定位于膜系统,而膜蛋白的正确折叠往往需要特定的疏水性环境,否则其结构和功能将会改变,蛋白相互作用也将会受到抑制。分离泛素酵母双杂交系统通过待检测蛋白质间的互作,将突变的泛素N 端NubG 和C 端Cub 融合,使报告蛋白LexA-VP16 解离入核,从而激活报告基因表达[20-23]。与传统的酵母双杂交技术相比,分离泛素系统的蛋白互作不再局限于核内,更适合于膜蛋白相互作用研究。GsCHX19.3 蛋白拓扑结构预测显示其具有11 个跨膜结构域,N 端位于胞外,C 端位于胞内(图2)。因此,研究采用针对膜蛋白的基于泛素分裂的酵母双杂交系统[16,24],选用pBT3-STE 作为诱饵载体,使GsCHX19.3 蛋白C 端与Cub-LexA-VP16 融合表达(图3)。酵母生长实验表明GsCHX19.3 蛋白对于酵母细胞生长无毒性作用(图4),GsCHX19.3 蛋白C 端位于胞内。当GsCHX19.3-Cub-LexA-VP16 重组蛋白的Cub 与Ost1-NubI 中未突变的NubI 结合形成完整的泛素后,释放LexA-VP16,使其入核激活报告基因表达(图5)。
14-3-3 蛋白是一类广泛存在的、高度保守的蛋白家族,在植物生长发育和逆境应答中发挥重要作用[25-27]。大豆中共发现18 个14-3-3 蛋白,分别命名为GsGF14a-p[9]。其中,GsGF14c 和GsGF14l 在大豆根瘤形成的早期阶段和异黄酮合成过程中起着关键作用[28-29]。我们前期也发现GsGF14o 参与调控根毛和气孔发育,以及对ABA 和干旱胁迫的应答过程[30]。研究表明14-3-3 蛋白往往通过与其他蛋白相互作用,增强或抑制靶蛋白的催化活性,或调节靶蛋白在细胞内的定位,或影响靶蛋白与其他蛋白的相互作用,或保护靶蛋白被降解[31]。例如大豆中,GmMYB176、GmMYB62 和GmMYB173 能够与所有14-3-3 蛋白互作,且SGF14l 能通过蛋白互作,调控MYBs 在细胞内的定位[29,32]。拟南芥14-3-3λ 能够与SOS2 互作抑制SOS2 活性,使其无法激活Na+/H+反向转运体SOS1[33]。此外,14-3-3 蛋白也能够与H+-ATPase 相互作用调控植物对碱胁迫的应答过程[34-35]。
研究中我们鉴定了10 个野生大豆14-3-3 家族蛋白与GsCHX19.3 的相互作用,发现只有GsGF14f能够与GsCHX19.3 互作(图7)。这一结果说明与MYB 转录因子不同,GsCHX19.3 与14-3-3 蛋白的相互作用具有特异性。但我们未能在GsCHX19.3 氨基酸序列中找到14-3-3 蛋白结合的典型模序RSXpSXP 和RXY/FXpSXP[36]。因此推测GsGF14f 可能是通过识别非磷酸化的序列与GsCHX19.3 互作。
在今后的研究中,将进一步利用BiFc 或CoIP 技术验证GsGF14f 与GsCHX19.3 蛋白相互作用,并着重关注以下3 个问题:GsGF14f 与GsCHX19.3 互作的识别序列是什么?GsGF14f 对GsCHX19.3 蛋白的调控作用又是什么?GsGF14f 基因是否和GsCHX19.3一样,调控植物对盐碱胁迫的应答过程?
4 结论
构建了pBT3-STE-GsCHX19.3 诱饵载体,其在酵母体内表达的GsCHX19.3-Cub-LexA-VP16 融合蛋白对酵母菌没有毒性,且拓扑结构符合该酵母双杂交系统。酵母双杂交结果显示,只有GsGF14f 能够与GsCHX19.3 蛋白互作,其余14-3-3 蛋白与GsCHX19.3 均不存在相互作用,即GsCHX19.3 与野生大豆14-3-3 蛋白互作具有特异性。
