两种海藻寡糖促乳酸菌增长的转录组比较研究
2020-02-28陈兴麟林河通
陈兴麟,李 丽,林河通
(1.自然资源部第三海洋研究所,福建 厦门 361005; 2.福建农林大学食品科学学院,福建 福州 350002)
龙须菜(Gracilarialemaneiformis)是江蓠属(Gracilaria)下的一个种。琼胶是许多红藻类植物合成的一种生物高聚物,是其细胞壁的主要成分[1],在常温环境下呈凝胶状[2]。琼胶及其他海藻细胞壁多糖具有的生理学功能与陆生植物的半纤维素类似,但具有更大的柔韧性,因此能耐受强烈的波浪和涌流[3-4]。细胞壁中的琼胶不仅能帮助红藻抵抗病菌侵染、干旱环境,维持细胞内的离子平衡,还可以使红藻能在更广的温度、盐度和pH下生存[3, 5]。琼胶寡糖的制备通常分为化学法和酶解法。化学法的降解条件不易控制、容易带来环境污染,并且产物不均一。而酶解法制备琼胶寡糖,反应条件温和、能耗低。因为酶催化具有专一性,能够选择性地切断糖苷键,因此降解产物也较为均一[6]。琼胶寡糖由于分子量较小,水溶性比琼胶多糖好,更有利于人体吸收,并具有一些独特的生理功能,例如,较强的抗氧化活性[7]、改善人体肠道菌群[8-9]等,具有很强的开发潜力。肠道菌群对人体的健康有重要的作用,虽然目前在界定有益菌和有害菌的界线上仍然存在争议,但是乳酸菌Lactobacillus普遍认为是益生菌[10],可以广泛的应用于食品工业,例如酸奶[11]、烘焙[12]和谷物制品[13]等。
微生物转录组是指微生物在某一时刻所转录出的所有RNA的总和,可以进一步揭示微生物在特定条件下基因的动态表达与调控[14]。通过比较微生物在不同环境、不同刺激物作用下转录组的差异,可以研究其基因表达的变化,进而研究环境及刺激物对微生物的影响及作用机理[15]。转录组通常以RNA-seq进行测序,即通过反转录酶将样品中的RNA反转录成cDNA,再通过构建cDNA测序文库进行测序,从而获得相应的RNA序列信息及表达量[16]。基因本体(gene ontology,GO)是一套国际标准化的基因功能描述的分类系统,是生物医学领域最成功的本体之一[17]。GO可分为三大类,即生物过程(biological process,BP),分子功能(molecular function,MF)和细胞组分(cellular component,CC),分别用来描述基因编码的产物所参与的生物过程、所具有的分子功能及所处的细胞环境[18]。GO注释是将一个基因和一个GO条目联系起来,每个条目有唯一的标示符(由“GO:”加上7个数字组成)。目前,GO已经广泛应用在基因集合富集分析、基因功能比较与分析、蛋白质互作预测[19-20]和帕金森疾病的研究[21]等诸多领域。
福建省是中国龙须菜生产大省,龙须菜最主要的经济价值是提取琼胶。而由琼胶进一步降解而成的寡糖,具有更多的生理活性以及更高的价值。通过制备寡糖可以实现龙须菜产业链的高值化利用。本研究以深海琼胶酶制备的海藻寡糖为试验物,检测其促乳酸菌Lactobacillusplantarum增长的效果,并通过对乳酸菌转录组的测序来研究其促生长机理,进一步拓展海藻寡糖的应用领域,也为海洋生物制品在食品营养学领域的应用基础研究提供参考。
1 材料与方法
1.1 材料
新琼四糖(NA4)、新琼六糖(NA6)由本课题组利用深海琼胶酶,通过酶解琼胶制备而成,纯化后纯度达95%以上;混合寡糖(NAOS)由本课题组以深海细菌Flammeovirgasp.OC4发酵龙须菜制得,为新琼二糖、琼胶三糖、新琼四糖、琼胶五糖、新琼六糖的混合物;低聚果糖(FOS)购自成都艾科达化学试剂有限公司;低聚木糖(XOS)购自源叶生物科技有限公司;葡萄糖(GLC)购自Sigma公司;乳酸菌Lactobacillusplantarum由本课题组前期从酸奶中分离得到。
1.2 培养基
MRS肉汤培养基:Peptone 1%(质量分数,下同),Beef extract 0.8%,Yeast Extract 0.4%,葡萄糖 2%,K2HPO40.2%,柠檬酸二铵 0.2%,乙酸钠 0.5%,MgSO40.002%,MnSO40.000 4%,Tween-80 0.01%。
2×MRS肉汤培养基:Peptone 2%,Beef extract 1.6%,Yeast Extract 0.8%,葡萄糖 4%,K2HPO40.4%,柠檬酸二铵 0.4%,乙酸钠 1.0%,MgSO40.004%,MnSO40.000 8%,Tween-80 0.02%。
1.3 主要仪器及试剂盒
全自动生长曲线分析仪Bioscreen C(MBR, Oy Growth Curves Ab Ltd);Total RNA Extractor(Trizol)试剂盒(上海生工生物工程技术服务有限公司);Epicenter Ribo-zero magnetic kit(bacteria)购自Epicenter公司;VAHTSTMmRNA-seq V2 Library Prep Kit for Illumina©、VAHTSTMDNA Clean Beads购自南京诺唯赞生物科技有限公司。
1.4 方法
1.4.1 乳酸菌种子液的培养 将添加了1.5%琼脂的MRS肉汤培养基于118 ℃灭菌15 min,然后制备MRS平板。将乳酸菌Lactobacillusplantarum在平板上划线培养,取单菌落接种于MRS肉汤培养基中,37 ℃摇培20 h,4 ℃保存待用。另外,取部分菌液,于100 ℃煮沸30 min,作为灭活菌体对照。
1.4.2 各种寡糖促乳酸菌增长的效果 Bioscreen C可装载2个检测板,每个检测板有100个孔(10×10),每个孔分别装载100 μL的培养系统,包含:50 μL的灭菌2×MRS肉汤培养基,5 μL乳酸菌保存液(对照组为灭活液),相应浓度的各种寡糖和双蒸水(double distilled water,ddw)。各寡糖液均提前经过了0.22 μm微孔滤膜过滤除菌,适量加入使体系终浓度分别为0.2、0.4、0.6、0.8、1.0 g/L。如表1所示,101~200号在第一个检测板上,201~224号在第二个检测板上。根据加入寡糖种类的不同,一共可以分为7个组。第一组(101~120号)添加的寡糖为NAOS。第二组(121~140号)和第三组(141~160号)添加的寡糖分别为NA4和NA6。第四组(161~180号)、第五组(181~200号)、第六组(201~220号)添加的寡糖分别为FOS、XOS和GLC。第七组(221~224号)为对照组,不额外添加任何寡糖。两个检测板同时放入全自动生长曲线分析仪(Bioscreen C)中,37 ℃水平摇培48 h,每2 h采集一次OD600值数据。
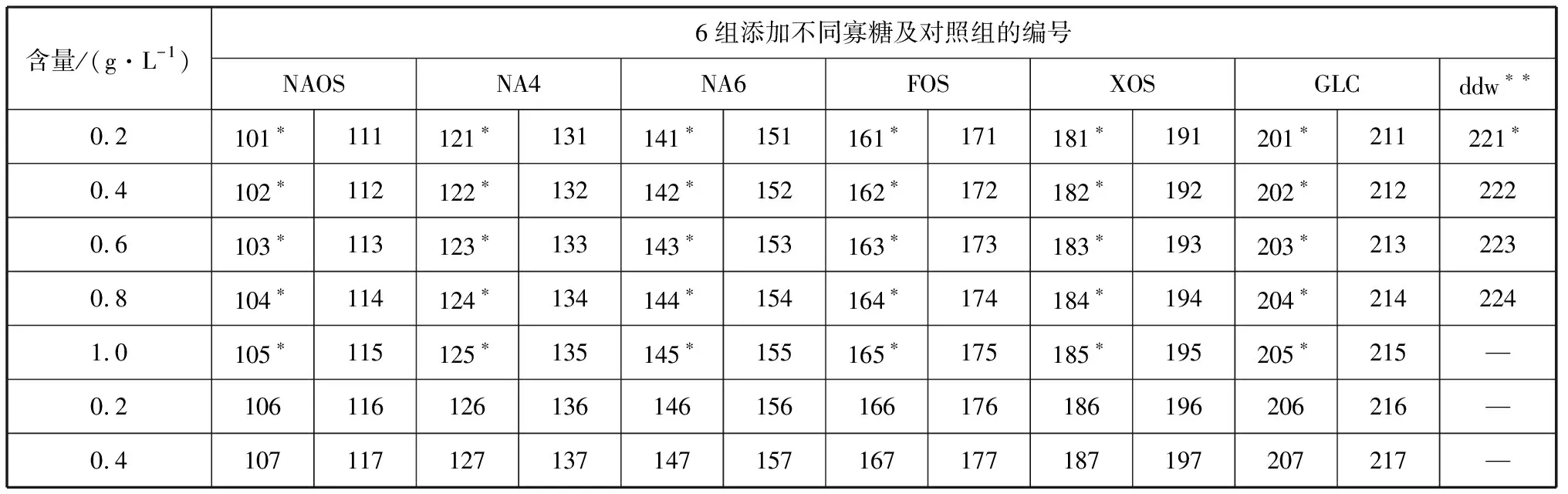
表1 乳酸菌Lactobacillus plantarum生长速率实验设计
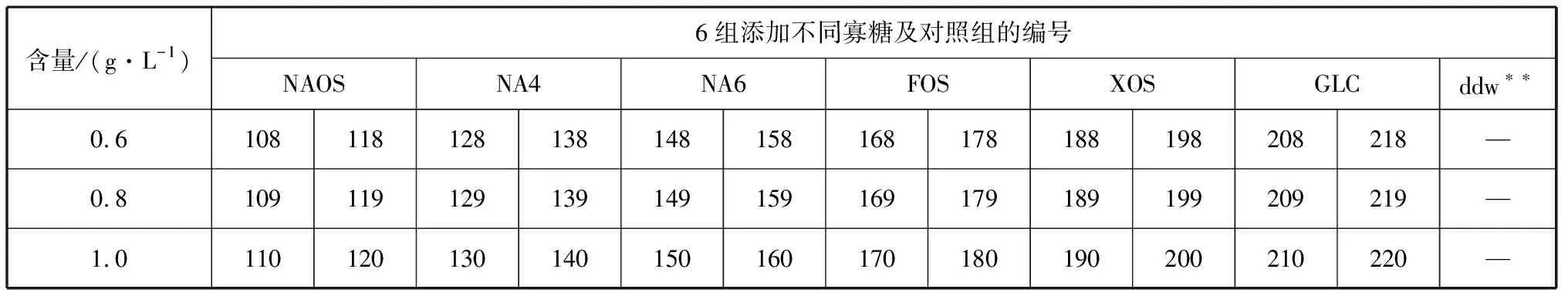
续表1
注:“*”表示空白对照:加入灭活菌体;“**”表示阴性对照:加入适量ddw替代寡糖液。
1.4.3 乳酸菌RNA的提取 如表2所示,设置5个处理组,每组2个重复,分别为:4H组,添加NA4至终浓度为0.6 g/L;6H组,添加NA6至终浓度为0.6 g/L;4L组,添加NA4至终浓度为0.2 g/L;6L组,添加NA6至终浓度为0.2 g/L;C组,不添加寡糖。分别取500 μL乳酸菌种子液,12 100 r/min离心去除上清,然后用100 μL ddw重悬菌体后,吸取100 μL菌液加入装有5 mL培养基的试管中,37 ℃摇培12 h。空白组添加等量的100 μL ddw。待摇培结束后,马上收集菌体,用Total RNA Extractor(Trizol)试剂盒提取RNA。
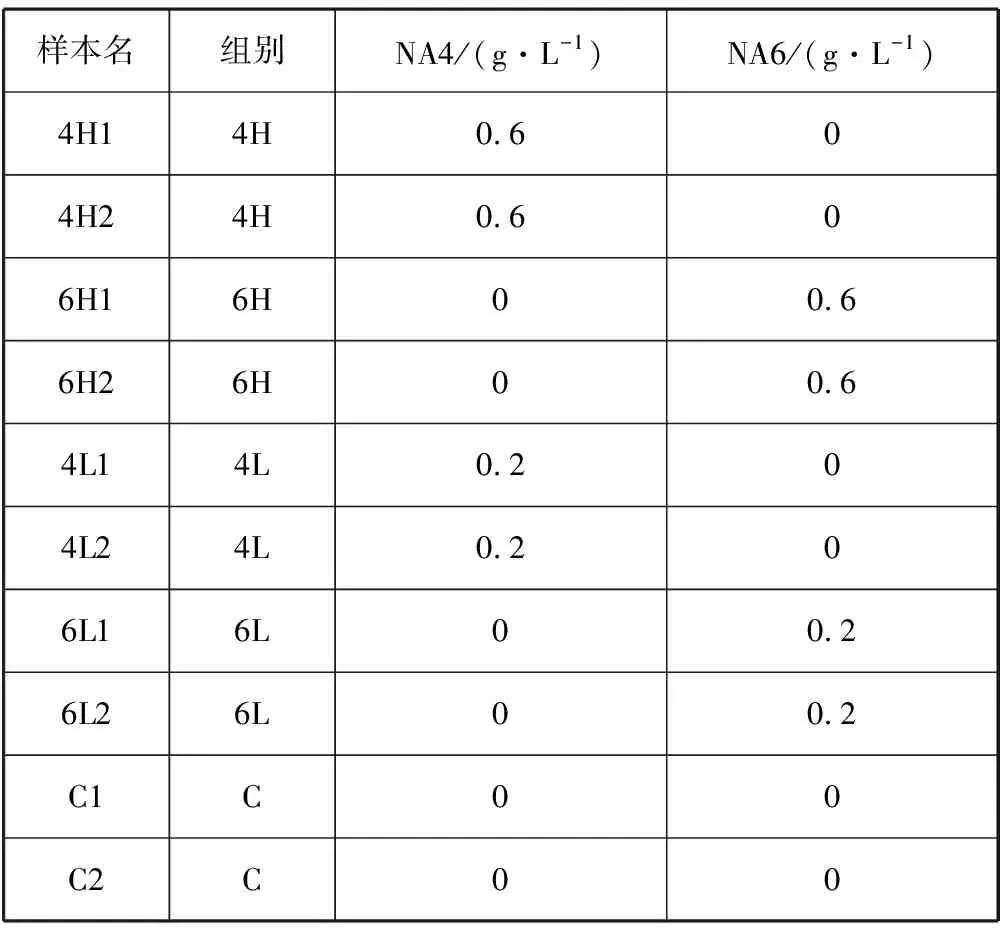
表2 Lactobacillus plantarum的培养条件及分组情况
1.4.4 乳酸菌转录组的测序与分析 依次用Epicenter Ribo-zero magnetic kit(bacteria)试剂盒纯化RNA,用VAHTSTMmRNA-seq V2 Library Prep Kit for Illumina®试剂盒构建测序文库,将构建好的文库在Illumina HiSeq X-ten测序平台完成测序,测序模式为2×150 bp双端测序。将测序仪得到的原始图像数据文件,通过CASAVA碱基识别(base calling)分析转化为原始测序序列(sequenced reads),即为Raw Data,以 FASTQ文件格式存储。对测序数据质量进行可视化评估,并使用Trimmomatic进行数据处理,依次去除带N碱基的序列、reads中的接头序列、Q值<20的低质碱基。为避免测序产物为污染,随机从Clean数据中抽取不低于9 800条序列与NCBI的NT数据库进行blastn比对,计算其物种分布。若与原始样品一致,则进行基因结构、表达水平、表达差异等分析,并进一步将差异表达基因进行GO功能分类。
2 结果与讨论
2.1 各种寡糖促乳酸菌增长的效果
如图1a所示,OD600值随着NA4、FOS、NA6、NAOS、XOS和GLC浓度的升高而升高,达到最高值后,再提高浓度会导致OD600值的降低,此结果与Lu等(2015)的研究结果相似[22]。这可能是由于过高的寡糖浓度会产生较高的渗透压,从而导致脱水作用而抑制菌株生长。不同寡糖的最适作用浓度并不一致,因此本研究均选取促乳酸菌增长效果最为显著的浓度进行进一步分析,即0.4 g/L的GLC,0.8 g/L的NAOS,以及0.6 g/L的NA4、NA6、FOS、XOS。在各自的最适浓度下进行比较,促乳酸菌增长效果最好的依次是NA4、FOS、NA6、NAOS、XOS、GLC,它们效果都好于对照组(图1b)。由于MRS肉汤培养基中已经含有GLC,额外的添加量产生的促生长效果有限。而其余各种寡糖显示了更好的促乳酸菌增长效果,说明不同的寡糖之间存在着协同增效作用。FOS被认为是一种重要的益生元,但在相同浓度下,NA4有着更好的促增长效果,说明NA4有进一步作为健康食品的应用潜力。
为了比较不同种类寡糖的促增长作用,通常需要先确定各自寡糖的最适作用浓度。根据传统方法,通常是比较一段时间内的OD600值的高低,选取数值高的浓度为合适的作用浓度[22]。但这不够严谨。如图1b所示,尽管在48 h时XOS和GLC组的OD600值一致,但从整体生长曲线上看XOS却是更好的生长促进剂。值得注意的还有,在不同实验组中达到OD600值最高点的时间并不一致,如FOS组用了26 h,而NA4组只需要20 h。因此,最优选浓度应该是通过测定不同组别生长曲线的最高点来确定。本实验中,通过Bioscreen C每2 h测定一次OD600值,同时筛选124组不同条件下乳酸菌的生长曲线,从而尽可能的消减误差值。相比于传统的摇瓶或试管培养,此方法所需的寡糖用量很小,可以在不进行大规模纯化的情况下研究寡糖促乳酸菌的增长效果。
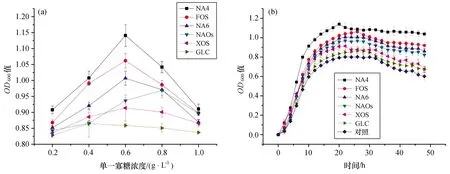
图1 寡糖促乳酸菌Lactobacillus plantarum的增长效果
2.2 乳酸菌RNA的提取
由于相比于NAOS,NA4和NA6成分单一、促乳酸菌增长的效果较好。而相比于FOS及XOS,其相关研究较少。故选取NA4和NA6做进一步研究。设置了4H组,它的NA4浓度为促乳酸菌生长效果最好的0.6 g/L。设置了6H组,它的NA6浓度也为促乳酸菌生长效果最好的0.6 g/L,以及4L组和6L组,处于三分之一的最适浓度下。在试管中培养12 h后,提取各自乳酸菌RNA。如图2所示,提取的Total RNA条带清晰,且大部分集中在1 000 bp以下的区域,符合后续实验的要求。
图2 乳酸菌Lactobacillus plantarum的RNA提取
2.3 RNA-seq测序数据分析
本次实验中,每一组高通量测序获得的原始测序总数据量都在4.55 Gb以上,其中碱基质量值在Q10(识别正确率为90%)的比例均在99.93%以上,质量值在Q30(识别正确率为99.9%)的比例也都高于94.39%。经过一系列严格的校正和过滤后,各组得到的高质量reads数在3 098万条与3 949万条之间,平均读长在145 bp以上。经校正后的各组数据碱基质量值在Q10的比例均达到99.99%以上,Q30的比例也都在97.23%以上。各组的GC含量相对一致,都在45.07%与45.66%之间,符合物种内GC含量相对一致的规律。综上所述,本次高通量测序的数据在测序深度和准确度上符合做进一步分析的要求。
为进一步确认样品无污染,随机从高质量数据中抽取不低于9 800条序列进行比对分析,比对结果如表3所示。从表中可以看出,各组之间的序列的比对结果较为一致,序列比对上的前7个物种分别是Lactobacillusplantarum,Lactobacilluspentosus,Lactobacillusparacasei,Lactobacillusbuchneri,Lactobacillusphage,Lactobacillushelveticus,Lactobacillusparaplantarum,都是乳杆菌属。其中,97%以上的序列比对结果为Lactobacillusplantarum,与实验室前期菌株鉴定结果一致,说明检测结果符合预期。
2.4 主成分分析
主成分分析(principal component analysis, PCA)被广泛用于处理多维度的基因表达数据[23]。随着高通量测序技术的发展,海量的组学数据越来越多的出现,需要使用PCA对数据进行降维处理,使结果可视化,更好的阐明其生物学机理[24]。为对各组样品的表达情况做一整体分析,采用PCA方法对其进行可视化处理(图3)。如图所示,除了4L1和4L2组内差异较大,其他的各组都按照其生物学重复聚集至相对较近的位置,说明试验重复性较好。各组之间分散至立方体的不同位置,说明寡糖的添加确实对其生理代谢产生了一定的影响。
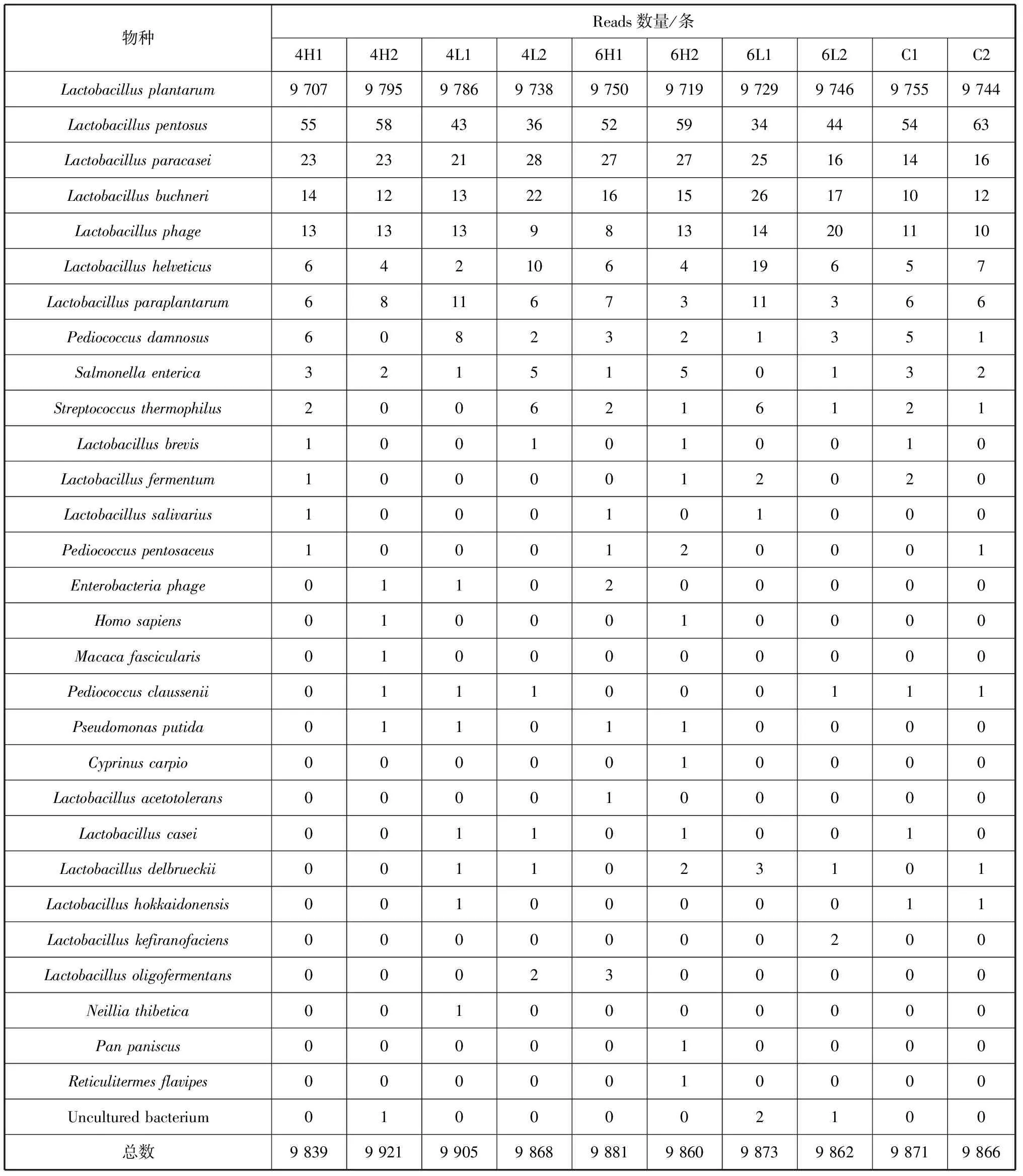
表3 不同实验组序列的blastn比对结果
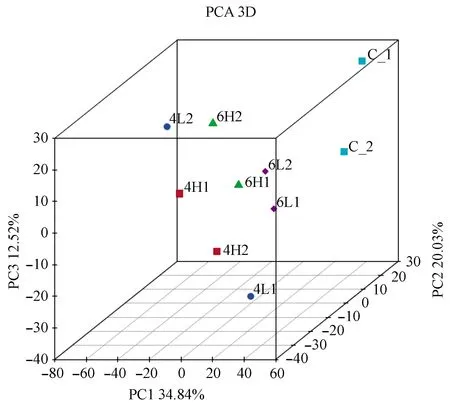
图3 各样本主成分分析
2.5 组间差异表达基因分析
基因表达水平的直接体现就是其转录的丰度情况,转录的丰度越高,则基因表达水平越高[25]。在RNA-seq分析中,我们可以通过定位到基因组区域或基因外显子区的测序序列(reads)的计数来估计基因的表达水平。Reads计数除了与基因的真实表达水平成正比外,还与基因的长度和测序深度成正相关。为了使不同实验组、不同基因之间的基因表达水平具有可比性,需要对各基因的表达情况进行标准化[26-27]。有研究表明,一些数据标准化方法,如RPKM等,反而使误差加大,从而对诊断的结果产生不利影响[28]。目前,许多研究采用TPM(transcripts per million)来计量在RNA池中某个基因转录的比例[29]。TPM同时考虑了测序深度和基因长度以及不同样本对reads计数的影响,是常用的基因表达水平估算方法。TPM计算公式[30]如下:
(1)
(2)
式(1、2)中:gl为基因外显子长度(gene length),readl为引入的均一化长度值(默认为1 000),nr为比对到该基因上的序列数(number of reads),G为求和符号的上界,即所有基因,T为求和值。
为了揭示不同处理对乳酸菌的基因表达的影响,利用DEGseq软件分析不同处理组(TPM-E)和对照组(TPM-C)样本间的差异表达基因(differentially expressed genes, DEGs)。筛选的差异表达基因必须达到以下2个要求:(i)log2(TPM-E/TPM-C)>1,即差异表达倍数至少为2倍;(ii)pValue<0.05。在本研究中,我们把在各处理组上调表达的基因定义为“上调基因”,相反地,在各处理组下调表达的基因即为“下调基因”。
如图4所示,4L组对比C组,有13个基因表达上调,22个基因表达下调。而随着NA4浓度的增高,4H组比C组,表达上调的基因增加至27个,表达下调的基因增加至28个。这说明随着NA4浓度增加至最适促生长浓度,其对乳酸菌Lactobacillusplantarum的生理代谢产生的影响逐步加大。而6L组和6H组也呈现出类似的趋势。6L组对比C组,有2个基因表达上调,12个基因表达下调。当NA6的浓度增加到0.6 g/L,表达上调的基因增加至18个,表达下调的基因仍为12个。如果NA4和NA6对比,NA4带给乳酸菌生理的影响更大,当我们比较相同浓度下乳酸菌的代谢情况时发现,浓度为0.2 g/L时,4L组比6L组,上调基因有9个,下调基因有1个。这可能是因为NA4分子量小,更容易被乳酸菌吸收。因此,这和前述实验检测的乳酸菌生长曲线,NA4有较好的促生长能力规律一致。
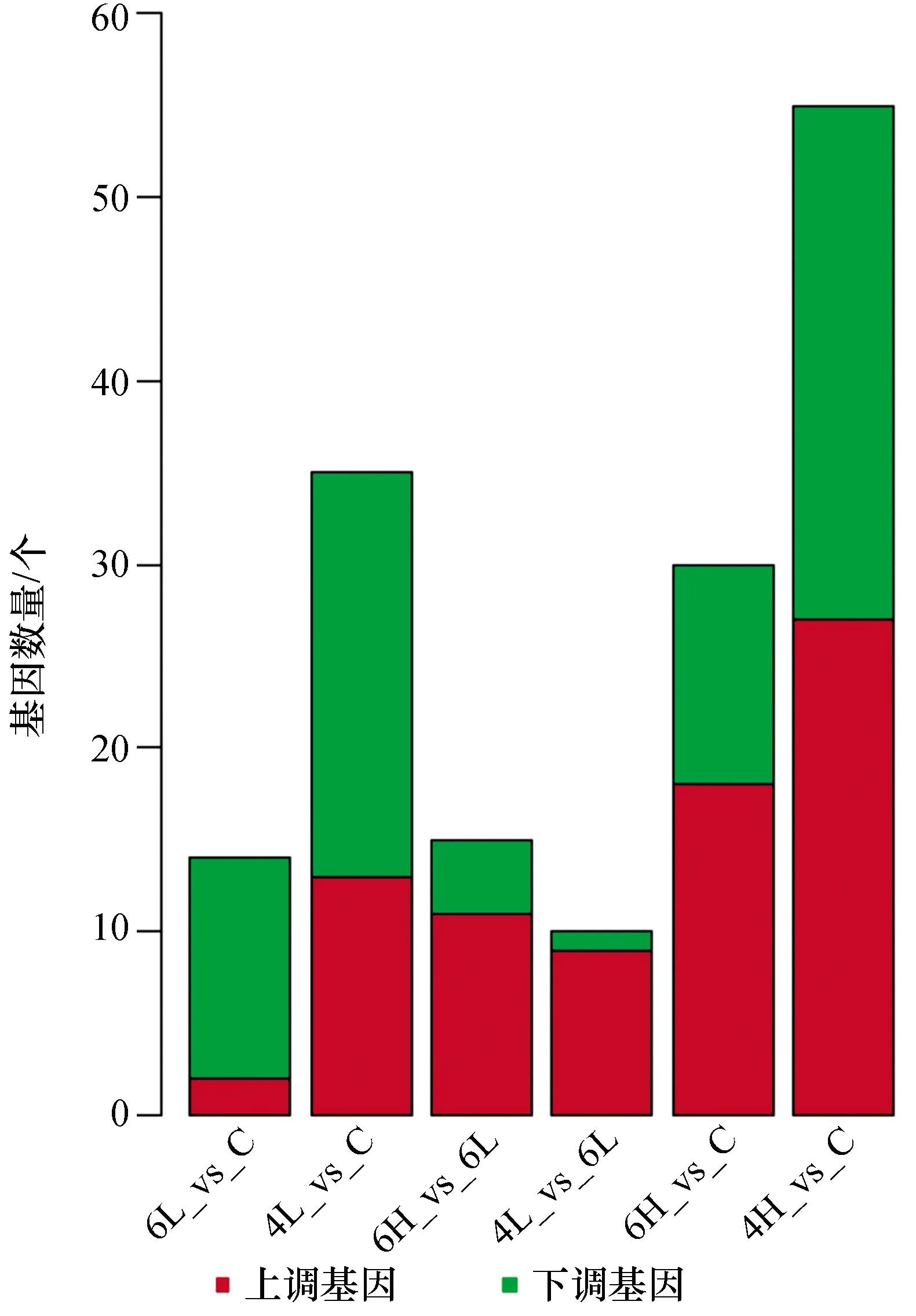
图4 不同组之间差异表达基因统计
2.6 差异表达基因的功能注释
2.6.1 4H组差异表达基因的注释 在对筛选出的DEGs相应蛋白进行GO功能注释分析中,利用topGO完成GO富集分析并绘制显著性GO有向无环图。GO分析结果显示,在55个DEGs中,有31个基因至少能关联到MF的一个GO功能条目,有24个基因至少能关联到CC的一个GO功能条目,以及27个基因至少能关联到BP的一个GO功能条目。基于GO二级分类的功能分类,已注释的差异表达基因可以划分为19个二级小类(图5),它们隶属于BP、MF及CC这三个分支。在MF中,基因数目最多的类别是“催化活性”(catalytic activity),该分类包括了22个差异表达基因,其次是“结合作用”(binding),其包含了17个差异表达基因,排在第三位的是“转运活性”(transporter activity),共有9个DEGs;在CC分支中,细胞相关组分(cell,cell part)和细胞膜组分(membrane)是DEGs对应的蛋白活动的两大主要场所;在BP分支中,“细胞内过程”(cellular process)、“代谢过程”(metabolic process)和“单组织过程”(single-organism process)这三个类别所占比例最多。
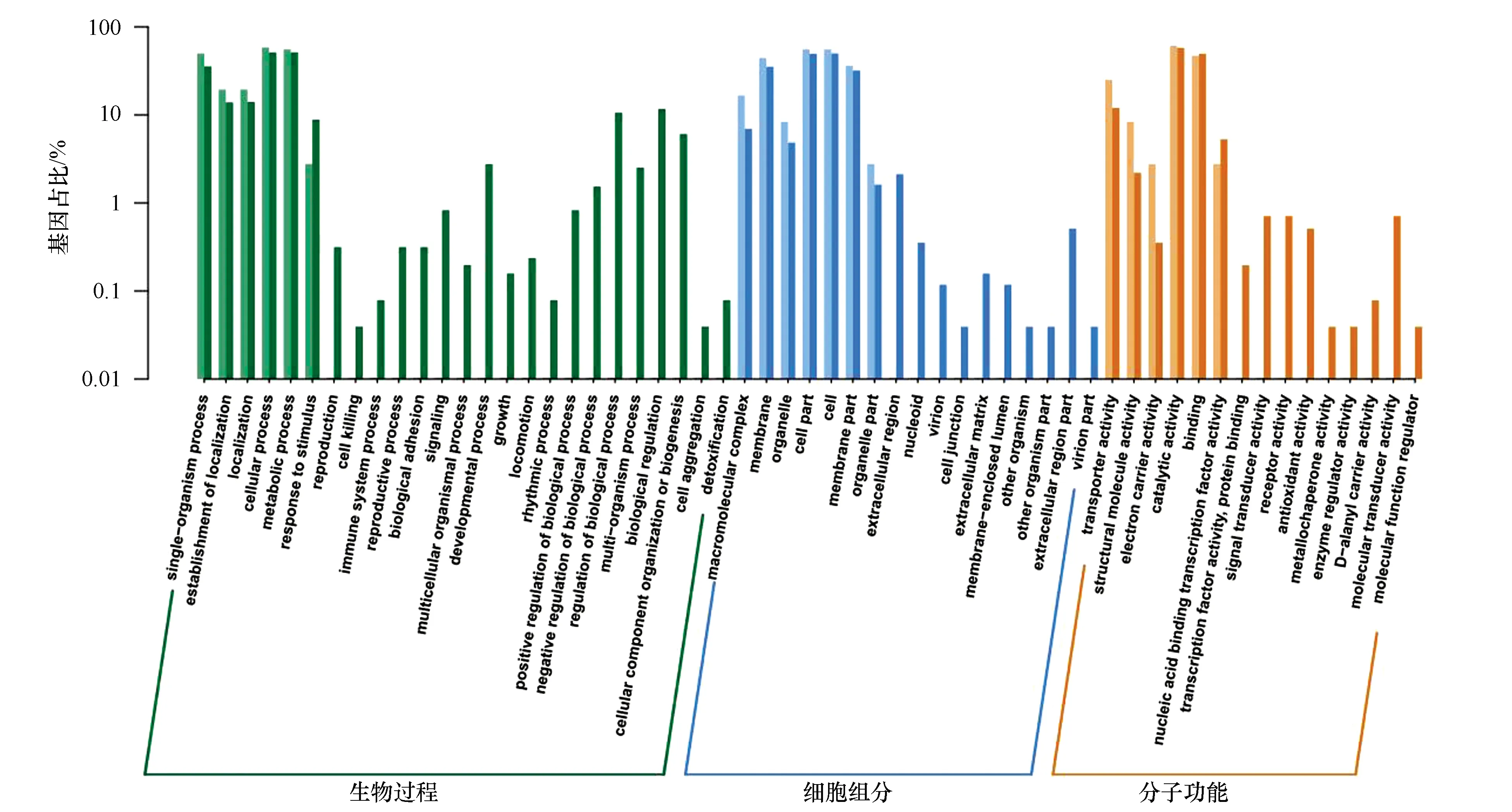
图5 4H组和C组转录组间差异表达基因的GO二级分类统计
2.6.2 6H组差异表达基因的注释 GO分析结果显示,在30个差异表达基因中,有16个基因至少能关联到MF的一个GO功能条目,有17个基因至少能关联到CC的一个GO功能条目,以及15个基因至少能关联到BP的一个GO功能条目。基于GO二级分类的功能分类,已注释的DEGs可以划分为17个二级小类(图6),它们隶属于BP、MF及CC这3个分支。在MF分支中,基因数目最多的类别是“催化活性”(catalytic activity),该分类包括了13个DEGs,其次是“结合作用”(binding),其包含了10个DEGs,排在第三位的是“转运活性”(transporter activity),共有6个DEGs;在CC分支中,细胞相关组分(cell,cell part)和细胞膜组分(membrane)是DEGs对应的蛋白活动的两大主要场所;在BP分支中,“细胞内过程”(cellular process)、“代谢过程”(metabolic process)和“单组织过程”(single-organism process)这3个类别所占比例最多。
2.6.3 4L组差异表达基因的注释 GO分析结果显示,在35个DEGs中,有25个基因至少能关联到MF的一个GO功能条目,有21个基因至少能关联到CC的一个GO功能条目,以及21个基因至少能关联到BP的一个GO功能条目。基于GO二级分类的功能分类,已注释的DEGs可以划分为21个二级小类(图7),它们隶属于BP、MF及CC这3个分支。在MF分支中,基因数目最多的类别是“结合作用”(binding),其包含了18个DEGs,其次是“催化活性”(catalytic activity),该分类包括了14个DEGs,排在第三位的是“结构分子活性”(structural molecule activity),共有6个DEGs;在CC分支中,细胞相关组分(cell,cell part)和细胞膜组分(membrane)是DEGs对应的蛋白活动的两大主要场所;在BP分支中,“细胞内过程”(cellular process)、“代谢过程”(metabolic process)和“单组织过程”(single-organism process)这3个类别所占比例最多。
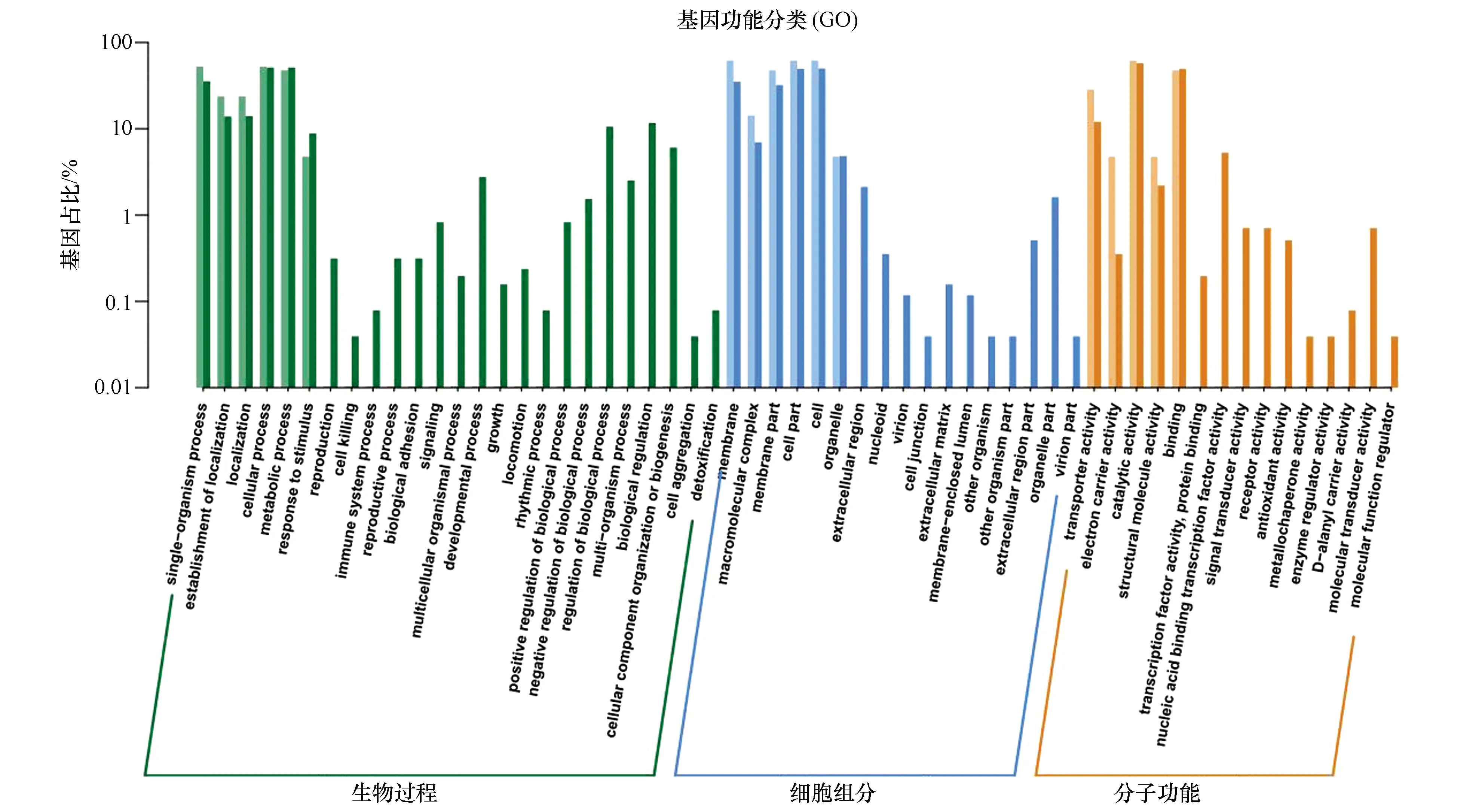
图6 6H组和C组转录组间差异表达基因的GO二级分类统计
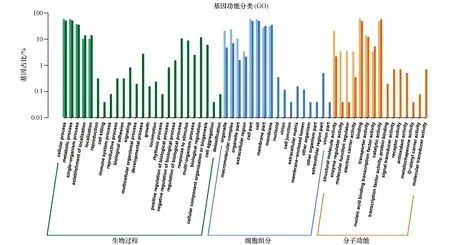
图7 4L组和C组转录组间差异表达基因的GO二级分类统计
2.6.4 6L组差异表达基因的注释 GO分析结果显示,在14个DEGs中,有8个基因至少能关联到MF的一个GO功能条目,有6个基因至少能关联到CC的一个GO功能条目,以及5个基因至少能关联到BP的一个GO功能条目。基于GO二级分类的功能分类,已注释的DEGs可以划分为17个二级小类(图8),它们隶属于BP、MF及CC这三个分支。在MF分支中,基因数目最多的类别是“结合作用”(binding),其包含了6个DEGs,其次是“催化活性”(catalytic activity),该分类包括了4个DEGs,排在第三位的是“结构分子活性”(structural molecule activity),共有3个DEGs;在CC分支中,细胞相关组分(cell,cell part)和细胞膜组分(membrane)等是DEGs对应的蛋白活动的两大主要场所;在BP中,“细胞内过程”(cellular process)、“代谢过程”(metabolic process)和“单组织过程”(single-organism process)这三个类别所占比例最多。
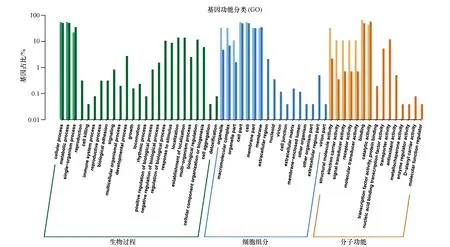
图8 6L组和C组转录组间差异表达基因的GO二级分类统计
2.6.5 讨论 从各组DEGs的GO注释结果来看,有着极大的相似性。在CC分支中,各组注释到的最相关条目均为“细胞相关组分”和“细胞膜组分”;在BP分支中,各组注释到的最相关条目均为“细胞内过程”、“代谢过程”、“单组织过程”。而在MF分支中,发现当处于最佳促生长的寡糖浓度时(即4H组和6H组),DEGs数目最多的类别依次为“催化活性”、“结合作用”、“转运活性”;而当寡糖浓度较低时(即4L组和6L组),DEGs数目最多的类别则变为“结合作用”、“催化活性”、“结构分子活性”。说明NA4和NA6在促乳酸菌生长的作用机理上有很大的相似性,对其影响最大的是“结合作用”以及“催化活性”。Lactobacillusplantarum在利用各类碳源时,乳酸菌中一些分解代谢物的蛋白与基因模块的结合起着重要的作用,例如,在FOS的利用中,LacI-GalR蛋白家族的Ccp A与基因位点cre的结合起了重要的调节作用[31]。而在利用多种碳源时,添加的寡糖会诱导乳酸菌先合成相关的催化酶[32],因此与“催化活性”相关的基因呈现上调表达。同时,当NA4和NA6浓度升高时,对细胞内的“转运活性”有更高的影响,将进一步通过基因敲除手段验证其作用机理。
3 结论
(1)通过全自动生长曲线分析仪筛选出乳酸菌Lactobacillusplantarum对各种寡糖的最适应用浓度,其中,GLC的最适应用浓度为0.4 g/L,NAOS为0.8 g/L,NA4、NA6、FOS、XOS均为0.6 g/L。
(2)在各个寡糖的最适浓度下,促乳酸菌增长的效果最好的依次是NA4、FOS、NA6、NAOS、XOS、GLC。
(3)完成了10组乳酸菌转录组测序,通过对差异表达基因的比较分析及GO注释,发现NA4和NA6在促乳酸菌生长的作用机理上有很大的相似性,对其影响最大的是“结合作用”以及“催化活性”。当寡糖浓度升高时对细胞内的“转运活性”有更高的影响。
