基于RNAi技术的转基因植物研究进展
2020-02-27黄春蒙朱鹏宇王智王晨光杜智欣魏霜张永江付伟
黄春蒙, 朱鹏宇, 王智, 王晨光, 杜智欣, 魏霜, 张永江, 付伟*
1.中国检验检疫科学研究院, 北京100176;
2.中国农业大学植物保护学院, 北京100193;
3.南宁海关技术中心, 南宁 530029;
4.广州海关技术中心, 广州 510623
进入21世纪以来,生物领域发展十分迅速,然而,我们仍然面临着许多复杂的挑战,其中包括在保持环境质量的同时保障人口迅速增长所带来的对粮食的需求。这不仅需要最大程度减少作物病虫害,也需要利用现代生物技术改进现有植物特性,特别是提高作物产量和抗逆性以适应气候变化。满足这些需求离不开包括RNA干扰(RNA interference,RNAi)技术在内的转基因技术、基因编辑技术以及合成生物学技术[1]。RNAi转基因植物是指基于RNAi技术,通过导入受体植物外源片段产生双链RNA(double strand RNA,dsRNA)并被加工成小RNA(small RNA,sRNA),进而引发与之互补的特定靶基因表达下降或沉默,从而获得的可用于农业生产或农产品加工的植物及其产品。目前,国内外研发人员已经利用RNAi技术开发出具备有害生物抗性、品质改良、有害物质减少等不同优良性状的生物技术产品。本文基于RNAi现象的发现和作用原理,阐述了RNAi转基因植物的设计、dsRNA或小干扰RNA(small interfering RNA,siRNA)的递送方式,并列举了RNAi转基因植物的研究实例以及全球部分商业化的RNAi转基因植物信息,旨在为RNAi技术的应用提供参考,以期能够充分利用RNAi技术助力发展高效绿色友好型农业。
1 RNAi的发现和作用机制
1.1 RNAi现象的发现
RNA沉默是由小分子非编码RNA(20~30 nt)引导的,包括多种沉默机制,如RNA降解、翻译抑制、染色质调控以及DNA缺失[2],几乎每个真核生物都存在某种形式的RNA沉默[3]。以siRNA和微小RNA(microRNA,miRNA)为主的各种类型的小RNA分子已得到鉴定并被证明参与了真核生物复杂的生命活动调控。如自然产生的反义RNA(antisense RNA)被发现参与了原核生物的DNA复制、RNA转录和翻译后调控[4],反义RNA通过与靶向mRNA杂交来抑制内源基因,由此产生的结构可能会阻碍细胞核的加工、细胞质输出,导致翻译抑制,进而使靶基因的mRNA快速降解[5]。而且,共抑制表达的正义RNA(sense RNA)与反义RNA一样,对靶基因同样具有抑制作用[6]。此外,在一个位点上的多拷贝转基因有可能会导致转录后基因沉默(post-transcriptional gene silencing,PTGS)。“异常RNA”的概念被引入作为RNA过丰度的结果,这些异常转录物可以用作引物,通过植物编码的RNA依赖的RNA聚合酶(RNA-dependent RNA polymerase,RdRp)来转化RNA或反义RNA,最终导致RNA降解[7]。这个概念同样适用于转基因重复序列易引发基因沉默的现象,转基因重复序列无论转录程度高低,都会直接导致RNA产物的异常从而触发RNA过丰度途径,因为转移DNA(transfer DNA,T-DNA)的插入往往导致直接重复(direct repeat,DR)或者倒置重复(inverted repeat,IR),而早期在马铃薯和矮牵牛花中的研究表明转基因低水平的表达与倒置重复紧密相关[8]。
同时,转基因重复也被认为涉及转录水平基因沉默(transcriptional gene silencing,TGS)。然而,这个过程需要内源性RdRp的存在,第一种植物编码RdRp在番茄中被发现,随后发现其在核糖核酸酶(ribonuclease,RNase)Ⅲ家族的Dicer酶或Dicer类似蛋白(Dicer like protein,DCL)作用下的产物是一种新的短单链RNA,被称为siRNAs[9]。siRNA在dsRNA和同源RNA的引导降解之间提供了联系。其实早在1991年,Cameron和Jennings[10]在观察到动物培养细胞的同源转基因可使转基因发生沉默后,便提出dsRNA可能参与同源性基因沉默的设想。1998年,Fire等[11]在研究秀丽隐杆线虫(Caenorhabditiselegans)时首次证明dsRNA是基因沉默的最主要的诱发因子,推测在植物中也存在类似机制,并将这种由dsRNA引发的抑制特定基因表达的现象称为RNAi。
除了这种典型的RNAi机制,还存在由动植物自身产生的miRNA调控基因表达的现象。1993年,研究者首次在线虫中发现miRNA途径,并在2000年被鉴定为Let-7miRNA,同时发现这种保守途径在线虫和哺乳动物中均存在[12],主要通过翻译抑制和/或降解同源mRNA来调节基因的表达。2001年首次报道了在哺乳动物细胞中采用RNAi技术抑制内源基因表达[13],进一步发展了RNAi技术的应用领域和方向,随后RNAi技术逐步应用于疾病治疗、基因功能研究和植物育种等领域。
1.2 RNAi的作用机制
在植物中,RNA沉默是一种抵御病毒、转座子和重复基因组等核酸入侵的防御机制,通常在转录后水平、转录水平、翻译起始水平调控基因表达。RNAi的核心机制分为3个阶段(图1):RNase Ⅲ Dicer酶将长dsRNA切割为siRNA双链体;选择siRNA双链体中的1条链,并将其装载到一种由Argonaute蛋白(AGO)组成的RNA诱导的沉默复合体(RNA-induced silencing complex,RISC);siRNA引导RISC识别并切割同源mRNA[14]。在某些物种中,RNAi还涉及RdRp,RdRp可以利用异常RNA或同源mRNA合成dsRNA,进一步参与到RNAi次级沉默信号扩大过程中[14]。
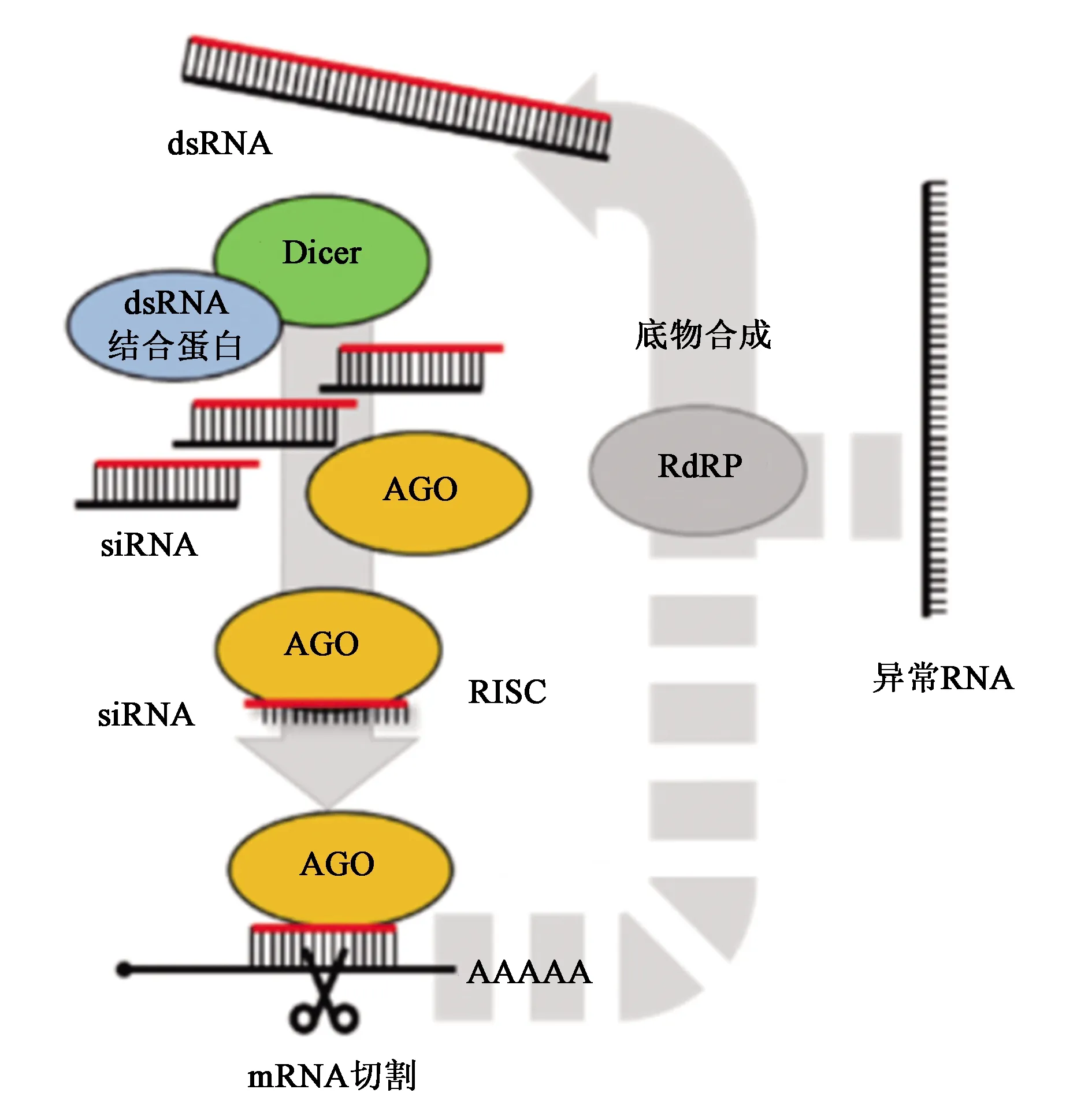
图1 RNAi机制示意图[14]
PTGS途径常常由转基因或病毒入侵引起,称为外源RNA沉默途径,分为倒置重复途径(IR-PTGS)和正义途径(S-PTGS)。IR-PTGS途径首先形成IR转录产物dsRNA,DCL4将dsRNA加工成21 nt的siRNA,经3′端甲基化转移酶HEN1甲基化修饰[15],装载到AGO蛋白形成RISC,在引导链RNA的引导下对同源mRNA进行切割降解。而S-PTGS途径主要负责异常RNA的降解。异常RNA分子通常缺少3′端polyA尾巴或5′端帽子,它们被认为是来源于高丰度的转基因正义/反义RNA、病毒RNA、截短型转录本以及复杂的基因插入、基因重复区域等。
TGS途径与PTGS途径具有共同的作用底物,并且都是通过靶向同源mRNA导致DNA甲基化,最终阻碍DNA转录。RNA介导的DNA甲基化(RNA-directed DNA methylation,RdDM)实际上是第一个发现的由sRNA控制的表观遗传机制。RdDM最初是在烟草植物中发现的,在存在病毒自主以RNA为模板进行RNA复制的情况下,其RNA被反转录成cDNA,并整合到基因组中的类病毒cDNA被特异性甲基化[16]。研究表明,与启动子区域互补的dsRNA可以诱导启动子甲基化和转录沉默[17]。RdDM由不同类型的序列诱导,有不同的靶标,常利用2种类型的dsRNA作为甲基化诱导物:具有反向重复序列的转基因发夹结构和dsRNA病毒[18]。RdDM不仅是控制重复序列和病毒入侵的响应机制,也是调控基因表达的调节网络的一部分。
除了siRNA所引起的RNA沉默现象,miRNA介导AGO1进行的靶基因切割也是一种重要的功能基因沉默模式,这在AGO1突变体互补实验中得到了证明。类似于其他真核生物,在miRNA的引导下,AGO1从mRNA碱基配对序列的中间进行切割[19],使被切割的mRNA暴露1个游离的5′端片段和1个3′羟基,再从3′端片段引发裂解,切割片段由末端尿苷酸转移酶HESO1在3′端对其进行尿酰化,最终降解。
植物的miRNA-靶基因相互作用并不总是导致AGO切割,也有可能导致翻译抑制。目前对植物miRNA介导的翻译抑制的分子机制的研究程度不如动物,但是二者存在相似之处。除2种常见的转录后调控模式外,植物miRNA也可以进入到RdDM机制中。在这种情况下,miRNA前体将由DCL 3处理产生较长的片段(24 nt),并被装载到AGO4/6/9系统以引导DNA序列发生甲基化修饰,进而在转录水平调控基因表达[20]。目前已在水稻中发现了这类miRNA的具体实例,其中有一类miRNAs由DCL3处理,装载到AGO4上引导DNA甲基化[21]。
2 RNAi转基因植物的研发现状
RNAi技术应用于植物育种多采用外源导入T-DNA的方式,将正义RNA、反义RNA或者倒置重复的靶基因序列放置于选定的启动子下形成dsRNA,并进一步加工成siRNA进入RNAi途径以调控植物发育,从而达到育种目的。此外,也有人工miRNA的途径和利用RdDM的途径,后者能够对靶基因的启动子进行表观遗传修饰从而调控靶基因表达以改变植物农艺性状。本文从转基因设计、sRNA递送方式、RNAi转基因植物的研究实例3个角度来阐述RNAi转基因植物的研发现状。
2.1 RNAi转基因设计
2.1.1正义或反义RNA 通过共表达mRNA靶标的正义和反义转录本,引入用于植物沉默的转基因dsRNA。该方法依赖于由2个独立转录的互补RNA杂交获得的用于RNA沉默的dsRNA。尽管与单独使用反义转录物相比,该方法的效率并未显著提高,且远小于使用IR的方法[22]。但是,正义和反义转录本可以在相反的启动子之间进行聚合转录,这种设计缩短了所需的T-DNA的长度,并避免重复使用序列。通过锥虫和果蝇的转基因实验证实了存在通过聚合转录的RNAi介导的基因抑制[23]。
2.1.2内含子嵌入的倒置重复序列 有时为了获得理想的表型,将内含子整合进反向重复区域作为一个保守的调控元件来保证基因时空特异性的表达和抑制。最常用的RNA沉默转基因结构是包含1个由间隔序列分隔的倒置重复IR序列。经过转录IR序列形成dsRNA并被加工成为成熟siRNA以有效地沉默靶基因。间隔序列可以是与靶标无关的任何序列,使用内含子衍生的可插入间隔可以增加沉默效率[24]。除了植物,基因内含子衍生的人工短发夹RNA(short hairpin RNA,shRNA)也能在哺乳动物细胞中诱导基因沉默[25]。有时在育种中追求更加理想的表型性状,会在表达一个基因的同时抑制另一个基因的表达。在高赖氨酸表型的基因工程玉米中,表达赖氨酸反馈不敏感的生物合成酶CordapA而抑制内源赖氨酸酮戊二酸还原酶/酵母氨酸脱氢酶(lysine ketoglutarate reductase/yeastine dehydrogenase,LKR/SDH)累积可以最大程度地提高成熟籽粒中游离赖氨酸的积累,从而获得高赖氨酸含量的玉米品种[26],在这个高赖氨酸含量的玉米中,抑制内源基因的设计则是采用插入内含子的倒置重复序列。
2.1.3人工miRNA 内源性miRNA序列可以被设计成人工miRNA(artificial miRNA,AmiRNA)。AmiRNA首次被报道是在人的细胞系中,内源性miRNA前体经修饰产生AmiRNA,从而选择性抑制互补靶序列[27]。在哺乳动物细胞中,尽管RNAi已经被证明是有效的,但dsRNA用于RNA沉默的途径是有限的。除少数胚胎细胞培养系统外,dsRNA还可诱导翻译抑制[28]。因此,由动物miRNA前体产生的shRNAs常被用于沉默动物系统中的内源性基因[29]。现有的研究已经给出使sRNA有效的发挥RNA沉默作用的途径和原则:在动物miRNAs中,来自5′端的第2~8位(种子区)的核苷酸位置可能是靶特异性的最重要的决定因素[30];sRNA双链中更倾向于将5′端处于不稳定状态的链装载到RISC[31];GC含量在30%~50%为佳[32];具有5-尿苷的sRNA可优先装载到AGO1[33]。同样地,miRNAs也能抑制报告基因和内源性基因在植物中的表达[34]。有学者基于以上原则,开发了一个用于设计miRNAs的平台,称为Web microRNA设计器(WMD)[35]。这个工具考虑到植物基因组序列信息,允许自动优化sRNA特性,以便有效地沉默靶基因,同时保持miRNAs在30多种植物中的靶向特异性。不像动物AmiRNA那样需要与靶基因完美配对,在设计植物AmiRNA时,需要考虑加入错配以避免产生siRNA沉默的移动性。已有研究表明可利用单个反向重复转基因敲除多个基因[36],同样地,将多个AmiRNA前体组合成转基因可以沉默多个靶基因。
2.1.4RdDM途径 农杆菌对瞬时表达基因的渗透、转基因砧木嫁接、反向育种和RdDM被归类为新植物育种技术(new plant breeding techniques,NPBTs)[37]。其中,RdDM方法可以在不改变基因组序列的情况下,通过转录基因沉默或启动子甲基化来修饰基因表达。甲基化可以由dsRNA诱导,该dsRNA需经RdDM介导的各种宿主酶处理,包括聚合酶Ⅳ和Ⅴ、AGO蛋白和胞嘧啶甲基转移酶[38];同时,siRNAs也可在靶DNA区诱导特定胞嘧啶残基(CG、CHG和CHH,其中H为A、C或T)的甲基化,导致TGS[39],这种改变可以稳定遗传数代。目的基因的TGS也可以通过人工导入与其启动子区域相对应的dsRNA来获得,这些表观遗传变化均可遗传。还有少数研究报道了由RdDM介导的内源基因的转录沉默,如在CaMV35S启动子控制下的绿色荧光蛋白(gteen fluorescent protein,GFP)基因[40]或水稻中的葡萄糖醛酸苷酶(glucuronidase,GUS)基因。此外,为了诱导水稻(OryzasativaL.)内源基因的TGS,Wakasa等[41]表达了不同启动子区域的dsRNA,从而诱导了RdDM,将来自谷蛋白多基因家族的GluB4基因作为靶标,与PTGS相比,TGS对靶标基因表现出特异性抑制。
2.2 sRNA递送方式
siRNA或dsRNA如何有效进入植物受体并针对靶基因发挥作用对于育种和基因功能研究十分关键。已经报道的技术手段是将合成的dsRNAs或siRNAs直接外源应用于植物表面,如直接喷雾、机械接种、高压喷涂以及采用促进RNA粘附的材料;也可采用间接方式,如纳米载入、蛋白质载体、病毒载入、T-DNA插入基因组等[42-44]。
目前采用最多的方式是构建转基因载体,如采用T-DNA插入、基因枪轰击将外源片段整合到植物基因组。已有研究报道了在拟南芥、矮牵牛花、番茄、马铃薯和烟草中均可利用T-DNA表达相应启动子区域的dsRNA从而诱导内源性基因的TGS[45-47]。
对于植物识别和吸收细胞外核酸的机制的研究是近年来才开始的。孟山都公司(现已并购入拜耳)的研究人员通过喷洒多核苷酸分子来改造转基因植物[48],研究表明,dsRNAs、siRNAs甚至单链DNA都能有效地促进植物的局部和系统沉默。与之类似的方法包括通过转入细菌表达dsRNAs的提取物[49]或含有载体肽的合成dsRNAs提取物[50]来沉默靶基因。除了dsRNAs,siRNAs也被采用纳秒脉冲激光诱导递送到细胞中[51]或与聚合物纳米粒子结合产生原生质体[52]以进行诱导培养。除孟山都公司在植物上直接应用siRNAs实现局部和全身性沉默外,Numata等[53]的研究表明,在含有蛋白质载体的复合物中喷洒合成的siRNAs可能会导致局部花青素的丧失,这可能是查尔酮合成酶基因沉默所致。将合成的GFP-siRNA溶液,在高压下通过常规压缩机和气刷手枪喷涂在GFP转基因烟草表面,可以沉默GFP基因的表达,使转基因烟草无荧光;在不施加高压的情况下,未观察到这种效果[54]。Numata等[53]将在体外合成的黄色荧光蛋白(yellow fluorescent protein,YFP)-siRNA与载体肽复合物渗透到杨树的叶片中,利用共聚焦激光扫描显微镜观察,结果发现YFP蛋白水平和荧光程度均降低。更简单的操作是利用软毛刷导入外源dsRNA,可以抑制转基因拟南芥中的新霉素磷酸转移酶Ⅱ(neomycin phosphotransferase,NPT Ⅱ)和GFP的转录水平[44]。上述研究表明,直接外源应用dsRNA、siRNA可以便捷高效地引起局部或系统沉默,并且可检测沉默效果。但是外源应用dsRNA、siRNA存在吸收率低的现象,推测生物损伤是植物RNA摄取的关键[54]。相比之下,利用T-DNA插入、基因枪轰击或病毒载入的方式更能引发稳定高效的RNA沉默效应,并且能够稳定遗传。
2.3 RNAi转基因植物的研究实例
目前,RNAi已广泛应用于植物育种领域,并已经研发出多种具有优良性状的作物,主要分为2大类:赋予植物抗性和调控植物代谢。其中,赋予植物抗性分为:抗虫、抗病毒、抗真菌、抗线虫、抗寄生性杂草等;调控植物代谢分为:产量性状改良、营养改良、消除过敏原等。在已有的RNAi技术中,抗虫作物的靶标包括西方玉米根虫(Diabroticavirgiferavirgifera)、南部玉米根虫(Diabroticaundecimpunctatahowardii)、科罗拉多马铃薯甲虫(Leptinotarsadecemlineata)、棉铃虫(Helicoverpaarmigera)等[55];抗真菌的靶标有致病疫霉(Phytophthorainfestans)、尖孢镰刀菌(Fusariumoxysporumf. sp.Cubense)和镰刀菌属(Fusariumsp.)[56-57];抗线虫的靶标为孢囊线虫、根结线虫、根腐线虫[58-60]。此外,抗病毒的作物有甜菜坏死黄脉病毒抗性烟草、马铃薯Y病毒抗性马铃薯、番茄黄化曲叶病毒抗性番茄、水稻东格鲁病毒抗性水稻、柑桔衰退病毒抗性墨西哥酸橙、马铃薯纺锤块茎类病毒抗性番茄[61];寄生杂草抗性的作物有抗独脚金杂草的转基因玉米[62]。产量性状改良的成功案例包括纤维品质优良的早熟丰产旱地品种棉花、种子含油量增加的拟南芥和麻风树、木质素含量降低的玉米[63-64]。营养改良的研究涉及同时增加类胡萝卜素和类黄酮含量的番茄、α-亚麻酸含量降低的大豆、β-胡萝卜素含量增加的柑桔、赖氨酸含量提高的玉米、油酸含量增加的花生、颜色改变的文心兰[65-67];消除过敏原的实例主要是沉默苹果中的Mald1、花生中的ARAH2、木质素中的P450、洋葱中的LFS、咖啡中的CaMXMT、烟草中的CYP82E4、番茄中的LeETR4和ACC、花生中的Arah2以及“无泪”洋葱中的黑麦因子合成酶基因[68]。此外,还有应用于医药的实例——利用RNAi技术生产抗乙肝病毒的生菜[69]。
国内外RNAi植物的研究已经取得长足发展,目前已经获得商业化批准、可用作食用或者加工原料的RNAi转基因植物的信息如表1所示。

表1 商业化应用的RNAi转基因植物
数据来源:欧洲食品安全局(European Food Safety Authority,EFSA);美国农业部(United States Department of Agriculture,USDA);国际农业生物技术应用服务组织(International Service for the Acquisition of Agri-biotech Applications,ISAAA)。
3 展望
RNAi技术作为2001年的十大科学成就之一,Science杂志在2002年又将其列为十大科学成就之首。同年,Nature杂志也将sRNA评选为重大科技成果之一。将RNAi技术应用于植物改良通常旨在减少害虫数量、提高产量、改良品质、降低种植成本、减少化学农药造成的环境污染,以期助力于实现农业的可持续发展。与基因编辑技术相比,RNAi技术也具有自身的不可替代性,RNAi技术能够克服物种的基因冗余和多倍性,允许一个基因家族的单个或多个成员或同源基因拷贝的沉默,如小麦和香蕉[70-71];RNAi抗虫作物的优点之一是几乎能够靶向任何基因,但是常规的转基因作物可能并没有商业化的杀虫蛋白可用;RNAi技术还可以提供广谱抗性。随着测序和生物信息学的发展,RNAi技术不仅可以利用转录后水平调控基因表达,也可以利用RdDM对基因组进行表观遗传修饰,给植物功能和育种研究带来了更多选择。而RNAi技术与合成生物学相结合可以更好地控制植物特定生长途径的代谢过程,能够达到精准改良育种的目的。值得注意的是,虽然RNAi技术在生物安全性方面的研究已经取得了一定的进展[72],但相关机制尚未明确,RNAi技术在作物改良中的应用潜力有待于进一步挖掘。
