不同发育时期亚麻茎秆中木质素积累相关miRNA及其靶基因的挖掘分析
2020-01-21谢冬微孙健
谢冬微 孙健


謝冬微(1982-),博士,主要从事亚麻种质资源优异基因挖掘研究工作,在作物种质资源创新和新品种选育方面积累了丰富的经验,理论基础夯实,科研业绩优异,被评为中国农业科学院麻类研究所科技创新工程“骨干专家”、中国农业科学院2018年度“优秀博士后”。先后主持国家自然科学基金青年项目1项,中国博士后自然科学基金面上项目1项,黑龙江省自然科学基金面上项目1项,以及其他地厅级科研项目5项。以第一作者在《Theoretical and Applied Genetics》《Frontiers in Plant Science》《Plant Science》《BMC Genomics》《Molecular Breeding》等SCI或EI收录期刊发表科技论文10篇,在《南方农业学报》《麦类作物学报》《大豆科学》《中国麻业科学》等国内核心期刊发表中文科技论文7篇。
摘要:【目的】深入挖掘分析亚麻茎秆不同发育时期木质素积累相关的miRNA及其靶基因,为解析亚麻纤维层木质素动态积累提供理论依据。【方法】以双亚4号(低木质素)和NEW(高木质素)花后20、30和40 d的茎秆为试验材料,基于miRNA测序和降解组测序结果分析miRNA转录水平动态变化及其靶基因注释功能,并采用实时荧光定量PCR检测2个亚麻品种间表达差异最显著的miRNA及其靶基因的表达模式。【结果】构建双亚4号和NEW花后20、30和40 d茎秆的miRNA文库,从中鉴定出305个miRNA,包括143个保守的miRNA和162个新鉴定的miRNA,且这些miRNA的靶基因不同转录本并非均受miRNA调控。将鉴定的305个miRNA与亚麻基因组序列信息进行靶基因预测,结果鉴定出286个miRNA的4807个靶基因,另外19个miRNA未预测到靶基因。通过对降解组测序数据进行整合分析,共获得21个miRNA的97个靶基因,共检测到97个降解位点,仅占鉴定出亚麻茎秆中miRNA靶基因总数(4807个)的2%,仍有大量的靶基因未被鉴定。对降解组中被降解的97个靶基因进行功能注释,发现有22个与植物生长发育有关,16个为转录因子,7个与植物次生代谢物积累有关。结合miRNA和降解组的测序结果,发现ama-miR156(Lus10003126)、bdi-miR397b-5p(Lus10017175)、lja-miR397(Lus10027782)、csi-miR160(Lus10021467)、aly-miR319c-p(Lus10008685)和gma-miR369h(Lus10011558)在双亚4号和NEW茎秆不同发育时期中最显著差异表达。实时荧光定量PCR检测结果显示,除gma-miR369h与其靶基因Lus10011558随亚麻茎秆的生长发育表达模式无明显规律外,其余5个miRNA与其靶基因表达模式恰好相反。【结论】亚麻茎秆木质素的积累过程与转录因子MYB、漆酶、生长素响应因子等编码基因密切相关,且这些基因可被其相应的miRNA负调控,并参与亚麻茎秆中木质素的积累。
关键词: 亚麻;木质素;积累;miRNA;靶基因;挖掘
中图分类号: S563.2 文献标志码: A 文章编号:2095-1191(2020)10-2321-10
Mining and analysis of miRNAs and target genes related to lignin accumulation in flax stalks at different developmental stages
XIE Dong-wei, SUN Jian*
(School of Life Science, Nantong University, Nantong, Jiangsu 226019, China)
Abstract:【Objective】Mining and analysis of miRNA and regulatory combination of target genes related to lignin accumulation in different development stage of stalk in flax could provide important reference for the analysis of dynamic lignin accumulation in flax fiber layer. 【Method】The stems of Shuangya 4 (low lignin) and NEW (high lignin) after flowering 20,30 and 40 d were used as research materials. Dynamic changes of miRNA transcription level and target gene annotation function were analyzed based on miRNA sequencing and degradation group sequencing. Real-time fluorescence quantitative PCR was used to detect the expression patterns of miRNAs and their target genes with the most significant differences between the two flax varieties. 【Result】Stalk miRNA libraries were constructed at 20,30 and 40 d after flowering of Shuangya 4 and NEW. 305 unique miRNAs were identified in this study,including 143 known conserved miRNAs and 162 newly miRNAs. Moreover,different transcripts of these miRNAs target genes were not all regulated by miRNAs. Target genes were predicted from 305 identified miRNAs and flax genome sequence information. A total of 286 miRNAs with 4807 target genes were identified,and another 19 miRNAs did not predict target genes. A total of 97 target genes of 21 miRNAs were obtained through integrated analysis of the sequencing data of the degradation group,and a total of 97 degradation sites were detected,accounting for only 2% of the total number of miRNA target genes(4807) identified in the flax stalk. There were still a large number of target genes that have not been identified. In the degradation group,97 target genes were functionally annotated,among which 22 were related to plant growth and development,16 were transcription factors and 7 were related to plant secondary metabolite accumulation. Combined with the sequencing results of miRNA sequencing and degradation group sequencing,it was found that ama-miR156(Lus10003126),bdi-miR397b-5p(Lus10017175),lja-miR397(Lus10027782),csi-miR160(Lus10021467), aly-miR319c-p(Lus10008685) and gma-miR369h (Lus10011558) were most significantly different in different development time of stalks between Shuangya 4 and NEW. The results of real-time quantitative fluorescence PCR showed that the expression pattern of gam-miR369h and its target gene Lus10011558 with the growth and development of stalk was not obvious, but the expression patterns of the other five miRNAs with their target genes were just the opposite. 【Conclusion】The process of lignin accumulation in flax stalks is closely related to MYB transcription factor,laccase,auxin response factor and SPL genes. These target genes can be negatively regulated by their corresponding miRNAs and participate in the accumulation of lignin in flax stalks.
Key words: flax; lignin; accumulation; miRNA; target gene; mining
Foundation item: National Natural Science Foundation of China(31801409); Heilongjiang Natural Science Foundation(C2018061)
0 引言
【研究意义】亚麻(Linum ustitatissimum L.)为亚麻科亚麻属一年生草本植物,是人类最早使用的天然植物纤维,其纤维纺织品具有透气、吸湿、抗菌及对人体无害等优点,日益受到人类的关注。但目前亚麻纺织业中优质亚麻纤维原料严重短缺,且其纤维品质和纺织性能易受木质素含量影响,究其原因是木质素会使纤维变硬、变脆(王玉富等,2008)。可见,木质素是制约纺织品质量的主要影响因子,且去除木质素工艺会产生大量废水从而造成环境污染。近些年来,已有学者对miRNAs在不同植物发育时期组织中的表达模式进行分析,发现miRNAs在植物的生长发育过程中发挥重要调控功能作用(Parizotto et al.,2004;Megraw et al.,2006;刘潮等,2019)。因此,深入挖掘亚麻不同发育时期茎秆中的miRNA及其靶基因,对揭示亚麻纤维层木质素动态积累的调控作用具有重要意义。【前人研究进展】目前,已有大量关于参与调控木质素合成的miRNA及其靶基因的研究报道。在拟南芥中,miR857通过作用于其靶基因AtLAC7来参与调控维管组织的次生生长,在miR857过表达植株中,AtLAC7基因表达量下调,漆酶活性下降10.8%,鲜重和茎直径分别下降6.5%和18.4%,茎拉伸力下降26.4%,木质素含量降低;而miR857敲除株系表现出漆酶活性增加33.3%,鲜重和茎直径分别增加14.7%和10.0%,茎拉伸力增加26.4%,植株木质素含量明显增加,次生木质部木质化程度增强(Zhao et al.,2015)。miR397b通过作用于其靶基因AtLAC4而调控木质素含量,miR397b过表达株系表现出木质素含量降低,次生壁厚度变薄,愈创木基和紫丁香基单体数目减少,但生殖生长期的植株表现出花序数目增多,致使果荚数也增多,产量提高,过表达AtLAC4基因的植株则表现出与之相反的表型(Wang et al.,2014)。在陆地棉中,GhLAC4基因mRNA的切割发生在miR397b及该基因的互补区域,说明GhLAC4基因是miR397b的靶基因,也暗示GhmiR397b可能参与棉花纤维中木质素的形成(丁妍等,2017)。在欧洲大叶杨中共发现49个漆酶基因(PtrLACs),其中有29个为Ptr-miR397a的靶基因,将Ptr-miR397a转入杨树中,结果发现Ptr-miR397a的靶基因中有17个PtrLACs下调表达,且转基因株系中木质素含量降低12%~22%,漆酶活性约下降40%,但木质素结构和组成未发生明显变化,证明漆酶参与木质素合成作用,且Ptr-miR397a是木质素合成的主要调节因子(Lu et al.,2013)。在玉米中,ZmmiR528作用于其靶基因ZmLAC3和ZmLAC5,当ZmmiR528过表达时,ZmLAC3和ZmLAC5基因表达量下降,木质素含量和茎杆穿刺强度降低;当敲减ZmmiR528或ZmLAC3过表达时,植株表现出相反的表型(Sun et al.,2018)。上述研究结果均表明,miRNA能通过调控其靶基因进而影响植株木质素含量。此外,转STTM164基因杨树的木质部细胞层数和细胞壁厚度较野生型明显增加,从而导致木质部加厚,木质素含量增加(梁澜,2017)。目前,关于亚麻木质素积累分子方面的研究报道相对较少。王进等(2009)以亚麻栽培品种白花为材料,采用半定量RT-PCR对亚麻木质素合成关键酶基因CCoAOMT1、4CL1、4CL2、COMT、F5H1、F5H2、F5H3在亚麻不同明显的组织及不同时期木质部和韧皮部的表达规律进行研究,结果发现这些基因呈组织特异性表达。龙松华等(2014)将已克隆获得的亚麻木质素合成关键酶咖啡酸-O-甲基转移酶基因(COMT)全长cDNA序列通过农杆菌介导转化亚麻,获得转基因植株,通过β-葡萄糖苷酸酶(GUS)染色和PCR检测,证实干扰载体已成功转入亚麻中,为推进亚麻纤维品质改良的分子育种打下基础。袁红梅等(2016)以亚麻茎总RNA为模版,通过RT-PCR扩增获得木质素合成关键酶糖基转移酶基因LuUGT72E1的全长开放閱读框(ORF)序列,并推测该酶参与木质素单体的糖基化修饰过程。【本研究切入点】至今,有关亚麻木质素积累分子调控方面的研究报道较少,且主要集中在上述少数几个木质素合成酶基因的克隆及其表达方面。虽然独立的miRNA数据分析可为解析不同发育时期亚麻茎秆中木质素积累提供重要信息,整合miRNA和降解组数据动态变化可为揭示miRNA调控基因表达提供较直接的证据,但目前针对参与亚麻茎秆木质素积累的miRNA及其靶基因挖掘分析的相关研究鲜见报道。【拟解决的关键问题】以双亚4号(低木质素)和NEW(高木质素)花后20、30和40 d的茎秆为试验材料,基于miRNA测序和降解组测序结果分析miRNA转录水平动态变化及其靶基因注释功能,采用实时荧光定量PCR检测2个亚麻品种间表达差异最显著的miRNA及其靶基因的表达模式,为解析亚麻纤维层木质素动态积累提供重要参考,也为利用生物技术手段培育低木质素的优质亚麻新品种提供科学依据。
1 材料与方法
1. 1 试验材料
供试材料为高木质素亚麻品种NEW(原产法国)和低木质素亚麻品种双亚4号(原产中国),由黑龙江省农业科学院经济作物研究所提供。于2019年4月中旬播种于黑龙江省农业科学院哈尔滨试验基地(东经126°68′,北纬45°65′),从播种到收获的年均降水量为350.2 mm,年均气温4.26 ℃。每份材料按行种植,行长10 m,行间距20 cm,设3次重复,10行区,小区面积20 m2,播种方式为条播,田间管理同大田生产。开花期对同一开花时间的亚麻植株进行挂牌标记,以备取样。主要试剂:Small RNA Sample Pre Kit、T4 RNA Ligase 1和T4 RNA Ligase 2购自天根生化科技(北京)有限公司;TRIzol(Invitrogen)购自北京雅安达生物技术有限公司;RevertAid First Strand cDNA Synthesis Kit(Fermentas)和SYBR Premix Ex Taq购自宝日医生物技术(北京)有限公司。主要仪器设备:Hiseq 2500(Illumina,美国)、2100 Bioanalyzer(Agilent,德国)和罗氏LightCycler 2.10 PCR仪(Roche,瑞士)等。
1. 2 试验方法
1. 2. 1 miRNA文库构建及测序分析 采集亚麻品种双亚4号和NEW花后20、30和40 d的茎秆下部5 cm(子叶节以上5~10 cm区段)进行RNA提取,设3次生物学重复,共18个样品。RNA检测合格后,取其1.5 μg作为RNA样本起始量,用ddH2O补充至6.0 μL,使用Small RNA Sample Pre Kit试剂盒进行mi-RNA文库构建。由于miRNA的5'端有磷酸基团、3'端有羟基,利用T4 RNA Ligase 1和T4 RNA Ligase 2分别在其3'端和5'端连接上接头,反转录合成cDNA,切胶回收得到的目的片段即为miRNA文库。使用Qubit 2.0对miRNA浓度进行检测,并稀释至1 ng/μL,使用Agilent 2100 Bioanalyzer对Insert Size进行检测,并以实时荧光定量PCR对miRNA的有效浓度进行准确定量,以保证miRNA文库质量。采用Illumina HiSeq2500进行高通量测序,测序读长为Single-end(SE)50 nt。基于测序结果,鉴定分析miRNA及其靶基因的转录本信息
1. 2. 2 降解组cDNA文库构建及测序分析 采集亚麻品种双亚4号花后20、30和40 d的茎秆下部5 cm(子叶节以上5~10 cm区段)进行RNA提取,通过磁珠捕获mRNA,5'-adaptor连接,以构建降解组cDNA文库,并用Hiseq 2500进行降解组测序。原始Tags经过去接头和过滤低质量,获得Clean Tags和Cluster Tags(Clean Tags聚类数据)。将Cluster Tags与参考基因组进行比对,分析其在参考基因组上的分布情况。将Cluster Tags与Rfam数据库进行比对,注释非编码RNA功能,未被注释的序列将用于降解位点分析。利用Cleaveland进行降解位点检测(Addo-Quaye et al.,2009),设置条件P-value<0.05。
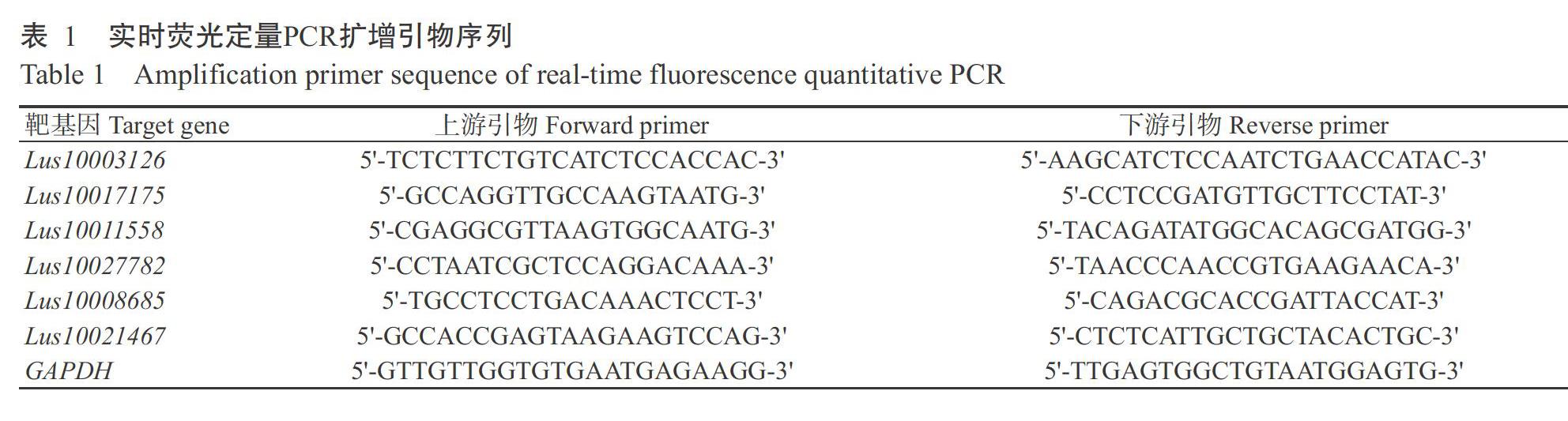
1. 2. 3 实时荧光定量PCR验证 选取长势一致且健康的双亚4号植株,采集花后0、10、20、30和40 d等5个时间点的茎杆下部5 cm(子叶节以上5~10 cm区段),设3次重复,液氮迅速冷冻后置于-80 ℃冰箱保存备用。用TRIzol提取上述样品的总RNA,RNA经DNA酶消化处理后反转录合成第一链cDNA。根据miRNA测序和降解组测序结果筛选出6个在双亚4号和NEW不同发育时期表达差异最显著的miRNA,在降解组找到其被降解的靶基因,并根据靶基因的CDS序列,采用Primer 5.0设计引物,内参基因为GAPDH(表1)。实时荧光定量PCR在罗氏LightCycler 2.10 PCR仪上进行,荧光染料为SYBR Premix Ex Taq,设3次生物学重复。反应体系20.0 μL:2×Hi SYBR Green qPCR Mix 10.0 μL,10 μmol/L上、下游引物各0.8 μL,cDNA模板2.0 μL,ddH2O补足至20.0 μL。扩增程序:95 ℃预变性15 min;95 ℃ 10 s,60 ℃ 30 s,65 ℃ 5 min,进行40个循环,4 ℃保存。以2-ΔΔCt法计算目的基因的相对表达量(Livak and Thomas,2001)。
1. 3 统计分析
试验数据采用Excel 2010进行统计分析,并以GraphPad Prism 6.01制图。
2 结果分析
2. 1 miRNA转录水平分析结果
试验前期对亚麻不同发育时期茎秆中木质素进行含量测定及扫描电镜观察,结果发现亚麻花后20~40 d是木质素急剧积累的重要时期。因此,本研究选取亚麻品种双亚4号和NEW花后20、30和40 d的茎秆构建miRNA文库,并鉴定出305个miRNA,其中包括143个保守的miRNA和162个新鉴定的miRNA。本研究还对部分miRNA(包括保守的miRNA和新鉴定的miRNA)的靶基因不同转录本进行详细分析,以期了解可变剪接是否造成转录本水平miRNA靶位点丢失,结果发现这些miRNA的靶基因不同转录本并非均受miRNA调控。如在保守的miRNA中,PB.11839可转录出5个转录本异构体,其中4个转录本可被miR160调控,另外丢失miR160靶位点的1个转录本是通过外显子跳跃产生(图1-A);PB.19964可轉录出7个转录本异构体,其中5个转录本可被miR168调控,其余2个转录本经外显子跳跃事件而丢失miR168靶位点(图1-B);在新鉴定的miRNA中,PB.16138可转录出2个转录本,1个可被unconservative_scaffold204_9948调控,1个经可变接受位点事件后丢失靶位点(图1-C)。为了更好的研究亚麻miRNA的功能,本研究将鉴定的miRNA与亚麻基因组序列信息进行靶基因预测,结果获得286个miRNA的4807个靶基因,另外19个miRNA未预测到靶基因,可能是因为亚麻基因组提交数据并不完整,造成靶基因预测不全面。
2. 2 降解组测序分析结果
利用降解组测序技术鉴定miRNA靶基因是目前最常用和最高效的方法。以亚麻品种双亚4不同发育时期(20、30和40 d)的茎秆构建3个降解组的cDNA文库(D01、D02和D03),并结合上述高通量测序获得的不同发育时期亚麻茎秆miRNA文库信息进一步鉴定亚麻茎秆miRNA靶基因。利用Cleaveland进行降解位点检测,结果发现,D01检测到86个靶基因被降解,共有86个靶基因降解位点;D02检测到92个靶基因被降解,共有92个靶基因降解位点;D03检测到83个靶基因被降解,共有83个靶基因降解位点。通过对3个降解组测序数据进行整合分析,共获得21个miRNA的97个靶基因,共检测到97个降解位点,仅占鉴定出亚麻茎秆中miRNA靶基因总数(4807个)的2%,仍有大量的靶基因未被鉴定。为了探索靶基因的功能,本研究将被降解的97个靶基因与同源物种比对进行功能注释,其中有22个靶基因与植物生长发育有关,如生长素响应因子(Auxin response factor)、生长调节因子(Growth regulate factor)和乙烯响应因子(Ethylene response factor)等;有16个靶基因为转录因子,如MYB、bHLH77和TCP2等;有7个靶基因与植物次生代谢积累有关,如铜氧化酶(Poly copper oxidase)、P450次生代谢酶(P450 secondary metabolism)和铜锌超氧化物歧化酶(Copper zinc superoxide dismutase)等(表2)。
2. 3 实时荧光定量PCR验证结果
结合miRNA测序和降解组测序结果筛选出6个在双亚4号和NEW不同发育时期表达差异最显著的miRNA,并根据降解组找到其被降解的靶基因:ama-miR156(Lus10003126)、bdi-miR397b-5p(Lus 10017175)、gma-miR396h(Lus1011558)、lja-miR397(Lus10027782)、csi-miR160(Lus10021467)和aly-miR319c-p(Lus10008685),共有6对miRNA-靶基因调控组合,最后采用实时荧光定量PCR检测其在亚麻茎秆不同发育时期的表达模式,结果如图2所示。6个miRNA及其对应靶基因均随茎秆发育呈相反的表达模式。ama-miR156的相对表达量随亚麻茎秆发育呈逐渐下降趋势,而其靶基因Lus10003126的相对表达量呈逐渐升高趋势,花后40 d相对表达量达最高,该靶基因被注释为启动子结合蛋白SBP,参与亚麻生长发育。bdi-miR397b-5p和lja-miR397的相对表达量均随亚麻茎秆发育呈逐渐升高趋势,均在花后40 d达最高,其靶基因Lus10017175和Lus10027782的表达模式恰好相反,整体上呈逐渐降低趋势,在花后40 d相对表达量达最低值,且这2个靶基因均被注释为漆酶基因,参与亚麻中次生代谢产物木质素的形成。aly-miR319c-p的相对表达量随亚麻茎秆发育呈逐渐下降趋势,其靶基因Lus10008685的表达模式恰好相反,在花后0~20 d的相对表达量缓慢升高,从花后30 d开始相对表达量迅速升高,该靶基因被注释为转录因子MYB,据报道该家族转录因子调控植物木质素生物合成过程(李维静等,2013),因此推断该靶基因对亚麻木质素积累及结构形成发挥重要作用。csi-miR160的相对表达量随亚麻茎秆发育呈逐渐下降趋势,在花后40 d时降至最低值,其靶基因Lus10021467的相对表达量在花后0~10 d无明显变化,从花后20 d开始相对表达量急剧增加,花后40 d达最高值,该靶基因被注释为生长素响应因子,说明该基因与亚麻的生长发育及木质素积累关系密切。gma-miR369h及其靶基因Lus10011558随亚麻茎秆的生长发育其表达模式无明显规律,gma-miR369h的相对表达量在花后0~30 d无明显变化,花后40 d明显下降,其靶基因Lus10011558的相对表达量在花后10~40 d均无明显变化。故推测这些miRNA及其靶基因在亚麻茎秆不同发育期相互作用,共同参与亚麻茎秆发育与次生代谢产物积累。
3 讨论
在纺织业、造纸业等工业生产中,木质素是造成纤维加工废弃物污染,影响纤维品质的主要因素。木质素是影响植物材料加工利用的主要限制因子。纤维中木质素含量过高会导致纤维偏硬脆,可纺性和服用性能差。由于木质素的去除难度大,木质素含量高则易导致脱胶成本高和工业污染(Vanholme et al.,2012)。因此,亚麻中木质素含量直接影响亚麻纤维的品质,直接决定其经济效益(Meagher and Beecher,2000)。Huis等(2012)将代谢组学和转录组学相结合对亚麻茎秆的木质部和茎外韧皮纤维部分的木质化进行研究,结果发现代谢中间体松柏醇最终可以产生木质素,但该研究未提及亚麻茎秆木质素积累过程中相关基因的表达情况。至今,未见有关调控亚麻茎秆木质素积累的相关miRNA及其靶基因的研究报道。本研究对亚麻茎秆不同发育时期的miRNA转录水平进行深入分析,结果发现亚麻茎秆木质素的积累过程与转录因子MYB、漆酶及生长素响应因子等编码基因密切相关。该结论推进了亚麻茎秆木质素积累方面的研究进展,并为利用生物技术手段培育低木质素优质纤维亚麻新品种提供了重要参考。
miR156是一类调控细胞生长的miRNA,其靶基因SPL10和SPL11能控制细胞分裂(Nodine and Bartel,2010),在胚发育早期(花后10 d)miR156可抑制SPL10和SPL11基因的表达,阻碍植株生长发育(Pa-latnik et al.,2003;Wang et al.,2012)。本研究也发现,ama-miR156在亚麻茎秆发育过程中的相对表达量呈逐渐下降趋势,且其靶基因Lus10003126在花后20~40 d的相对表达量明显升高,说明ama-miR156在亚麻茎秆发育中后期对其靶基因的抑制作用越来越弱,从而促进茎秆纤维层的正常发育。此外,杨树中Ptr-miR397a过表达会引起17个漆酶基因的相对表达量下调,导致木质素含量降低(Lu et al.,2013);拟南芥AtmiRNA397b过量表达会致使AtLAC4基因的相对表达量降低,导致木质素含量降低(Wang et al.,2014)。本研究中,bdi-miR397b-5p和lja-miR397的相对表达量随亚麻茎秆发育呈逐渐升高趋势,均在花后40 d达最高值,其靶基因Lus10017175和Lus10027782表达模式恰好相反,整体上呈逐渐降低趋势,由于这2个靶基因均被注释为漆酶,故推测这2个miRNA负调控亚麻木质素合成,且在不同物种中的功能具有一定的保守性。
本研究中,aly-miR319c-p的相对表达量随亚麻茎秆发育呈不断下降趋势,其靶基因Lus10008685被注释为转录因子MYB,相对表达量不断升高。Shen等(2012,2013)研究发现,柳枝稷中过表达R2-R3-MYB转录因子基因家族成员PvMYB4,在未经酸预处理的条件下即可使糖化效率增加300%,导使纤维素乙醇产量比普通植株提高2.6倍。由于miR319c-p及其靶基因在植物木质素发育过程中的作用未见相關研究报道,因此aly-miR319c-p及其靶基因Lus10008685调控木质素合成的功能是本研究新发现,但其具体作用机理还需进一步研究验证。miR160是植物中一类较重要的miRNA家族,具有组织特异性表达的特点,能在模式植物拟南芥中反馈调节生长素响应因子的表达,从而影响植株对生长素的敏感性,进而调控植物的生长发育(Mallory et al.,2005)。吴书昌(2016)利用深度测序技术发现miR160在棉花胚珠的发育及纤维起始过程中特异表达,推测其可能会影响棉花胚珠的发育和纤维的起始。本研究也发现,随着亚麻茎秆的发育csi-miR160相对表达量不断下降,其靶基因Lus10021467的相对表达量不断上升,该靶基因被注释为生长素响应因子,推测csi-miR160及其靶基因Lus10021467在亚麻茎秆纤维发育和木质素积累方面扮演重要调控角色,进一步验证前人的相关研究结果。
虽然在纺织业、造纸业等工业生产中,木质素是造成纤维加工废弃物污染及影响纤维品质的主要因素,但木质素在植物组织中具有增强细胞壁及黏合纤维的作用,对植物的生长起机械支持抗压的作用。因此,在今后培育低木质素亚麻品种时要充分利用miRNA及靶基因在亚麻韧皮部(纤维层)特异表达这一特性进行有目标地降低亚麻韧皮部(纤维层)的木质素含量,而不影响其植株的抗倒伏性,从而培育抗倒伏性强、纤维品质好的亚麻新品种。
4 结论
亚麻茎秆木质素的积累过程与转录因子MYB、漆酶、生长素响应因子等编码基因密切相关,且这些靶基因可被其相应的miRNA负调控,并参与亚麻茎秆中木质素的积累。
参考文献:
丁妍,王燕美,刘进元. 2017. 陆地棉miR397b的生化功能验证[J]. 上海农业学报,33(1):10-14. [Ding Y,Wang Y M,Liu J Y. 2017. Identification of the biochemical function of miR397b in Gossypium hirsutum[J]. Acta Agriculturae Shanghai,33(1):10-14.]
李维静,苏衍菁,苏相亭,卜仕金. 2013. MYB转录因子在植物木质素合成中的调控机理[J]. 山东畜牧兽医,34(11):69-71. [Li W J,Su Y J,Su X T,Bu S J. 2013. Regulation mechanism of MYB transcription factor in plant lignin synthesis[J]. Shandong Journal of Animal Husbandry and Veterinary Science,34(11):69-71.]
梁澜. 2017. PtrmiR164a在杨树次生细胞壁合成过程中的功能研究[D]. 重庆:西南大学. [Liang L. 2017. Functional characterization of PtrmiR164a invoved in the regulation of secondary cell wall in Populus[D]. Chongqing:Southwest University.]
刘潮,褚洪龙,韩利红,杨云锦,高永,唐利洲. 2019. 桑树NBS-LRR类基因家族的全基因组鉴定及其调控micro-RNAs分析[J]. 江苏农业学报,35(3):544-553. [Liu C,Chu H L,Han L H,Yang Y J,Gao Y,Tang L Z. 2019. Genome-wide identification of NBS-LRR genes and regulation analysis by microRNAs in mulberry[J]. Jiangsu Journal of Agricultural Sciences,35(3):544-553.]
龙松华,乔瑞清,李翔,陈信波,邓欣,邱财生,郭媛,郝冬梅,王玉富. 2014. 亚麻木质素合成相关基因COMTRNAi表达载体构建及转化[J]. 中国麻业科学,36(4):169-173. [Long S H,Qiao R Q,Li X,Chen X B,Deng X,Qiu C S,Guo Y,Hao D M,Wang Y F. 2014. Construction of RNAi vector caffeic acid O-methyltransferase gene and transformation in flax[J]. Plant Fiber Sciences in China,36(4):169-173.]
王进,陈信波,高原,张彦红,龙松华,邓欣,何东锋,王玉富. 2009. 亚麻木质素合成关键酶基因表达分析[J]. 作物学报,35(8):1468-1473. [Wang J,Chen X B,Gao Y,Zhang Y H,Long S H,Deng X,He D F,Wang Y F. 2009. Expression of critical lignin metabolism genes in flax(Linum usitatissimum)[J]. Acta Agronomica Sinica,35(8):1468-1473.]
王玉富,黄海燕,薛召东,邱财生,郝冬梅. 2008. 亚麻CAD基因克隆及反义表达载体的构建[J]. 中国麻业科学,30(6):289-292. [Wang Y F,Huang H Y,Xue Z D,Qiu C S,Hao D M. 2008. Cloning of partial sequences of cinnamyl alcohol dehydrogenase gene from flax and construction of its antisense expression vector[J]. Plant Fiber Sciences in China,30(6):289-292.]
吴书昌. 2016. MiR160在棉花胚珠发育中功能分析[D]. 武汉:华中农业大学. [Wu S C. 2016. Functional characteri-zation of miR160 in cotton ovule development[D]. Wuhan:Huazhong Agricultural University.]
袁紅梅,郭文栋,赵丽娟,于莹,吴建忠,程莉莉,赵东升,康庆华,黄文功,姚玉波,宋喜霞,姜卫东,刘岩,马廷芬,吴广文,关凤芝. 2016. 亚麻糖基转移酶基因LuUGT72E1的克隆与表达分析[J]. 作物杂志,(4):62-67. [Yuan H M,Guo W D,Zhao L J,Yu Y,Wu J Z,Cheng L L,Zhao D S,Kang Q H,Huang W G,Yao Y B,Song X X,Jiang W D,Liu Y,Ma T F,Wu G W,Guan F Z. 2016. Cloning and expression analysis of the glycosyltransferase gene LuUGT72E1 in flax[J]. Crops,(4):62-67.]
Addo-Quaye C,Miller W,Axtell M J. 2009. CleaveLand:A pipeline for using degradome data to find cleaved small RNA targets[J]. Bioinformatics,25(1):130-131.
Huis R,Morreel K,Fliniaux O,Lucau-Danila A,Fenart S,Grec S,Neutelings G,Chabbert B,Mesnard F,Boerjan W,Hawkins S. 2012. Natural hypolignification is associated with extensive oligolignol accumulation in flax stems[J]. Plant Physiology,158(4):1893-1915.
Livak K J,Thomas D S. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods,25(4):402-408.
Lu S,Li Q,Wei H,Chang M J,Tunlaya-Anukit S,Kim H,Liub J,Song J Y,Sun Y H,Yuan L,Yeh T F,Peszlen I,Ralph J,Sederoff R R,Chiang V L. 2013. Ptr-miR397a is a negative regulator of laccase genes affecting lignin content in Populus trichocarpa[J]. Proceedings of the National Academy of Sciences of the United States of America,110(26):10848-10853.
Mallory A C,Bartel D P,Bartel B. 2005. microRNA-directed regulation of Arabidopsis AUXIN RESPONSE FACTOR17 is essential for proper development and modulates expression of early auxin response genes[J]. The Plant Cell,17(5):1360-1375.
Meagher L P,Beecher G R. 2000. Assessment of data on the lignan content of foods[J]. Journal of Food Composition and Analysis,13(6):935-947.
Megraw M,Baev V,Rusinov V,Jensen S T,Kalantidis K,Hatzigeorgiou A G. 2006. microRNA promoter element discovery in Arabidopsis[J]. RNA,12(9):1612-1619.
Nodine M D,Bartel D P. 2010. microRNAs prevent precocious gene expression and enable pattern formation du-ring plant embryogenesis[J]. Genes Development,24(23):2678-2692.
Palatnik J F,Allen E,Wu X,Schommer C,Schwab R,Carrington J C,Weigel D. 2003. Control of leaf morphogenesis by microRNAs[J]. Nature,425(6955):257-263.
Parizotto E A,Dunoyer P,Rahm N,Himber C,Voinnet O. 2004. In vivo investigation of the transcription,proces-sing,endonucleolytic activity,and functional relevance of the spatial distribution of a plant miRNA[J]. Genes Development,18:2237-2242.
Shen H,He X,Poovaiah C R,Wuddineh W A,Ma J,Mann D G J,Wang H,Jackson L,Tang Y,Chen F,Dixon R A. 2012. Functional characterization of the switchgrass(Pani-cum virgatum) R2R3-MYB transcription factor PvMYB4 for improvement of lignocellulosic feedstocks[J]. New Phytologist,193(1):121-136.
Shen H,Poovaiah C R,Ziebell A,Tschaplinski T J,Pattathil S,Gjersing E,Engle N L,Katahira R,Pu Y,Sykes R. 2013. Enhanced characteristics of genetically modified switchgrass(Panicum virgatum L.) for high biofuel production[J]. Biotechnology for Biofuels,6(1):71-86.
Sun Q,Liu X G,Yang J,Liu W W,Du Q G,Wang H Q,Fu C X,Li W X. 2018. microRNA528 affects lodging resistance of maize by regulating lignin biosynthesis under nitrogen-luxury conditions[J]. Molecular Plant,11(6):806-814.
Vanholme R,Storme V,Vanholme B,Sundin L,Christensen J H,Goeminne G,Halpin C,Rohde A,Morreel K,Boerjan W. 2012. A systems biology view of responses to lignin biosynthesis perturbations in Arabidopsis[J]. The Plant Cell,24(9):3506-3529.
Wang C Y,Zhang S,Yu Y,Luo Y C,Liu Q,Ju C,Chen Y Q. 2014. miR397b regulates both lignin content and seed number in Arabidopsis via modulating a laccase involved in lignin biosynthesis[J]. Plant Biotechnology Journal,12(8):1132-1142.
Wang S K,Wu K,Yuan Q B,Liu X Y,Liu B,Lin X Y,Zeng R Z,Zhu H T,Dong G J,Qian Q,Zhang G Q,Fu X D. 2012. Control of grain size,shape and quality by OsSPL16 in rice[J]. Nature Genetics,44(8):950-954.
Zhao Y Y,Lin S,Qiu Z B,Cao D C,Wen J L,Deng X,Wang X H,Lin J X,Li X J. 2015. microRNA857 is involved in the regulation of secondary growth of vascular tissues in Arabidopsis [J]. Plant Physiology,169(4):2539-2552.
(責任编辑 陈 燕)
