文蛤过氧化氢酶的生物信息学分析
2020-01-02张志东陈爱华吴杨平陈素华李秋洁
张志东,张 雨,陈爱华*,吴杨平*,曹 奕,陈素华,田 镇,2,李秋洁,2
(1. 江苏省海洋水产研究所,江苏 南通 226007;2. 上海海洋大学 水产与生命学院,上海 201306)
文蛤(Meretrixmeretrix)隶属于软体动物门(Mollusca)、瓣鳃纲(Lamellibrabchia)、帘蛤目(Veneroida)、帘蛤科(Veneridae)、文蛤属(Meretrix),是我国重要的经济贝类之一[1]。近年来,随着海水养殖业的发展,文蛤养殖面临着污染、疾病等问题的困扰,因此选育抗逆性强的文蛤良种是目前行之有效的手段之一。过氧化氢酶(CAT)在文蛤抗逆性方面具有重要的指示作用。机体在逆境中,会大量产生过氧化氢(H2O2),其具有强烈的氧化作用,对细胞产生毒害[2]。过氧化氢酶(Catalase, CAT)是一类广泛存在于生物体内的活性酶,可与生物体内的过氧化氢特异性结合,使之分解为水和氧气,维持机体氧化还原平衡,使机体免受氧化应激(Oxidative Stress,OS)的损伤[3]。十九世纪初,Thenard首次发现过氧化氢酶。到了20世纪,Oscar Loew将该酶正式命名为“Catalase”,即过氧化氢酶,又称触酶[4]。以前主要研究过氧化氢酶活性及作为生物抗逆性的标志物[5-7]。近年来,许多水生生物过氧化氢酶基因陆续被克隆出来[8-11],Wang等[12](2012)首次克隆出文蛤过氧化氢酶基因(MmeCAT),进一步推动了过氧化氢酶在分子生物学领域的研究。目前关于文蛤过氧化氢酶蛋白结构的研究尚未有报道,本研究在已公布的MmeCAT序列的基础上,利用生物信息学方法对其表达产物理化性质、亲水性、跨膜结构域、磷酸化位点、糖基化位点、信号肽、亚细胞定位、功能结构域、二级结构以及三级结构等进行预测,旨在为文蛤过氧化氢酶的结构与功能的研究提供理论基础。
1 材料与方法
1.1 数据来源
文蛤MmeCAT基因来源于GenBank,其登录号为JQ005875.1。对该基因完整编码蛋白质序列进行 BLAST 分析;与其同源性较高的有织锦巴非蛤(Paphiatextile),登录号为KF673103.1;河蚬(Corbiculafluminea),登录号为KX211962.1;硬壳蛤(Mercenariasp.),登录号为JN671460.1;合浦珠母贝(Pinctadafucata),登录号为HQ703465.1;厚壳贻贝(Mytiluscoruscus),其登录号为KX957929.1等。以上物种CAT基因均由NCBI网站(https: / /www.ncbi.nlm.nih.gov /)GenBank数据库下载。
1.2 实验方法
首先用BioEdit7.0软件将文蛤CAT基因完整编码蛋白序列翻译成氨基酸序列待用。然后将氨基酸序列输入ExPASy ProtParam在线工具(https://web.expasy.org/protparam/)分析该蛋白质的理化性质。理化性质包括氨基酸组成、分子式、相对分子质量、理论等电点、消光系数、半衰期、不稳定系数、脂肪系数等,并用Excel2007整理所得数据。通过ProtScale工具(https://web.expasy.org/protscale/)预测该蛋白质疏水性、亲水性。通过TMHMM Server v 2.0(http://www.cbs.dtu.dk/services/TMHMM/)预测跨膜结构区域。通过NetPhos 3.1 Server在线工具(http://www.cbs.dtu.dk/services/NetPhos/)对该蛋白质的磷酸化位点进行预测。利用NetOGlyc 4.0 Server在线工具(http://www.cbs.dtu.dk/services/NetOGlyc/)对该蛋白质进行糖基化位点预测。通过SignalP 5.0 Server软件判断该蛋白质信号肽存在与否。通过PSORT Ⅱ Prediction在线软件(https://psort.hgc.jp/form2.html)对该蛋白质进行亚细胞定位。使用BlastP对该蛋白质保守结构域进行分析。使用SOPMA在线工具(https://npsa-prabi.ibcp.fr)分析该蛋白质二级结构,并用SWISS-MODEL在线工具(https://www.swissmodel.expasy.org/)预测该蛋白质三级结构。运用STRING数据库(https://string-db.org/),设置为高置信度0.7,不限制数量,构建与CAT相互作用的蛋白网络。
2 结果与分析
2.1 文蛤过氧化氢酶理化性质分析
通过ExPASy ProtParam在线工具(https://web.expasy.org/protparam/)获得文蛤过氧化氢酶的理化性质。文蛤过氧化氢酶由511个氨基酸组成,其中甘氨酸(Gly)有42个,含量最多占8.2%;色氨酸(Trp)和半胱氨酸(Cys)均有6个,含量最少占1.2%(见图1)。相对分子质量为58 181.29 Da;理论等电点为8.05;带负电荷氨基酸残基数(Asp+Glu)为63个,带正电荷氨基酸残基数(Arg+Lys) 为65个;其分子式为C2588H3928O767N730S20;假设所有半胱氨酸全部形成胱氨酸,其消光系数为63 175 mol /L, 相应的吸光度为 1.086; 假设所有的半胱氨酸均未形成胱氨酸时,消光系数为 62 800 mol /L,相应的吸光度为 1.079;其半衰期为30 h,脂肪族氨基酸指数为57.28,不稳定系数为27.77(<40),可知文蛤过氧化氢酶为稳定蛋白质。
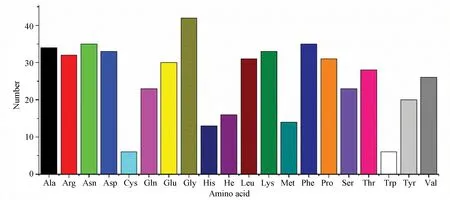
图1 文蛤过氧化氢酶氨基酸组成Fig.1 Amino acid composition of catalase in M.meretrix
2.2 文蛤过氧化氢酶疏水性与亲水性分析
通过ProtScale工具得到文蛤过氧化氢酶疏水性与亲水性见图2。图2中的负值表示亲水性,该值越小说明该蛋白质亲水性越强;正值为疏水性,正值越大说明该蛋白质疏水性越强。文蛤过氧化氢酶第311位的精氨酸(Arg)分值最高为1.767 ;第 500位的缬氨酸(Val) 和 491 位的丙氨酸(Ala)分值最低,为-3.200 。亲水性总平均值为-0.732,表明过氧化氢酶为亲水性蛋白。
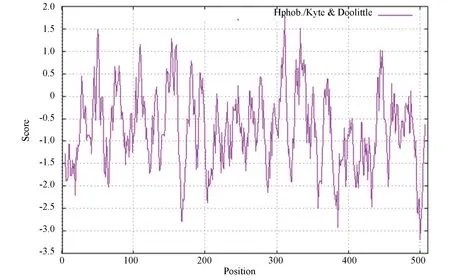
图2 文蛤过氧化氢酶亲水性与疏水性Fig.2 Hydrophilic and hydrophobic properties of catalase in M.meretrix
2.3 文蛤过氧化氢酶糖基化与磷酸化位点预测
通过NetOGlyc 4.0 Server与NetPhos 3.1 Server在线工具对该蛋白质进行糖基化和磷酸化位点预测。结果显示文蛤过氧化氢酶糖基化位点有51个,磷酸化位点有71个。其中位于丝氨酸(Ser)残基上的有23个,位于苏氨酸(Thr)残基上的有28个,位于酪氨酸(Tyr)残基上的有20个(见图3)。
2.4 文蛤过氧化氢酶跨膜区域与信号肽预测
运用TMHMM Server v 2.0软件预测文蛤过氧化氢酶跨膜区域,结果显示,该酶为膜外蛋白。通过SignalP 5.0 Server软件判断文蛤过氧化氢酶不存在信号肽。
2.5 文蛤过氧化氢酶亚细胞定位和保守结构域分析
通过PSORT Ⅱ Prediction在线软件对文蛤过氧化氢酶进行亚细胞定位。结果表明:文蛤过氧化氢酶有69.6%可能存在于过氧化氢酶体中,有21.7%存在于细胞核中,存在于线粒体和细胞骨架的可能性均为4.3%,说明文蛤过氧化氢极可能存在于过氧化物酶体中。用Blastp在线工具比对文蛤过氧化氢酶蛋白质序列,结果显示该蛋白质拥有血红素结合的保守结构域(Heme binding pocket)、NADPH结合位点(NADPH binding site)以及四聚体界面(Tetramer interface)三个结构功能域,属于典型单功能过氧化氢酶的第三分支(见图4),可能属于catalase like超家族中clade 3 分支的成员。
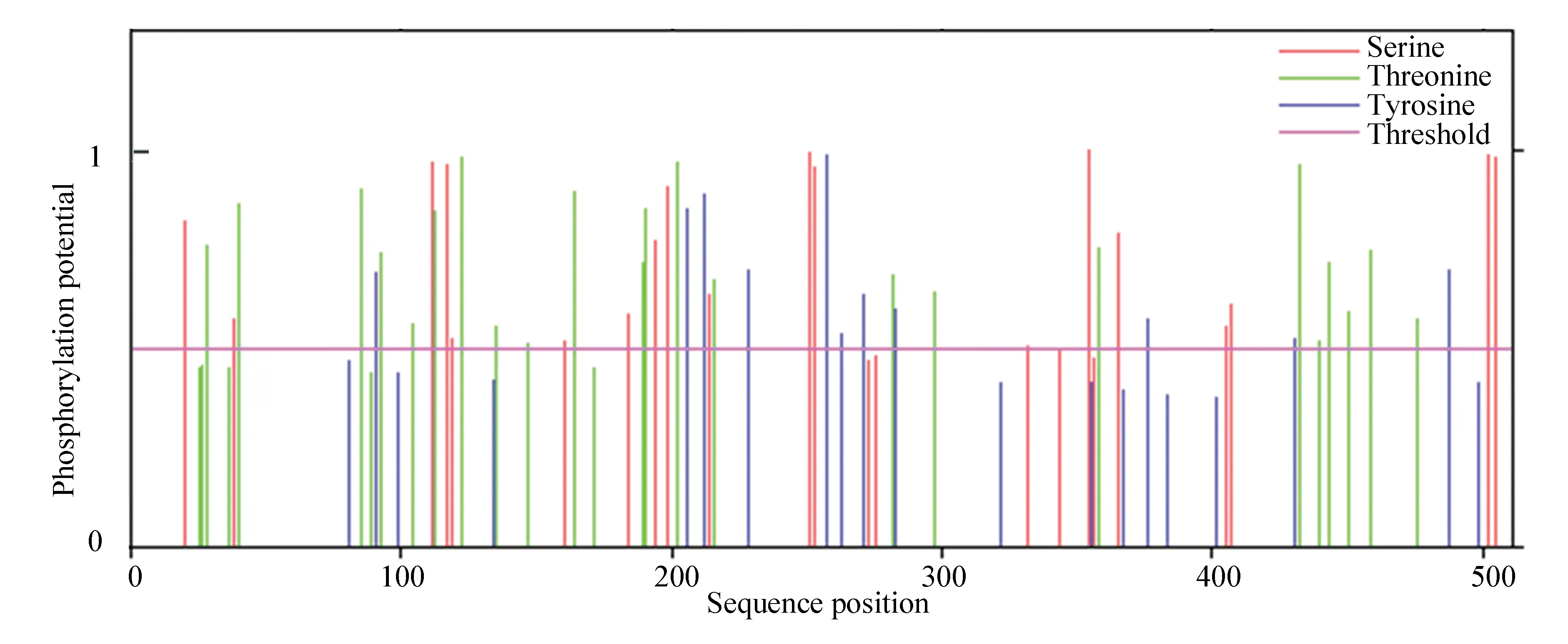
图3 文蛤过氧化氢酶磷酸化位点预测Fig.3 Prediction of catalase phosphorylation sites in M.meretrix

图4 文蛤过氧化氢酶保守结构域分析Fig.4 Conservative structure domain analysis of catalase in M.meretrix
2.6 文蛤过氧化氢酶二级结构和三级结构预测
使用SOPMA在线工具预测文蛤过氧化氢酶二级结构,结果表明无规卷曲(Random coil)和α-螺旋(Alpha helix)是其主要构成元件。其中无规卷曲由266个氨基酸构成,占总氨基酸的52.05%;α-螺旋由145个氨基酸构成,占总氨基酸的28.38%;78个氨基酸形成延伸链(Extended strand),占比为15.25%;β-转角(Beta turn)由22个氨基酸构成,占比最小(4.31%)。通过SWISS-MODEL在线工具预测得出文蛤过氧化氢酶三级结构(见图5),可见其扭曲和折叠较多,结构丰富,这对其生物学功能的发挥具有重要作用。
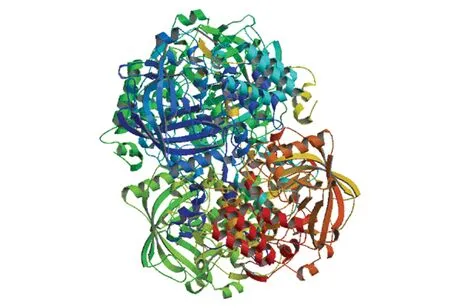
图5 文蛤过氧化氢酶三级结构Fig.5 Three-dimensional model of catalase in M.meretrix
2.7 文蛤过氧化氢酶蛋白质相互作用分析
运用STRING数据库(https://string-db.org/),设置为高置信度0.7,不限制数量,构建与CAT相互作用的蛋白网络(见图6)。结果发现:与CAT相互作用的蛋白质主要包括SOD1(Superoxide Dismutase 1 )、SOD2(Superoxide Dismutase 2 )、SOD3(Superoxide Dismutase 3 )、GSR(Glutathione Reductase)、AKT1(Protein Kinase B)、SCP2(Single Cell Protein 2)、HSD17B4(17β-Hydroxysteroid dehydrogenase Ⅳ)、ACOX1(Acyl Coenzyme A Oxidase 1)、HAO1(Hydroxyacid Oxidase 1)、DAO(Diamine Oxidase)。
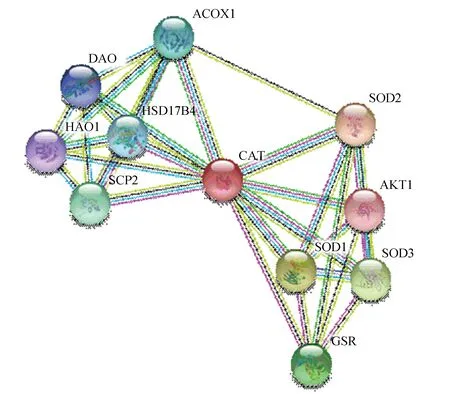
图6 CAT蛋白相互作用网络Fig.6 Protein-protein interaction network for CAT
对以上互作蛋白的基因进行GO分析,结果表明:有4个互作基因表达产物具有超氧化物歧化酶活性,有6个基因参与表达氧化还原酶,有3个基因参与过氧化物代谢过程,3个基因参与去除超氧自由基,3个基因表达产物作为蛋白质复合体,还有3个基因表达产物可能是磷蛋白质,还有3个基因表达产物可能存在于细胞质中(见图7)。KEGG分析表明:互作基因在过氧化物酶体的形成过程显著富集,当文蛤受氧化应激时,ACOX、SCPX、CAT等基因有上调趋势,PRDX1基因则有下调趋势(见图8)。
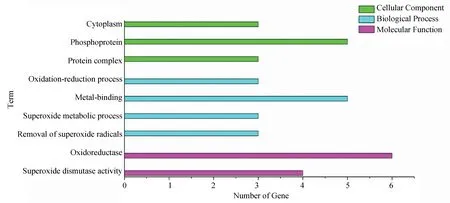
图7 GO注释结果Fig.7 Results of GO annotation
3 讨论与结论
利用生物信息学的方法,对文蛤过氧化氢酶蛋白结构进行分析,结果显示文蛤过氧化氢酶由511个氨基酸组成,相对分子质量为58 181.29 Da,为亲水蛋白,与栉孔扇贝(ChlamysFarreri)[13]、褶纹冠蚌(Cristariaplicata)[14]、盘鲍(Haliotisdiscusdiscus)[15]、仿刺参(Apostichopusjaponicus)[16]等过氧化氢酶的研究结果非常相似,这些酶的编码蛋白序列都在 500~520 个氨基酸之间,说明了该酶在物种进化上具有高度的保守性。文蛤过氧化氢酶的主要二级结构是无规卷曲,属于catalase like超家族。 根据GO分析、KEGG通路分析结果,结合亚细胞定位情况和结构分析结果,推测文蛤过氧化氢酶在文蛤细胞内主要参与过氧化物酶体的合成及抗氧化作用。根据文蛤过氧化氢酶互作蛋白的基因GO分析和KEGG通路分析结果可知,互作基因表达产物具有超氧化物歧化酶活性、参与去除超氧自由基、参与过氧化物代谢过程等生物过程。当文蛤受氧化应激时,CAT等基因显著上调(见图8),与苏恩萍[17]等文蛤过氧化氢酶活力研究结果一致,该结果说明文蛤过氧化氢酶的活力不仅受外界环境的影响,同时也受基因的调控。有研究表明SOD和CAT是机体防御过氧化损害系统中的两个关键酶,其能有效地消除活性氧,防止细胞膜系统过氧化作用的发生[3]。因此,CAT基因与互作蛋白中的SOD基因之间的调控关系值得深入研究。
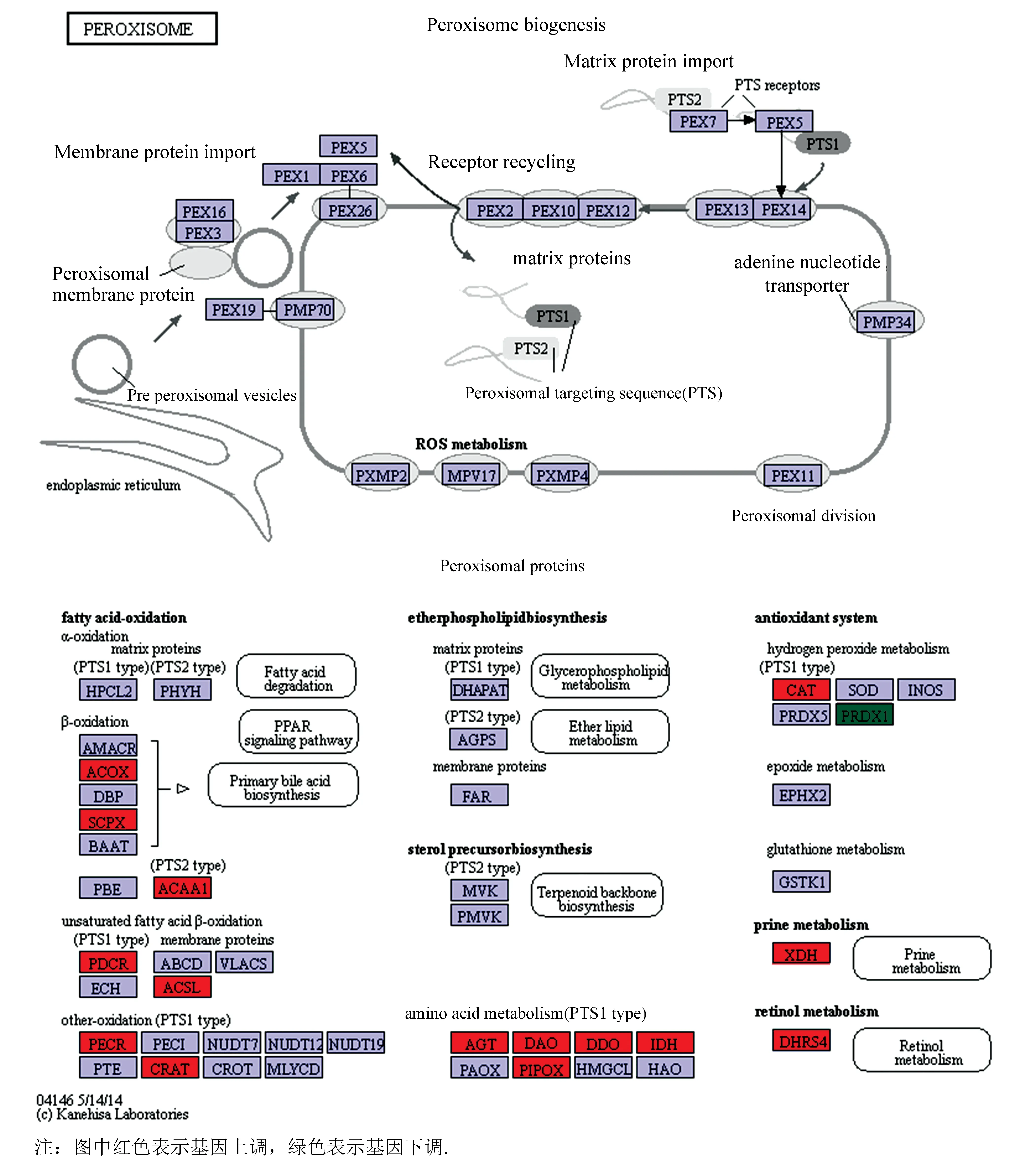
图8 互作基因信号通路Fig.8 Interacting gene signaling pathways
对文蛤过氧化氢酶理化性质、亲水性、磷酸化位点、糖基化位点、跨膜结构域、信号肽、亚细胞定位、二级结构、三级结构以及功能域等生物学信息进行了整理,为研究文蛤MmeCAT基因及其编码产物的功能提供了更多的信息。文蛤过氧化氢酶属稳定性蛋白,在生物进化上具有高度的保守性,其基因可以稳定遗传,且具有使机体免受氧化应激的生物学功能[18]。因此,在今后文蛤抗逆性品种选育上,可将文蛤过氧化氢酶含量作为目标性状之一。研究文蛤过氧化氢酶结构,有助于进一步研究文蛤的生物应激作用,同时为文蛤抗逆性品种选育提供理论基础。
