基于宏基因组技术分析自然发酵高粱菌群结构
2020-01-01葛云飞赵舒婷刘德志王维浩曹龙奎
葛云飞,赵舒婷,刘德志,王维浩,2,曹龙奎,2,*
(1.黑龙江八一农垦大学食品学院,黑龙江大庆163319;2.黑龙江八一农垦大学国家杂粮工程技术研究中心,黑龙江大庆163319)
高粱又名蜀黍,因其高产性、高抗逆性及其在发酵、制糖、饲料行业中用途广泛而被大量种植,其种植面积及产量仅次于小麦、玉米、水稻和大麦,成为世界上第五大粮食作物[1],是一种富含膳食纤维,蛋白质,脂肪,叶酸,铁和其他微量元素的天然高营养功能性食品。高粱具有凉血、解毒之功,常吃高粱粥和高粱米饭,可治积食、消化不良等症,可用于防治多种疾病[2]。在西方国家通常将高粱进行发酵生产“高粱粥”、“高粱发酵饮料”[3]及“高粱断奶粥”等产品[4],而在国内由于高粱的适口性较差,膳食纤维的含量较高而不易被人体消化吸收,通常将其应用于饲料行业中,使其经济、营养价值受到限制,发酵是我国常见改善谷物口感及理化性质的方法,因此通过将高粱进行自然发酵处理,不仅可以扩大高粱的应用范围,更能提高高粱的食用品质。目前国内外对发酵高粱研究主要集中在发酵高粱淀粉的理性化质、凝胶特性[5]及体外淀粉消化率[6]等,但对发酵高粱的微生物菌群结构变化涉略较少。发酵过程中微生物代谢产生的有机酸、淀粉酶类、醇类等小分子物质作用于高粱淀粉颗粒,改善高粱淀粉的理化性质,使高粱的营养价值提高,并赋予发酵高粱产品特殊风味改善其口感。
对于传统发酵过程中的微生物研究通常采用实验室分离培养,生理生化反应定向鉴定菌种,但由于实验室可培养的微生物种类仅占样品环境的1%~10%左右,因此通过实验室纯培养不能进一步分析传统发酵过程中的微生物群落构成、群落演替以及各种微生物的代谢特性[7],随着分子生物学的发展,发酵微生物菌落变化研究不在需要通过纯培养技术进行,大大减少了试验工作量并为不可培养微生物的研究提供契机。宏基因组学利用现代基因技术,涵盖生物信息统计分析和基因组学两方面的意义和技术[8],宏基因组学以基因组技术为基础通过对环境中全部DNA 系统全面的研究,以揭示发酵过程中微生物的菌落交替、相互作用关系及生理生化功能[9]。因此本文利用宏基因组技术研究自然发酵高粱过程中的菌群结构变化,代谢通路,为工厂化自发酵高粱产品调控提供理论基础与数据支持。
1 材料与方法
1.1 材料与试剂
高粱:山东临沂;DNA 提取试剂盒:上海科兴责任有限公司;Tris 硼酸、琼脂糖凝胶、核酸染料、TE 缓冲液和DNA Marker:青岛高科园海博生物技术有限公司;AxyPrep DNA 凝胶回收试剂盒:AXYGEN 公司;蒸馏水:国家杂粮技术研究中心重点实验室自制。
1.2 仪器与设备
Dgg-9053A 型电热鼓风干燥箱:上海森信实验仪器有限公司;MJ-10A 型磨粉机:上海市浦恒信息科技有限公司;PTC-200 型梯度扩增PCR 仪、CDS-8000 型凝胶成像分析系统:美国Bio-Rad 公司;GS-FLX 型高通量测序仪:美国Roche454 公司;DYY-12 型电泳仪:北京六一仪器厂;Qubit2.0 荧光计:沃德生物医学仪器公司;LS-3781L-PC 型高压灭菌锅:日本松下健康医疗器械株式会社。
1.3 试验方法
1.3.1 样品处理
将高粱进行自然发酵0~15 d,取发酵前期(3 d)、发酵中期(8 d)、发酵后期(14 d),装入无菌离心管中,送去宏基因组高通量测序。
1.3.2 样品总DNA 的提取
根据DNA 试剂盒说明书所示标准步骤,进行提取,具体操作步骤如下:
1)称取发酵液样品1 g 于2 mL 离心管中,加入0.8 mL SLX Mlus 缓冲溶液,振荡混匀5 min。
2)加入80 μL DS 缓冲液并且振荡混匀。
3)恒温金属浴70 ℃裂解10 min。
4)25 ℃室温下离心5 min(13 000 r/min)。
5)吸取离心后上清液600 μL 于2 mL 无菌离心管中,加入200 μL SP2 缓冲液,振荡混匀。
6)加入100 μL HTR 试剂,混匀10 s 后进行冰浴5 min。
7)25 ℃室温离心5 min(13 000 r/min)。
8)吸取离心后上清液400 μL 于2 mL 无菌离心管中,加入450 μL Binding 缓冲液和40 μLMagsi Particles溶液,振荡混匀,25 ℃下静置2 min。
9)将2 mL 离心管放置在磁力架上吸附5 min,小心吸弃上层清液,并且移开。
10)加入500 μL Binding 缓冲液,振荡混匀,25 ℃室温放置2 min。
11)将2 mL 离心管放置在磁力架上吸附5 min,小心吸弃上层清液,并且移开。
12)加入1 000 μL PHB 缓冲液,振荡混匀磁珠,放置在磁力架5 min,弃上清。
13)加入1 000 μL SPM Wash 缓冲液,混匀磁珠,放置在磁力架5 min,弃上清。
14)重复步骤(13)即加入1 000 μL SPM 缓冲液,混匀磁珠,放置在磁力架5 min,弃上清。
15)将2 mL 离心管于55 ℃烘箱中烘10 min,使残留酒精完全挥发。
16)加入60 μL Elution 缓冲液到离心管中,充分震荡混匀,65 ℃金属浴10 min。
17)磁力架吸附5 min,小心吸取上清DNA 液体到新的1.5 mL 离心管中。
1.3.3 样品DNA 测序文库构建
1.3.3.1 样品基因组DNA 片段化
将不同发酵时期发酵液样品进行DNA 片段化,制备插入片段长度为500 bp 左右的文库,初始DNA 总量为800 ng,使用Elution 缓冲液将DNA 稀释到130 uL,装入0.5 mL 的DNA 打断管中,将DNA 片段化,即使最后打断的DNA 片段大小集中在300 bp~500 bp 范围内。使用核酸纯化磁珠将破碎后的DNA 片段浓缩回收。
1.3.3.2 样品基因组DNA 浓度测定
1.3.3.3 样品基因组DNA 片段末端修复及连接接头
通过Illumina 公司试剂盒进行建库,其具体操作步骤如下:
1)将超端准备酶和末端修复反应缓冲液置于冰上解冻。
2)分别加入(绿色)端预酶混合物试剂3.0 μL、(绿色)末端修复反应缓冲试剂6.5 μL、片段化DNA 试剂55.5 μL 于不同发酵时期样品片段化后DNA 中。
3)使用移液器轻轻吹打或振荡混匀,并短暂离心将反应液离心至管底。
4)将上述聚合酶链式反应(polymerase chain reaction,PCR)管置于PCR 仪,热盖设置为≥75 ℃,并运行以下程序:20 ℃保持30 min、65 ℃保持30 min。
5)将DNA 连接酶,连接增强剂和转接酶置于冰上解冻。
6)分别加入(红色)DNA 连接酶试剂15 μL、(红色)连接增强剂试剂1 μL、转接酶试剂2.5 μL。
7)使用移液器轻轻吹打或振荡混匀,并短暂离心将反应液离心至管底。
8)将上述PCR 管置于PCR 仪,并运行以下程序:20 ℃保持15 min。
1.3.3.4 纯化后的接头连接产物进行PCR 扩增富集
1)涡旋振荡混匀核酸纯化磁珠。
2)根据DNA 片段长度要求,向100 μL DNA 上清液中分两次加入磁珠,涡旋混匀或移液器吹打10 次混匀,25 ℃孵育5 min。
3)将PCR 管短暂离心并置于磁力架中,待溶液澄清后(约5 min),小心移除上清,加入200 μL 新鲜配制的80%乙醇漂洗磁珠两次。
4)将PCR 管从磁力架中取出,加入适量21 μL 双蒸水,涡旋振荡充分混匀,25 ℃静置5 min。
5)将PCR 管短暂离心并置于磁力架中分离磁珠和液体,将20 μL 上清液转移至干净无菌管中。
6)向无菌管中加入DNA 片段连接酶试剂20 ng、(蓝色)热启动聚合酶链反应母液试剂20 μL、Index Primer/i7 Primer(50 μmol/L)和Universal PCR Primer/i5 Primer(50 μmol/L)试剂各1 μL。
7)使用移液器轻轻吹打或振荡混匀,并短暂离心将反应液收集至管底后将PCR 管置于PCR 仪中进行扩增。
8)所得PCR 产物用2 g/100 mL 的琼脂糖凝胶电泳检测。
1.3.4 数据分析
通过不同软件对微生物多样性、基因组装及基因功能注释技术路线将样品测序得到的原始序列经过数据质控之后,进行组装、注释公共数据库等分析。
2 结果与分析
2.1 发酵样品总DNA 的提取
发酵样品总DNA 琼脂糖凝胶电泳检测结果如图1 所示。
由图1 可知发酵样本主条带清晰,无明显拖尾且浓度较高,说明发酵样品不存在降解,污染,3 个样品对应位置的条带大小正确,浓度合适,为后续试验的准确性提供了保障,由此可证明扩增片段可用于Illumina双末端测序。
发酵样品的DNA 浓度检测结果如表1 所示。
发酵样品总DNA 的浓度均达到了测序标准,因此可以构建二代测序文库,可以进行下一步试验。
2.2 发酵样品Illumina 测序数据处理
发酵样品的高通量测序检测结果如表2 所示。

图1 发酵样品总DNA 的提取Fig.1 Extraction of total DNA from fermentation samples

表1 发酵样品总DNA 的浓度Table 1 Total DNA concentration of fermentation sample
不同发酵时期所得序列数分别为4.7×107、7.9×107、4.5×107,为了保证信息分析质量,将测序得到的原始数据进行过滤,得到清晰数据,将所得优质reads 利用拼接软件IDBA_UD 进行拼接组装,根据序列间的重叠关系,获得一定数目的重叠数,并对多个组装结果进行了综合评定,选择最佳组装结果[10]。对拼接的拼接重叠数序列进行开放阅读框(open reading frame,ORF)预测,选择长度大于等于100 bp 的基因,并将其翻译成蛋白序列以便后续物种注释及京都基因百科全书与基因组(Kyoto Encyclopedia of Genes and Genomes,KEGG)功能注释进行研究分析。

表2 高通量测序结果Table 2 High throughput sequencing results
基因在不同样本间会出现基因个数及丰富度有差异的现象,香农维纳(Shannon-Wiener)指数与辛普森(Simposon)指数被用来表征微生物基因多样性。香农维纳指数为物种种类数目,即丰富度又称为种类中个体分配上的平均性,样本种类数目多,其样本的多样性越高。同样,种类之间个体分配的均匀性增加也会是多样性的提高。如果每一个个体都属于不同的种,多样性指数就最大;如果每个个体都属于同一种,则多样性指数就最小。而辛普森指数为随机取样的两个个体属于不同种的概率,群落中种属越多,各种个体分配越均匀,指数越高,表明多样性越好。由表2 可知在发酵不同时期其香浓维纳指数呈先增加后降低的趋势,而辛普森指数为先降低后增加,说明随着发酵时间的延长,发酵液中的微生物群落中种属类别呈先增加后降低的趋势,在发酵中期微生物种属较多、菌属的丰富度较大即发酵样本多样性较高。
2.3 发酵样品菌群结构及菌种比例分析
将基因集蛋白序列与数据库进行同源性比对,得到功能注释和同源物种信息。同时根据美国国家生物技术信息中心(National Center of Biotechnology Information,NCBI)的微生物分类学信息数据库,获得基因的物种分类注释信息,其中高粱在不同发酵时期的微生物菌群在物种分类学水平上的相对丰度如图2所示。

图2 自然发酵过程中菌群结构变化Fig.2 Changes in bacterial structure during natural fermentation
在高粱自然发酵前期过程中,丰富度大于1%的菌群结构如下:乳酸乳球菌的丰富度较高,达到26.54%、乳杆菌属含量约为22.68 %、其次是肠杆菌属含量1.59%、固氮菌株丰富度为5.27%,随着发酵时间的延长当发酵时间为8 d 即发酵中期时酵母菌的丰富度高达36.47%、植物乳杆菌及戊糖片球菌为发酵中期乳酸菌的主要菌种,其中植物乳杆菌的丰富度较发酵前期高出7.26%,肠杆菌属丰富度上升至2.65%、而在发酵后期即自然发酵14 d 时葡糖醋杆菌属丰富度最高,其中Gluconacetobacter diazotrophicus(固氮葡糖醋杆菌)菌种含量为45.54%,醋杆菌属的丰富度随发酵时间的延长而增加至11.38%,此时发酵液产生较大的刺激性酸味、酸度增加,说明高粱在自然发酵过程中优势菌为乳酸菌及酵母菌,Michael G 等[11]通过将高粱进行发酵生产酸面团研究发现其中优势菌种为罗伊氏乳杆菌,植物乳杆菌和发酵乳杆菌及干酪乳杆菌,棒状乳杆菌等,而Sedjro Emile Tokpohozin 等[12]通过基质辅助激光解吸离子飞行质谱获得的蛋白质组分析高粱啤酒中的优势菌为发酵乳杆菌,植物乳杆菌,瑞士乳杆菌,副干酪乳杆菌和短乳杆菌等,Jialiang Xu 等[13]通过宏基因组分析高粱米酒中优势菌属为寡养单胞菌属,蛭弧菌属,溶藻属,硫磺菌属和无色杆菌等,本文通过将高粱进行自然发酵研究发现其发酵过程中的优势菌种为胚芽乳杆菌(Lactobacillus plantarum)、乳酸乳球菌(Lactococcus lactis)、费比恩毕赤酵母(Cyberlindera fabianii)、醋酸杆菌(Acetobacter malorum)、固氮葡糖醋杆菌(Gluconacetobacter diazotrophicus)等,说明在高粱自然发酵的过程中酵母菌是慢慢富集的过程,在发酵过程之中乳酸菌的大量存在增加了酵母菌的代谢潜能[14],而在发酵后期醋杆菌属的大量富集使酵母菌丰富度下降,说明毕赤酵母等酵母菌耐酸性较差。
2.4 发酵样品菌群主要代谢通路分析
发酵样品中主要代谢通路及主成分分析结果如表3 所示。
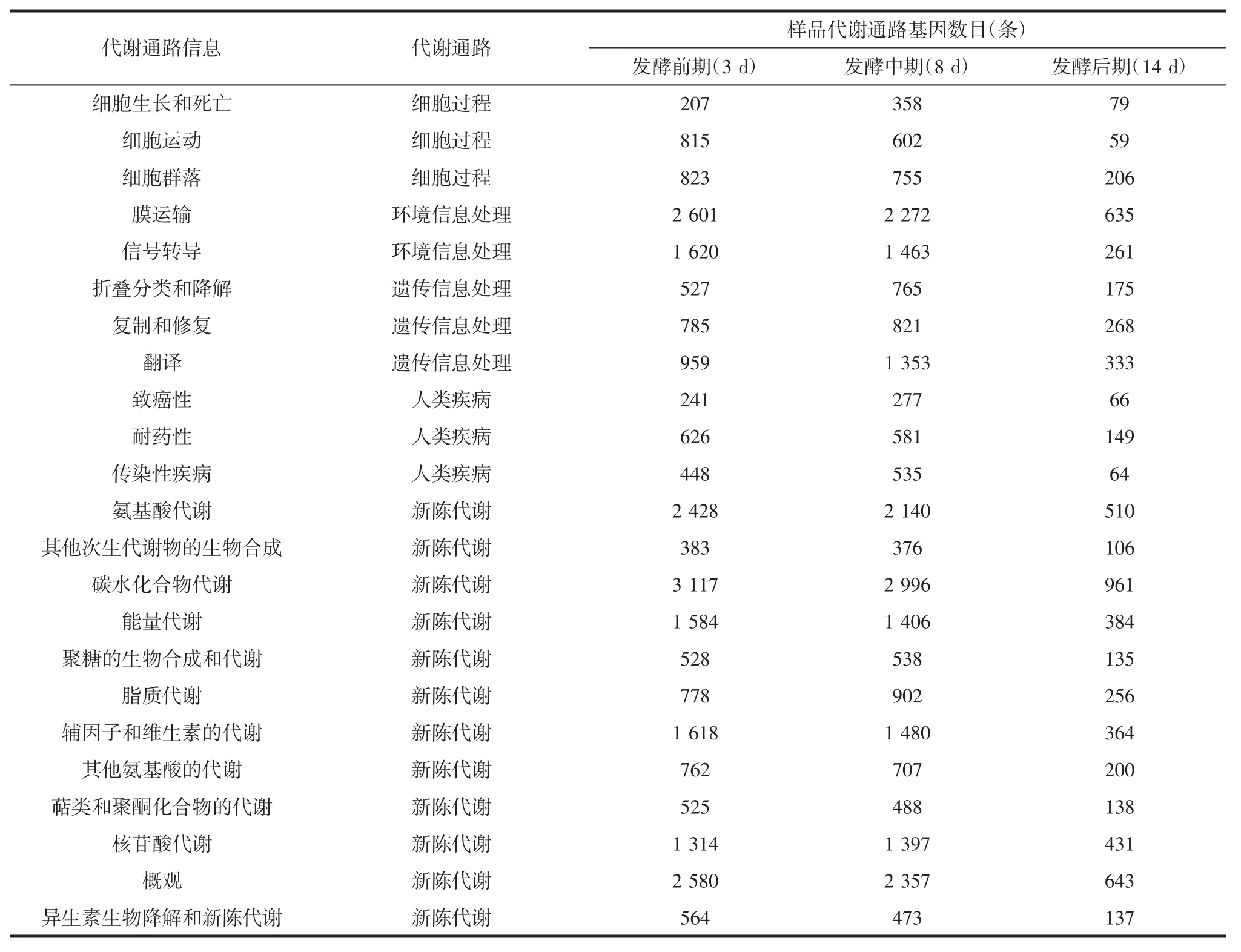
表3 发酵样品中主要代谢通路及主成分分析Table 3 Main metabolic pathways in fermentation samples
使用GhostKOALA[15]将基因集蛋白序列与KEGG数据库进行比对,得到序列对应的基因数据库(KEGG orthology,KO)基因号,根据KO 与通路和模块的联系得到序列的通路,模块注释信息,包括代谢通路、合成通路、膜转运、信号传递、细胞周期以及疾病相关通路等,并统计KEGG 各功能层级在各个样本中的丰富度。其结果如下:
在高粱自然发酵的过程中,微生物菌群的代谢结构发生变化,随着发酵时间的延长,氨基酸代谢、碳水化合物代谢等基因数目较大,而发酵后期基因数目有一定程度的降低,通过宏基因组序列的代谢重建,揭示了蛋白质和碳水化合物异养发酵的特征,与检测到乙醇和pH 值下降相符即发酵随着发酵时间的延长醋酸菌属的丰富度增加,使整个发酵体系的酸度增加pH值下降,使微生物生长的抑制效应逐渐增强,其中乳酸菌和醋酸菌自身具有一定的醋酸耐受力,逐渐成为丰度较高的主体微生物,而通过代谢途径可知,碳水化合物代谢主要途径为三羧酸循环、丙酮酸代谢、磷酸戊糖和糖酵解途径等[16],其中间代谢产物所产生大量的柠檬酸、乙酸、丁酸等有机酸亦使溶液的pH 值逐渐降低。因此,除加工技术(麦芽化,糖化),高粱麦芽汁组成之外,发酵产生的代谢产物(有机酸,醇)和pH值等因素决定了发酵剂微生物菌群结构。
3 结论
通过红宏基因组技术分析高粱在自然发酵过程中菌群演替变化及优势菌群的代谢途径和特定功能的基因数目,结果发现在发酵过程中优势菌种为胚芽乳杆菌(Lactobacillus plantarum)、乳酸乳球菌(Lactococcus lactis)、费比恩毕赤酵母(Cyberlindera fabianii)、醋酸杆菌(Acetobacter malorum)、固氮葡糖醋杆菌(Gluconacetobacter diazotrophicus)等,其中酵母菌的丰富度随着发酵时间的延长而增加,而发酵后期醋杆菌属的大量富集使整个发酵体系显酸性,说明发酵过程中的优势菌种为耐酸性菌种,其中发酵体系中醋酸菌的丰度较高,由于其具有较强的氧化能力,能够较快地将乙醇氧化为醋酸[17],使发酵液pH 值降低。通过功能注释聚类分析结果得知,高粱自然发酵过程中主要功能信息包括微生物代谢通路、合成通路、膜转运、信号传递、细胞周期以及疾病相关通路等,其中间代谢产物有机酸等为微生物的繁殖生长提供了有利条件。本试验通过高通量测序的宏基因学分析高粱发酵过程中的微生物菌群结构变化,进一步明确发酵高粱中微生物的群落结构演替、相互作用,同步确定各种微生物的丰度,通过了解高粱自然发酵过程中的代谢机理,并挖掘出新的功能基因,对高粱自然发酵的工厂化生产及品质调控提供理论基础与数据支持。
