高粱苗期耐盐性转录组分析和基因挖掘
2019-12-19董明再吐尼古丽库尔班吕芃杜瑞恒叶凯侯升林刘国庆
董明,再吐尼古丽·库尔班,吕芃,杜瑞恒,叶凯,侯升林,刘国庆
高粱苗期耐盐性转录组分析和基因挖掘
董明1,再吐尼古丽·库尔班2,吕芃1,杜瑞恒1,叶凯2,侯升林1,刘国庆1
(1河北省农林科学院谷子研究所/河北省杂粮重点实验室,石家庄 050035;2新疆农业科学院生物质能源研究所,乌鲁木齐 830091)
【】探究高粱耐盐胁迫响应机制,挖掘高粱耐盐胁迫基因,为高粱耐盐育种提供理论基础。以高粱感盐品种L甜和耐盐品种石红137为供试材料,采用水培试验。待高粱植株长至三叶一心期,使用2%NaCl溶液对幼苗进行盐胁迫,分别设置0(对照)、1和24 h处理,每个处理3次重复。测定不同处理样品株高、根长、干物重、Na+含量和叶绿素相对含量(SPAD值),并依托Illumina HiSeq 2000平台进行转录组测序分析。利用FPKM方法计算基因表达量,在差异表达基因检测过程中,将差异表达倍数(fold change)≥2且FDR<0.001作为筛选标准。通过Gene Ontology和KEGG Pathway数据库对参与高粱不同时间盐胁迫差异表达基因进行分析注释。盐胁迫处理对高粱株高、根长、干物重等性状无显著影响,对钠离子含量和SPAD值影响显著。石红137株高、根长、钠离子含量和SPAD值均高于L甜。转录组测序结果鉴定得到已知基因26 628个,新基因866个。石红137中的差异基因数目高于L甜。石红137中,0 h vs 1 h、0 h vs 24 h、1 h vs 24 h三组的差异基因数目分别为375、4 206和3 750个。感盐品种L甜中,0 h vs 1 h、0 h vs 24 h、1 h vs 24 h三组的差异基因数目分别为167、2 534和1 612个。GO分析共获得25个功能注释,分别为光合作用、细胞物质代谢、翻译过程以及激素合成等与盐胁迫相关的差异表达基因。KEGG分析发现盐胁迫1 h表达差异基因富集在植物激素信号转导途径,涉及脱落酸(abscisic acid,ABA)、生长素(auxin,AUX)、细胞分裂素(cytokinin,CTK)、赤霉素(gibberellins,GS)、乙烯(ethylene,ETH)过程等共71个基因。盐胁迫24 h表达差异基因富集于光合作用相关途径,涉及Lhca、Lhcb、磷酸烯醇式丙酮酸羧化酶(phosphoenolpyruvate carboxylase,PPC)、磷酸核酮糖激酶(phosphorylribonucleic kinase,PRK)等20个基因。类黄酮生物合成代谢途径差异可能是引起石红137和L甜的耐盐能力差异的原因之一,花青素还原酶(anthocyanidin reductase,ANR)和黄酮醇合成酶(flavonol synthase,FLS)参与类黄酮生物合成途径。高粱的盐胁迫过程是一个复杂的生物过程,依赖于多个基因在复杂网络中的平衡表达。盐胁迫条件下,高粱应对环境刺激受到激素信号转导和光合作用的控制。类黄酮生物合成途径在耐盐品种中起到了重要作用。
高粱;盐胁迫;转录组;基因挖掘
0 引言
【研究意义】盐胁迫是一类重要的非生物胁迫,严重制约了农业的可持续发展[1]。盐胁迫下,植物的光合作用、呼吸速率以及物质代谢受到严重危害,最终导致产量的降低。高粱作为一种重要的粮食兼经济作物,具有较强的抗旱、耐盐碱能力[2],但不同品种间耐盐性存在较大差异。高粱耐盐基因的鉴定与挖掘能为探讨高粱耐盐的分子机理、培育耐盐品种提供坚实基础。【前人研究进展】目前,全球范围内约有10亿hm2的土地存在不同程度的盐渍化[3]。盐胁迫对高粱的生长发育有很大影响,根据田间土壤盐分运行规律,作物在萌发和幼苗生长期间受盐胁迫的危害尤为严重。盐胁迫除了使植物受到离子胁迫之外,同时使植物受到水胁迫和低氧胁迫等。在盐渍土上种植耐盐品种是减轻土壤渍化危害的有效方法。早期大量研究表明盐胁迫条件下,盐害主要通过改变土壤溶液渗透势和离子浓度影响植物根系对矿质元素的吸收[4],从而对植株地上部分产生影响,包括植株的形态发育、水分平衡、质膜透性、光合作用、呼吸作用以及物质代谢等途径[5-8]。植物对抗盐胁迫是一个复杂的过程,涉及多个与发育和生理相关的途径[9]。耐盐植物的耐盐性主要体现在植株体内的离子平衡,Na+和Cl-被贮藏在液泡内,保持细胞渗透势的稳定[10]。部分有机溶质,例如蛋白质、氨基酸等物质也起到稳定细胞渗透势的作用。目前,在水稻中鉴定出对盐胁迫反应的基因有280个,耐盐相关QTL有332个,遍布水稻的整个基因组,耐盐相关miRNA有29个[11]。RNA-Seq技术可以筛选耐盐与盐敏感植株的差异表达基因,鉴定植株响应盐胁迫应答基因及表达特性,以期更好地理解植物对盐胁迫响应的分子机制,为进一步鉴定和克隆重要的耐盐基因,提高植物耐盐性状奠定基础。利用该技术对玉米[12]、小麦[13]和高粱[14-16]等耐盐影响因子和机理进行了初步探讨,对作物耐盐调控网络的解析起到了促进作用。王海莲等[17]利用石红137和L甜杂交衍生181个重组自交系,发现长时间低盐胁迫会抑制高粱幼苗生长。在盐胁迫下控制高粱苗高、苗鲜重和苗干重QTL的表达具有较强的环境特异性,而控制高粱苗高的qSH1-1和qSH7-2在高粱耐盐遗传改良中将发挥重要作用。同时研究发现6个主要QTL和5个染色体区域在高粱耐盐过程中起到关键作用[18]。【本研究切入点】目前,对高等植物耐盐性分子机制的研究主要集中在拟南芥和水稻,在高粱中报道较少,且主要集中在个别转录因子的调控分析或赤霉素和多效唑对高粱耐盐的影响等,对于高粱苗期耐盐的转录组测序分析较少。【拟解决的关键问题】本研究对高粱耐盐品种和感盐品种进行盐胁迫处理,通过转录组测序研究高粱对抗盐胁迫的分子机制,同时比较感盐和耐盐品种间响应盐胁迫的差异,为探究高粱耐盐胁迫机制提供丰富理论途径,并为高粱耐盐育种奠定坚实基础。
1 材料与方法
1.1 试验材料
根据Wang等[18]研究结果选用感盐品种L甜和耐盐品种石红137为供试材料。试验材料选取水培方式种植,种子消毒后摆放在培养盒中,在28℃长日照(16 h/8 h)条件下培养。待长至一叶一心,用Hoagland培养液代替水进行培养。在三叶一心期利用2% NaCl溶液进行盐胁迫处理,处理时间分别为0(对照)、1和24 h,每个处理3次重复。处理结束之后将叶片迅速剪下放入液氮中冷冻,样品放于-80℃贮藏。干样取下之后放入105℃烘箱10 min杀青,之后80℃烘干至恒重备用。
1.2 钠离子含量测定
样品钠含量采用HNO3-H2O2方法消煮。称取样品0.2 g至消煮管中,加入HNO3-H2O2(4﹕1)混合液5 mL,静置12 h以上,用江苏宜兴的LNK-872型多功能快速消化器消煮至溶液蒸干。冷却至室温,加入5% HNO3溶液8 mL,70℃封口水浴2—3 h,涡旋、静置,至溶液澄清后转移至10 mL离心管,用ICP-OES(PerkinElmer OPTIMA 210DV)对样品进行测定。
1.3 叶绿素相对含量测定
高粱叶片叶绿素相对含量通过日本产SPAD-502型叶绿素仪进行测定。
1.4 cDNA文库的构建及转录组测序
样品RNA由百迈客生物科技有限公司制备。RNA样品通过质量检测进入Illumina HiSeq 2000平台进行转录组测序。下机所得原始数据(raw data)经过过滤得到纯净数据(clean data),纯净数据再与指定参考基因组(Sbicolor_v2.1)比较得到比对数据(mapped data)。
1.5 差异基因的筛选
将样品处理两两比较得到差异基因。在差异表达基因检测过程中,将Fold Change≥2且FDR<0.001作为筛选标准。差异倍数(fold change)表示两样品(组)间表达量的比值。错误发现率(false discovery rate,FDR)是通过对差异显著性值(-value)进行校正得到。采用了公认的Benjamini-Hochberg校正方法对原有假设检验得到的显著性值(-value)进行校正,并最终采用FDR作为差异表达基因筛选的关键指标。
1.6 差异基因的注释和分类
将比较得到的差异基因依次和Nr(NCBI non-redundant protein,NCBI非冗余蛋白)数据库、Swiss-Prot(Swiss-Prot proteinsequence,瑞士蛋白序列)数据库、KEGG(Kyoto encyclopedia of genes and genomes,京都基因与基因组百科全书)数据库、COG(clusters of orthologousgroups of proteins,蛋白直系同源聚类)数据库和GO(gene ontology,基因本体论数据库)数据库中的蛋白序列进行比对,从而获得与差异基因对应的蛋白功能注释及功能分类统计。
1.7 实时荧光定量PCR分析(qRT-PCR)
为了验证高粱耐盐胁迫转录组结果的准确性,从差异基因数据库中随机挑选4个基因,利用Primer 6.0设计引物(表1),分析实时荧光定量PCR结果是否与转录组结果一致。
2 结果
2.1 盐胁迫对高粱的影响
盐胁迫处理能够影响高粱叶片的钠离子含量和SPAD值(图1)。对株高、根长、干物重等性状无显著影响,石红137株高和根长高于L甜(表2)。盐胁迫处理1 h,植株表型未出现明显变化。盐胁迫处理24 h时,植株样品出现明显萎蔫(图2)。与对照相比,2个品种的钠离子含量均在盐胁迫处理24 h达到最高,且差异显著。SPAD值在盐胁迫处理24 h显著降低。石红137在2个时期盐胁迫处理的钠离子含量和SPAD值均高于L甜。

表1 实时荧光定量PCR引物
2.2 转录组测序结果评估
取2个高粱品种盐胁迫0、1和24 h叶片样品,分别提取各个样品RNA,进行质检、构建文库之后,利用Illumina HiSeq平台进行测序。6个处理18个样本高通量测序得到序列标签数在42 082 348—56 174 902,比对到参考基因组的序列标签数在33 795 395—44 513 494,各样品的序列标签与参考基因组的比对效率在76.27%—79.74%。经过测序质量控制,共得到130.80 Gb处理后的数据量,各样品Q30碱基百分比均不小于92.40%(表3)。将所有的处理后序列标签组装并与参考基因组进行比对,鉴定出已知基因26 628个,新基因866个。

不同小写字母表示处理间差异达5%显著水平 Different lowercase letters indicate a significant difference at a 5% level between different treatments

A:盐胁迫0 h;B:石红137盐胁迫处理 A: 0 h salt stress of Shihong137 and L tian; B: Salt stress treatment and control treatment of Shihong137

表2 盐胁迫不同时间农艺性状统计
g:感盐品种L甜;n:耐盐品种石红137;gCK:L甜对照处理;g1h:L甜盐胁迫1h处理;g24h:盐胁迫24 h处理;nCK:石红137对照处理;n1h:石红137盐胁迫1 h处理;n24h:石红137盐胁迫24 h处理。下同
g: Salt sensitive variety L tian; n: Salt tolerant variety Shihong 137; gCK: L tian control; g1h: L tian salt stress for 1 h; g24h: L tian salt stress for 24 h; nCK: Shihong 137 control; n1h: Shihong 137 salt stress for 1 h; n24h: Shihong 137 salt stress for 24 h. The same as below
2.3 盐胁迫不同时期差异基因筛选
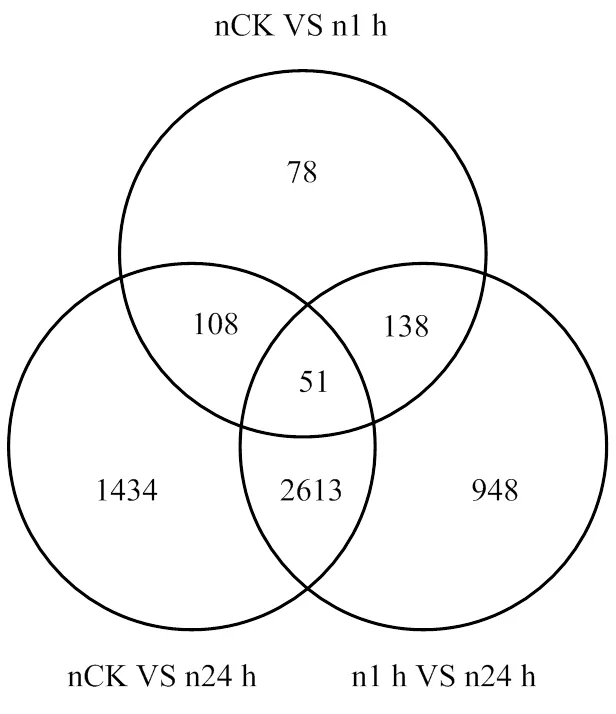
不同处理两两比较,得到差异基因数目(DEGs)(表4)。耐盐品种石红137中,0 h vs 1 h、0 h vs 24 h和1 h vs 24 h的差异基因数目分别为375、4 206和3 750个。感盐品种L甜中,0 h vs 1 h、0 h vs 24 h和1 h vs 24 h的差异基因数目分别为167、2 534和1 612个。相同处理时间2个品种之间比较,gCK vs nCK、g1h vs n1h和g24h vs n24h的差异基因数目分别为1 240、1 184和1 910。石红137在盐胁迫后的差异基因数目明显高于L甜,盐胁迫后1 h高出124.55%,24 h高出65.98%。2品种都是在盐胁迫24 h后DEG数目迅速升高,达到最高值。盐胁迫24 h DEG数目高于1 h DEG数目,石红137增加数量较多,增幅1000.00%,而L甜增幅较小,增加965.27%。

表3 高粱叶片转录组测序统计

表4 盐胁迫不同时期差异基因数目
在3组比较中,L甜有13个差异基因共表达,石红137有51个差异基因共表达。有727个基因在2个品种中共表达。不同品种之间的差异基因表达数目比单个品种中不同处理比较的差异基因表达数目多(图3)。
2.4 与盐胁迫相关差异基因
根据GO功能注释,nCK vs n1h和gCK vs g1h富集差异最为显著的前10个生物过程可分为3类,且仅有一个不同(表5)。分别为:1)与激素相关:生长激素的响应、吲哚乙酸生物合成;2)与细胞物质代谢相关:谷胱甘肽代谢过程、色氨酸分解代谢过程、脯氨酸转运、甲硫氨酸生物合成硫代葡萄糖苷过程;3)对胁迫的直接响应:对缺水反应的负调节、细胞对磷缺乏的反应、细胞高渗盐度反应(nCK vs n1h)/脱黄化(gCK vs g1h)。

表5 高粱叶片中盐胁迫差异基因的GO富集分析
g:感盐品种L甜;n:耐盐品种石红137;gCK:L甜对照处理;g1h:L甜盐胁迫1 h处理;g24h:盐胁迫24 h处理;nCK:石红137对照处理;n1h:石红137盐胁迫1 h处理;n24h:石红137盐胁迫24 h处理。下同
nCK vs n24h和gCK vs g24h富集差异最为显著的前10个生物过程有4个过程不同。nCK vs n24h中GO富集差异最显著的前十个生物过程分为:1)与光合作用相关过程:光系统Ⅱ组件、类囊体膜组织、光系统Ⅰ中的光合电子运输、对红光的反应;2)与细胞物质代谢相关的生物过程:麦芽糖代谢过程、淀粉生物合成、半胱氨酸生物代谢过程、色氨酸分解代谢过程;3)与翻译相关生物过程:rRNA过程;4)与激素相关的生物过程:对karrikin的响应。gCK vs g24h富集差异最为显著的前10个生物分为:1)与光合作用相关过程:光系统Ⅱ组件、对红光的响应、光系统Ⅰ中的光合电子运输、类囊体膜组织;2)与细胞物质代谢相关的生物过程:戊糖磷酸支路、异戊烯二磷酸生物合成,4-甲基季戊四醇途径、麦芽糖代谢过程、淀粉生物合成;3)与激素相关生物过程:对karrikin的响应、生长激素的响应。
KEGG富集分析表明,gCK vs g1h和nCK vs n1h差异基因主要富集在植物激素信号传导途径。gCK vs g24h和nCK vs n24h差异基因的前五个富集通路中有4个相同:分别为光合器官中的碳固定、碳代谢、氨基酸生物合成和光合作用-天线蛋白。另外一个不同通路在gCK vs g24h和nCK vs n24h分别为丙酮酸代谢和光合作用。g1h vs g24h和n1h vs n24h差异基因的前五个富集通路有4个通路相同,且与gCK vs g24h和nCK vs n24h一致。两类蛋白与高粱叶片盐胁迫相关:盐胁迫早期与植物激素信号相关、盐胁迫后期主要涉及到光合作用。2个品种的同一时期比较发现CK之间没有显著的富集通路,而比较盐胁迫1 h处理和24 h处理有共同的富集通路类黄酮代谢。
2.5 植物激素信号转导途径
植物激素不仅能够调控植株的生长发育,还参与的植物的非生物胁迫。植物激素信号转导途径包含了生长素(IAA)、脱落酸(ABA)、细胞分裂素(CTK)、乙烯(ETH)和赤霉素(GS)(图4),分别鉴定出基因22、24、11、9和5个。IAA途径包括AUX、ARF、GH3和SAUR 4个基因家族,ABA途径包括:ABF、SnRK2、PP2C和PYR/PYL 4个基因家族。SAUR和ABF同源基因全部表达上调,其他同源基因在不同处理中表达模式不同。
2.6 光合作用相关途径
Lhca、Lhcb、PPC和PRK等20个光合作用相关途径基因参与耐盐胁迫过程(表4)。PPC(磷酸烯醇式丙酮酸羧化酶)是参与植物碳固定的关键酶,参与光合作用的暗反应。Sobic.002G167000在2个品种中均表达上调,分别上调3.80和5.19倍。Sobic.010G160700在2个品种中均下调表达。Sobic.004G106900和Sobic.007G106500的表达模式在2个品种中表现不同。LHC是一类能够捕获光能并能将能量迅速传至反应中心引起光化学反应的色素蛋白[19],分为2个亚类:Lhca和Lhcb,2个品种中Lhca和Lhcb的同源基因均表达下调。PRK(磷酸核酮糖激酶)参与碳固定和碳代谢2个途径,PRK 2个同源基因在2个品种中均表达下调(表6),下调基因在2个品种中分别达到1.24—128.71倍和9.99—863.12倍。下调基因倍数在石红137中高于L甜。
2.7 类黄酮途径
类黄酮是植物体内重要的次生代谢物之一。通过KEGG富集图比较分析发现,2个品种在对照不理中无差异显著的富集通路(图5)。盐胁迫1和24 h处理中存在类黄酮生物合成途径,且差异显著。花青素还原酶(ANR)Sobic.006G226400和黄酮醇合成酶(FLS)Sobic.004G310100参与到类黄酮生物合成途径,2个基因在石红137中的表达量高于L甜(图6)。


A:gCK vs nCK;B:g1h vs n1h;C:g24h vs n24h

表6 涉及光合作用与盐胁迫相关的基因

图6 ANR(A)和FLS(B)在2个品种的FPKM值
2.8 实时荧光定量PCR验证
为了验证转录组测序结果的可靠性,随机选取4个与激素信号转导和光合作用途径相关基因进行实时荧光定量PCR验证(图7)。将qRT-PCR数据和转录组数据进行相关性分析,其中有3个基因qRT-PCR结果与转录组测序结果显著相关(<0.05),表明转录组测序结果真实可靠。

A:Sobic.006G18400;B:006G034300;C:004G300300
3 讨论
高粱叶片对盐胁迫的响应受到多种途径的调控,转录组的整体分析有利于系统性了解高粱的耐盐机制。为了筛选高粱耐盐基因,本研究对2个高粱品种的2个盐胁迫时期进行了转录组测序并对差异基因进行注释。本研究所选2个高粱品种代表了对盐胁迫的不同反应。L甜对盐胁迫的反应要弱于石红137对盐胁迫的反应,了解2个品种对盐胁迫响应的差异,可以揭示控制石红137的耐盐基因,从而对高粱耐盐基因表达形成全面了解。
在2个品种中,total reads最低达到42 082 348,最高达到56 174 902。平均有78.33%的reads比对到了参考基因组(表3)。结果表明,所选数据库相对完整。其余的注释基因未在参考基因组中检测到,鉴定为新基因。
3.1 2个品种中的差异基因
为确定响应盐胁迫的相应基因,对盐胁迫处理3个时期的转录组差异表达基因进行比较。结果表明,gCK vs g24h和g1h vs g24h差异基因数目明显高于gCK vs g1h的差异基因数目,与L甜的形态指标变化一致。nCK vs n24h和n1h vs n24h的差异基因数目高于nCK vs n1h,这与Na+离子含量变化相对应。石红137在盐胁迫3个时期差异基因的数目均高于L甜的差异基因数目,这表明与感盐品种L甜相比,耐盐品种石红137的有更多的基因参与到盐胁迫中,为进一步阐明高粱耐盐的调控机制提供了研究基础。
GO富集分析显示,激素响应、光合作用以及碳代谢相关途径参与到高粱叶片的盐胁迫中来,有利于高粱及时响应盐胁迫。KEGG分析还发现差异基因涉及激素信号传导、光合作用以及类黄酮代谢途径,这些过程都在高粱应对盐胁迫过程中起着重要作用。
3.2 植物激素信号转导途径
植物激素是重要的次级信号分子,能够调节多种外界环境刺激[20]。本研究通过转录组分析发现生长素(IAA)、脱落酸(ABA)、细胞分裂素(CTK)、乙烯(ETH)、赤霉素(GA)、都参与到高粱叶片的盐胁迫,这些基因耐盐胁迫过程中表达模式不同。
ABA在非生物胁迫中起着重要作用,有研究表明高盐条件下植物会诱导产生ABA,并且ABA信号通路在植物对抗盐胁迫过程中是必需的[21]。ABA能够响应干旱、盐胁迫等多种非生物胁迫[22]。植物体内ABA信号分为依赖ABA途径和非依赖ABA途径[23],ABA信号途径由PYR/PYL/PCAR(ABA受体)、PP2C(蛋白磷酸酶2C家族)、SnRK2(SNF1相关蛋白激酶)和ABF(ABA响应转录因子)组成,通过4个部分的调节响应盐胁迫信号。PYR/PYL能够通过抑制PP2C,释放SnRK2激酶,以磷酸化的形式调节ARF活性[24-25]。本研究中ARF的2个同源基因在2个品种中均表达上调,表明ABA含量升高,与Zhu等[22]研究结果一致,表明高粱通过提高植株体内ABA活性来对抗盐胁迫。
生长素作为调节植物生长发育的植物激素,近年来发现对非生物胁迫也具有重要的调节作用[26-27]。有研究分析发现在非生物胁迫中生长素原初反应相关基因、、、的表达量发生改变[28]。盐胁迫会使植物因为水分吸收受阻而导致渗透胁迫,研究表明外源增施生长素能够提高拟南芥叶片的保水能力[29]。本研究获得参与生长素途径的基因家族,这些基因在盐胁迫过程中被显著调节。AUX/IAA的同源基因Sobic.003G291200在应对盐胁迫过程中显著提高。表明高粱应对外界盐胁迫依赖于AUX/IAA介导的信号过程。
3.3 光合作用相关途径
后期参与盐胁迫的途径为光合作用相关途径,这些途径包括光合器官中的碳固定、碳代谢和光合作用-天线蛋白途径。光合作用是植物生产的能量的主要方式,对植物的生长发育起到至关重要的作用。
光合作用包括2个步骤:光反应阶段和暗反应阶段。光反应又分为2个阶段:原初反应、电子传递和光合磷酸化。原初反应包含光能的吸收、传递和转化。本研究中涉及到原初反应的途径为光合作用-天线蛋白。LHC是一类捕光蛋白复合体,能够捕获光能并把能量传递至反应中心[19,30]。本研究发现在2个品种中Lhca和Lhcb 2组蛋白的表达量下调。可能是因为在盐胁迫条件下,细胞内渗透势升高,为了维持植物细胞内正常的生理活动,高粱叶片LHC多个蛋白下调表达,以降低光合速率,而使高粱适应盐胁迫环境。PPC是C4作物进行光合作用碳固定的关键酶[31],本研究2个PPC同源基因(Sobic.002G167000、Sobic.007G106500)在石红137中上调表达,一个PPC同源基因(Sobic.010G160700)在石红137中下调表达,表明高盐环境对高粱叶片的碳固定产生了影响。PRK是碳代谢的关键酶,将5-磷酸核酮糖催化合成1,5-二磷酸核酮糖(RuBP),PRK在2个品种中的下调表达表明,RuBP的再生受到抑制,从而影响光合作用的正常进行。
3.4 类黄酮生物合成途径
类黄酮是一类植物次生代谢产物,常见的类黄酮包括查尔酮、黄酮醇、黄酮、黄烷醇、黄烷酮、花青素等。比较不同品种对照样本未发现差异显著代谢途径,但对2个时期的盐胁迫处理样品进行比较发现共同的差异代谢途径,即类黄酮生物合成途径。对参与到此代谢途径的基因进行比较发现石红137中花青素还原酶(ANR)和黄酮醇合成酶(FLS)的表达量均高于L甜。结果表明,石红137在次级代谢产物类黄酮合成过程要强于L甜。研究表明耐盐植物的耐盐性主要体现在植株体内的离子平衡,Na+和Cl-被贮藏在液泡内,保持细胞渗透势的稳定。部分有机溶质,例如蛋白质、氨基酸等物质也起到稳定细胞渗透势的作用[10]。则次级代谢产物的合成能力可能是石红137和L甜是否耐盐的重要原因。
4 结论
高粱的盐胁迫过程是一个复杂的生物过程,依赖于多个基因在复杂网络中的平衡表达。盐胁迫条件下,高粱应对环境刺激受到激素信号转导和光合作用的控制。类黄酮生物合成途径在耐盐品种中起到了重要作用。
[1] KAUSHAL S S. Increased salinization decreases safe drinking water ., 2016, 50(6): 2765-2766.
[2] KHALID N, AQSA T, IQRA, KHALID H, ABDUL M. Induction of salt tolerance in two cultivars of sorghum (L.) by exogenous application of proline at seedling stage., 2010, 10(1): 93-99.
[3] GUZM N-MURILLO M A, ASCENCIO F, LARRINAGA- MAYORAL J A. Germination and ROS detoxification in bell pepper (L.) under NaCl stress and treatment with microalgae extracts., 2013, 250(1): 33-42.
[4] MUNNS R. Comparative physiology of salt and water stress., 2002, 25(2): 239-250.
[5] 高玉红, 闫生辉, 邓黎黎. 不同盐胁迫对甜瓜幼苗根系和地上部生长发育的影响. 江苏农业科学, 2019, 47(3): 120-123.
GAO Y H, YAN S H, DENG L L. Effects of different salt stress on root and above-ground growth and development of muskmelon seedlings., 2019, 47(3): 120-123. (in Chinese)
[6] GULZAR S, KHAN M A, UNGAR I A. Salt tolerance of a coastal salt marsh grass., 2003, 34(17/18): 2595-2605.
[7] KERKEB L, DONAIRE J P, VENEMA K, RODR GUEZ-ROSALES M P. Tolerance to NaCl induces changes in plasma membrane lipid composition, fluidity and H+-ATPase activity of tomato calli., 2001, 113(2): 217-224.
[8] PARIDA A K, DAS A B, MITTRA B, MOHANTY P. Salt-stress induced alterations in protein profile and protease activity in the mangrove bruguiera parviflora., 2004, 59(5/6): 408-414.
[9] HARE P D, CRESS W A. Metabolic implications of stress-induced proline accumulation in plants., 1997, 21(2): 79-102.
[10] SERRAJ R, SINCLAIR R T. Osmolyte accumulation: can it really help increase crop yield under drought conditions., 2002, 25(3): 33-41.
[11] GANIE S A, MOLLA K A, HENRY R J, BHAT K V, MONDAL T K. Advances in understanding salt tolerance in rice., 2019, 132(4): 851-870.
[12] WANG M, WANG Y, ZHANG Y, LI C, GONG S, YAN S, LI G, HU G, REN H, YANG J, YU T, YANG K. Comparative transcriptome analysis of salt-sensitive and salt-tolerant maize reveals potential mechanisms to enhance salt resistance., 2019, 41(7): 781-801.
[13] AMIRBAKHTIAR N, ISMAILI A, GHAFFARI M R, NAZARIAN FIROUZABADI F, SHOBBAR Z S. Transcriptome response of roots to salt stress in a salinity-tolerant bread wheat cultivar., 2019, 14(3): e0213305.
[14] YANG Z, ZHENG H, WEI X, SONG J, WANG B, SUI N. Transcriptome analysis of sweet sorghum inbred lines differing in salt tolerance provides novel insights into salt exclusion by roots., 2018, 430(1): 423-439.
[15] AKBUDAK M A, FILIZ E, KONTBAY K. DREB2 (dehydration- responsive element-binding protein 2) type transcription factor in sorghum (): genome-wide identification, characterization and expression profiles under cadmium and salt stresses., 2018, 8(10): 426.
[16] FORGHANI A H, ALMODARES A, EHSANPOUR A A. Potential objectives for gibberellic acid and paclobutrazol under salt stress in sweet sorghum ([L.] Moench cv. Sofra)., 2018, 61(1): 113-124.
[17] 王海莲, 张华文, 刘宾, 杨延兵, 秦岭, 陈二影, 管延安. 低度盐胁迫下高粱苗期相关性状的QTL定位. 分子植物育种, 2017, 15(2): 604-610.
WANG H L, ZHANG H W, LIU B, YANG Y B, QIN L, CHEN E Y, GUAN Y A. QTL mapping for traits related to salt tolerance at seedling stage of Sorghum under low salt stress., 2017, 15(2): 604-610. (in Chinese)
[18] WANG H L. CHEN G L, ZHANG H W, LIU B, YANG Y B, GUAN Y A. Identification of QTLs for salt tolerance at germination and seedling stage ofL. Moench.,2014, 196: 117-127.
[19] 汪仁, 李晓丹, 江玉梅, 贺佳, 彭峰, 夏冰. 石蒜捕光叶绿素a/b结合蛋白基因的克隆和序列分析. 江苏农业科学, 2011, 39(2): 42-44.
WANG R, LI X D, JIANG Y M, HE J, PENG F, XIA B. Cloning and sequence analysis of light-harvesting chlorophyll a/b binding protein gene from., 2011, 39(2): 42-44. (in Chinese)
[20] DUAN L, DIETRICH D, NG C H, CHAN P M, BHALERAO R, BENNETT M J, DINNENY J R. Endodermal ABA signaling promotes lateral root quiescence during salt stress inseedlings., 2013, 25(1): 324-341.
[21] ACHARD P, CHENG H, DE GRAUWE L, DECAT J, SCHOUTTETEN H, MORITZ T, VANDER S D, PENG J, HARBERD N P. Integration of plant responses to environmentally activated phytohormonal signals., 2006, 311(5757): 91-94.
[22] ZHU J K. Salt and drought stress signal transduction in plants., 2002, 53(1): 247-273.
[23] YAMAGUCHI-SHINOZAKI K, SHINOZAKI K. Transcriptional regulatory networks in cellular responses and tolerance to dehydration and cold stresses., 2006, 57(1): 781-803.
[24] CUTLER S R, RODRIGUEZ P L, FINKELSTEIN R R, ABRAMS S R. Abscisic acid: emergence of a core signaling network., 2010, 61(1): 651-679.
[25] FUJITA Y, NAKASHIMA K, YOSHIDA T, KATAGIRI T, KIDOKORO S, KANAMORI N, UMEZAWA T, FUJITA M, MARUYAMA K, ISHIYAMA K, KOBAYASHI M, NAKASONE S, YAMADA K, ITO T, SHINOZAKI K, YAMAGUCHI-SHINOZAKI K. Three SnRK2 protein kinases are the main positive regulators of abscisic acid signaling in response to water stress in., 2009, 50(12): 2123-2132.
[26] YUAN H, ZHAO K, LEI H, SHEN X, LIU Y, LIAO X, LI T. Genome-wide analysis of the GH3 family in apple (a)., 2013, 14(1): 297.
[27] CHEONG Y H, CHANG H-S, GUPTA R, WANG X, ZHU T, LUAN S. Transcriptional profiling reveals novel interactions between wounding, pathogen, abiotic stress, and hormonal responses in., 2002, 129(2): 661-677.
[28] SONG Y, WANG L, XIONG L. Comprehensive expression profiling analysis of OsIAA gene family in developmental processes and in response to phytohormone and stress treatments., 2009, 229(3): 577-591.
[29] LIU X, ZHANG H, ZHAO Y, FENG Z, LI Q, YANG H Q, LUAN S, LI J, HE Z H. Auxin controls seed dormancy through stimulation of abscisic acid signaling by inducing ARF-mediated ABI3 activation in Arabidopsis ., 2013, 110(38): 15485-15490.
[30] 李安节, 柳振峰. 植物光系统Ⅱ捕光过程的超分子结构基础. 生物化学与生物物理进展, 2018, 45(9): 935-946.
LI A J, LIU Z F. Supramolecular structural basis of light harvesting in plant photosystem II., 2018, 45(9): 935-946. (in Chinese)
[31] O'LEARY B, PARK J, PLAXTON WILLIAMC. The remarkable diversity of plant PEPC (phosphoenolpyruvate carboxylase): recent insights into the physiological functions and post-translational controls of non-photosynthetic PEPCs., 2011, 436(1): 15-34.
Transcriptome Analysis and Gene Mining of Salt Tolerance in Sorghum Seedlings (L. Moench)
DONG Ming1, KUERBAN Zaituniguli2, Lü Peng1, DU RuiHeng1, Ye Kai2, HOU ShengLin1, LIU GuoQing1
(1Institute of Millet Crops, Hebei Academy of Agriculture & Forestry Sciences/The Key Minor Cereal Crops Laboratory of Hebei Province, Shijiazhuang 050035;2Institute of Bioenergy, Xinjiang Academy of Agriculture Sciences, Urumqi 830091)
【Object】The primary aim of this study was to identify salt tolerance genes and explore the tolerance response mechanism under salt stress in sorghum, which may provide a theoretical basis for sorghum salt tolerance breeding. 【Method】Two sorghum varieties, L-Tian, salt-sensitive and Shihong 137, salt-tolerant were employed as plant materials. The sorghum seedlings were treated with 2% NaCl solution at three-leaf and one heart stage. Three treatments including 0 h (CK), 1 h and 24 h were conducted. The plant height, root length, dry matter weight, Na+content and relative content of chlorophyll (SPAD value) were determined, and the transcriptome sequencing was performed on Illumina HiSeq 2000 platform. The FPKM method was employed to calculate the gene expression level. Both the differential expression fold (Fold Change) ≥ 2 and FDR<0.001 were used as screening criteria to detect the differentially expressed genes. The Gene Ontology and KEGG Pathway databases were used to analyze the differentially expressed genes involved in salt stress at different time points. 【Result】Salt stress had no significant effect on plant height, root length and dry matter weight of sorghum, but had significant effect on Na+content and SPAD value. The plant height, root length, Na+content and SPAD value of Shihong 137 were higher than those of L-tian. Totally 26628 known genes and 866 new genes have been identified from RNA-seq, of which, the number of differentially expressed genes from Shihong 137 is higher than that from L-tian. 375, 4206 and 3750 differentially expressed genes in 0 h vs 1 h, 0 h vs 24 h and 1 h vs 24 h groups had been identified from Shihong 137 respectively. The number of differentially expressed genes of 0 h vs 1 h, 0 h vs 24 h, and 1 h vs 24 h of L-tian was 167, 2534 and 1612, respectively. GO and KEGG analysis revealed that plant hormones such as abscisic acid, auxin, cytokinin, gibberellin and ethylene were involved in the salt tolerance of sorghum at an early stage of salt stress (1h), while Lhca, Lhcb, phosphoenolpyruvate carboxylase, and ribulose phosphate kinase were involved in the salt tolerance at a late stage of salt stress (24 h). The difference in salt tolerance between Shihong 137 and L-tian was mainly caused by the flavonoid biosynthetic metabolic pathway. 【Conclusion】The sorghum response to salt stress is a complex biological process that relies on the balanced expression of multiple genes in complex networks. Under salt stress, sorghum response to environmental stimuli was controlled by both hormonal signal transduction and photosynthesis. The flavonoid biosynthesis pathway played an important role in salt-tolerant varieties.
sorghum (L.); salt stress; transcriptome; gene mining
10.3864/j.issn.0578-1752.2019.22.005

2019-06-17;
2019-09-24
国家自然科学基金(31660435)、国家现代农业产业技术体系(CARS-06-13.5-A10)
董明,E-mail:dddongming@126.com。再吐尼古丽·库尔班,E-mail:zaytungul@sohu.com。董明和再吐尼古丽·库尔班为同等贡献作者。通信作者侯升林,E-mail:shenglinhou@aliyun.com;刘国庆,E-mail:guoqingliu@hotmail.com
(责任编辑 李莉)
