香樟SRAP分子标记引物筛选
2019-12-10张丽华韩浩章王晓立李素华王芳董蓉刘宇
张丽华 韩浩章 王晓立 李素华 王芳 董蓉 刘宇


摘 要:以香樟嫩叶为试验材料,采用SRAP-PCR分子标记体系对420对引物组合进行了筛选。结果表明:共选出19对引物组合,扩增出367条带,平均19.3条,范围在15~25条。其中,多态性带230条,平均12.11条,范围在6~17条;多态性的比例62.16%,范围在40%~81%。SRAP-PCR分子标记体系稳定,筛选出的引物组合可用于不同种源香樟遗传多样性及品种鉴定的研究。
关键词:香樟;SRAP分子标记;引物筛选
中图分类号 S79文献标识码 A文章编号 1007-7731(2019)20-0025-04
Abstract:Using camphor leaves as material,420 primer combinations were screened by SRAP-PCR molecular marker system. The results showed that:a total of 19 pairs of primers were selected,and 367 bands were expanded,with an average of 19.3 and a range of 15~25,of which 230 polymorphic belts,an average of 12.11,a range of 6~17,a polymorphic ratio of 62.16 %,and a range of 40~81 %. The SRAP-PCR molecular marker system is stable,and the selected primer combination can be used for genetic diversity and variety identification of different species of camphor.
Key words:Camphor;SRAP molecular marker;Primer screening
香樟[Cinnamomum camphora(L.) Presl],又名小叶樟、樟、芳樟,为樟科樟属常绿乔木,主要分布于中国长江以南地区,适宜深厚、肥沃的酸性土壤环境和温暖、湿润气候,不耐干旱、贫瘠和盐碱土壤环境,是我国重要的园林绿化树种、林用树种和经济树种。近年来,香樟在我国长江以北地区的栽培面积越来越广,但由于引种地与种源生产地之间的气候和土壤条件差异较大,冬季低温和土壤盐碱环境已成为了限制香樟在我国北方地区引种栽培成功的关键因素,选育抗寒、耐盐碱品种成为了当务之急[1]。目前,关于香樟优良品种选育的研究主要集中在种质资源、田间子代生长特性分析以及优良家系早期筛选等方面[2],而关于香樟耐盐碱、耐寒品种选育研究则相对较少。香樟的生长发育受环境的影响很大,地理类型较多,实生后代变异程度大[3-4],形态标记、同功酶和染色体核型分析等传统标记方法很难区分辨别香樟种间或种内关系,而DNA分子标记技术能在基因组水平上探究植物种间及无性系间的遗传变异,不受气候和土壤环境因素的干扰,所得的信息内容丰富,更能体现出植物种内和种间的遗传变异。
相关序列扩增多态性(Sequence-related amplifiedpolymorphism,SRAP)是Li和Quiros[5]开发的分子标记技术。在观赏南瓜(Cucurbita moschata)[6]、野牛草(Buchloe dactyloides)[7]等植物上的研究表明,SRAP比ISSR(Inter-simple Sequence Repeat)、SSR(Simple Sequence Repeat)、RAPD(Random Amplified Polymorphic DNA)等幾种分子标记表现出更丰富的遗传多样性,具有操作简单、多态性丰富、引物利用率高、重复性好、费用低等优点[8]。SRAP标记技术已广泛应用于品种鉴定、遗传多样性分析、遗传变异和亲缘关系分析和指纹图谱构建等的研究中[9]。关于香樟植物分子标记方面,宋爱云等[4]于2003年将RAPD分子标记技术应用于鉴定香樟优选株和普通株,吕昕谣等[10]也建立了普陀樟的SRAP反应体系,而关于香樟SRAP分子标记方面的研究尚未见报道。为此,本研究采用课题组成熟的SRAP-PCR反应体系,对420对引物进行了筛选,以期为进一步开展香樟的遗传多样性及相关遗传研究提供技术支撑,也可为香樟基因组分析、分子标记辅助育种等研究奠定基础。
1 材料和方法
1.1 试验材料 本试验所选香樟试材共14个样品(表1),包括浙江、江苏、江西、安徽等地香樟分布地区,于2018年2—4月份采集并移栽于宿迁学院樟属植物种苗基地,2019年4月进行基因组DNA提取。所用SRAP正反向引物参照前人研究公布的引物序列[5-10],由深圳华大基质科技有限公司合成,PCR反应相关试剂10X Buffer、Mg2+、Taq DNA聚合酶、dNTPs和DNA marker均购自天根生化科技有限公司。
1.2 试验方法
1.2.1 基因组DNA的提取与检测 采用试剂盒法(购自原平皓生物技术有限公司)提取香樟基因组DNA,采用1% 琼脂糖凝胶电泳测基因组DNA的质量(电泳仪电源为美国BIO-RAD Powerpac Basic型,电泳仪为北京君意JY-SPCT型),电泳结束后,在自动凝胶图像分析仪(上海培清JS-780R全自动凝胶成像分析仪)上观测,确定DNA纯度并拍照。用核酸蛋白测定仪(BioPhotometer D30)确定其浓度以及A260/A280的比值,最后将样品DNA质量浓度稀释到50ng/μL,于-80℃保存备用。
1.2.2 PCR扩增体系 SRAP-PCR反应体系为:2.0mmol/L的Mg2+,0.2mmol/L的dNTPs,DNA模板60ng,Taq DNA聚合酶1.5U,正反引物0.5μmol/L,10X loading Buffer 2μL,加ddH2O至20μL。
1.2.3 PCR反应程序 PCR反应程序为:94℃预变性5min,94℃变性1min,35℃复性1min,72℃延伸1min反应5个循环,94℃变性1min,51℃复性1min,72℃延伸1min反应35个循环,72℃延伸5min,4℃保存。PCR扩增产物采用8%非变性聚丙烯酰胺凝胶电泳分离,银染法染色后,在可见光灯箱上照相记录。
1.2.4 引物筛选 从供试材料中选取外观形态差异较大的3个香樟材料DNA模板对420对SRAP引物进行筛选,最终筛选出清晰度高、稳定性好、多态性丰富的SRAP引物用于所有DNA样品扩增(表2)。
1.3 数据处理与分析 对凝胶电泳结果中清晰、易于辨认的条带根据其有无进行统计,同一位点有带记为“1”,无带记为“0”,建立二元原始数字矩阵,并计算扩增产物的多态位点数和多态性百分率。
2 结果与分析
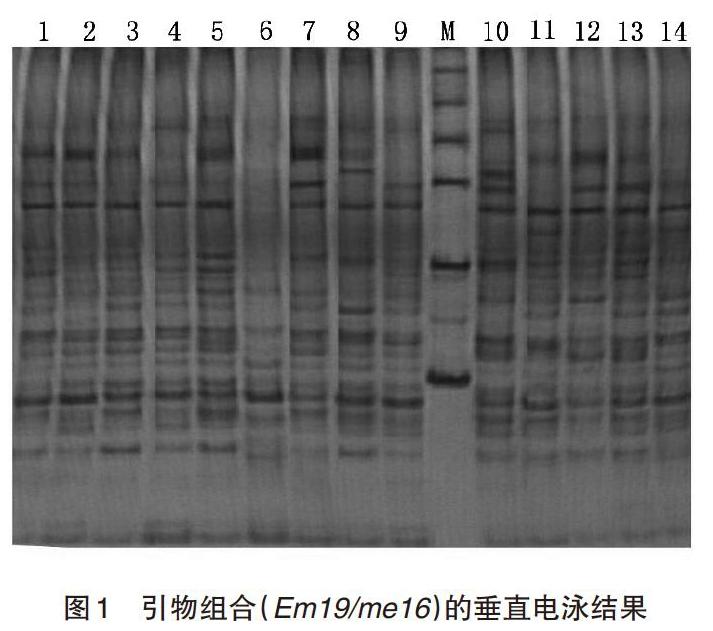
2.1 香樟DNA的提取检测 通过测定,14份香樟的DNA样品的A260/A280的值均数在1.7~2.0,经过0.8%琼脂糖电泳检测,其条带清晰,符合扩增的基本要求(图1)。
2.2 SRAP分子标记引物组合的筛选及多态性分析 以14份香樟种源DNA为材料,利用SRAP分子标记体系对420对引物进行筛选,淘汰那些模糊的、条带较少的引物,选择出清晰且明亮、条带数目多的引物。最终从420对引物组合中选出19对特异性强的引物组合,共获得367条带,平均19.3条,范围在15~25条。其中,多态性带230条,平均12.11条,范围在6~17条;多态性比例为62.16%,范围在40%~81%(表3)。
3 讨论与结论
SRAP-PCR反应的引物序列和扩增程序都是特定的,所反映的遗传多态性能直接揭示不同植物基因组DNA间的差异[11]。研究认为,SRAP-PCR试验的结果与温度以及Mg2+、dNTPS、引物、Taq DNA聚合酶、模板DNA这5个因素的浓度有关[12],不同物种的SRAP-PCR研究影响因素的差别也很大,这可能与不同植物本身的遗传差异有关,也可能与研究者在设定正交设计时不同因素的浓度设定不同有关。此外,不同研究者采用的仪器设备、凝胶的质量和浓度均有差异,可能也是造成SRAP-PCR试验结果存在差异的原因。因此,适宜的SRAP-PCR反应体系必须针对不同植物种类、不同试验条件、不同试验仪器及试剂对主要的影响因素进行优化,以保证试验结果的稳定性和可靠性。
目前,关于SRAP分子标记引物筛选的研究较多。郭大龙等[8]从100对SRAP引物组合中,筛选出19对能在葡萄中扩增出条带清晰且多态性好的引物组合。安佰义等[12]从176对SRAP引物组合中,筛选出25对适用于白檀的引物组合。李芳芳等[13]从96对SRAP引物组合中,筛选得到18对条带清晰、稳定且多态性丰富的引物作为枫香树基因组的扩增引物组合。本研究中,将20条正向引物和21条反向引物随机组合得到420对引物组合,其中19个组合能在香樟DNA中扩增出清晰明亮的条带。由此可见,SRAP引物组合在不同植物的基因组上具有不同的结合位点及数量,这导致扩增效果及适用引物存在差异。引物序列是SRAP-PCR扩增过程成功的关键,即使建立了最优PCR扩增体系,倘若引物的选择没有特异性,往往实验结果并不理想[11]。
在获得最优PCR扩增体系的基础上,本研究对420对引物进行了大量的筛选工作,从中初步筛选出19对引物。通过对这些引物进行SRAP-PCR的多态性数据分析,得出总条带数为367条。其中,多态性条带数达230条,其扩增产物片段大小均在2000bp以内且分布较均匀,多态率为62.16%。这为香樟的遗传多样性研究、品种鉴定、亲缘关系分析、遗传图谱构建等研究奠定了基础。
参考文献
[1]韩浩章,王晓立,刘宇,等.香樟黄化病现状分析及其治理研究[J].北方园艺,2010(13):232-235.
[2]张谦,曾令海,蔡燕灵,等.樟树自由授粉家系生长与形质性状的遗传分析[J].中南林业科技大学学报,2014,34(1):1-6.
[3]姚小華,任华东,吴柯久,等.樟树苗期遗传变异研究[J].江西农业大学学报,1999,21(3):320-328.
[4]宋爱云,陈辉,董林水.RAPD分子标记在鉴定香樟优选株和普通株中的应用[J].应用与环境生物学报,2003,9(3):263-265.
[5]Li G,Quiros C F. Sequence-related amplified polymorphism(SRAP),A new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica[J]. Theor Appl Genet.,2001(103):455-461.
[6]Ferriol M,Pic B,Nuez F. Genetic diversity of a germplasm collection of Cucurbita pepousing SRAP and AFLP markers[J]. Theor Appl Genet.,2003(107):271-282.
[7]Budak H,Shearman R C,Parmaksiz I,et al. Molecular characterization of Buffalo grass germplasm using sequence-related amplified polymorphism markers[J]. Theor Appl Genet.,2004(108):328-334.
[8]郭大龙,张君玉,李猛,等.葡萄SRAP反应体系优化及引物筛选[J].基因组学与应用生物学,2010,29(2):379-384.
[9]郭丽琴,李友丽,饶国栋,等.利用SRAP分子标记分析杨属遗传变异和亲缘关系[J].林业科学研究,2019,32(3):88-96.
[10]吕昕谣,赵颖,赵国淼,等.普陀樟SRAP-PCR反应体系的建立[J].浙江林业科技,2014,34(3):23-27.
[11]Zhu S.,Liu T.,Tang Q.,et al. Evaluation of bamboo genetic diversity using morphological and SRAP analyses[J].Genetika,2014,50(3):306-313.
[12]安佰义,于慧颖,吴双,等.白檀SRAP-PCR体系优化及引物筛选[J].北方园艺,2018,24:15-20.
[13]李芳芳,杨少宗,柳新红,等.枫香树DNA提取及SRAP-PCR反应体系的建立与优化[J].河南农业大学学报,2015,49(1):46-52.
(责编:张宏民)
