普通烟草碳酸酐酶家族基因的生物信息学分析
2019-12-06赵希胜曾鼎宸吕承承蒋垚童铸吴润生李立芹鲁黎明
赵希胜 曾鼎宸 吕承承 蒋垚 童铸 吴润生 李立芹 鲁黎明

摘 要:为挖掘普通烟草碳酸酐酶基因的信息并探讨其功能,本研究利用生物信息学方法,在烟草基因组数据库中对普通烟草碳酸酐酶家族成员进行了检索,并对其理化性质、遗传进化、基因结构、蛋白保守基序、顺式作用元件和组织表达模式进行了分析。结果显示,在普通烟草中至少含有9个α和6个β亚家族成员。在进化关系上,不同亚家族成员之间,序列同源性较低;与水稻相比,拟南芥和普通烟草两个亚家族间的亲缘关系较近。亚细胞定位分析结果显示,烟草α亚家族成员在细胞壁、细胞膜、线粒体、叶绿体、细胞质等细胞器中均有分布,而β亚家族成员均存在于叶绿体中。基因结构和保守基序分析说明,各亚家族成员的基因结构和蛋白保守基序呈现较高的一致性。启动子顺式作用元件分析显示,烟草碳酸酐酶基因的启动子中,均含有多个参与光反应、激素响应、非生物胁迫和机械损伤等相关的顺式作用元件。基因组织表达模式分析表明,烟草的碳酸酐酶基因不同成员在不同组织表达水平存在差异,并呈现出时空特异性。本研究结果为烟草碳酸酐酶基因的功能研究奠定了基础。
关键词:烟草;碳酸酐酶;生物信息学;基因结构;表达
中图分类号:S572.03 文章编号:1007-5119(2019)05-0068-09 DOI:10.13496/j.issn.1007-5119.2019.05.010
Abstract:In order to find out the information of carbonic cnhydrase (CA) genes in tobacco and explore their functions, the members of common tobacco CA family were identified from the tobacco genome database, and the physical and chemical properties, genetic evolution, gene structure, conserved motifs of proteins, cis-acting elements and tissue expression patterns were analyzed. The results showed that there were at least 9 alpha and 6 beta subfamily members in tobacco. On evolutionary relations, lower sequence homology was observed between different subfamily members. Compared with rice, CA members betweenArabidopsis thalianaand common tobacco are more closely related. Subcellular localization analysis showed that tobacco alpha subfamily members were distributed in cell wall, cell membrane, mitochondria, chloroplast, cytoplasm and other organelles, while beta subfamily members were present in chloroplast. The analysis of gene structure and conserved motifs showed that the gene structure and conserved motifs of proteins of each subfamily were highly consistent. Analysis of the cis-acting elements of the promoter showed that the promoter of the carbonic anhydriase genes in tobacco contains several cis-acting elements involved in light reaction, hormone response, abiotic stress and mechanical damage. The expression pattern analysis confirmed that the expression levels of carbonic anhydrase genes in different subfamilies of tobacco were different in different tissues and showed the specificity of time and space. The results of this study laid a foundation for the functional study of tobacco carbonic anhydrase genes.
Keywords: tobacco; carbonic anhydrase; bioinformatics; gene structure; expression
碳酸酐酶(Carbonic Anhydrase; CA; EC 4.2.1.1)是一種与光合作用密切相关的金属酶,能够高效可逆地催化CO2与HCO3-之间的转化,是CO2浓缩机制的重要组分[1]。生物体内的碳酸酐酶是一个多基因家族,目前依据晶体结构和亚基组成,可将其分为α、β、γ、δ、ε及θ等6个亚家族,尽管这些亚家族成员之间的氨基酸序列差异较大,但其活性中心均含有催化作用必不可少的锌离子[2-3]。在高等植物中,主要含有α、β、γ三个亚家族[4]。其中α亚家族主要以单体的形式存在,大部分成员由10条β链围成一个大的β片区活性中心,其外围被7个α螺旋所包围。而β亚家族最早在植物中被鉴定[5],β亚家族成员的Zn2+与一个组氨酸、两个半胱氨酸和一个水分子配位形成不规则的四面体位于活性中心部位[6],其主要鉴别模式为C-[SA]-D-S-R-[LIVM]-x-[AP][7]。截至目前,在拟南芥中已克隆了8个α、6个β、3个γ和2个γ-like基因[8-9];水稻、玉米、高粱和谷子中各鉴定了2个、5个、5个和4个与C4光合通路相关的β亚家族基因[10-11];此外,小黑杨、红藻和衣藻等植物的CA家族基因也相继被鉴定和克隆[12-14]。
高等植物碳酸酐酶的主要功能是参与光合作用,如将衣原体CA基因过表达于烟草后显著提高了转基因植株的光合速率和生物学产量[15]。水稻OsβCA1突变体植株和RNAi株系均表现出生物学产量和CA活性明显降低[16]。除此之外,CA还参与了植物呼吸作用、pH调节、氨基酸及脂质的代谢、气孔运动、非生物胁迫等生理过程[17]。如玉米碳酸酐酶突变体植株的气孔导度明显高于野生型,并随着CO2分压的增加,气孔关闭速度变慢[18],HU等[19]在研究拟南芥CA突变体时也得到了类似的结果。FROMM等[20]和PERALES等[21]研究发现,拟南芥γ-CAs在线粒体电子传递和光呼吸中起着重要作用。HOANG等[22]将棉花碳酸酐酶活性抑制后,发现其脂质的合成速率显著降低。在拟南芥βca2βca4双突变体叶片中,氮的同化受到阻遏[23]。烟草叶绿体碳酸酐酶基因在酵母中表达后,其抗氧化活性显著增强[24]。TIANPEI等[25]将水稻OSCA基因异源表达后也发现,其活性氧含量明显降低,并增强了对热应激的耐受性。水稻OsCA1基因过表达后,转基因幼苗的耐盐性显著增强[26]。
普通烟草(Nicotiana tabacum)是一种重要的叶用经济作物,因此,深入探讨碳酸酐酶在烟草光合作用中的地位与作用,具有重要的理论意义与实践
价值。然而,作为一个多功能基因家族,烟草的碳酸酐酶研究却较为薄弱。所以,本研究采用生物信息学手段,对普通烟草碳酸酐酶α和β两个亚家族进行了检索,并对其理化性质、亚细胞定位、进化关系、基因结构、保守motif、启动子顺式作用元件以及组织表达模式等进行了分析,以期为烟草碳酸酐酶基因的功能研究提供借鉴。
1 材料与方法
1.1 普通烟草CA家族成员的鉴定
从TAIR(https://www.arabidopsis.org/)数据库下载拟南芥已鉴定的α-CA和β-CA蛋白全长序列,通过本地Blast检索普通烟草蛋白数据库(https://solgenomics.net/,e-value值为1e-5)[27-28]。从pfam数据库(http://pfam.xfam.org/)分别下载α-CA和β-CA的隐马尔可夫(HMM)模型(α-CA:PF00484;β-CA:PF00494),利用HMMER 3.0 检索烟草蛋白数据库(e-value值为1e-5)。将以上两种方式获得的蛋白序列,利用Perl去除冗余后,通过pfam、CDD和SMART软件进一步验证结构域信息,剔除不完全序列,最终获得普通烟草α-CA和β-CA基因家族成员。
1.2 基本理化性质分析
利用ProtParam(http://web.expasy.org/protparam/),在线预测普通烟草α和β碳酸酐酶蛋白的分子量、等电点。利用ProtScale(http://web.expasy.org/protscale/)网站,进行疏水性分析。利用Cell-PLoc 2.0(http://www. csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)进行亚细胞定位预测。
1.3 同源性分析
从Phytozome(https://phytozome.jgi.doe.gov/pz/ portal.html)下载水稻已鉴定的α-CA与β-CA亚家族成员。利用MEGA7.0软件将获得的普通烟草与拟南芥和水稻所有的α-CA与β-CA两个亚家族成员的蛋白序列进行比对后,采用邻接法构建发育树(Bootstrap值为1000)。
1.4 蛋白保守基序和基因结构分析
将烟草α和β亚家族蛋白序列利用MEGA 7软件进行聚类,从茄科基因组数据库下载普通烟草基因组数据文件,利用TBtools(https://github.com/ CJ-Chen/TBtools)提取α-CA与β-CA基因结构信息并进行可视化。利用MEME软件(http://meme- suite.org/tools/meme)预测α-CA和β-CA家族蛋白保守基序(参数设置:motif最大值为10,motif长度范围在6~100之间)。
1.5 启动子顺式作用元件分析
从NCBI数据库截取普通烟草两个CA亚家族成员起始密码子(ATG)上游2000 bp,利用PlantCARE在线工具[29],预测各基因启动子顺式作用元件,并进行绘图。
1.6 基因表达模式分析
在GEO(https://www.ncbi.nlm.nih.gov/gds/)数据库,下载普通烟草K326根(Root)、芽(Shoot)、茎尖(Shoot Apex)分别在0、6、12、18 h的表达数据(GSE95717)。利用TopHat2软件,将本研究获得的普通烟草α和β碳酸酐酶成员依次检索并提取相应表达数据,进行均一化处理后利用Mev软件作图。
2 结 果
2.1 普通煙草CA家族基因的鉴定
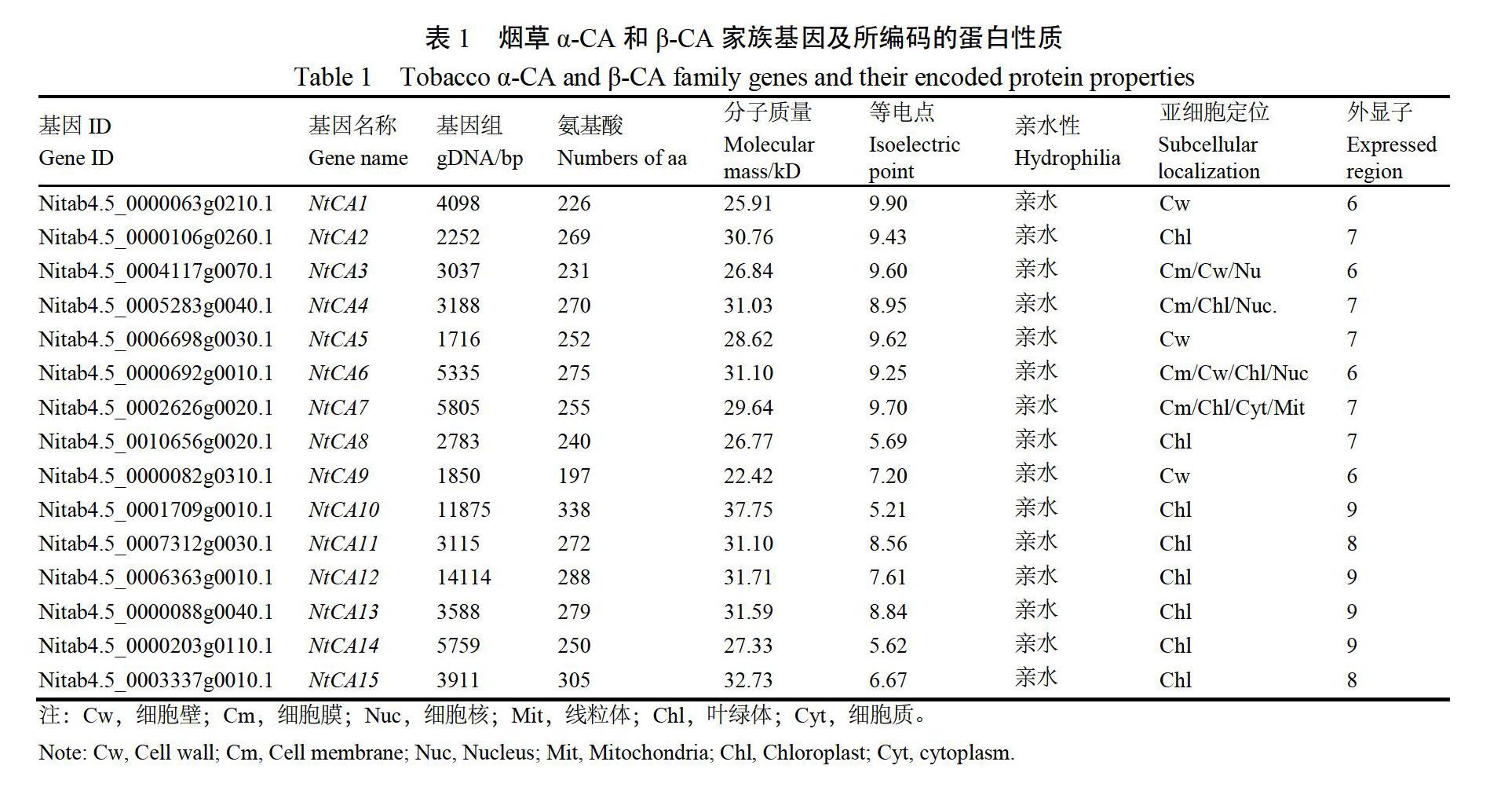
利用本文1.1所述的两种方法获得烟草CA候选序列,利用CDD、SMART和Pfam等蛋白结构预测工具进一步验证,最终在烟草基因组数据库中检索出了9个α-CA和6个β-CA基因,分别命名为NtCA1~9及NtCA10~15(表1),其DNA序列长度介于1716~14114 bp之间。
2.2 基本理化性质分析
由表1看出,α-CA亚家族的NtCA1~9编码的氨基酸残基数量为197~275个,分子量在22.42~31.10 kDa,理论PI值均大于7.0。β-CA亚家族的NtCA10~15所编码的氨基酸数量为250~340个,分子量在27.33~37.75 kDa,其理论PI值3个成员大于7.0,另外3个成员均在5~7之间。
亲/疏水性分析显示,两个亚家族所有成员均为亲水性蛋白。亚细胞定位预测表明,α-CA家族成员分布于细胞壁、叶绿体、细胞膜、线粒体等细胞器上。有趣的是,NtCA3、NtCA4、NtCA7和NtCA8在多个细胞器中均有分布,而β-CA主要分布在叶绿体上。
2.3 序列相似性与同源性分析
由表2看出,α-CA与β-CA亚家族之间的序列相似度较低,如NtCA1和NtCA11序列相似度仅为7.7 %;而亚家族内各成员的序列相似度较高,如α-CA亚家族中,NtCA2和NtCA5的相似度高达87.7%。
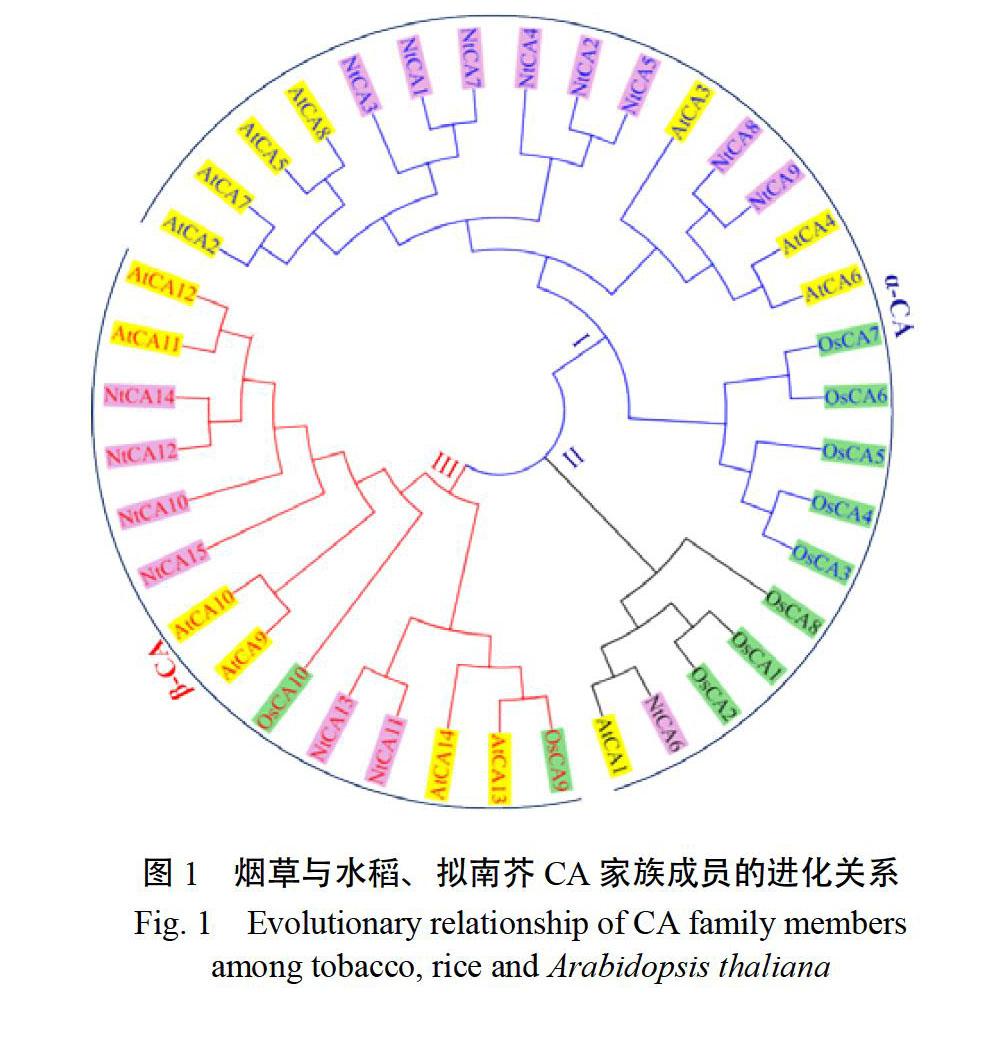
为了明确烟草15个CA成员与水稻及拟南芥CA成员的同源性,将拟南芥8个α-CA(AtCA1-8)和6个β-CA(AtCA9-14)的氨基酸序列,及从Phytozome
下载的水稻8个α-CA(OsCA1-8)和2个β-CA(OsCA9-10)的氨基酸序列,连同这15个普通烟草的CA成员一起比对,并构建NJ树(图1)。可以明显看出,参与NJ树构建的所有β-CA成员,均很好地聚为一类,而α-CA亚家族成员根据自举值被分为两类,说明不同CA亚家族成员在进化上具有相对独立性。此外,整体上看,普通烟草与拟南芥两个CA亚家族成员之间的亲缘关系更近一些。
2.4 保守基序和基因结构分析
聚类分析的结果表明(图2A),烟草CA15个成员聚为两类,其中,NtCA1至NtCA9为一类,NtCA10至NtCA15聚为另外一类。Motif分析表明,烟草所有α-CA家族成员均含有Motif1、Motif2和Motif5。所有β-CA亚家族成员均包含Motif3、Motif4和Motif6(图2B、2D)。
基因结构分析显示(图2C),烟草α-CA亚家族各成员外显子个数在6~7之间,而β-CA成员则在8~10之间,β-CA亚家族成员的外显子数目要多于α-CA亚家族,说明β-CA亚家族成员的基因结构较α-CA亚家族复杂,其中所蕴藏的生物学意义值得探索。
2.5 启动子顺式作用元件分析
烟草15个CA基因的启动子顺式作用元件分析结果表明,在这些基因的启动子中,含有多个参与光响应、激素、非生物胁迫等相关的顺式作用元件及多种转录因子结合位点。比如,控制光反应的相关元件ACE、I-box、Box4、G-Box和TCT-motif等[30];参与干旱诱导的MBS元件[31];参与低温应答的LTR元件[32];参与MeJA响应的CGTCA-motif和TGACG-motif;参与ABA相关的ABRE调控元件;参与GA响应的GARE-motif和P-box元件[31];参与SA和IAA响应的TCA-element[33];参与乙烯响应的ERE元件[32];与厌氧诱导调控相关的ARE元件[31];与机械损伤相关的WUN-motif元件[34];以及分生组织表达相关元件CAT-box和参与多种胁迫应答的MYC元件和W-box等[35](图3)。此结果暗示,烟草的碳酸酐酶基因可能广泛参与了烟草的生长发育过程及外界胁迫响应。
2.6 基因表达模式分析
基因表达数据的结果显示(图4),总体上看,NtCA15、NtCA14和NtCA12在所有组织及所有时间点的表达水平均较高;而NtCA2、NtCA3、NtCA5、NtCA8及NtCA9的表达水平均较低。在α-CA亚家族中,NtCA1和NtCA6在18 h内不同组织中均有表达,在根部表达量较其他组织高;而NtCA4和NtCA7也主要在根部表达,在其他组织中表达量较低。在β-CA成员中,虽然各基因在不同组织、不同时间段均有表达,但其表达水平却存在差异,比如NtCA15在芽和茎端的表达量显著高于根部,NtCA10在根部12 h的表达量显著高于0 h。本结果表明,烟草CA基因的表达具有组织及时空特异性,也更进一步反映出烟草CA功能的多样性与广泛性。
3 讨 论
本研究通过生物信息学方法,从烟草基因组数据库中,检索出了分属α及β碳酸酐酶亚家族的15个基因。同源性分析显示,普通烟草α及β-CA亚家族基因,相对于水稻,与拟南芥相应基因的同源性更高。此外,从烟草碳酸酐酶编码蛋白的氨基酸序列相似度看,α及β亚家族之间的相似度较低,而亚家族内相对较高,也同样表明在进化上亚家族内基因的变异要小于亚家族间。在基因结构方面,烟草碳酸酐酶α亚家族基因的外显子个数在6~7,而β亚家族大多数在8~10,这与水稻、玉米和拟南芥的CA家族基因结构类似[36]。同时,Motif分析结果显示,烟草碳酸酐酶α亚家族基因均含有Motif1、Motif2和Motif5。这些Motif中都存在3个与锌离子配位的组氨酸和形成活性位点相关的保守氨基酸残基;而β亞家族基因则均含有Motif3、Motif4和Motif6,该保守结构域包含了两个半膀氨酸和一个组氨酸残基及β类碳酸酐酶所特有的保守性基序C-[SA]-D-S-R-[LIVM]-x-[AP][7-8]。此结果一方面表明了植物碳酸酐酶基因在进化上的保守性,另一方面也表明了其基因结构的多样性与复杂性。而这种结构上的多样性与复杂性,为植物碳酸酐酶超家族基因的功能多样性奠定了基础。
基因的启动子区域包含有不同的顺式作用元件。这些元件的种类与数量,在很大程度上影响基因的转录调控,从而使其在不同生理过程中行使着不同的功能。在本研究中,烟草15个CA基因的启动子区域,均鉴定出了多个与光反应相关的调控元件,暗示着烟草的CA基因可能参与了光合作用。此外,还鉴定出了一些响应环境胁迫相关的元件以及参与激素响应的顺式作用元件。本结果表明,普通烟草的碳酸酐酶基因可能广泛参与了烟草的生长发育与胁迫响应过程。
基因在植物不同组织中的表达水平,反映了其主要发挥功能的场所。本研究通过烟草转录组数据,分析了烟草15个CA基因18 h之内在不同组织中的表达量。与拟南芥的研究结果相类似[8],烟草的CA基因在不同组织中表达水平不一。其中,属于β亚家族的NtCA12、NtCA14和NtCA15这3个基因在所有组织中的表达均较强,暗示着其在烟草碳酸酐酶基因中的重要地位。与此相对应的是属于α亚家族的NtCA2、NtCA3、NtCA5、NtCA8及NtCA9,它们在所有组织中的表达水平均较低。
对普通烟草碳酸酐酶家族基因的鉴定与分析,只是其功能鉴定的第一步。其在烟草生长发育及逆境响应中发挥作用的分子机制,有待于更进一步的深入研究。
4 结 论
在普通烟草中检索出属于碳酸酐酶家族的9个α-亚家族与6个β-亚家族成员。所有成员均分别含有α-、β-亚家族功能结构域,其启动子中包含多个参与光、激素和胁迫响应相关的顺式作用元件。烟草碳酸酐酶基因的表达具有组织特异性,并可能广泛参与了烟草的生长发育及逆境胁迫响应。本研究结果为烟草碳酸酐酶基因的功能研究奠定了基础。烟草碳酸酐酶基因在光合作用以及非生物胁迫响应中的功能与作用机制,则成为下一步研究的重点。
参考文献
[1] MELDRUM N U, ROUGHTON F J. Carbonic anhydrase its preparation and properties[J]. Journal of Physiology, 1933, 80(2): 113.
[2] MAFRA D, COZZOLINO S M F. Erythrocyte zinc and carbonic anhydrase levels in nondialyzed chronic kidney disease patients[J]. Clinical Biochemistry, 2004, 37(1): 67-71.
[3] SMITH K S, FERRY J G. Prokaryotic carbonic anhydrases[J]. Fems Microbiology Reviews, 2000, 24(4): 335-366.
[4] MORONEY J V, BARTLETT S G. Carbonic anhydrases in plants and algae[J]. Plant Cell & Environment, 2001, 24(2): 141-153.
[5] 郭敏亮,高煜珠. 植物的碳酸酐酶[J]. 植物生理学报,1989(3):75-80.
GUO M L, GAO Y Z. Plants of Carbonic anhydrase[J].
Plant Physiology Communications, 1989(3): 75-80.
[6] KIMBER M S, PAI E F. The active site architecture of Pisum sativum beta-carbonic anhydrase is a mirror image of that of alpha-carbonic anhydrases[J]. Embo Journal, 2014, 19(7): 1407-1418.
[7] 何宣,王白羽,張晓磊,等. 小黑麦碳酸酐酶蛋白质三维结构预测[J]. 生物技术通报,2012(2):151-158.
HE X, WANG B Y, ZHANG X L, et al. Structure prediction of carbonic anhydrase from triticale (triticosecale wittmack)[J]. Biotechnology Bulletin, 2012(2): 151-158.
[8] FABRE N, REITER I N, GENTY B, et al. Characterization and expression analysis of genes encoding alpha and beta carbonic anhydrases in Arabidopsis[J]. Plant Cell & Environment, 2010, 30(5): 617-629.
[9] PERALES M, PARISI G, FOMASARI M S, et al. Gamma carbonic anhydrase like complex interact with plant mitochondrial complex I[J]. Plant Molecular Biology, 2004, 56(6): 947-957.
[10] MUTHUSAMY S K, LENKA S K, KATIYAR A, et al. Genome-wide identification and analysis of biotic and abiotic stress regulation of C4 photosynthetic pathway genes in rice[J]. Applied Biochemistry & Biotechnology, 2018, 187(1): 221-238.
[11] WILLIAMS B P, AUBRY S, HIBBERD J M. Molecular evolution of genes recruited into C4 photosynthesis[J]. Trends in Plant Science, 2012, 17(4): 213-220.
[12] 曲贊霜,钱婷婷,侯聪,等. 小黑杨碳酸酐酶家族基因表达特性分析[J]. 北京林业大学学报,2015,37(2):94-99.
QU Z S, QIAN T T, HOU C, et al. Expression analysis of carbonic anhydrase gene family in Populus simonii×Pnigra[J]. Journal of Beijing Forestry University, 2015, 37(2): 94-99.
[13] RAZZAK M A, LEE J M, LEE D W, et al. Expression of seven carbonic anhydrases in red alga Gracilariopsis chorda and their subcellular localization in a heterologous system, Arabidopsis thaliana[J]. Plant Cell Reports, 2019, 38(2): 147-159.
[14] ASPATWAR A, HAAPANEN S, PARKKILA S. An update on the metabolic roles of carbonic anhydrases in the model alga chlamydomonas reinhardtii[J]. Metabolites, 2018, 8(1): 22.
[15] NO?LKE G, BARSOUM M, HOUDELET M, et al. The integration of algal carbon concentration mechanism components into tobacco chloroplasts increases photosynthetic efficiency and biomass[J]. Biotechnology Journal, 2018, 14(3): e1800170.
[16] CHEN T, WU H, WU J, et al. Absence of OsβCA1 causes CO2 deficit and affects leaf photosynthesis and stomatal response to CO2 in rice[J]. Plant Journal for Cell & Molecular Biology, 2017, 90(2): 344.
[17] IVANOV B N, IGNATOVA L K A K. Diversity in forms and functions of carbonic anhydrase in terrestrial higher plants[J]. Russian Journal of Plant Physiology, 2007, 54(2): 143-162.
[18] KOLBE A R, BRUTNELL T P, COUSINS A B, et al. Carbonic anhydrase mutants in zea mays have altered stomatal responses to environmental signals[J]. Plant Physiology, 2018, 177(3): 980-989.
REN A Y, KONG Y Z. Genome-wide identification and expression pattern analysis of the ERF gene subfamily in Nicotiana tabacum[J]. Chinese Tobacco Science, 2017, 38(1): 15-22.
[29] MAGALI L, PATRICE D, GERT T, et al. PlantCARE, a
database of plant cis-acting regulatory elements and a
portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research, 2002, 30(1): 325-327.
[30] 李英华,王阔,郑艳红,等. 大豆GmGBP1基因启动子的光周期响应元件TCT-motif功能分析[J]. 中国油料作物学报,2018,40(4):592-596.
LI Y H, WANG K, ZHENG Y H, et al. TCT-motif function of photoperiod response element of soybean GmGBP1 gene promoter[J]. Chinese Journal of Oil Crop Sciences, 2018, 40(4): 592-596.
[31] 马婧,刘群,王晓斌,等. 2个蜡梅nsLTP基因启动子的克隆及其在烟草中的瞬时表达分析[J]. 植物遗传资源学报,2012,13(4):601-608.
MA J, LIU Q, WANG X B, et al. Cloning and transient expression assay of two nsLTP gene promoters from chimonanthus praecox in tobacco[J]. Journal of Plant Genetic Resources, 2012, 13(4): 601-608.
[32] 于兰芳,周硕,张双喜,等. 葡萄F-box基因VvF-box5的基因结构与表达分析[J]. 植物遗传资源学报,2018,19(2):361-369.
YU L F, ZHOU S, ZHANG S X, et al. Gene structure and expression analysis of F-box gene VvF-box5 in grapevine(Vitis vinifera L.)[J]. Journal of Plant Genetic Resources, 2018, 19(2): 361-369.
[33] 范貝,刘明晓,李慧杰,等. 拟南芥AAP基因家族的生物信息学分析[J]. 生命的化学,2016,36(3):372-378.
FAN B, LIU M X, LI H J, et al. Bioinformatics analysis of AAP gene family in Arabidopsis thaliana[J]. Chemistry of Life, 2016, 36(3): 372-378.
[34] 郑晓瑜,郭晋艳,张毅,等. 植物非生物胁迫诱导启动子顺式作用元件的研究方法[J]. 植物生理学报,2011,47(2):129-135.
ZHENG X Y, GUO J Y, ZHANG Y, et al. Research methods of cis-acting elements in plant abiotic stress inducible promoters[J]. Plant Physiology Communications, 2011, 47(2): 129-135.
[35] 杨献光,梁卫红,齐志广,等. 植物非生物胁迫应答的分子机制[J]. 麦类作物学报,2006(6):158-161.
YANG X G, LIANG W H, QI Z G, et al. Molecular mechanisms of plant responses to abiotic stresses[J]. Journal of Troticeae Crops, 2006(6): 158-161.
[36] 臧丹丹,刘文进,王玉成. 柽柳ThERF1基因上游调控因子W-box互作蛋白[J]. 东北林业大学学报,2014,42(2):120-123,132.
ZANG D D, LIU W J, WANG Y C. Proteins interacting with up-stream regulators of w-box in an ERF gene (ThERF1) from Tamarix hispida[J]. Journal of Northeast Forestry University, 2014, 42(2): 120-123, 132.
[37] TEMS U, BUMELL J N. Characterization and expression of the maize β-carbonic anhydrase gene repeat regions[J]. Plant Physiology & Biochemistry, 2010, 48(12): 945-951.
