山西与内蒙产蒙古黄芪的ISSR体系优化及遗传多样性分析
2019-12-05王丹丹王倩玉陈彤垚张福生秦雪梅
崔 洁,王丹丹,王倩玉,陈彤垚,张福生*,秦雪梅*
1山西大学中医药现代研究中心;2山西大学化学化工学院;3山西大学地产中药功效物质研究与利用山西省重点实验室,太原 030006;4石家庄四药有限公司,石家庄 050021
黄芪为传统大宗中药材之一,豆科植物蒙古黄芪Astragalusmembranaceus(Fisch.) Bge.var.mongholicus(Bge.)Hsiao或膜荚黄芪Astragalusmembranaceus(Fisch.)Bge.的干燥根,具有补气升阳、固表止汗、利水消肿、生津养血、行滞通痹、托毒排脓、敛疮生肌之功效[1],素有“十药八芪”之称。黄芪为多基原植物,目前市场上的流通商品药材主要为蒙古黄芪的栽培品,且栽培品主要来源于山西、甘肃、内蒙古等地,其中,山西、内蒙古主要为种子直播,多采取仿野生的生长方式,而甘肃则为育苗移栽的生长方式。目前,黄芪的栽培品存在以下几个问题:首先,黄芪的种子来源不一,人为过度干预造成种质退化;其次,生长方式不同使得黄芪的外观性状[2-4]与内在化学成分含量发生了较明显的变化[5-7],而这些又可能会对临床上的药效强弱产生一定影响。因此,我们需要对黄芪的遗传变异进行分析,便于对黄芪的种质进行复壮,并对生长方式进行考察,以期达到规范栽培管理。本文以山西与内蒙古的8批蒙古黄芪为例,优化了蒙古黄芪ISSR反应体系;同时对传统道地产区的山西与内蒙古产蒙古黄芪进行了种质遗传多样性与遗传距离的分析,为黄芪种质资源的保护及蒙古黄芪优良品种的挖掘奠定基础。
简单重复序列间扩增(inter-simple sequence repeat,ISSR)分子标记是在1994年建立的一种分子标记技术,可用于种质资源遗传多样性分析、亲缘关系、品种鉴定与品种选育等[8],如Tang等[9]菊科的40份种质进行了ISSR遗传多样性研究;Wang等[10]运用ISSR分子标记结合相关分析技术,设计了快速预测远志药材样品中tenuifoliside C含量高低的特异性引物;Yang等[11]根据psbA/trnH基因间区域的核酸序列与ISSR扩增结果,设计了SCAR标记的引物15F/15R,其可用于鉴别韩国黄芪以及从中国进口的黄芪。
Zhang[12]优化了影响黄芪ISSR体系的以下因素:DNA用量、dNTPs浓度、Taq酶用量、引物浓度。本实验不仅对以上因素进行了优化,还对激活Taq酶和影响退火温度的Mg2+浓度,PCR扩增的循环次数进行了优化,同时在此优化的体系下,对8批蒙古黄芪进行了遗传多样性分析。以期为蒙古黄芪种质资源的评价提供技术支持,促进蒙古黄芪种质保护及良种选育等工作的进一步开展。
1 仪器与试剂
1.1 仪器
TC-XP-D基因扩增仪(杭州博日科技有限公司),DYY-6B型稳压稳流电泳仪(北京市六一仪器厂),ZF-258全自动凝胶成像分析系统(上海嘉鹏科技有限公司),TGL-16高速台式冷冻离心机(湘仪离心机),XW-80A旋涡混合器(上海精科实业有限公司),NanoDrop 2000 超微量分光光度计(Thermo公司),移液器(Eppendorf)。
1.2 试剂
Biowest琼脂糖、β-巯基乙醇均购于太原灏洋生物科技有限公司;GoldView I型核酸染色剂,Solarbio公司;DL 2000 DNA Marker、6×Loading Buffer、PCR试剂盒,TaKaRa公司;CTAB植物基因组DNA快速提取试剂盒(货号:DN14,50次),北京艾德莱生物科技有限公司;实验所用到的引物根据大不列颠哥伦比亚大学(UBC)公布的100对ISSR引物序列进行引物合成,由大连宝生物(TaKaRa)公司合成;三氯甲烷、异戊醇、无水乙醇、硼酸等试剂,国产分析纯。
1.3 药材样本采集
本实验所用黄芪样本,主要采集于山西省以及内蒙古自治区,经山西大学中医药现代研究中心的秦雪梅教授鉴定为蒙古黄芪(Astragalusmembranaceus(Fisch.) Bge.var.mongholicus(Bge.) Hsiao),存放于山西大学中医药现代研究中心标本室。详细信息见表1。

表1 黄芪样品信息表Table 1 Information of A.membranaceus sample source
续表1

编号No.采集地Collection place简称Abbreviation生长方式Growth mode6山西省忻州市五寨县丘陵坡地山西五寨丘陵坡地半野生7山西省忻州市五寨县山西五寨栽培(平地移栽)8山西省大同市浑源县官儿乡木沟村麻黄沟山西浑源官儿乡野生
2 方法
2.1 基因组DNA的提取与检测
依据CTAB植物基因组DNA快速提取试剂盒的操作步骤进行实验,提取所有黄芪样本的DNA,并用1.5 %的琼脂糖凝胶电泳检测提取DNA的完整性,同时用NanoDrop 2000 超微量分光光度计检测其浓度和纯度。
2.2 ISSR-PCR初始反应条件及扩增程序
参考相关文献[12-17],确定黄芪ISSR-PCR初始反应条件为:总体积20 μL,包含10×PCR Buffer 2 μL、2.5 mmol/L MgCl2、0.4 mmol/L dNTPs、0.4 μmol/L引物、1.5 UTaqDNA聚合酶、DNA模板约20 ng,以ddH2O补齐体积至20 μL。反应程序为:94 ℃充分变性5 min,94 ℃变性45 s,50 ℃退火60 s,72 ℃延伸90 s,30个循环后,最后72 ℃延伸10 min。反应结束后,取PCR反应液5 μL,加2 μL的6×Loading Buffer,在1.5 %琼脂糖凝胶上电泳,电压105 V,电泳30~40 min,在凝胶成像仪上观察并照相记录。
2.3 ISSR-PCR反应体系优化设计
以2号黄芪的基因组DNA为模板,用ISSR-PCR初始反应体系及扩增程序对引物UBC825进行扩增,结果出现多态性条带,但条带不够清晰,甚至出现拖尾及非特异性扩增的条带,因此,在此初始反应体系的基础上采用单因素实验设计,依据①dNTP→②引物→③MgCl2→④Taq酶用量→⑤DNA用量→⑥退火温度→⑦循环次数的顺序对影响黄芪ISSR-PCR反应的各个因素分别设置了不同水平的处理,具体实验过程如下所示:(1)dNTPs:0.2、0.4、0.47、0.54、0.6 mmol/L;(2)引物:0.2、0.25、0.3、0.35、0.4、0.6 μmol/L;(3)MgCl2:1.2、2.4、3.0、3.5、4.0 mmol/L;(4)Taq酶:1.0、1.5、2.0 U;(5)DNA:20、40、50、60、70、80 ng;(6)退火温度:根据Tm=4(G+C)+2(A+T)确定该值,以该值为基础上下浮动6 ℃,确定出退火温度的范围,在PCR仪上进行梯度退火筛选;(7)循环次数:25、28、30次。
2.4 ISSR-PCR引物的筛选
参照大不列颠哥伦比亚大学(University of British Columbia,UBC)所设计的ISSR引物序列,将8批样品的DNA等质量混合后进行引物的筛选以及退火温度的优化,最终确定14条扩增重复性好、条带清晰的引物,用于全部样本的扩增。
2.5 8批黄芪样品的遗传一致度及遗传距离分析
遗传一致度(I)反映的是两个样品亲缘关系的远近程度,亲缘关系越接近,种群在所有位点的等位基因就越多,遗传一致度就越接近1,反之,遗传一致度就越接近0。遗传距离(D)反映的是两个样品的遗传变异程度,遗传变异越大,遗传距离越接近1,反之,遗传距离越接近0。当I=1,D=0时,表示两样品的所有等位基因位点完全一致;当I=0,D=∞时,表示两样品的等位基因位点完全不一致。本研究所采用的遗传一致度I与遗传距离D均由软件POPGENE version 1.32采用Nei的标准计算方法而得。
3 结果与分析
3.1 ISSR-PCR反应体系优化的结果
3.1.1 dNTPs浓度的优化结果
本实验先对dNTPs设置了3个浓度,分别为0.2、0.4、0.6 mmol/L,见图1A。dNTPs在低浓度(0.2 mmol/L)的情况下,扩增产率低且条带模糊;dNTPs浓度为0.4和0.6 mmol/L时条带较清晰;所以在0.4-0.6 mmol/L之间设置了梯度进行进一步细化,细化浓度分别为0.4、0.47、0.54、0.6 mmol/L,见图1B。结果发现在0.54 mmol/L时条带最为清晰,之后将其浓度设置为0.5 mmol/L时也能达到0.54 mmol/L相同的效果,本着节约的原则,所以dNTPs的浓度最终确定为0.5 mmol/L。
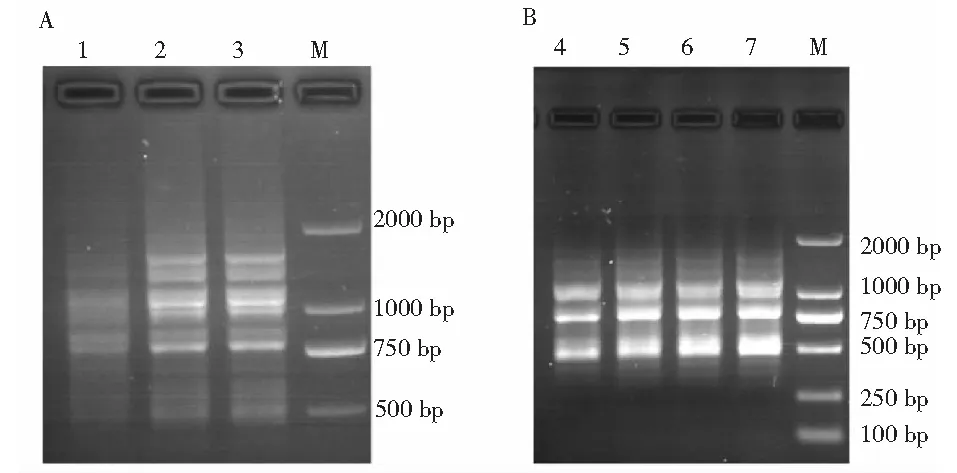
图1 不同浓度dNTPs的ISSR-PCR扩增电泳图Fig.1 Electrophoresis of ISSR-PCR with different concentrations of dNTPs 注:1~3 dNTPs的浓度分别为0.2、0.4、0.6 mmol/L;4~7 dNTPs的浓度分别为0.4、0.47、0.54、0.6 mmol/L;M:DL 2000 DNA Marker。Note:The concentrations of 1-3 dNTPs were 0.2、0.4、0.6 mmol/L,respectively;the concentrations of 4-7 dNTPs were 0.4、 0.47、0.54、0.6 mmol/L,respectively.M:DL 2000 DNA marker.
3.1.2 引物浓度的筛选结果
本实验对引物浓度先设置了3个梯度,分别为0.2、0.4、0.6 μmol/L,见图2A。PCR结果发现在0.2、0.4 μmol/L时条带差不多且清晰,高浓度(0.6 μmol/L)时条带变模糊。所以在0.2、0.4 μmol/L之间设置了不同浓度进行细化,细化浓度分别为0.2、0.25、0.3、0.35、0.4 μmol/L,见图2B。结果发现引物浓度0.25 μmol/L时条带清晰且扩增稳定。所以引物最终浓度为0.25 μmol/L。
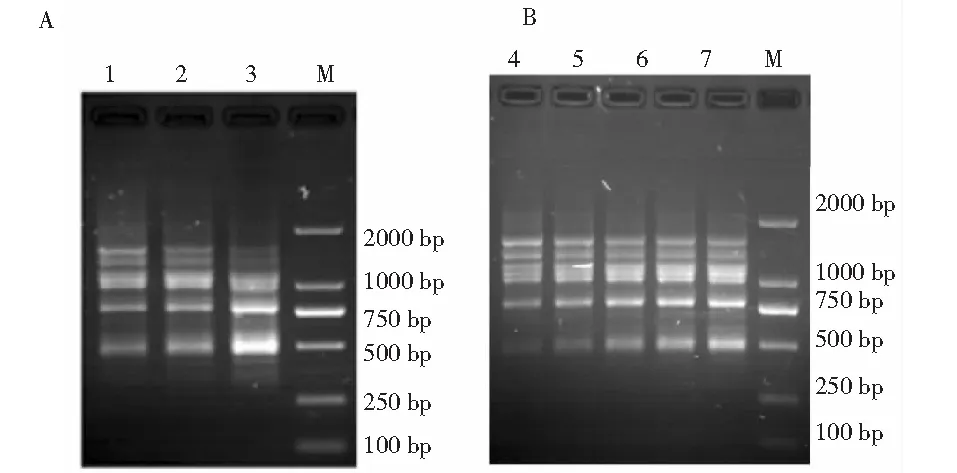
图2 引物(UBC825)不同浓度的ISSR-PCR电泳图Fig.2 Electrophoresis of ISSR-PCR with different concentrations of primer UBC825 注:1~3 引物浓度分别为0.2、0.4、0.6 μmol/L;4~8 引物浓度分别为0.2、0.25、0.3、0.35、0.4 μmol/L;M:DL 2000 DNA marker。Note:1-3 primer concentrations were 0.2、0.4、0.6 μmol/L;4-8 primer concentrations were 0.2、0.25、0.3、0.35、0.4 μmol/L, respectively.M:DL 2000 DNA marker.
3.1.3 MgCl2浓度的优化结果
本实验对MgCl2设置了3个浓度,分别为1.2、2.4、3.6 mmol/L,见图3A。结果发现,当MgCl2浓度为低浓度(1.2 mmol/L)时PCR产物条带少且模糊,当浓度为2.4 mmol/L时条带比1.2 mmol/L清晰;当MgCl2浓度为3.6 mmol/L时PCR扩增条带多且较清晰。所以接下来在2.4-3.6 mmol/L之间分别设置了以下浓度:2.5、3.0、3.5、4.0 mmol/L,结果如图3B。当MgCl2浓度为高浓度(4.0 mmol/L)时,条带变模糊;MgCl2浓度为2.5 mmol/L时条带数少且不清晰;3.5 mmol/L时条带清晰度比3.0 mmol/L时的好,所以选择3.5 mmol/L作为MgCl2的终浓度。

图3 不同MgCl2 浓度的ISSR-PCR 扩增结果琼脂糖凝胶电泳图Fig.3 Agarose gel electrophoresis map optimized for the concentration of different MgCl2 注:1~3 MgCl2浓度分别为1.2、2.4、3.6 mmol/L;4~7 MgCl2浓度分别为:2.5、3.0、3.5、4.0 mmol/L;M:DL 2000 DNA marker。Note:1-3 The concentrations of MgCl2 were 1.2、2.4、3.6 mmol/L,respectively;the concentrations of 4-7 MgCl2 were 2.5、3.0、3.5、4.0 mmol/L,respectively.M:DL 2000 DNA marker.
3.1.4Taq酶用量的优化结果
在确定了dNTPs,引物,MgCl2浓度的情况下,对Taq酶量进行了考察,设置浓度为1.0、1.5、2.0 U,结果如图4所示,1.0、1.5、2.0 UTaq酶条带相似,但出于经济考虑最终确定Taq酶量为1.0 U。

图4 不同用量Taq酶的 ISSR-PCR 扩增电泳图Fig.4 Electrophoresis of ISSR-PCR with different amounts of Taq polymerase 注:1~3 Taq酶用量分别为1.0、1.5、2.0 U;M:DL 2000 DNA marker。Note:1-3 Taq enzyme amounts were 1.0、1.5、2.0 U, respective-ly.M:DL 2000 DNA marker.
3.1.5 DNA模板量的优化结果
DNA加入量分别为20、40、60 ng,如图5A所示。结果发现,DNA用量在20 ng是扩增条带模糊;当DNA用量为40和60 ng时条带较清晰,所以在40、60 ng附近进行DNA细化,设置浓度为40、50、60、70、80 ng,结果如图5B。在20 μL体系中,50~80 ng扩增出的条带最为清晰,基于相同条件下模板量少原则,最终选择DNA模板量为50 ng。

图5 不同用量DNA模板的ISSR-PCR扩增结果Fig.5 Electrophoresis of ISSR-PCR with different amounts of DNA template 注:1~3 DNA模板用量分别为20、40、60 ng;4~8 DNA模板用量分别为40、50、60、70、80 ng;M:DL 2000 DNA marker。Note:1-3 DNA template amounts is 20、40、60 ng;4-8 DNA template amounts is 40、50、60、70、80 ng.M:DL 2000 DNA marker.
3.1.6 ISSR-PCR退火温度的筛选结果
以引物UBC825进行梯度退火温度筛选为例,其Tm=4(G+C)+2(A+T),为50 ℃,设置中心温度为51 ℃,梯度宽度为6 ℃,选择47.6、50.5、53.2、55.8 ℃进行PCR扩增,结果如图6。由图6可知,引物UBC825在47.6和50.5 ℃扩增条带模糊,53.2 ℃扩增条带清晰,55.8 ℃条带变模糊,所以UBC825退火温度最终选择53.2 ℃。

图6 UBC825引物不同退火温度下的ISSR-PCR电泳图Fig.6 Electrophoresis of ISSR-PCR with different concentrations of Primer UBC825 注:1~4退火温度分别为47.6、50.5、53.2、55.8 ℃;M:DL 2000 DNA marker。Note:1-4 Annealing temperatures are 47.6、50.5、 53.2、55.8 °C.M:DL 2000 DNA marker.
3.1.7 ISSR-PCR循环次数的筛选结果
如图7所示,本实验分别进行了25、28、30次的PCR扩增,结果循环次数为25次时扩增产物条带模糊,28次,30次循环时,产物条带基本相同,为节约时间,最终循环次数选择28次。
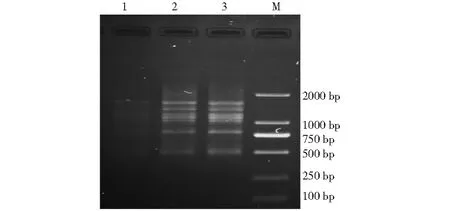
图7 不同循环次数下的ISSR-PCR扩增电泳图Fig.7 Electrophoresis of ISSR-PCR with different cycles 注:1~3 ISSR-PCR循环次数分别为25、28、30次;M:DL 2000 DNA marker。Note:1-3 ISSR-PCR cycle times are 25、28、30 times; M:DL 2000 DNA marker.
最终确定反应体系为:总体积20 μL,包含10×PCR Buffer 2 μL、0.5 mmol/L dNTPs、1.0 UTaq酶、3.5 mmol/L MgCl2、0.25 μmol/L引物、50 ng DNA,加ddH2O水补足至20 μL。反应程序为:94 ℃充分变性5 min,94 ℃变性45 s,53 ℃退火60 s,72 ℃延伸90 s,28个循环后,最后72 ℃延伸7 min。
3.2 优化体系对样本的扩增结果
运用优化好的体系对8批蒙古黄芪样本进行ISSR-PCR扩增,图8为部分引物在优化好的体系下的扩增结果。由此可以看出本实验建立的黄芪ISSR-PCR体系稳定,可靠。

图8 部分引物的ISSR-PCR扩增结果图Fig.8 ISSR-PCR amplification results of some primers

图9 引物UBC825对8批蒙古黄芪ISSR-RCR扩增结果图Fig.9 Amplification results of primers UBC825 on 8 batches of A.membranaceus ISSR-RCR
3.3 引物筛选扩增结果
将琼脂糖凝胶电泳图导入ZF-258全自动凝胶成像分析系统,对其进行数据统计,记录扩增条带的多态性。根据DNA条带的有无进行区分标记,对条带清晰的记为“1”,对缺失或模糊不清的标记为“0”,对数据进行统计得到01矩阵。将统计结果01矩阵按照POPGENE 1.32 软件的格式要求修改,导入数据,进行多态性条带百分率计算,以及遗传一致性与遗传距离分析。结果如表2。筛选的14条引物对8批蒙古黄芪样本进行扩增,共扩增出126条带,其中多态性条数118条,多态性位点百分数为93.65%,平均每个引物扩出8.43个DNA片段,其多态性为93.6%,说明黄芪遗传多样性丰富。
3.4 遗传相似系数与聚类结果分析
分别用14条引物对8批蒙古黄芪样本进行扩增,扩增结果用软件POPGENE version 1.32采用Nei的标准计算方法(Nei,1978)计算遗传一致度I与遗传距离D,所得结果见表3。遗传一致度I与遗传距离D均是对样品间遗传分化程度的描述。由表3可以看出,8批黄芪样品的遗传一致度在0.492 1~0.730 2之间,遗传距离在0.314 5~0.709 1之间,说明黄芪样品的遗传分化程度较大。其中,2号内蒙古南洼村与6号山西五寨丘陵坡地样品的亲缘关系最近,遗传一致度为0.730 2,遗传距离为0.314 5;1号内蒙古兴和县与4号山西浑源样品的亲缘关系最远,遗传一致度均为0.492 1,遗传距离均为0.709 1。山西不同产地的遗传距离变幅为0.347 6~0.559 6,差异比较明显。

表2 黄芪ISSR扩增反应的引物及扩增的条带数Table 2 Primers and amplified bands of ISSR amplification reaction
注:B=(T,C,G); H=(A,T,C); R=(A,G); Y=(C,T); V=(A,C,T);D=(A,G,T)。
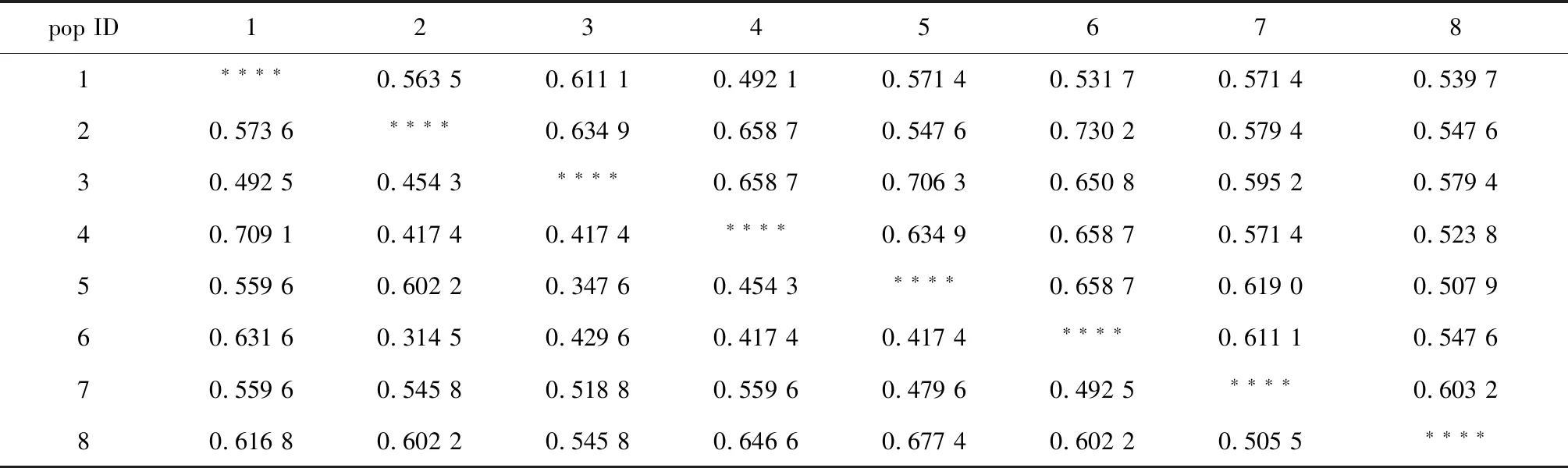
表3 8批黄芪样品的遗传一致度与遗传距离Table 3 Genetic consistency and genetic distance of 8 samples of A.membranaceus
3.5 8批蒙古黄芪样品的多态性条带聚类分析
利用NTSYS-pc 2.1软件,通过非加权类平均法(unweighted pair-group method arithmetic averages,UPGMA),对8批蒙古黄芪样品的多态性条带进行聚类分析,并构建树状聚类图(见图10)。由图可知,当匹配系数为0.50时,黄芪样品明显分为三类。其中,1号内蒙古兴和县跟8号山西浑源官儿乡样品分别单独聚为一类,2号内蒙古南洼村、6号山西五寨丘陵坡地、3号、4号山西浑源、5号山西五寨芦芽山、7号山西五寨聚为一类。2号内蒙古南洼村与6号山西五寨丘陵坡地样品间的遗传距离最近,二者亲缘关系最近。而1号内蒙古兴和县与8号山西浑源官儿乡的距离最远,说明二者亲缘关系较远。通过比较发现,UPGMA聚类结果与遗传一致度与遗传距离的分析结果(表3)基本是一致的。
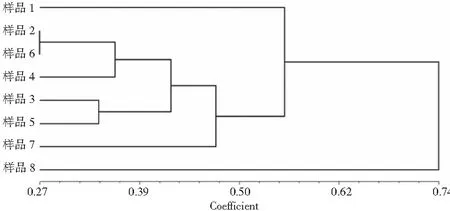
图10 8批蒙古黄芪样品的UPGMA聚类图Fig.10 UPGMA clustering map of 8 samples of A.membranaceus
4 讨论
本研究采用单因素实验对影响蒙古黄芪的ISSR反应体系的因素进行了优化,并对山西与内蒙古2个产地蒙古黄芪的遗传多样性进行了分析。而目前,植物基因多态性的研究多采用DNA分子标记,DNA间的差异可以通过分子标记呈现。DNA分子标记技术有多种,其中ISSR技术具有操作简单、多态性和重复性好等特点,但是ISSR反应受到PCR反应中各因素的影响,不同的样本需要建立不同的ISSR反应体系[18]。因此,本研究对影响ISSR-PCR反应的dNTPs、引物、MgCl2、Taq酶、DNA、退火温度进行了优化。其中dNTPs、MgCl2、Taq酶、引物浓度对ISSR-PCR扩增影响较大,另外,退火温度会影响模板与引物的结合,退火温度低容易发生错配,所以适当提高退火温度会降低错配率,但太高又会影响复性,所以选择合适的退火温度是必要的。
通过对蒙古黄芪的聚类分析发现,虽然1号和2号蒙古黄芪样本均来自内蒙古兴和县,且二者地理位置较近,但在聚类图上却分为了2大类,这可能与它们生长的小环境有较大差异有关,使得它们逐渐适应了当地的气温、海拔高度、降水等,这与Wang等[19]对长白山地区安图与和龙的聚类分析是相似的。2号内蒙古南洼村与山西产地的3-7号蒙古黄芪样本虽然地理位置较远,但是聚为了一类,可能是产地之间相互引种造成了基因交流,使得遗传背景一致,这与Zhang[20]对不同产地蒙古黄芪聚类分析一致。聚类分析中2号内蒙古南洼村野生品、6号山西五寨丘陵地半野生品与4号山西浑源野生品先聚在了一起,而后与山西其他产地栽培品聚为一类,这说明栽培品与野生品之间存在较高的遗传相似度,体现出它们亲缘关系更为接近,这可能与它们生长气候、降水、光照等有关,这与Zhang[12]对6个蒙古黄芪居群的聚类结果“先以生存方式野生、栽培聚类”分析一致;如果后续实验能对不同生长方式蒙古黄芪的化学成分含量进行分析和生长物候条件对蒙古黄芪的影响,以及建立化学成分与分子标记间的联系,这将对蒙古黄芪种植资源的挖掘与保护提供帮助。
本实验仅对山西与内蒙古的8批蒙古黄芪进行了遗传多样性分析,后续会采用本实验建立的ISSR-PCR体系对大规模不同产地蒙古黄芪进行进一步验证,收集更多数据,同时结合样本表型数据对蒙古黄芪进行种质评价;其次,对本次收集样本进行化学成分含量测定,将此次的PCR结果与黄芪化学成分含量建立联系,为蒙古黄芪的种质复壮及挖掘品种潜力提供帮助。
