分子对接技术在昆虫化学感受研究中的应用进展
2019-11-27李敏郭美琪相伟芳
李敏 郭美琪 相伟芳

摘要 分子对接是利用生物信息分析手段预测配体和受体的结构模型,模拟出配体复合物的结构并通过结合自由能来判断结合强度的一种技术。分子对接包括蛋白质和小分子的前期准备,识别结合位点,搜索配体化合物的构象,评估对接结果四部分。昆虫的化学感受途径主要通过化感蛋白与小分子化合物的结合,实现对化学信息素的接收。分子对接可以精确模拟昆虫化感蛋白和信息化合物的结构以及二者的结合形式,可作为研究昆虫化学感受途径的有效技术手段,从而有利于研究开发昆虫抑制剂,用于农林病虫害的防治。本文综述了分子对接技术在昆虫化学感受研究中的应用进展,并详细介绍了分子对接的相关软件,举例说明了分子对接的过程,可为该领域的相关研究提供理论支持和方法指导。
关键词 分子对接; 昆虫; 化学感受基因; 化学信息素; 配体复合物
中图分类号: Q 965 文献标识码: A DOI: 10.16688/j.zwbh.2018464
Abstract Molecular docking is a technique for predicting the structural models of the ligands and receptors, simulating the structures of ligand complexes, and determining the strength of binding via combining free energies by means of bioinformatic analysis. Molecular docking involves the preparation of proteins and small molecules, the recognition of binding sites, the search for the conformation of ligand compounds, and the evaluation of the docking results. The reception of chemical pheromones by insects is mainly achieved by the combination of chemosensory proteins and small molecule compounds. Molecular docking accurately simulates the structures and the combined forms of insect chemosensory proteins and information compounds, which can be applied efficiently to the study of insect chemosensory pathways. This review summarized the research progresses in molecular docking technique in the field of insect chemosensory reception, introduced the related softwares of molecular docking, and illustrated the process of molecular docking, which provides solid theoretical supports and logical methodological guidance for the related research in this field.
Key words molecular docking; insect; chemosensory gene; chemical pheromone; ligand complexes
分子對接技术,即通过构建并优化蛋白质与小分子化合物的三维结构,将小分子匹配到蛋白质的结合位点上,并评估结合力强弱的一种生物分析技术[1]。目前,分子对接应用于医药研发和生物大分子设计等方面,在昆虫化学感受领域中,分子对接主要被应用于两种小分子结合蛋白:气味结合蛋白 (odorantbinding protein, OBP) 和化学感受蛋白(chemosensory protein, CSP) 的功能预测以及与小分子物质的结合中。研究昆虫嗅觉和味觉感受蛋白的结合机制,对农林病虫害的防治,利用天敌防治虫害等工作有着很重要的意义。
1 分子对接的涵义
分子对接的理论最早可以追溯到Fisher 提出的“锁-钥模型”。“锁-钥模型”将配体与受体的结合视为刚性过程,即在结合过程中,配体和受体的三维结构不发生改变。由于刚性对接本身的局限性,1958年Koshland 提出了“诱导契合学说”[2],该学说认为在分子对接过程中,应将受体与配体视为柔性结构,即构象可以在一定范围内发生改变以实现契合。随着分子对接理论的进一步深入,目前在分子对接过程中常用半柔性对接作为对接方法,即在对接过程中,将受体构象视为刚性,而小分子构象可以在一定范围内发生变化,此类对接比较适宜处理大分子与小分子之间的结合,例如蛋白质和配体之间的结合[3]。
2 分子对接的过程
分子对接的过程可以概括为四部分:1)蛋白质和小分子配体的准备,在进行分子对接之前,需在结构数据库中搜索蛋白质和小分子配体的三维结构以进行后续操作。2)结合位点的识别,即在进行对接之前,需要识别蛋白质三维结构中可结合小分子的位点[1]。1994年, Collins 等第一次利用多尺度算法确定了蛋白质表面可结合配体的位点,并成功进行了柔性分子对接,促进了分子对接的发展[4]。3)配体化合物的构象搜索,即在分子对接过程中,由于分子结构具有一定的柔性,配体会相对于受体发生位置及结构的改变,因此,需要搜寻到所有的配体结构以及配体与受体的结合模式[5]。4)评估对接结果,即对构象搜索过程中,搜索到的大量配体复合物结构中配体的位置摆放的合理性和受体、配体结合的亲和性进行评估,从而确定结合力强的配体复合物[6]。
精确识别结合位点,提高构象搜索能力,改善评估功能都有助于增强分子对接结果的准确性[7]。
3 蛋白质受体结构预测的技术进展
目前,X射线衍射,核磁共振已成为解析生物分子结构的两大主要技术。X射线衍射研究常常是利用同步辐射的手段获得蛋白质三维结构,更适合于较大分子量的样品,核磁共振则适用于分子量相对较小的蛋白样品[8-10]。这两种技术经常联合用于蛋白质结构的研究[11-12]。近年来,随着冷冻电镜技术兴起,其逐渐被应用于解析生物大分子结构的研究中。冷冻电镜技术能够将生物大分子的静态结构在原子级分辨率下进行解析[13]。由于生物大分子并不是稳定不变的,常表现出亚稳状态,甚至会表现出连续构象变化的非平衡态,因此冷冻电子显微镜也具备了对生物大分子中的每一个子状态进行动态解析的能力[14]。Joel等利用冷冻电镜技术预测了榕小蜂Apocrypta bakeri的嗅觉系统中高度保守的Orco受体的三维结构[15]。随着技术的发展,对蛋白质结构的了解更加深入,可以对蛋白质的三维构象进行更为准确的预测。
4 分子对接的应用
随着分子对接的进一步发展,目前分子对接主要应用于医药研发以及生物大分子设计等方面,并日渐广泛地应用于昆虫化感基因领域的研究。
4.1 医药研发
利用分子对接可以预测小分子化合物和靶蛋白之间的结合亲和力,因此,分子对接一经出现,就被迅速应用于医药研发领域[16-20]。分子对接技术不仅可以用于筛选化合物,还可用于虚拟筛选预测候选药物的靶蛋白[21-22]。
在癌症的治疗中,微管蛋白抑制剂已被证明是一种消除癌细胞的有效策略[23]。研究人员利用AutoDock 4.2软件,结合分子动力学模拟等相关技术,筛选出了新型微管蛋白抑制剂,对后续的研究提供了具有价值的抑制剂数据集[24]。
4.2 生物大分子设计
利用大分子物质和小分子配体的结合能力,可以设计出所需的生物大分子。目前,分子对接已经被广泛应用于生物催化[25-26]、生物传感器[27]以及生物降解[28]的研究中。
近年来,随着纳米技术的日益发展,纳米复合材料也被广泛应用于生物领域的研究中[29-31]。以蛋白质-聚电解质复合物形式的聚合物为基础可以生产纳米复合材料[32]。研究人员利用AutoDock Vina进行分子对接研究并构建了酶-聚电解质复合物模型,并将该模型应用于有机磷的现代解毒剂的研发中[33]。
4.3 昆虫化学感受领域的应用
随着多个物种基因组及转录组测序完成,昆虫化学感受途径机理的研究也取得了很大的进展[34]。研究昆虫化学感受途径有利于掌握昆虫对信息化合物的接收机制,从而为深入研究昆虫的嗅觉及味觉机制,研究开发害虫抑制剂和利用益虫新技术等提供理论支持[35]。
目前,分子对接技术被广泛应用于化学感受途径中重要的化学感受蛋白OBP和CSP与小分子结合的功能预测中。对于蛋白CSP的研究已经被应用于膜翅目蜜蜂科的中华蜜蜂Apis cerana Fabricius[36-39];半翅目粉虱科的烟粉虱Bemisia tabaci (Gennadius)[40]、蝽科[41]等类群的研究中。对于蛋白OBP的研究已经被应用于膜翅目蜜蜂科的中华蜜蜂Apis cerana[37]; 半翅目飛虱科的褐飞虱Nilaparvata lugens Stl [42]、蝽科[41]、蚜科[43];鳞翅目夜蛾科的斜纹夜蛾Spodoptera litura (Fabricius)[44-46]、螟蛾科的欧洲玉米螟Ostrinia nubilalis(Hübner)[47]、菜蛾科的小菜蛾Plutella xylostella (Linnaeus)[48];直翅目蝗科的东亚飞蝗Locusta migratoria manilensis (Meyen)[49];鞘翅目叶甲科[50]等类群的研究中。
5 分子对接技术常用软件
分子对接技术常用软件列于表1。
6 应用举例
6.1 分子对接前蛋白质和小分子配体准备
6.1.1 蛋白质准备
6.1.1.1 同源建模
常用SWISSMODEL[57],MODELLER 9.9[58]对蛋白质进行同源建模,在线提交蛋白的一级序列,以蛋白模型数据库中同源蛋白的三维结构为模板来构建所需的蛋白模型,一般模型相似度达到30%及以上,便可获得相对比较合理的构象[59]。
6.1.1.2 模型评估
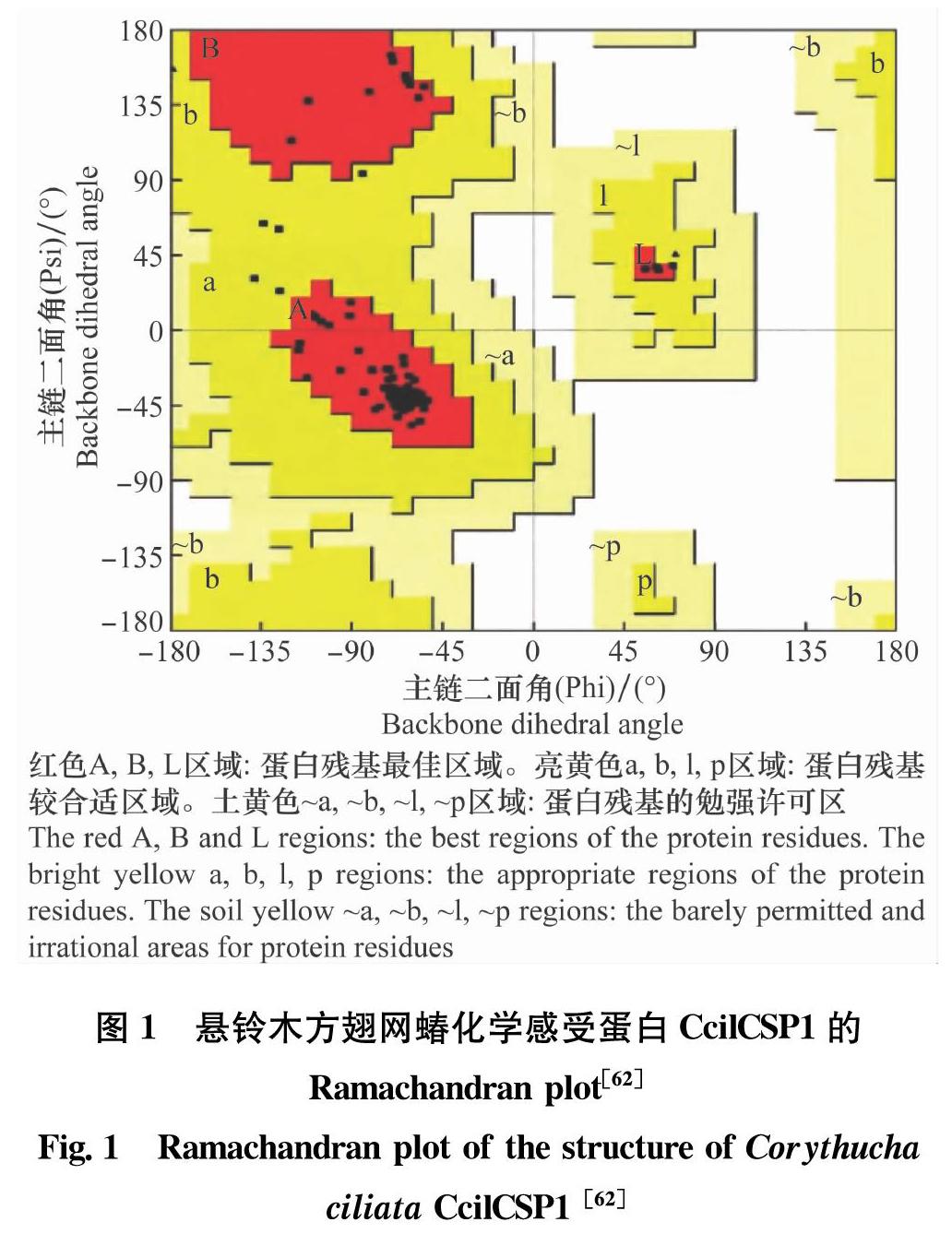
经同源建模得到的蛋白质的三维结构,可通过两种方式进行模型质量评估。1)利用Ramachandran plot(拉氏图)进行评价[60]。拉氏图用于阐述蛋白质或肽立体结构中肽键内α碳原子和羰基碳原子之间的键的旋转度ψ对α碳原子和氮原子之间的键的旋转度φ的关系,主要说明肽类或蛋白质的氨基酸的允许区和不允许区。很多软件都可以生成Ramachandran plot,不同软件生成的Ramachandran plot形式不同。一般落在允许区和最大允许区的氨基酸残基占蛋白总体氨基酸残基的比例高于90%,即可认为同源建模所得模型的构象合理[61]。2)对同源建模所得蛋白质结构模型还可以进行能量评价。利用PROSA(https:∥prosa.services.came.sbg.ac.at/prosa.php),提交蛋白质三维结构的PDB文件,分析后显示评价的结果图像。评价结果会显示出在PDB数据库中所确定的与目标模型大小相似的蛋白结构链的Zscore,并形成一个分布区,若建模所得的模型的Zscore落在分布区内,即代表模型合理。一般Zscore为负值即为合理[52]。
图1为悬铃木方翅网蝽化学感受蛋白CcilCSP1经同源建模获得的蛋白质结构模型的Ramachandran plot[62]。
在Ramachandran plot中,CcilCSP1经同源建模所得蛋白模型的94.7%的氨基酸残基位于最大允许区,有5.3%的氨基酸残基位于较合适区域,基本没有氨基酸残基落在勉强许可区和不合理区。另一方面,用PROSA评价得到Zscore打分为-6.26,落在了较好的结构蛋白的Zscore分布范围。经两种方法评估,CcilCSP1经同源建模获得的模型构象合理[62]。
6.1.2 小分子准备
小分子配体结构的下载:小分子结构可以在pubchem (https:∥pubchem.ncbi.nlm.nih.gov/) 小分子数据库中获取[63]。
6.2 小分子结合位点的识别
将蛋白质三维结构的PDB文件导入Discovery Studio 4.5中,若晶体结构中不含有氢原子,则Chemistry/Hydrogens/Add进行加氢操作。在工具栏中,展开ReceptorLigand Interactions/Define and Edit Binding Site,单击Define Site一栏下的From Receptor Cavities,通过寻找受体蛋白的“空腔”来寻找蛋白中可能结合小分子配体的位置。
6.3 配体复合物的构象搜索
由于Discovery Studio中的对接模块为非开源部分,因此,利用Schrdinger 2015-2软件包内的对接程序进行对接。将蛋白结构导入Schrdinger 2015-2,利用Maestro程序来观察对接过程。Tasks/protein preparation,删除蛋白质“空腔”内存在的水分子和杂原子基团,以保证蛋白受体和小分子配体对接充分,单击Preprocess,蛋白质准备完成。Tasks/Docking/Gride Docking 对蛋白质上可能存在的小分子配体的结合位点进行坐标定义,并生成网格文件,此处可对照Discovery Studio中识别出的“空腔”位置进行定义。Tasks/Ligand preparation/LigPrep 在Schrdinger 2015-2中导入并优化小分子结构,Task/Conformational search /Bioactive search/Standard,搜索小分子化合物可能存在的构象,以在对接过程中存在小分子所有三维构象。Task/Docking/Glide Docking 进行分子对接,搜索出所有可能存在的配体复合物的结构模型。
6.4 对接结果的评估
对接结束后,得到所有配体复合物的对接分数,其中Dockingscore为对接分数,数值越小,代表小分子配体与蛋白质结合程度越强。在没有金属存在的情况下,该数值应与Glidescore相一致。
选中与蛋白受体亲和力最强的小分子配体粘贴到蛋白结构上,小分子自动填充至蛋白“空腔”内。单击ReceptorLigand Interactions工具栏中的View Interactions,定义蛋白质和小分子配体,单击Show receptorligand interactions on a 2D diagram 下的Show 2D Diagram 指令,即在新页面中显示配体-蛋白相互作用的二維平面图,便于更加直观地观察受体和配体之间的相互作用以及关键的氨基酸基团。
7 分子对接技术准确性验证
在利用分子对接技术对昆虫化学感受基因相关的研究中常常运用分子动力学模拟[64],荧光竞争结合分析[45,47]以及体外试验等方法来验证并进一步确认蛋白质和小分子配体的结合位点和结合强度。
为探索昆虫气味结合蛋白和小分子化合物识别过程,Xin等构建了斜纹夜蛾的气味结合蛋白OBP1的三维结构,通过Discovery Studio 2.1进行OBP1与小分子配体的对接,并进行了荧光竞争结合试验,荧光竞争结合分析结果与分子对接结果相同,验证了分子对接技术结果的准确性[45]。
化学感受蛋白CSP在昆虫化学感受途径中起重要作用,为了探究昆虫解毒和防御机制,Liu等以烟粉虱Bemisia tabaci为研究对象,测定编码CSP1、CSP2和CSP3的基因序列,将CSP与可能相关基因的表达联系起来,并试图找到CSP1、CSP2和CSP3与真正的挥发性或非挥发性同源化学配体的相互作用。利用荧光竞争结合分析和分子对接技术进行研究,结果显示CSP1与亚油酸的亲和力较高,而CSP2和CSP3蛋白则与α戊基肉桂醛结合较好。在试验中分子对接和荧光竞争结合分析的结果表现出了极大的一致性[40]。
8 挑战及展望
虽然分子对接技术已经被广泛应用到各种领域,但依旧面临着巨大的挑战。例如:1)许多蛋白结构尚未完全明确,缺乏用于建模的相关模型;2)评分函数过于简单化,不能精确评估蛋白受体和配体间的相互作用。因此,构建更多的蛋白晶体结构,使得同源建模的模型精准度进一步提高,并在对接过程中,开发一种用来准确预测结合亲和力并能够同时筛选数据库中成千上万的分子的方法是亟待解决的任务[65]。
随着大分子结构分析技术的丰富,蛋白质晶体结构的增加以及优化,数据库的进一步扩充,评分函数的进一步优化,分子对接技术也将进一步革新,从而更具有准确性,将被广泛应用于环境保护、虫害防控以及利用昆虫嗅觉机制开发新型抑制剂[66]。
参考文献
[1] S 'LED Z ' P, CAFLISCH A. Protein structurebased drug design: from docking to molecular dynamics [J]. Current Opinion in Structural Biology, 2017, 48:93-102.
[2] KOSHLAND D E. Application of a theory of enzyme specificity to protein synthesis [J]. Proceedings of the National Academy of Sciences of the United States of America, 1958, 44(2): 98-104.
[3] BONVIN A M J J. Flexible proteinprotein docking [J]. Current Opinion in Structural Biology, 2006, 16(2): 194-200.
[4] COLLINS J G, SHIELDS T P, BARTON J K.1HNMR of Rh (NH3)4phi3+ bound to d(TGGCCA)2: classical intercalation by a nonclassical octahedral metallointercalator [J]. Journal of the American Chemical Society, 1994, 116(22): 9840-9845.
[5] SOUSA S F, FERNANDES P A, RAMOS M J. Proteinligand docking: current status and future challenges[J]. Proteins: Structure, Function, and Bioinformatics, 2010, 65(1):15-26.
[6] HUANG Shengyou, ZOU Xiaoqin. Advances and challenges in proteinligand docking [J]. International Journal of Molecular Sciences, 2010, 11(8): 3016-3034.
[7] BAN L, SCALONI A, BRANDAZZA A, et al. Chemosensory proteins of Locusta migratoria[J]. Insect Molecular Biology, 2010, 12(2):125-134.
[8] ROSENZWEIG R, KAY L E. Solution NMR spectroscopy provides an avenue for the study of functionally dynamic molecular machines: the example of protein disaggregation [J]. Journal of the American Chemical Society, 2016,47(20):1466-1477.
[9] KAY L E. New views of functionally dynamic proteins by solution NMR spectroscopy [J]. Journal of Molecular Biology, 2016, 428(2): 323-331.
[10] TUGARINOV V, KANELIS V, KAY L E. Isotope labeling strategies for the study of highmolecularweight proteins by solution NMR spectroscopy [J].Nature Protocols,2006,1(2):749-754.
[11] PERRY J J, TAINER J A. Developing advanced Xray scattering methods combined with crystallography and computation[J]. Methods, 2013, 59(3): 363-371.
[12] GRANT T D, LUFT J R, WOLFLEY J R, et al. Small angle Xray scattering as a complementary tool for highthroughput structural studies [J]. Biopolymers, 2011, 95(8):517-530.
[13] DASHTI A, SCHWANDER P, LANGLOIS R, et al. Trajectories of the ribosome as a Brownian nanomachine [J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(49):17492-17497.
[14] LU Ying, WU Jiayi, DONG Yuanchen, et al. Conformational landscape of the p28bound human proteasome regulatory particle [J]. Molecular Cell, 2017, 67(2): 322.
[15] BUTTERWICK J A, MRMOL J D, KIM K H, et al. CryoEM structure of the insect olfactory receptor Orco [J]. Nature, 2018, 560: 447-452.
[27] SIVASUBRAMANIAN A, MAYNARD J A, GRAY J J. Modeling the structure of mAb 14B7 bound to the anthrax protective antigen [J]. Proteins Structure Function & Bioinformatics, 2010, 70(1): 218-230.
[28] SURESH P S, KUMAR A, KUMAR R, et al. An Insilco approach to bioremediation: Laccase as a case study [J]. Journal of Molecular Graphics & Modeling, 2008, 26(5): 845-849.
[29] BABY T T, ARAVIND S S J, AROCKIADOSS T, et al. Metal decorated graphene nanosheets as immobilization matrix for amperometric glucose biosensor [J]. Sensors and Actuators B: Chemical, 2010, 145(1): 71-77.
[30] MAO Kexia, WU Dan, LI Yan, et al. Labelfree electrochemical immunosensor based on graphene/methylene blue nanocomposite [J].Analytical Biochemistry, 2012, 422(1): 22-27.
[31] SONG Yang, LUO Yanan, ZHU Chengzhou, et al. Recent advances in electrochemical biosensors based on graphene twodimensional nanomaterials [J]. Biosensors & Bioelectronics, 2016, 76:195-212.
[32] BUWALDA S J, VERMONDEN T, HENNINK W E. Hydrogels for therapeutic delivery: current developments and future directions [J]. Biomacromolecules, 2017, 18(2):316-330.
[33] LYAGIN I V, EFREMENKO E N. Biomolecular engineering of biocatalysts hydrolyzing neurotoxic organophosphates[J]. Biochimie, 2017, 144: 115-121.
[34] 柳曉磊, 翁群芳, 任珍珍,等.昆虫化学感受基因家族研究新进展[J]. 江苏农业科学, 2010(5):1-5.
[35] PELOSI P, ZHOU J J, BAN L P, et al. Soluble proteins in insect chemical communication[J]. Cellular & Molecular Life Sciences, 2006, 63(14):1658-1676.
[36] LIU Qingjun, WANG Hua, LI Hongliang, et al. Impedance sensing and molecular modeling of an olfactory biosensor based on chemosensory proteins of honeybee [J].Biosensors & Bioelectronics,2013, 40(1): 174-179.
[37] LI Hongliang, ZHANG Linya, NI Cuixia, et al. Molecular recognition of floral volatile with two olfactory related proteins in the Eastern honeybee (Apis cerana) [J]. International Journal of Biological Macromolecules, 2013, 56(5):114-121.
[38] LI Hongliang, NI Cuixia, TAN Jing, et al. Chemosensory proteins of the eastern honeybee, Apis cerana: Identification, tissue distribution and olfactory related functional characterization[J]. Comparative Biochemistry & Physiology Part B, 2016, 194/195(4):11-19.
[39] LI H, TAN J, SONG X, et al. Sublethal doses of neonicotinoid imidacloprid can interact with honey bee chemosensory protein 1 (CSP1) and inhibit its function [J].Biochemical & Biophysical Research Communications, 2017, 486(2):391-397.
[40] LIU Guoxia, MA Hongmei, XIE Hongyan, et al. Biotype characterization, developmental profiling, insecticide response and binding property of Bemisia tabaci chemosensory proteins: role of csp in insect defense [J/OL]. PLoS ONE, 2016, 11(5): e0154706.DOI:10.1371/journal.pone.0154706.
[41] LIU Naiyong, ZHU Jiaying, JI Mei, et al. Chemosensory genes from Pachypeltis micranthus, a natural enemy of the climbing hemp vine [J]. Journal of AsiaPacific Entomology, 2017, 20(2):655-664.
[42] GOPAL J V, KANNABIRAN K. Studies on interaction of insect repellent compounds with odorant binding receptor proteins by in silico, molecular docking approach [J]. Interdisciplinary Sciences Computational Life Sciences, 2013, 5(4): 280-285.
[43] DU Shaoqing, YANG Zhaokai, QIN Yaoguo, et al. Computational investigation of the molecular conformationdependent binding mode of (E)βfarnesene analogs with a heterocycle to aphid odorantbinding proteins [J]. Journal of Molecular Modeling, 2018, 24(3):70.
[44] LIU Naiyong, YANG Ke, LIU Yan, et al. Two generalodorant binding proteins in Spodoptera litura are differentially tuned to sex pheromones and plant odorants [J]. Comparative Biochemistry & Physiology Part A Molecular & Integrative Physiology, 2015, 180: 23-31.
[45] XIN Yi, ZHANG Yanbo, WANG Peidan, et al. Ligands binding and molecular simulation: the potential investigation of a biosensor based on an insect odorant binding protein [J]. International Journal of Biological Sciences,2015,11(1):75-87.
[46] ZHANG Yali, FU Xiaobin, CUI Hongchun, et al. Functional characteristics, electrophysiological and antennal immunolocalization of general odorantbinding protein 2 in tea Geometrid, Ectropis oblique[J]. International Journal of Molecular Sciences,2018, 19(3):425.
[47] AHMED T, ZHANG Tiantao, WANG Zhenying, et al. Three amino acid residues bind corn odorants to McinOBP1 in the polyembryonic endoparasitoid of Macrocentrus cingulum Brischke [J/OL]. PLoS ONE, 2014, 9(4): e93501.DOI:10.1371/journal.pone.0093501.
[48] ZHU Jiao, PAOLO P, LIU Yang, et al. Ligandbinding properties of three odorantbinding proteins of the diamond backmoth Plutella xylostella[J]. Journal of Integrative Agriculture, 2016, 15(3): 580-590.
[49] 張龙.飞蝗嗅觉的细胞与分子机制研究进展[J]. 生命科学, 2010, 22(12):1215-1228.
[50] WANG Yinliang, JIN Yincan, CHEN Qi, et al. Selectivity and ligandbased molecular modeling of an odorantbinding protein from the leaf beetle Ambrostoma quadriimpressum (Coleoptera: Chrysomelidae) in relation to habitatrelated volatiles [J]. Scientific Reports,2017,7:15374.DOI:10.1038/541598017155388.
[51] LIU Zhifeng, LIU Yujie, ZENG Guangming, et al. Application of molecular docking for the degradation of organic pollutants in the environmental remediation: A review [J]. Chemosphere, 2018, 203:139-150.
[52] 张亮仁.常用计算机辅助药物设计软件教程[M].北京:中国医药科技出版社, 2017.
[53] MORRIS G M, HUEY R, LINDSTROM W, et al. AutoDock 4 and AutoDock Tools 4: Automated docking with selective receptor flexibility [J]. Journal of Computational Chemistry, 2010, 30(16): 2785-2791.
[54] RUPPERT J, WELCH W, JAIN A N. Automatic identification and representation of protein binding sites for molecular docking [J]. Protein Science, 1997, 6(3):524-533.
[55] JAIN A N. Scoring noncovalent proteinligand interactions: a continuous differentiable function tuned to compute binding affinities[J]. Journal of ComputerAided Molecular Design, 1996, 10(5):427-440.
[56] NIKOLICHZUGICH J, SLIFKA M K, MESSAOUDI I. The many important facets of Tcell repertoire diversity [J].Nature Reviews Immunology, 2004, 4(2):123-132.
[57] SCHWEDE T, KOPP J, GUEX N, et al. SWISSMODEL: An automated protein homologymodeling server[J]. Nucleic Acids Research, 2003, 31(13): 3381-3385.
[58] WEBB B, SALI A. Comparative protein structure modeling using MODELLER[J]. Current Protocols in Bioinformatics,2014, 47(36):5.6.1-32.
[59] XIANG Zhexin. Advances in homology protein structure modeling [J]. Current Protein and Peptide Science, 2006, 7(3): 217-227.
[60] HOLLINGSWORTH S A, KARPLUS P A. A fresh look at the Ramachandran plot and the occurrence of standard structures in proteins[J]. Biomolecular Concepts, 2010, 1(3/4): 271-283.
[61] CARUGO O, DJINOVI C 'CARUGO K. Half a century of Ramachandran plots [J]. Acta Crystallographica, 2013, 69(8):1333-1341.
[62] 付寧宁,刘佳,渠成等. 悬铃木方翅网蝽化学感受蛋白CcilCSP1的结构及其结合寄主挥发物的预测分析[J]. 林业科学, 2017, 53(10):109-117.
[63] WANG Yanli, CHENG Tiejun, BRYANT S H, et al. PubChem BioAssay: A decades development toward open highthroughput screening data sharing [J].Nucleic Acids Research, 2017, 22(6):655-666.
[64] GELIN B R, MCCAMMON J A, KARPLUS M. Dynamics of folded proteins [J]. Nature, 1977, 267(5612): 585-590.
[65] ADENIYI A A, MES S. Implementing QM in docking calculations: is it a waste of computational time?[J]. Drug Discovery Today, 2017, 22(8):1216-1223.
[66] PARAMASIVAN R, SIVAPERUMAL R, DHANANJEYAN K J, et al. Prediction of 3dimensional structure of salivary odorantbinding protein2 of the mosquito Culex quinquefasciatus, the vector of human lymphatic filariasis[J]. Silico Biology, 2007,7(1):1-6.
(责任编辑: 田 喆)
