转录组测序技术在鉴定小麦染色体臂置换系染色体组成上的应用
2019-11-25缪娜娜丁明全杨思晴戎均康
缪娜娜,丁明全,杨思晴,戎均康
(1.浙江农林大学林业与生物技术学院,浙江临安 311300; 2.浙江农林大学农业与食品科学学院,浙江临安 311300)
小麦(TriticumaestivumL.)是世界上播种面积最大、产量仅次于玉米(ZeamaysL.)和水稻(OryzasativaL.)的第三大粮食作物,提供人类约20%的能量,其蛋白质和脂肪含量比其他农作物更丰富[1]。由于当前推广的栽培小麦品种大多来源于少数核心种质,使得小麦育种资源的遗传多样性显著下降[2-3]。从小麦野生近缘种中挖掘和利用新的优质基因资源是丰富普通小麦遗传背景的有效途径。
野生二粒小麦是普通小麦A和B染色体组的供体,具有粒大、蛋白质含量高和贮藏蛋白遗传多样性丰富等优良性状,并含有丰富的遗传变异,能够抵抗多种生物和非生物胁迫[4]。Garvin等[5]在野生二粒小麦中定位了Qfhs.ndsu-3AS和Qfhs.fcu-7AL等多个抗赤霉病相关的基因,可用于小麦赤霉病抗性的改良。Uauy等[6]在野生二粒小麦中发现籽粒蛋白质含量基因GPC-B1,能够显著提高小麦的蛋白质含量及锌、铁等微量元素含量,提升幅度超过10%~15%,对小麦品质改良具有重要价值。以上研究均表明,利用野生二粒小麦改良栽培小麦具有非常重要的理论和现实意义。
以普通小麦品种中国春(Chinese Spring,CS)为背景的野生二粒小麦材料TTD140染色体臂置换系(chromosome arm substitution line,CASL)是由以色列Weizmann科学院Feldman教授利用常规杂交结合细胞学鉴定等方法创制的[7]。利用这套材料已鉴定到很多优良或特异性状,如CASL7AS/CASL4BS/CASL7AL的千粒重明显高于CS[8],CASL2BS晚熟且与光周期基因Ppd-B1紧密连锁[9]。由于CASL是通过CS端体与TTD140杂交后再和相应CS端体多次回交产生,因此在非置换染色体臂上仍然会有野生二粒小麦片段残余。Zhou等[9]通过749个SSR标记鉴定该套CASL材料,其中277个在CS与TTD140间存在多态性,在非置换染色体上检测到25个没有被CS置换的TTD140染色体片段。由于小麦基因组巨大,常规分子标记在小麦材料之间多态性较低,因此不能有效监测所有置换区段[7-9];本研究利用转录组测序结合SNP分析技术,不仅可以从全基因组水平研究基因的表达,还可以更准确地了解染色体臂置换系每条染色体的遗传组成。因此,本研究利用三个CASL材料和中国春进行RNA测序,比对CS参考基因组序列,分析纯合SNP在染色体上的分布,最终确定野生二粒小麦置换区段的大小和位置,以期为CASL的鉴定提供新的有效手段。
1 材料与方法
1.1 试验材料
中国春(CS)及其染色体臂置换系材料CASL3AL、CASL7BS和CASL4AL由Feldman等[7]创制。2016年10月将试验材料种植于浙江农林大学试验田,每个材料连续种两行,行间距约30 cm。拔节期每个材料取10株最上部展开叶混合,用锡箔纸包好,置于液氮中冷冻保存待用。
1.2 试验方法
1.2.1 RNA提取
使用RNAprep Pure Plant Kit试剂盒(TIANGEN,北京)提取叶片组织总RNA,选取28S∶18S=2∶1,OD260/280值1.8~2.0,总量大于1 μg的样品进行建库。
1.2.2 转录组测序
1.2.3 RNA-Seq分析步骤
通过分析变异位点在染色体上的分布来判断野生二粒小麦的置换区段。分析步骤如下:
(1)去除测序原始读段raw reads的测序接头序列(fastp 0.19.5[10]),过滤掉低质量reads,并去除3'末端和5'末端低质量的碱基,得到高质量clean reads;
(2)以CS基因组“IWGSC RefSeq v1.0”(https://urgi.versailles.inra.fr/download/iwgsc/IWGSC_RefSeq_Assemblies/v1.0/)[1]为参考基因组,使用Hisat2[11]建立索引,并将clean reads比对到参考基因组上,提取唯一比对short reads,将比对后的SAM文件进行排序(GATK4.0:SortSam);
(3)过滤标记PCR扩增过程中产生的重复reads(GATK4.0:Markduplicates[12]),添加接头文件(Picard:Add Or Replace Read Group);
(4)建立索引,检测SNP变异位点(Samtools,GATK4.0:Haplotype Caller);
(5)对上述SNP位点进行硬过滤(GATK 4.0:Variant Filtration[13]),过滤标准为QUAL<60.0,QD<2.0,MQ<40.0,FS>60,SOR> 3.0,MQRankSum<-12.5,ReadPosRankSum< -8.0,DP<10;
(6)重复上述流程对每个CASL材料进行SNP变异的检测,使用Excel软件统计每个材料纯合(基因型为1/1)SNP数量及其在染色体的 分布;
(7)获取CASL材料每个基因的表达量FPKM(Fragments per kilobase of transcript per million fragments mapped)(HTseq[14]:htseq-read),阈值为FPKM≥1。
1.2.4 SSR引物设计与标记多态性验证
SSR分子标记来源于GrainGenes 3.0网站(https://wheat.pw.usda.gov/GG3/),或根据发布的中国春和野生二粒小麦Zavitan的参考基因组(https://wheat.pw.usda.gov/graingenes_downloads/Zavitan/)信息,通过MISA[15](MicroSAtellite identification tools,http://pgrc.ipk-gatersleben.de/misa/misa.html)软件对上述检测出的置换区段的序列进行SSR位点的预测,标准为二核甘酸重复次数不低于6次,三、四、五核苷酸重复次数不低于5次。然后,用Primer 3.0(http://primer3.ut.ee)根据鉴定出的SSR位点批量设计引物,并通过e-PCR 2.3.12验证引物的特异性。设计好的引物由上海华大生物公司合成。
使用CTAB法提取TTD140、CS和CASL3AL的叶片基因组DNA。PCR反应体系为15 μL,包含11.25 μL ddH2O、1.5 μL 10×Buffer、1 μL DNA、0.40 μL上下游引物、0.25 μL dNTPs、0.2 μL TakaraTaq酶。PCR反应程序为: 94 ℃预变性5 min;94 ℃变性30 s,58 ℃复性 30 s,72 ℃延伸 30 s,30个循环;72 ℃延伸 7 min, 4 ℃保存。使用10%聚丙烯酰胺凝胶电泳检测TTD140和CS之间的SSR标记多态性。
1.2.5 SNP位点验证
在3A染色体SNP突变位点密集区域设计两对特异性引物(表1)并进行PCR扩增。PCR体系为50 μL,包含19.5 μL ddH2O、25 μL GCbuffer、2 μL DNA、1 μL上下游引物、1 μL dNTPs、0.5 μL LaTaq酶。PCR反应程序为: 94 ℃预变性7 min;94 ℃变性30 s,62 ℃复性 30 s,72 ℃延伸1 min,35个循环;72 ℃延伸 7 min, 4 ℃保存。将TTD140、CS和CASL3AL基因组中的扩增产物直接割胶回收送生工生物工程(上海)股份有限公司测序。PCR产物目的片段约650 bp左右,使用MEGA7.0对这三个材料的扩增序列和CS/TTD参考基因组目标序列进行比对。

表1 SNP验证引物序列Table 1 Primer sequence for SNP verification
2 结果与分析
2.1 样品RNA提取及测序短片段(short reads)比对
经检测,三个CASLs和CS的RNA总量为164~344 ng·μL-1,18S和28S条带清晰且条带亮度比值28S∶18S=2∶1(图1),OD260/280在 1.8~2.0之间。本研究4个样品的RNA在测序后共产生315 290 370个raw reads,过滤后得到 303 606 610个clean reads。质控后四个材料Q20都大于96%,Q30约91%,说明测序原始数据质量可靠。将clean reads与中国春参考基因组比对,发现四个样品93.17%~95.56%的short reads能比对到参考基因组上,唯一比对(Unique mapped)率为81.60%~84.24%,非唯一比对(Multiple mapped)率为6.22%~9.00%,8.19%~10.57%的short reads未比对上(表2)。比对结果说明绝大多数的short reads可以比对到参考基因组上进行后续分析。
泳道M:Marker; 泳道1~4:CASL3AL、CASL4AL、CASL7BS和CS。
Lane M:Makers; Lanes 1-4:CASL3AL, CASL4AL, CASL7BS and CS.
图1 CASLs与CS总RNA质量琼脂糖凝胶检测结果
Fig.1Detection of total RNA from CASLs and CS with agarose gel
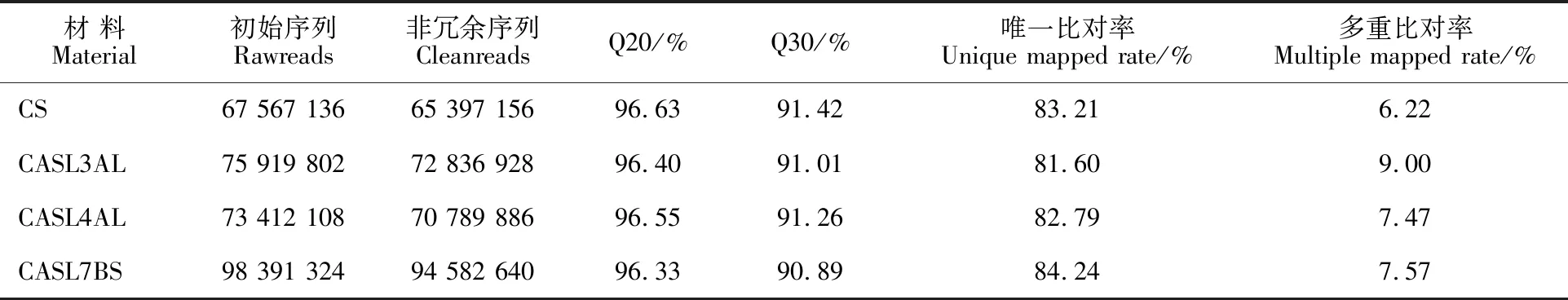
表2 高通量测序短片段(short reads)与参考基因组序列的比对率Table 2 Mapping rate of the short reads to the CS reference genome sequences
2.2 CASL间SNP在染色体上的数量和位置分布分析
CASL间SNP数量分析发现,每个CASL材料的表达基因数量不同,但每条染色体上表达基因数量占表达基因总数的百分比大致相同(表3)。表达基因在染色体上的分布呈现从着丝粒向端粒逐渐增加的趋势(图2d,f,h和j)。但不同CASL材料之间SNP的数量和其在染色体分布存在很大不同(图2c,e,g和i)。CASL3AL、CASL7BS和CASL4AL分别有1 940、2 594和 6 037个SNP。SNP主要集中在CASL材料的置换染色体3A,7B和4A上(图2)。其中CASL3AL的79.6%SNP分布于3A染色体的108-750 Mb区间,表明不仅3A染色体的整个长臂被置换,同时短臂上靠近着丝粒约200 Mb的区段也被TTD140置换(图2e);CASL7BS的 85.20%SNP主要集中在7B的0~510 Mb和5A上的30~460 Mb之间(图2g),表明除置换染色体7B外,5A上也有430 Mb的区段来自于TTD140。CASL4AL的SNP分布复杂(图2i),34.6%的SNP分布在染色体4A的40~710 Mb区段。余下的65.4%SNP位点分布在其他11条染色体,如7B上的713个SNP分布于0~570 Mb区段,5B的696个SNP分布在长臂近末端410~675 Mb的区段;还有9条染色体含有超过100个SNP,分布于染色体端部100 Mb内。有趣的是,CASL4AL较多的SNP分布在D组染色体亚组上,如475个SNP分布于5D上的530~566 Mb,347个SNP分布于7D上的13~46 Mb之间。
2.3 利用SSR分子标记验证CASL3AL置换 区段
为了验证SNP分析鉴定出的CASL染色体组成的可靠性,在3A染色体上新设计222对SSR标记,其中77对在CS和TTD140间存在多态性;加上20对具有多态性的公共分子标记(barc、wmc、gwm、cfa系列),共计97对。这些标记覆盖整条3A染色体,标记间平均距离约7.83 Mb。用这些标记验证CASL3AL的置换区段,检测到TTD140带型的84对标记集中在短臂上靠近着丝粒的P0409标记和长臂上的P2089标记之间区段(108~750 Mb)(图3),没有检测到TTD140条带的标记则集中在P0008-P0400标记之间(0~108 Mb),说明3A染色体的108~750 Mb区段来自于TTD140,检测到TTD140带型的SSR标记分布区间与上述RNA-seq分析出的SNP分布区域(图2e:3A)相吻合,证明使用RNA-seq结合SNP分析技术鉴定CASL材料的染色体组成是可靠的。
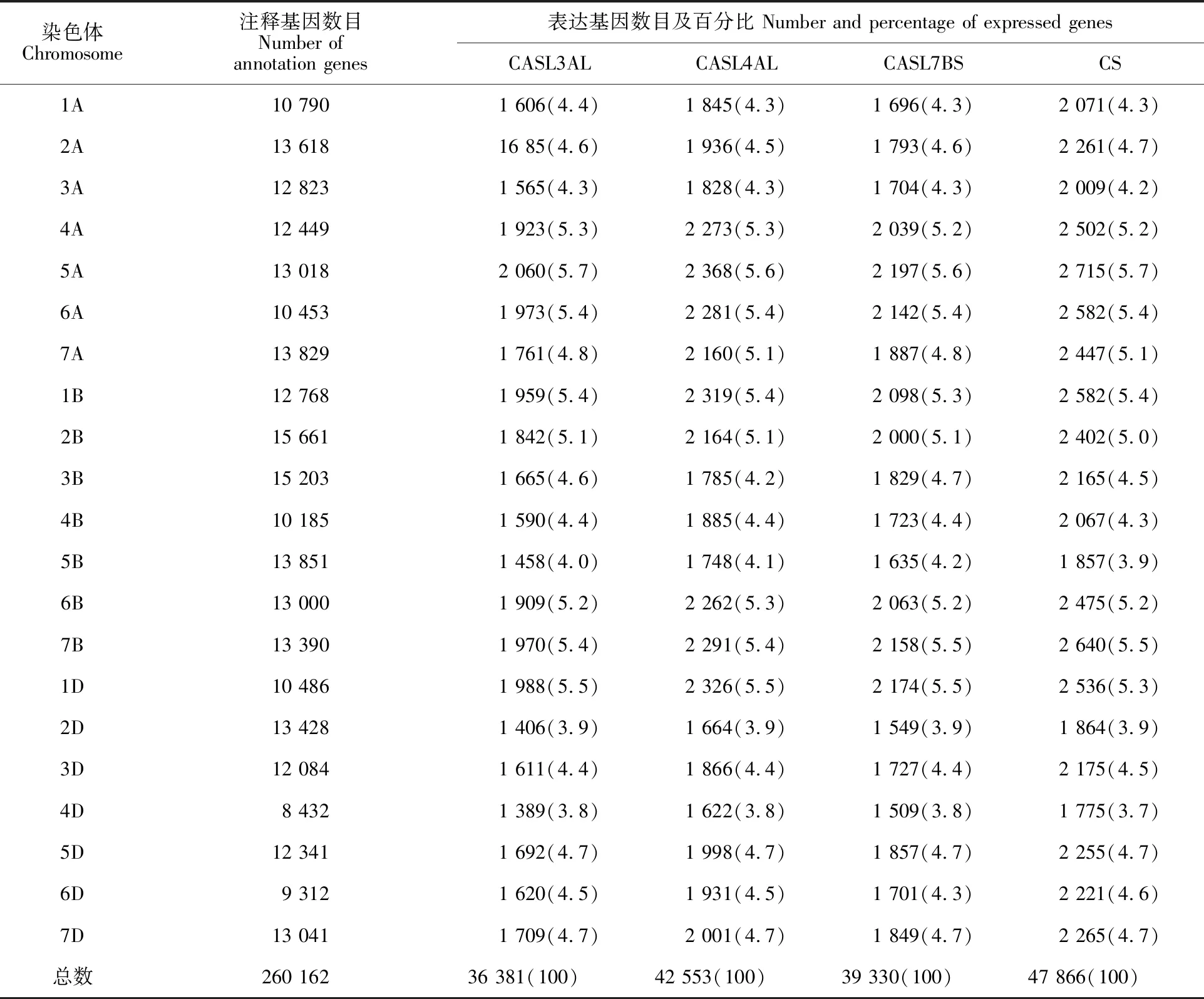
表3 表达基因在染色体上的分布Table 3 Distribution of expressed genes on chromosomes
括号内的数据为每条染色体上表达基因数量占表达基因总数的百分比。
The data in bracket are the percentage of the number of expressed genes on each chromosome to the total number of expressed genes.
2.4 SNP突变位点验证
为验证SNP位点,设计了两对引物对SNP富集区域进行扩增,其中引物3A_SNP_1在CS和CASL3AL的扩增产物测序结果显示重叠峰,可能是该引物特异性不高导致PCR产物不纯引起的,引物3A_SNP_2测序结果正常。将引物3A_SNP_2在CS、CTTD140和CASL3AL基因组中扩增产物的序列与TTD和CS参考基因组进行比对,其中320 bp序列中存在7个SNP(图4),本研究所用CS的序列和CS参考基因组一致,CASL3AL和TTD140的序列与Zavitan参考基因组一致,证明CASL3AL的该段DNA序列来自于TTD140。
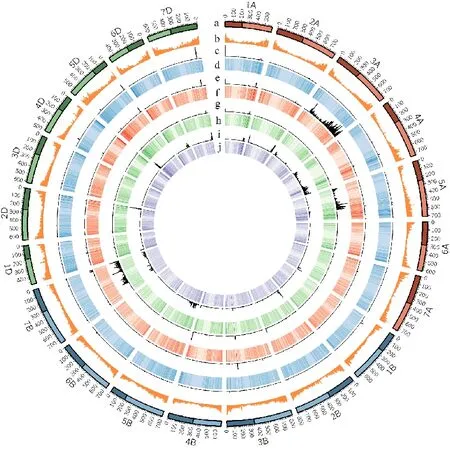
a:小麦染色体长度及其着丝粒位置(颜色较浅的为长臂,颜色较深的为短臂);b:小麦注释基因在染色体上的分布;c:中国春材料的纯合SNP分布;d:中国春材料表达基因分布(FPKM≥1);e:CASL3AL上纯合SNP的分布;f:CASL3AL材料表达基因的分布(FPKM≥1);g:CASL7BS上纯合SNP的分布;h:CASL7BS料表达基因的分布(FPKM≥1);i:CASL4AL上纯合SNP的分布;j:CASL4AL材料表达基因的分布(FPKM≥1)。
a:Length and centromeric position of chromosome in wheat(lighter colors indicate the long arm and dark colors indicate short arm); b:The distribution of annotated genes on chromosomesin wheat; c:The distribution of homozygous SNP in Chinese Spring; d:The distribution of genes expressed in Chinese Spring(FPKM≥1); e:The distribution of homozygous SNP in CASL3AL; f:The distribution of gene expressed in CASL3AL( FPKM≥1 ); g:The distribution of homozygous SNP in CASL7BS; h:The distribution of gene expressed in CASL7BS(FPKM≥1); i:The distribution of homozygous SNP in CASL4AL; j:The distribution of gene expressed in CASL4AL(FPKM≥1).
图2 表达基因和SNP在染色体上的分布
Fig.2 Distribution of SNPs and expressed genes on chromosomes
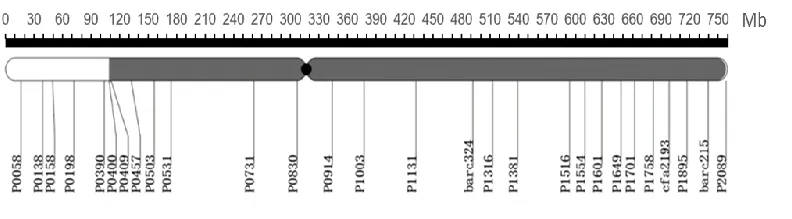
P0058等为SSR标记,标记间间距为物理距离,深灰色区域代表CASL3AL中来自TTD140的染色体片段。
P0058,etc. are SSR markers. Intervals between SSRs are physical distance. The bar with dark grey represents the fragment from TTD140 in CASL3AL.
图3 CASL3AL与CS间多态性SSR分子标记在3A染色体上的分布
Fig.3 Distribution of SSR polymorphic molecular markers between CASL3AL and CS on chromosome 3A

CS_reference为IWGSC Ref Seqv1.0参考序列;CS为中国春测序结果;TTD_reference为Zavitan参考序列;TTD为TTD140;CASL3AL为染色体臂置换材料。
CS_reference is IWGSC Ref Sqv1.0 reference sequence; CS is the sequencing result of Chinesse Spring;TTD_reference is the Zavitan reference sequence; TTD is TTD140; CASL3AL is a chromosome arm substitution material.
图4 CS/TTD140/CASL3AL测序片段与参考基因组的比对结果
Fig.4 Comparison of sequencing fragments of CS/TTD140/CASL3AL with reference genome
3 讨 论
CASL材料是由CS端体与TTD140杂交,然后经CS端体多次回交选育而成,理论上非置换臂还会存在少量的TTD染色体片段没有被CS替换。Millet等[7]使用RFLP和SSR分子标记对以普通小麦品种Bethlehem(BL)为背景的CASL染色体组成进行了鉴定,发现在非置换染色体上确实有一定量的TTD存在;Zhou等[8]利用多态性SSR标记对以CS为背景的CASL材料的染色体组成进行了鉴定,也在非置换染色体上发现了少量TTD片段。本研究采用转录组测序结合SNP分析法,检测三个CASL材料中的TTD置换片段。三个CASL材料的大多数SNP存在于置换染色体臂和靠近着丝粒附近约200 Mb的另一条臂上,此外,CASL7BS在非置换臂5A染色体上检测到约430 Mb的TTD片段,CASL4AL在11条非置换染色体上也检测到TTD片段。本研究在置换染色体上检测到的TTD片段范围比Millet等[7]和Zhou等[8]检测到的片段范围更精确,同时在非置换染色体上检测到了更多的TTD片段。这可能是因为Millet等[7]和Zhou等[8]所用的SSR标记检测具有一定的局限性,如Zhou等[8]用749对SSR标记只筛选到277对多态性标记用于TTD置换片段的检测,平均每条染色体13对,明显偏低。这表明使用传统的SSR分子标记检测置换区段存在一些不足,如标记多态性低、鉴定过程费工费时、标记覆盖密度低等。而利用转录组数据获得的SNP分布广且相对均匀,检测更可靠。但该方法也存在一些不足,如转录组数据具有时间、组织、空间特异性等特点,可能还有部分TTD置换区段无法被检测到。为此,可在取样时取多株不同时期不同部位的样品混合测序,以达到全面鉴定置换区段的效果。综上所述,本研究对置换材料的供体片段的鉴定提供了快速、准确的方法,具有较高的应用价值。
