野桑蚕scalloped同源基因的克隆和序列分析
2019-11-05朱文东
朱文东


摘要:Hippo通路与昆虫的生长、发育和变态等过程密切相关,在动物不同物种之间高度保守。scalloped基因是Hippo通路元件Yki的偶联因子之一,参与调控基因的表达。克隆了野桑蚕(Bombyx mandarina)scalloped基因的完整开放阅读框(ORF)及其上下游的部分非编码序列。序列联配分析表明,与家蚕相比,野桑蚕scalloped基因序列存在多处的核酸片段缺失、插入及位点差异。结构域分析表明,野桑蚕Scalloped蛋白与家蚕、黑腹果蝇、小鼠和人的蛋白结构类似,均含有完整的TEAD蛋白家族典型TEA结构域和PDB结构域,但野桑蚕编码蛋白缺失蛋白C末端序列。系统发生树分析表明,昆虫Scalloped蛋白与脊椎动物TEF3可能存在共同的起源;家蚕Scalloped在进化中出现晚于野桑蚕且经历较多遗传选择过程。
关键词:野桑蚕(Bombyx mandarina);Hippo通路;scalloped基因
中图分类号:Q785 文献标识码:A
文章编号:0439-8114(2019)17-0123-06
DOI:10.14088/j.cnki.issn0439-8114.2019.17.033 开放科学(资源服务)标识码(OSID):
Abstract: Hippo pathway is closely related to the physiological and biochemical processes of insect growth, metamorphosis and development, and highly conserved among different species. Scalloped is one of terminal action factor of Hippo pathway, which regulates the expression of a large number of genes. In this study, the complete ORF and partial UTR sequence of scalloped in wild silkworm (Bombyx mandarina) were cloned. Indels and variable sites were discovered after sequences analysis between Bombyx mori and Bombyx mandarina. The domain structure analysis shown that the encoding protein of scalloped in wild silkworm, domestic silkworm, fruit fly, mice and human have the similar domain architecture, while partial of the C-terminal of wild silkworm scalloped protein absent. The phylogenetic analysis showed that the Scalloped in insects and TEF3 in vertebrate could have a common origin, and the Scalloped of domestic silkworm could be appeared later than that in wild silkworm, and experienced more genetic selection process.
Key words: wild silkworm(Bombyx mandarina); Hippo pathway; scalloped gene
Hippo通路是最早在果蠅中发现的一条细胞信号转导途径[1],也是一条极为保守的信号通路,在昆虫生长和发育过程中起着十分重要的作用[2,3]。果蝇Hippo通路核心部分是由Hpo(Hippo)、Wts(Warts)、Sav(Salvador)、Mats(Mob as tumor suppressor)和Yki(Yorkie)组成的,Yki(Yorkie)是该通路的末端作用因子。Hippo通路通过调控Yki的辅转录调控作用,起到控制细胞生长和形态发生等作用。该通路不同组分的功能缺陷均可导致果蝇胚胎、复眼、翅成虫盘等器官组织过生长,甚至产生致死作用[2-5]。。哺乳动物Hippo通路由相应的同源物MST1/2、hSAV1、LATS1/2、MOB1构成[6-8]。果蝇Hippo通路核心激酶级联反应首先是在上游信号输入因子的作用下,Hpo-Sav复合体磷酸化,并依次磷酸化Wts-Mats复合物而激活,活化后的Wts-Mats复合物促使Yki磷酸化并与14-3-3蛋白结合,抑制Yki的促增殖和抗凋亡活性,促使细胞凋亡[7,9]。如果Hippo通路被阻断或失活,Yki不能被磷酸化,则Yki可迁移进入细胞核,并与一系列转录因子结合,促进下游靶基因的表达,最终启动Hippo通路的促增殖和抗凋亡作用[10]。在众多转录因子中,TEAD同源蛋白是该通路末端作用因子YAP在细胞核内的主要偶联蛋白之一[11,12]。YAP与TEAD的相互作用在不同物种之间表现出极高的保守性。果蝇Yki与Sd蛋白作用,哺乳动物YAP与Sd同源物TEF/TEAD蛋白相互作用,Sd/TEAD提供DNA结合活性,可以促进非磷酸化的YAP核定位。Hippo通路的功能响应最终有赖于YAP和TEAD蛋白在细胞核内的偶联并与靶基因识别位点的结合[8,9]。TEAD在果蝇中的同源蛋白被命名为Scalloped。研究发现scalloped基因的功能受到破坏后可导致果蝇形态产生严重异常。
在哺乳动物中,TEAD家族蛋白在胚胎发育、营养代谢、压力应答等事件中起到重要作用[9,13-16]。研究还发现,TEAD还与乳腺癌、输卵管肿瘤、胃肠肿瘤等密切相关。抑制TEAD-YAP活性可以控制癌症的发生发展[11]。因此,TEAD蛋白作为一种肿瘤标志物的同时,也是肿瘤靶向治疗的潜在位点之一。
野桑蚕(Bombyx mandarina)是家蚕(Bombyx mori)的驯化祖先[17,18]。长期的人工驯化使得家蚕和野桑蚕在体形、器官尺寸、发育模式等方面均表现出显著差异。目前,已有20余个家蚕Hippo通路成员基因获得克隆和鉴定[19,20]。鉴于Hippo通路在动物生长发育中的重要作用,在野桑蚕中开展对scalloped的研究将有助于深入研究驯化作用对桑蚕体形及器官影响的分子机制,同时还为桑蚕生产性能改良、农林害虫防控提供可利用的分子靶标。本试验依据scalloped基因的保守和同源性,克隆了野桑蚕scalloped基因开放阅读框(ORF)及部分上下游非翻译区(UTR)序列,通过生物信息学方法分析野桑蚕scalloped编码基因的序列特征,为深入研究Hippo通路与野桑蚕变态发育提供理论基础。
1 材料与方法
1.1 供试昆虫
所使用的昆虫为野桑蚕材料1份,由陕西省蚕桑重点实验室提供。
1.2 野桑蚕总RNA的抽提和cDNA第一链的合成
利用Trizol试剂(Invitrogen)抽提5龄第三天的野桑蚕总RNA,超微量分光光度计(NanoDrop 2000,Thermo)检测RNA质量和浓度是否合格,使用M-MLV反转录试剂盒(Invertrogen)并按照试剂盒使用说明书反转录并合成cDNA。
1.3 基因克隆和測序
根据家蚕scalloped基因序列(XM_004927964.1)设计引物,p1:5′-TCTTTGTGCCATTCTTCGCAT-3′,5′-TCTTTTGAAGTGATATGATGTCC-3′;p2:5′-CAA
TTTTGGCAGCCAGGCCTACA-3′,5′-ATAGCTATAG
AATCATAAGGC-3′。
以野桑蚕cDNA作为模板,克隆scalloped基因全长。PCR条件:94 ℃ 4 min;94 ℃ 40 s,56 ℃ 40 s,72 ℃ 100 s,26次循环;72℃ 10 min,结束反应。PCR产物采用1.20%(m/V)的琼脂糖凝胶电泳鉴定,切胶回收目的片段,TA克隆后测序。测序结果使用Lasergene软件包下的SeqMan子程序去除接头污染,之后进行序列拼接,组装成单一序列。
1.4 信息学分析
从CNBI数据库获取家蚕及其他物种的scalloped同源基因的核酸和蛋白序列;利用MUSLE软件包进行核苷酸、氨基酸序列的同源性比对和相似性分析;采用MEGA6.0软件以邻接法(Neighbor-joining)构建系统发育树(Bootstrap=1 000,Poisson model,pairwise deletion);利用ExPASY(http://wed.expasy.org/compute_pi)、SMART(http://small.embl-heidelberg.de/)、PSIPRED(http://bioinf.cs.ucl.ac.uk/psipred/)、ORF finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)等在线工具分析蛋白理化性质、序列特征、保守基序、二级结构特征和开放阅读框。
2 结果与分析
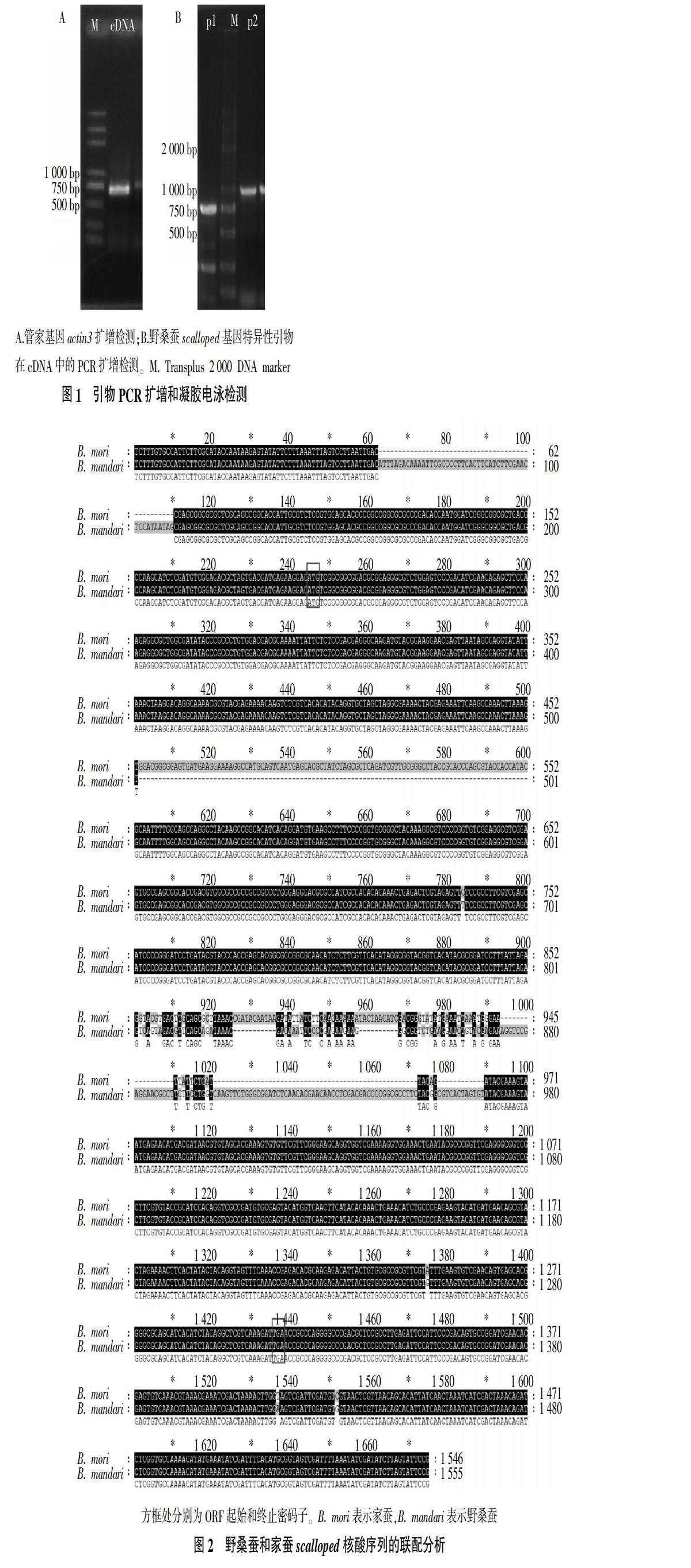
2.1 PCR扩增和凝胶电泳检测
抽提野桑蚕RNA后,反转录合成cDNA。利用管家基因actin3对cDNA进行检测,结果表明在500~750 bp出现单一清晰的目的条带(图1A),表明反转录成功,合成的cDNA可以用于后续试验。利用设计的2对野桑蚕scalloped基因特异性引物(p1、p2),以合成的cDNA为模板进行PCR扩增,电泳检测结果显示2对引物可扩增获得明亮、单一的条带(图1B)。将p1、p2扩增获得的凝胶条带切胶、回收,提取扩增产物并转入载体中,进行TA克隆,最后抽提单一菌落的载体质粒进行测序分析。
2.2 野桑蚕scalloped基因克隆和测序
经克隆测序和序列拼接后,获得一段长度为1 555 bp的核酸序列。将获得的核酸序列提交至NCBI数据库中进行Blast检索,结果与昆虫scalloped基因及哺乳动物TEAD基因具有较高同源性,其中与家蚕scalloped基因序列比对覆盖度为82%,匹配序列的相似性为99%。ORF框预测结果表明,野桑蚕scalloped基因ORF长度为1 074 bp,起始密码子和终止密码子分别为ATG和TGA,ORF编码357个氨基酸残基,编码蛋白预测分子质量为40.2 ku,等电点为4.72(图2)。结果表明,试验扩增所得的序列为野桑蚕scalloped基因,并成功克隆得到该基因的ORF编码序列及其上下游的部分非翻译区(UTR)。
2.3 序列特征分析
将克隆得到的野桑蚕scalloped基因与家蚕同源核酸序列进行比对。结果(图2)表明,两者在多个位点及片段上存在差异。与家蚕相比,这些差异包括野桑蚕scalloped基因在5′端UTR序列存在一处长度为48 bp的序列插入,在ORF内存在多处序列插入或缺失,以及在多个位点上出现碱基替换。蛋白序列比对(图3)表明,相比于家蚕,野桑蚕scalloped基因编码蛋白显著变短,其主要差异位点在于蛋白C端和141~176 aa位点之间出现氨基酸残基的缺失,同时在282~283 aa位点之间出现48个氨基酸残基的插入。暗示该基因伴随桑蚕的驯化过程出现显著的序列变异。
二級结构分析(图4)表明,野桑蚕Scalloped蛋白在N端(1~86 aa区域)存在3处α螺旋和1处β折叠,在N端(204~357 aa)存在1处α螺旋和6处β折叠,而在两区域之间是一段较长的无规卷曲结构,与TEA结构域和PDB结构域在该蛋白上的分布相吻合,表明TEA结构域主要由α螺旋构成,PDB结构域主要由β折叠及无规卷曲结构组成。
结构域分析(图5)表明,野桑蚕Scalloped蛋白与家蚕(Bombyx mori)、黑腹果蝇(Drosophila
melanogaster)、小鼠(Mus musculus)和人(Homo
sapiens)的蛋白结构类似,均含有完整的TEAD蛋白家族典型TEA结构域和PDB结构域,但野桑蚕缺失Scalloped的同源蛋白C端序列。野桑蚕与其他物种在TEA结构域和PDB结构域相应蛋白序列上具有高度的相似性,但在两结构域之间的连接区域与果蝇、赤拟谷盗(Tribolium castaneum)等存在一定程度的区别,且与家蚕存在明显差异。暗示野桑蚕Scalloped蛋白在与家蚕等具有类似功能的同时,可能还存在其他生物学功能,或存在行使功能过程的差异性。
2.4 系统发生树分析
为了进一步分析scalloped基因编码蛋白亲缘关系,利用赤拟谷盗、家蚕、黑腹果蝇等昆虫及人、小鼠、斑马鱼(Danio rerio)等脊椎动物的同源蛋白序列构建了系统进化树(图6)。脊椎动物中存在4种Scalloped蛋白的同源蛋白(TEF1、TEF3、TEF4和TEF5),但物种之间存在数量差异,如在斑马鱼中未获得TEF3和TEF4同源蛋白,表明scalloped在脊椎动物中可能存在功能上的多样性。TEF1、TEF4和TEF5各自构成亚群,同时三者可聚类共同构建成一个大支。昆虫中仅存在一种Scalloped同源蛋白,且与脊椎动物中的TEF3聚为一大类群,其中脊椎动物TEF3又单独聚为亚群,暗示TEF3与昆虫Scalloped蛋白亲缘关系更为接近,可能存在共同的起源,在进化过程中出现分化。野桑蚕同家蚕聚为一个亚群,其中家蚕支长显著长于野桑蚕,表明该基因在家蚕进化过程中经历较长的进化选择事件。
3 小结与结论
Hippo信号通路是一条极为保守的信号转导途径,参与对胚胎、器官、组织发生等过程的调控,与昆虫生长发育、变态发育、生殖、营养代谢等许多生理生化过程密切相关。有关Hippo通路在桑蚕中的研究较少,野桑蚕中尚未见相关的研究报道。因此,考察Hippo通路组成元件在家蚕和野桑蚕之间的差异,可以为桑蚕发育性状差异研究提供理论基础。
scalloped基因编码蛋白是Hippo通路的主要终端作用因子,调控了大量发育密切关联基因的表达。本研究克隆了野桑蚕scalloped基因的完整ORF及其上下游的非编码序列,序列分析表明野桑蚕scalloped基因编码蛋白与昆虫及哺乳动物同源蛋白具有高度相似性,与家蚕相比存在多处的核酸片段缺失和插入及位点差异,暗示野桑蚕scalloped基因在可能存在类似的调控功能的同时,还可能存在其他作用及功能行使方式。
系统发生树分析表明,脊椎动物之间存在Scalloped同源蛋白数量差异,可能与基因功能分化有关;昆虫Scalloped蛋白与脊椎动物TEF3可能存在共同的起源;家蚕Scalloped在进化中出现晚于野桑蚕且经历较多遗传选择过程,可能与家蚕起源于野桑蚕且经过快速的人工选择有关。
本研究通过对野桑蚕Hippo通路末端效应元件scalloped基因进行克隆和分析,有利于深入研究桑蚕Hippo通路的作用机理,为深入开展家蚕、野桑蚕的性状差异分析及相关功能基因发掘提供理论基础。
参考文献:
[1] BADOUEL C,GARG A,MCNEILL H. Herding Hippos:Regulating growth in flies and man[J].Current opinion in cell biology,2009,21(6):837-843.
[2] PANTALACCI S,TAPON N,LEOPOLD P. The Salvador partner Hippo promotes apoptosis and cell-cycle exit in Drosophila[J]. Nat Cell Biol,2003,5(10):921-927.
[3] UDAN R S,KANGO-SINGH M,NOLO R,et al. Hippo promotes proliferation arrest and apoptosis in the Salvador/Warts pathway[J].Nat Cell Biol,2003,5(10):914-920.
[4] XU T,WANG W,ZHANG S,et al. Identifying tumor suppressors in genetic mosaics:The Drosophila lats gene encodes a putative protein kinase[J].Development,1995,121(4):1053-1063.
[5] HARVEY K F,PFLEGER C M,HARIHARAN I K.The Drosophila Mst ortholog, hippo,restricts growth and cell proliferation and promotes apoptosis[J].Cell,2003,114(4):457-467.
[6] WU S,HUANG J,DONG J,et al. hippo encodes a Ste-20 family protein kinase that restricts cell proliferation and promotes apoptosis in conjunction with salvador and warts[J].Cell,2003, 114(4):445-456.
[7] KANGO-SINGH M,NOLO R,TAO C,et al. Shar-pei mediates cell proliferation arrest during imaginal disc growth in Drosophila[J].Development,2002,129(24):5719-5730.
[8] TAPON N,HARVEY KF,BELL D W,et al. salvador promotes both cell cycle exit and apoptosis in Drosophila and is mutated in human cancer cell lines[J].Cell,2002,110(4):467-478.
[9] ZHANG L. Control of growth and beyond:A special issue on Hippo signaling[J].Acta Biochim Biophys Sin(Shanghai),2015, 47(1):1.
[10] DONG J,FELDMANN G,HUANG J,et al. Elucidation of a universal size-control mechanism in Drosophila and Mammals[J].Cell,2007,130(6):1120-1133.
[11] ZHOU Y,HUANG T,CHENG A S,et al. The TEAD family and its oncogenic role in promoting tumorigenesis[J].International journal of molecular sciences,2016,17(2):138.
[12] LAMAR J M,STERN P,LIU H,et al. The Hippo pathway target,YAP,promotes metastasis through its TEAD-interaction domain[J].Proceedings of the national academy of sciences,2012,109(37):E2441-E2450.
[13] YIN M X,ZHANG L. Hippo signaling in epithelial stem cells[J].Acta biochimica biophysica sinica,2015,47(1):39-45.
[14] SHI P,FENG J,CHEN C. Hippo pathway in mammary gland development and breast cancer[J].Acta biochimica biophysica sinica,2015,47(1):53-59.
[15] MO J S,MENG Z,KIM Y C,et al. Cellular energy stress induces AMPK-mediated regulation of YAP and the Hippo pathway[J].Nature cell biology,2015,17(4):500-510.
[16] MAO B,GAO Y,BAI Y,et al. Hippo signaling in stress response and homeostasis maintenance[J].Acta biochimica biophysica sinica,2015,47(1):2-9.
[17] XIANG H,LIU X,LI M,et al. The evolutionary road from wild moth to domestic silkworm[J].Nature ecology & evolution,2018,2(8):1268-1279.
[18] XIA Q,GUO Y,ZHANG Z,et al. Complete resequencing of 40 genomes reveals domestication events and genes in silkworm(Bombyx)[J].Science,2009,326(5951):433-436.
[19] XU X,BI H L,ZHANG Z J,et al. BmHpo mutation induces smaller body size and late stage larval lethality in the silkworm,Bombyx mori[J].Insect science,2018,25(6):1006-1016.
[20] ZENG W,LIU R,ZHANG T,et al. BmYki is transcribed into four functional splicing isoforms in the silk glands of the silkworm Bombyx mori[J].Gene,2017,646:39-46.
