微生物DNA提取通用试剂盒的研究
2019-10-16李新欣候珺淇代振玉
张 杰,李 研,杨 琳,李新欣,赵 新,候珺淇,代振玉
(周口师范学院 生命科学与农学学院,河南 周口 466001)
DNA是遗传信息的载体,是重要的生物信息分子,是生物学研究的主要对象. 微生物DNA提取过程中遇到许多纯度不够高、方法通用性不强、提取速度慢的问题. 在过去的五年内,总共有3 000多篇文章报道过利用试剂盒或CTAB法提取微生物DNA或 RNA的研究,文章中有160 000多条基因或基因组序列被公布[1].
微生物DNA的提取有很多方法,例如细菌可以采用SDS-酶裂解法,霉菌和酵母菌采用改良的氯化苄法,而放线菌采用改良酶法. 这些方法的简要步骤如下:通过对培养的微生物菌落进行比较和筛选,选择出长势良好的菌落(细菌、霉菌、酵母菌和放线菌菌落),然后利用改进的CTAB法对不同微生物菌落的DNA进行提取. DNA提取过程中细胞破壁的方法有液氮研磨法、石英砂振荡研磨法和高温加热法等[1-2]、改良酶或化学法破壁[3]. 国内外已报道了多种酵母菌总DNA制备的方法,但它们都存在各自的优缺点[4-6]. 关于真菌DNA提取的方法,国内外已经有很多报道. 目前存在和使用的真菌DNA提取方法步骤大体相似[7-9],主要差别在于破坏真菌菌丝、孢子的细胞壁的方式不同和提取缓冲液的配方有差异.
常采用试剂盒提取微生物DNA,从细胞中提取的DNA数量较少,很难满足Southern印迹等试验的要求. CTAB法可在短时间内制备大量DNA样品,但是提取过程中常用一些对人体有害的试剂,如氯仿、酚和巯基乙醇,通用性较弱,需要针对不同的样品改变配方[10-12].
本研究通过对微生物的4种代表菌种(霉菌、酵母菌、细菌和放线菌)的DNA提取方法的比较,获得了微生物DNA提取通用试剂盒的配制方法,该方法不使用对人体有害的试剂,从而为微生物DNA快速、方便和廉价的提取提供了科学依据,促使CTAB法在微生物DNA提取时更具通用性和方便性.
1 材料与方法
1.1 实验材料
从宋河酒业提供的酒糟中分离出来的细菌、霉菌、放线菌和酵母菌为研究对象,采用笔者制备的DNA extraction kit试剂盒提取其DNA.
1.2 实验方法
1.2.1 试剂盒的操作流程
改进的 CTAB法操作步骤如下: 样品破碎(多样品冷冻研磨仪PS-24研磨)→取50 mg样品粉末,加入800 μL的dissociation buffer(CTAB抽提液,内含1% PVP),剧烈涡旋 →65 ℃水浴10 min,该期间充分混合样品两次 →加入140 μL purification buffer(酚、碳酸二甲酯、异戊醇的比例为25∶24∶1)涡旋混合样品. 10 000×g离心10 min →小心吸取上清液转移到另一个新离心管中,而后加0.7倍体积的异丙醇并涡旋以便沉淀DNA →10 000 × g离心2 min沉淀DNA →小心吸取或轻轻倒出上清液并弃去,确保DNA不被丢掉 →加300 μL C液(无菌去离子水)重悬DNA,无菌去离子水提前预热到65 ℃. 加入4 μL RNase A 并混匀 →将整个样品 (包括形成的沉淀) 转移到放有收集管的HiBind© DNA column中,10 000× g离心1 min,弃滤液. 转移柱子到一个新收集管上→加入700 μL DNA wash buffer(70%乙醇),10 000× g离心1 min,弃滤液 →将柱子最大转速离心2 min至干燥 →将柱子转移到干净的1.5 mL的离心管中,加入50~80 μL的C液(C液提前65 ℃预热) 室温静置20 min,10 000 × g 离心3~5 min →琼脂糖凝胶电泳和微量核酸检测仪测其浓度和纯度,-80 ℃保存备用.
备注:新的提取过程中,用dissociation buffer与CTAB抽提液相比增加了1% PVP,purification buffer(酚、碳酸二甲酯、异戊醇的比例为25∶24∶1)代替了原有的纯化溶液(酚、氯仿、异戊醇的比例为25∶24∶1),DNA的纯化过程增加了DNA收集柱,DNA溶解液用去离子的elution buffer代替了双蒸水.
1.2.2 菌株样品的制备
(1)分别将细菌、霉菌、放线菌和酵母菌接种到相应的培养皿上培养,收集菌体放置于-80 ℃冰箱中保存备用.
(2) 分别将细菌、霉菌、放线菌和酵母菌接种到液体培养基中培养,抽虑收集菌体放置于-80 ℃冰箱中保存备用.
1.2.3 DNA质量检测
(1)DNA的感官评定: DNA沉淀物自然干燥后,通过对其颜色的观察,感官上评定糖、酚等一些杂质的污染程度. 含糖比较多的DNA沉淀时,沉淀物常常呈胶状,纯DNA则为絮状等沉淀;含有酚类杂质的DNA沉淀物多呈黄色至深褐色,难以溶解或溶液颜色深,纯DNA为类似石棉样的白色纤维状物.
(2)微量核酸检测仪Agilent 2100检测DNA质量曲线图:全波长的分光光度计核酸检测仪NanoDrop ND-1000(波长范围为 220 nm~750 nm)测定提取DNA的质量和浓度. DNA的纯度主要指标包括蛋白质、糖、RNA及其他杂质污染的程度,可通过OD260/OD280比值判断. 通常OD260/OD280比值越接近1.8,表明DNA越纯;在1.8~2.0之间表明有RNA污染;大于2.0表明DNA有降解或RNA比较多;小于1.8有蛋白质污染. OD260/OD230比值应在2~2.5之间,偏小则表明有盐残余.
(3)琼脂糖凝胶评定:通过琼脂糖凝胶电泳评定DNA的质量和浓度.
2 结果与分析
2.1 DNA质量的感官评定

表1 提取孢子DNA的感官分析
由表1可知,通过该试剂盒提取到的4种微生物孢子的DNA状态和颜色均符合高质量DNA的要求,说明该提取方法提取到的DNA符合感官要求.

表2 提取菌体DNA的感官分析
由表2可知,通过该试剂盒提取到的4种微生物菌体的DNA状态和颜色均符合高质量DNA的要求,说明该提取方法提取到的DNA符合感官要求.
由图1A可知,该试剂盒提取到的4种微生物孢子的DNA在液体沉淀中出现白色絮状沉淀,且无明显的杂质. 由图1B可知,该试剂盒提取到的4种微生物菌丝体的DNA在液体沉淀中出现白色絮状沉淀,且无明显的杂质.
上述实验数据表明该试剂盒能够提取到4种微生物的孢子或菌丝体的DNA,且提取到的DNA在感官上符合要求.

图1 提取的DNA在液体中的形态
2.2 DNA质量的理化检测
为了检测试剂盒提取到的DNA质量,用Agilent 2100和NanoDrop检测了所提样品的片段大小、纯度和浓度. 由图2和图3可知,试剂盒所提取到的DNA主峰片段大小在3 000~7 000 bp范围之内,而次峰的片段大小在200~400 bp和12 000 bp左右,表明提取的DNA能够满足下游实验需求. 由图2和图3的主峰比较可知,提取到的孢子DNA的浓度比菌丝体DNA的浓度稍低.
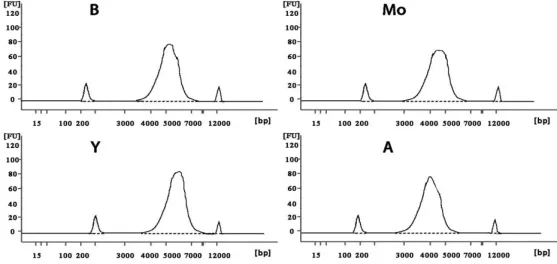
图2 Agilent 2100检测提取孢子DNA质量曲线效果图
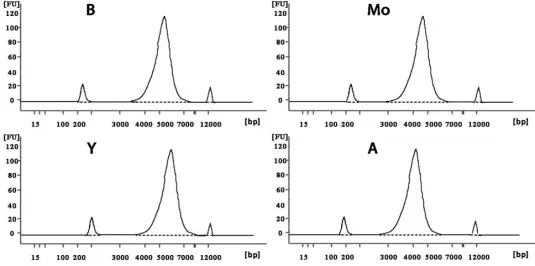
图3 Agilent 2100检测提取菌体DNA质量曲线效果图
用NanoDrop分别检测了提取的孢子和菌丝体DNA的OD260/280和OD260/230的比值. DNA的OD260/280比值大于1.8,小于2.1;OD260/230的比值均大于2.0,小于2.5 (见表3和表4). 上述结果表明所提样品DNA均符合下游实验的要求,该试剂盒提取DNA时具有通用性,提取的DNA质量较高.
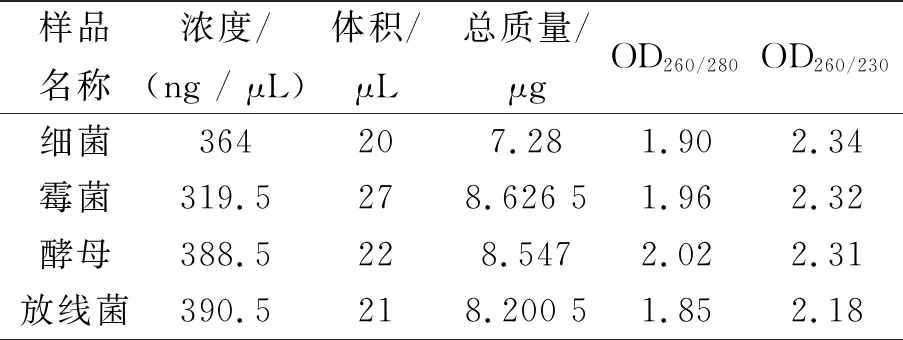
表3 NanoDrop检测提取孢子DNA质量结果
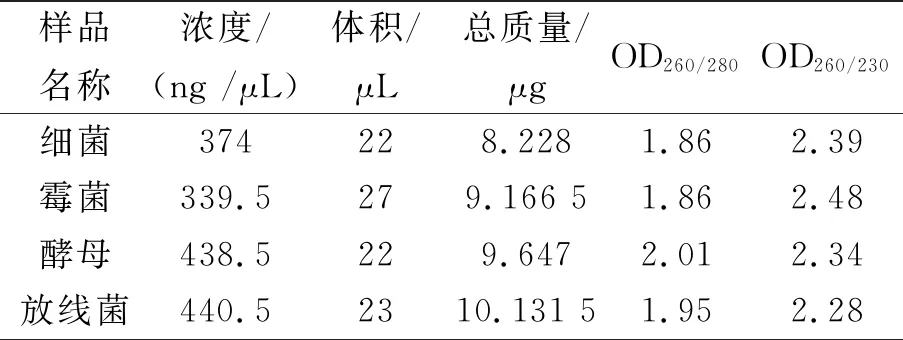
表4 NanoDrop检测提取菌体DNA质量结果
2.3 DNA质量的琼脂糖凝胶电泳检测
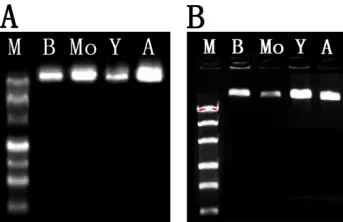
A固体培养基上的孢子DNA B液体培养基的菌丝或菌体DNA图4 琼脂糖凝胶检测的DNA质量
琼脂糖凝胶电泳条带的亮度和整齐度能直观评定DNA的完整性和高质量性. 由图4可知,提取的孢子和菌丝体DNA的电泳图像与Marker相比较,可以看出试剂盒提取的DNA电泳的条带整齐、明亮和Marker主条带无差异. 上述结果表明:该试剂盒提取的DNA质量和浓度均能满足下游实验的需要.
3 讨论
CTAB法是传统的提取DNA的方法,它操作过程相对简单,价格低廉. 但是传统的CTAB法使用了氯仿和β-巯基乙醇等不利于人体健康的试剂[13]. 市场上和临床试验中应用的800个微生物源天然化合物中[8],来源于放线菌的抗生素占整个抗生素的67%(链霉菌占52%,稀有放线菌占15%). 故利用分子生物学的方法研究这些微生物是必然的趋势[14]. 分子生物学研究方法的首要步骤就是提取目的DNA[9].
各种微生物细胞壁的结构成分差别很大,例如真菌细胞壁主要成分为几丁质,与细菌相比其结构要坚固得多. 在提取DNA时该菌类的培养时间尽可能短,一般以20 h左右为最佳. 而本试剂盒,从4种菌的孢子和菌丝体中均提取到了高质量的DNA,说明了该试剂盒的通用性. 含多糖聚合物 DNA 的提取比较困难[15],本试剂盒在传统的CTAB法上增加了HiBind© DNA column吸附DNA,然后再收集DNA,这样降低了多糖聚合物的污染,获得了高质量的DNA.
