基于三维全卷积网络的肝脏和肝癌分割算法研究
2019-09-25凌彤辉
凌彤辉
(1.中国科学院 上海技术物理研究所,上海 200083; 2.中国科学院大学,北京 100049)
0 引言
肝癌是仅次于肺癌的第二大高致死率癌症,也是中国和全世界发病率和死亡率增长最快的癌症之一。虽然计算机断层扫描(computed tomography,CT)已广泛应用于肝癌的筛查、诊断和治疗评估,但对CT图像中的肝癌进行定性和定量分析需要花费医生大量时间,且医生的分析结果具有主观性。在计算机技术和人工智能的推动下,计算机辅助诊断(computer aided diagnosis,CAD)系统快速发展。CAD可以对医学影像进行自动化处理,对医学影像中的肝脏和肝癌进行智能化分析,为医生提供诊断依据。在对肝脏和肝癌智能分析中,肝脏分割和肝癌分割是必不可少的一步。然而,由于肝脏与周边组织的对比度低,相邻器官边界模糊,形状变化大,自动分割肝脏是一项具有挑战性的任务。同时,肝癌形态呈弥漫性、不均匀性和稀疏性,大小不一,特征不明显,自动分割肝癌是更加艰巨的任务。
肝脏分割算法主要分为传统肝脏分割算法和神经网络的方法。传统肝脏分割算法主要有三种:基于图像灰度的肝脏分割算法[1-3],基于模型的肝脏分割算法[4-6]和基于纹理特征的肝脏分割算法[7-9]。例如Bae等(1993)[3]提出了一种基于图像灰度的肝脏分割算法。Wu等(2016)[6]提出了一种基于图割的肝脏分割算法。Luo等(2009)[7]提出了一种基于纹理特征和SVM的肝脏分割算法。传统肝脏分割算法都存在不足之处: 当存在边界模糊时,基于灰度的算法准确度较低;基于统计形状模型的算法对存在形变的肝脏无法准确分割;基于纹理特征的算法依赖于特征分类器和人工提取的特征。神经网络的方法主要分为二维全卷积网络[10-11]和三维全卷积网络[12-13]。例如郭树旭等(2017)[14]采用了改进的二维全卷积网络对肝脏进行分割;孙明建等(2018)[15]针对肝脏分割问题,则采用三维全卷积网络结合条件随机场的方式,取得了较高的准确率。二维全卷积网络的分割准确率一般低于三维全卷积网络,因为二维全卷积网络无法利用CT影像中的三维空间信息。
肝癌分割是在肝脏分割的基础上进行计算。肝癌分割算法目前主要采用神经网络的算法,例如Kaluva等(2018)[16]采用一个新型二维全卷积神经网络网络进行肝脏分割和肝癌分割,通过加入简单的形态学操作去除假阳性。Yuan等(2018)[17]采用了3个二维全卷积网络,前两个网络用于对肝脏进行粗分割和细分割,第三个网络用于对肝癌进行分割。
为了解决肝脏和肝癌的准确分割问题,本文提出了基于三维全卷积网络的肝脏分割算法和肝癌分割算法。肝脏分割和肝癌分割算法都采用Vnet[13]网络搭建而成。本文在Vnet网络中加入了批归一化(Batch Normalization, BN)[18],提高了模型的收敛速度。在肝脏分割中,通过对Vnet网络的分割结果进行后处理,显著提高准确率。在肝癌分割中,采用了组合损失函数来训练Vnet网络,让模型更好地收敛,并加入后处理提高了肝癌分割准确率。本文针对肝脏分割和肝癌分割进行了实验,利用公开数据集验证了算法的性能。
1 Vnet网络
Vnet网络是在全卷积神经网络[10]基础上提出的一个新型三维全卷积网络。Vnet 网络结构成编码器-解码器结构, 左半部分为编码过程,由卷积层和池化层组成,右半部分为解码过程,由卷积层和反卷积层组成。Vnet网络编码部分和解码部分拥有相同的卷积层数。网络结构如图1所示。其中卷积层的作用是提取图像特征,池化层的作用是降低数据维度,反卷积的作用是恢复图像尺寸。
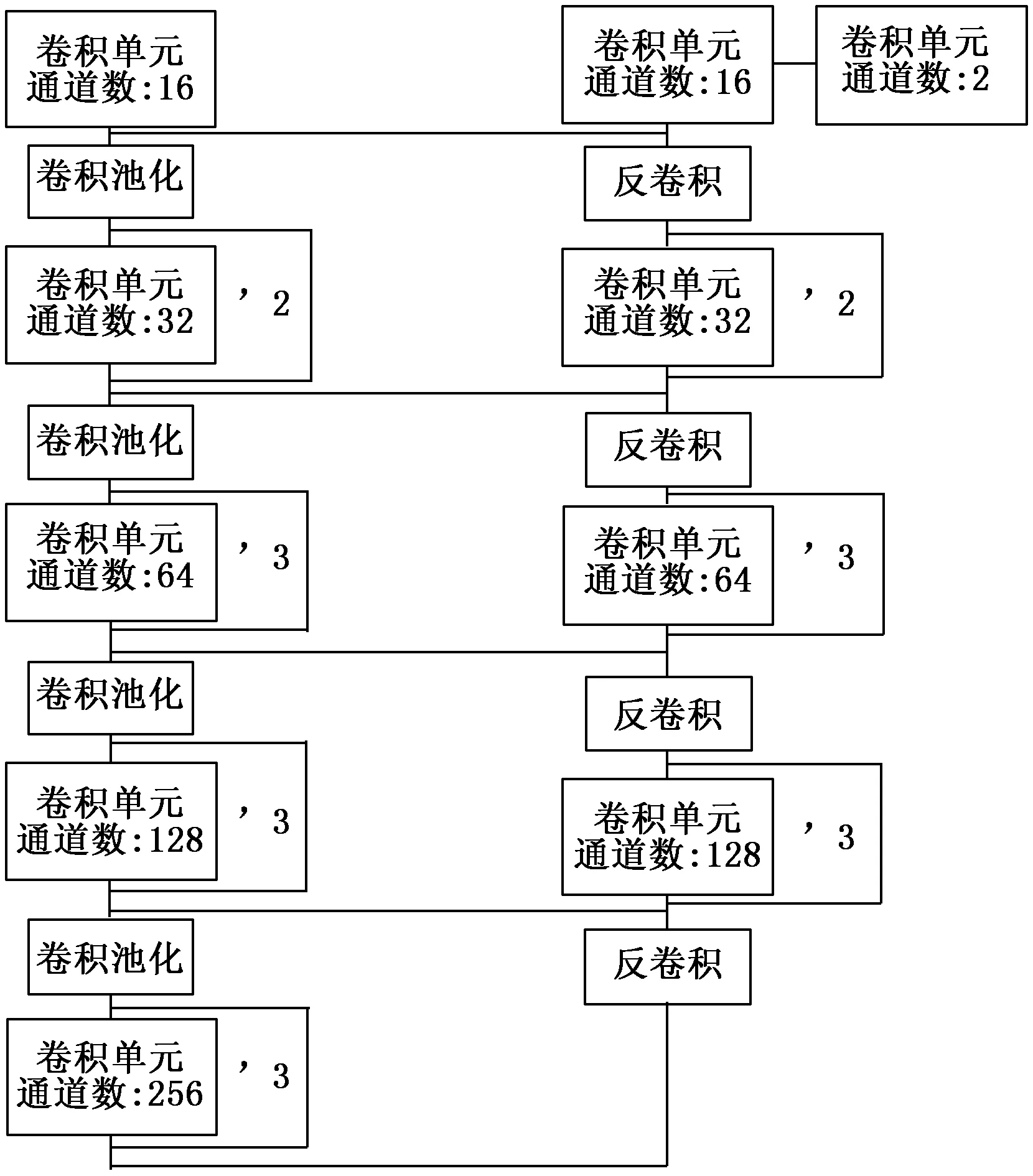
图1 Vnet网络结构
Vnet网络在卷积层当中采用了Resnet(He等,2016)[19]的残差学习结构,即将卷积层的输入与卷积层最后的输出进行加和,再进行后续计算,从而缓解了梯度消失的问题。每个卷积层包含1到3个卷积单元。每个卷积单元由两部分组成:卷积和激活函数。Vnet网络中的卷积采用5×5×5的卷积核进行卷积,padding大小为2,stride为1。因此卷积后得到的特征图大小不变。Vnet网络采用的激活函数为PReLU函数。本文为了提高模型的收敛速度在卷积单元中加入了BN。BN就是通过计算均值和标准差把神经元的数值分布变回均值为0,方差为1的标准正态分布,从而达到加快模型收敛和缓解过拟合的效果。
编码和解码中间为级联层。级联层的作用是将编码部分的特征图与解码部分中等大小的特征图进行拼接,再进行卷积,从而弥补了下采样过程中丢失的细颗粒度图像特征和位置信息。Vnet网络的池化层采用了卷积池化的方式。即使用2×2×2 的卷积核对特征图进行卷积,步伐大小为2,从而达到池化的效果。卷积池化的好处在于训练网络的时候占用更少的内存。
Vnet网络整体属于端到端的网络结构,输入图像便可直接得到分割结果。Vnet网络在最后一个卷积层后,使用卷积核大小为1×1×1的卷积将特征图像进行组合压缩,得到两个和原图像尺寸大小相同的特征图,然后对这两个特征图使用Softmax函数,得到原图像中每个体素属于前景和背景的概率,如果前景概率大于背景概率,则预测为前景目标,标记为1,如果前景概率小于背景概率,则预测为背景,标记为0。
2 肝脏分割
本文采用Vnet网络对CT影像中的肝脏进行分割。算法流程如图2所示。具体流程如下:首先对CT影像数据进行预处理,然后将预处理后的图像尺寸缩小4倍,之后将缩小后的图像尺寸裁剪为大小128×128×128,再使用训练好的Vnet网络对图像数据进行计算,最后使用后处理优化分割结果并还原为原图像尺寸,从而得到肝脏的分割结果。

图2 肝脏分割流程图
2.1 预处理
预处理主要包括以下三步:分辨率标准化,调窗处理和灰度值标准化。CT影像由于机器的差异或者参数设置的差异,其空间分辨率大相径庭,如果没有进行分辨率标准化,将会影响肝脏分割准确度。针对肝脏分割,本文采用了分辨率0.69×0.69×1.0 mm3进行标准化。分辨率标准化的方式是根据标准分辨率和自身分辨率,计算缩放倍数进行缩放。缩放的方式为线性插值。调窗处理即选择灰度值范围,所有大于窗口最大值的灰度值修改为窗口最大值,所有小于窗口最小值得灰度值修改为窗口最小值。该操作可以过滤CT影像中不相关的组织和器官,从而降低算法分割难度,提高分割准确率。针对肝脏分割,选择灰度值范围为[-250,250]。灰度值标准化是指将灰度值减去灰度值的均值,再除以灰度值的标准差,灰度值标准化的目的是为了方便数据处理和加快模型收敛。灰度值的均值和标准差是通过统计训练数据计算得到的。
2.2 模型训练
Vnet网络属于有监督学习算法,因此网络模型的权重参数需要通过训练得到。训练Vnet网络分为两部分:制作训练集和训练模型。制作训练集就是将提供的CT数据预处理,然后缩放裁剪为大小128×128×128。训练模型需要选择梯度下降算法、选择损失函数和设置相关的超参数。本文选择了Adam自适应梯度下降算法[20]来拟合参数,损失函数选择为Dice损失函数。Dice损失函数描述的是图像分割结果中预测区域和真实区域之间的重合程度。公式如下:
(1)
其中:N代表体素块的体素数量;pi代表网络的预测结果;gi代表对应体素真实标记。
训练Vnet网络的学习率设置为0.001,梯度系数设为0.9,平方梯度的系数设为0.999,权重衰减系数设置为1×10-8。为了提高模型的泛化能力,加入了数据增强。增强方式包括:在制作训练数据时,加入随机平移;在训练模型中加入动态的随机旋转和动态B样条插值。
2.3 后处理
后处理是为了优化分割结果。这里采用了形态学方法来进行后处理:首先对分割结果中标记为1的连接通量进行腐蚀,然后只保留分割结果中的最大连接通量,最后再对最大连接通量进行膨胀。通过上述操作可以有效减少分割结果中的假阳性。
3 肝癌分割
本文采用Vnet网络来对肝脏中的肝癌进行分割。算法流程如图3所示。具体流程如下:首先是根据肝脏分割结果,提取肝脏区域,然后再对数据进行预处理,之后将CT图像横断面尺寸缩小为256×256,再采用训练好的Vnet网络对肝脏区域进行扫描计算,最后进行后处理并还原为原图像尺寸,从而得到肝癌的分割结果。肝癌分割的Vnet网络输入大小为256×256×16。

图3 肝癌分割流程图
3.1 预处理
肝癌分割的预处理包括调窗处理,三维区域直方图均衡化和灰度值标准化。肝癌分割不需要分辨率标准化,是因为分辨率标准化会导致部分CT影像中的小型肝癌丢失。直方图均衡化的用途是为了提高肝癌和肝组织之间的对比度,从而提高肝癌的分割准确度。
3.2 模型训练
肝癌分割和肝脏分割都采用Vnet网络结构。不同的地方在于:肝癌分割的Vnet网络的输入大小为256×256×16;计算的图像数据只包含肝脏区域;需要采用扫描计算的方式对整个肝脏区域进行计算。肝脏分割只需要Vnet网络对整体计算一次。
肝癌分割的Vnet网络计算的图像数据只包含肝脏区域,因此训练数据也只需要包含肝脏区域。在根据真实标记提取肝脏区域后,对训练数据进行预处理。为了减少类别不平衡,只采用数据中包含肝癌的切片做成训练集,因为肝癌分割和肝脏分割都采用Vnet网络结构,因此在训练肝癌分割的时候,采用训练好的肝脏分割Vnet网络的权重参数作为肝癌分割Vnet网络的初始化参数,并在此基础上进行微调,相应的学习率设置为0.0001。梯度下降算法同样采用Adam算法。
在CT影像中,肝癌形态呈弥漫性、不均匀性和稀疏性,因此对肝癌分割会出现严重的类别不平衡问题。图像分割本质上是像素级的分类问题,当出现类别不平衡问题,分割准确率会大幅下降。为了能让Vnet网络在类别不平衡的情况依然能够被有效地训练,本文采用了组合损失函数。该损失函数由Dice函数和带权重的交叉熵损失函数组成,公式如下:
E=EDice+ECrossEntropy
(2)
交叉熵损失函数的权重是通过统计训练数据中肝癌和背景的比例得到的。Dice损失函数本身对类别不平衡问题较为不敏感,带权重的交叉熵损失函数则利用了阈值移动的原理来应对类别不平衡问题。通过组合这两个损失函数可以让模型更好地收敛,提高模型性能。
训练肝癌分割的Vnet网络也加入了数据增强,包括:在制作训练数据时,加入随机平移;在训练模型中加入动态的随机旋转。
3.3 后处理
肝癌分割结果中,部分被标记为肝癌的体素点位于肝脏区域外部。而肝癌必然位于肝脏区域内,基于这个先验,对这部分肝癌体素点直接剔除。
4 实验与讨论
4.1 实验数据
本文采用的数据集为MICCAI 2017 Liver Tumor Segmentation Challenge(LiTS)的数据集。LiTS数据集提供了130例带标注的腹部CT数据。这些腹部CT数据的轴向分辨率最小为0.55 mm,最大为1.0 mm,层厚最小为0.45 mm,最大为6.0 mm。数据提供了肝脏和肝癌的真实标记,可用于算法的训练和测试。
4.2 实验环境
实验所使用的硬件环境如下:Intel(R) Core(TM) i7-6700K CPU @ 4 GHz,内存(RAM)64 GB,显卡 GeForce GTX 1080两块。开发系统为Ubuntu 14.04,开发工具为python和pytorch。
4.3 实验评估方法
为了评估器官分割实验中算法的分割准确度,本文采用以下五个指标进行全面地评估,分别为:
A代表真实属于器官的区域,B代表预测属于器官的区域;S代表表面体素点,S(A)和S(B)代表A和B的表面体素点。
1)Dice系数:
(3)
2)体积重叠误差(Volume Overlap Error, VOE):
(4)
3)相对体积偏差(Relative Volume Difference, RVD):
(5)
4)平均对称面距(Average Symmetric Surface Distance, ASSD):
(6)
d(v,S(A))表示体素v到表面体素S(A)的最短欧式距离。公式如下:
(7)
5)最大对称面距(Maximum Symmetric Surface Distance , MSSD):
(8)
Dice系数的值越接近1,则分割越精准。VOE数值越小代表分割准确度越高。RVD值为0时,分割算法是最优的。ASSD和MSSD都是值越小,表明分割效果良好,当值为0时,分割结果最好。
4.4 实验设计
为了保证实验结果的准确性,本文采用5折交叉验证进行实验,即随机将130例数据分为5组,每组包含26例数据。每次挑选其中的1份作为测试集,剩下的4份作为训练集。然后进行5轮训练和测试。最后统计5轮测试结果的平均值作为最终测试结果。为了防止训练过拟合,每次训练需要从训练集抽取15例数据作为验证集。
在训练肝脏分割和肝癌分割的Vnet网络中,batch size的大小都设置为4,训练迭代次数都设置为200,在出现明显过拟合的时候停止训练。
4.5 实验结果
利用LiTS数据集完成5折交叉验证实验,平均测试结果如表1所示。

表1 测试结果
通过表1可以看出基于Vnet网络的肝脏分割算法在交叉实验中平均Dice系数达到0.951,说明算法可以准确地对肝脏进行分割。基于Vnet网络的肝癌分割算法的平均Dice系数达到0.712,说明算法可以对较为准确地对肝癌进行分割。
随机从测试集中抽取一例CT数据,并选择其中一层切片。对该切片的算法分割结果和真实标记进行可视化并对比。对比图如图4所示。通过该图可以发现,肝脏分割算法的分割结果和真实标记基本相同,而肝癌分割算法的分割结果中大型肝癌可以较为准确地分割,小型肝癌则存在一处漏检,原因在于小型肝癌特征不明显,分割难度较大,容易出现漏检。

图4 分割结果对比图
4.6 讨论
本文提出了基于Vnet网络的肝脏分割算法,采用了Vnet网络分割结合后处理的方式。为了对比肝脏分割算法的性能,同样采用5折交叉验证实验验证了Unet[11]网络和3D Unet[12]网络的性能。Unet网络为二维全卷积网络,在二维图像分割上准确度较高,被广泛应用。Unet结构上也为级联对称结构,通过级联层补充压缩过程中丢失的信息。3D Unet网络则是在Unet网络基础上提出的三维全卷积神经网络,和Unet网络不同的地方在于数据维度,网络深度和卷积核的数量。Unet网络和3D Unet网络的平均测试结果如表2所示。表中还罗列了未加后处理的Vnet网络的平均测试结果。

表2 肝脏分割测试结果对比
从测试结果可以看出,Vnet网络加后处理的方式在测试集上的各项指标都优于单一Vnet网络,说明后处理的加入有效去除了分割结果中的假阳性,提高了分割准确率。3D Unet网络在测试集的各项指标低于Vnet网络,主要原因在于3D Unet网络的网络深度和感受野较都小于Vnet网络。Unet网络的在测试集的表现则明显低于Unet网络和Vnet网络。其原因在于Unet网络作为二维全卷积网络无法利用CT影像中的空间信息,即切片间的相关性。三维全卷积神网络则可以很好的利用空间信息,因此三维全卷积神经网络准确率是更高的。
本文在Vnet网络结构中加入了BN来加快模型收敛。经过实验,加入了BN后的Vnet网络平均在第96次迭代时,验证集Dice系数达到最高值,而训练未加入BN的Vnet网络平均在第121次迭代才达到最高值,说明Vnet网络在加入BN后可以更快收敛。
本文在训练肝癌分割Vnet网络中,在训练好的肝脏分割Vnet网络的权重参数的基础上进行微调。使用微调的方式训练Vnet网络,验证集上Dice系数平均最高为0.741,而采用随机初始化参数的方式训练Vnet网络,验证集上Dice系数平均最高为0.728。说明采用微调的方式,肝癌分割的Vnet网络收敛效果更好。
本文在肝癌分割算法中采用了组合损失函数,目的是为了应对类别不平衡问题。使用组合损失函数相比于使用单一的Dice损失函数和单一的交叉熵损失函数,模型的收敛效果会更好。本文分别使用单一的Dice损失函数、单一的交叉熵损失函数和组合损失函数训练Vnet网络。在验证集没有出现过拟合的情况下,采用单一的Dice损失函数在验证集上Dice系数平均最高为0.727,,单一的交叉熵损失函数为0.725,组合损失函数为0.741。说明组合损失函数相比单一损失函数可以让模型更好地收敛。
在肝癌分割算法中加入了简单的后处理:剔除不在肝脏区域的肝癌体素点。经过测试,不加后处理的肝癌分割准确率为0.707,加入后处理后肝癌分割准确率为0.712。说明该后处理有效提高了肝癌分割准确率。
5 结束语
本文提出了基于三维全卷积网络的肝脏分割和肝癌分割算法。采用了加入BN的Vnet网络进行肝脏分割和肝癌分割。通过加入后处理有效提高了肝脏分割的准确度。通过采用微调的方式和组合损失函数,更好地训练肝癌分割的Vnet网络。利用公开数据集验证了肝脏分割和肝癌分割的准确度。测试结果表明肝脏分割算法可以准确地对肝脏进行分割,肝癌分割算法达到较高准确度。后续需要针对小型肝癌的分割进行改进。
