植物DNA条形码研究领域文献计量学及可视化分析
2019-09-10熊勇李文义杨淬罗斌圣杨青松
熊勇 李文义 杨淬 罗斌圣 杨青松






摘 要:为全面了解植物DNA条形码研究领域的发展和最新动态,探讨中国DNA条形码发展的状态和前景,该文利用Web of Science 数据库对该研究领域进行文献计量学统计,并对引用频次、研究热点和研究前沿进行了可视化分析。结果表明:(1)中国、美国、加拿大学者在该领域文献贡献率最大,中国研究机构发文量领先,但美国、加拿大科研机构论文质量较高,影响力较大。(2)2009年是该领域研究的高峰期,该研究领域的前沿和研究热点主要集中在物种的识别和生物多样性应用、DNA条形码候选序列筛选和鉴定技术的规范化。(3)中国学者在植物DNA条形码领域研究具有领军作用和很高的影响力,国家提倡中药产业的发展也推动了我国DNA条形码蓬勃发展,但论文的质量和影响力与美国、英国、加拿大等发达国家研究还有一定差距,应加大与发达国家科研机构合作,提高研究能力,DNA条形码技术在植物的鉴定、分类和生物多样性的保护起到非常重要的作用。这表明建立一个更全面、通用的全球植物DNA条码库以及开发新的标记并采用新的测序技术是植物DNA条形码研究的未来前景。
关键词:植物DNA条形码, 文献计量学, 生物多样性, Citespace, 可视化分析
中图分类号:Q949.9
文献标识码:A
文章编号:1000-3142(2019)04-0557-12
Abstract:In order to obtain the latest information anddevelopment research filed ofdNA barcoding of plants, and todiscuss its role in biodiversity conservation, we used bibliometrics, Histcite and Citespaces to analyze literatures ofdNA barcoding in plants based on Web of Sciencesdatabase, and analyzed citation counts, research hot and research front visualization. The results were as follows:(1) The rate of contribution of literatures from Chinese, American, Canadian authors was the highest. Chinese institutions published literatures counted more than others countries, while literatures from American and Canadian institutions had much higher quality and influence. (2) This research field reached the peak at 2009, and the research front and hot focused on species identify and biodiversity application, find the suited candidatesdNA barcoding sequences and refining the identical technology. (3) Chinese authors played a leading role and had strength influence atdNA barcoding of plants. Chinese government encouraged traditional Chinese medicine industry to promote thedevelopment ofdNA barcoding technology at China. But the quality and influence of published literatures had a gap between China anddeve-loped countries, and Chinese institutions should increase cooperation with thedeve-loped countries institutions to promote itself research ability. It is very important thatdNA barcoding technology at species identify and biodiversity conversation. Tomorrow’s outlook for plantdNA barcoding is to build a global plantdNA barcode library, newdNA markers and new sequencing technologies.
Key words:DNA barcoding of plants, bibliometric, biodiversity, Citespace, visualization analysis
DNA條形码技术是利用标准的基因片段对物种进行快速鉴定(Hebert et al., 2003), 该技术提供了可信息化的分类学标准和有效的分类学手段,已经被成功用于生物物种鉴定和分类(Liu et al., 2011),生物多样性调查(Lahaye, 2008)和生态学研究(Valentini, 2009)等领域,并成为进展最迅速的学科前沿之一。生命条形码数据 (BOLD) 系统提供了主要针对动物类群dNA 条形码研究的技术规范,由于植物本身的生物学特性与所使用的条形码不同,植物DNA条形码研究相对滞后些,然而其作为一个DNA条形码的研究领域重要内容之一,植物DNA条形码不仅受到植物学研究的影响,又同时受到了分子生物学和生物信息学相互渗透和影响。DNA条形码在植物中研究及应用不断增多, Blaxter(2004)对DNA条形码的在分类中应用前景进行了分析,Kress et al.(2005)利用DNA条形码技术鉴定开花植物,Chase et al.(2007)提出关于陆生植物条形码的标准化方法的建议,2009年CBOL Plant Working Group开展DNA条形码技术在陆生植物的应用,2017年Bashir Mohammed Abubakar 对DNA条形码在草药产品中的鉴定应用进行回顾和总结(Mohammed et al., 2017),DNA条形码技术在植物上的研究和应用在源源不断的增加。
文献计量学是利用数学和统计学方法来分析相关知识载体,从而获取有价值信息的一门交叉学科(赵蓉英和许丽敏, 2010)。一个学科或一个研究领域的发展情况,必然表现在相关论文的发表和被引用方面,因此文献计量学可以用于分析、描述学科发展状况和预测学科发展趋势,再通过绘制知识图谱将结果以图形的形式直观、形象地表达出来(顾洪涛和王筠, 2013)。目前用文献计量学对DNA条形码在植物上的研究领域总体分析见报道的虽然不多,但国内外已经有很多基于Web of Science数据库对文献进行文献计量学统计分析,如孙秀焕和路文如(2012)基于Web of Science分析了水稻研究态势,杨华等(2013)进行了国际茶多酚类研究文献发展态势研究,Garfield(2009)利用Histcite软件对科学史进行可视化分析。本研究基于Web of Science 数据库,对2003—2016年植物DNA条形码国际文献进行文献计量学统计和分析,再利用Histcite和Citespace软件对该研究领域进行了可视化分析,达到对植物DNA条形码研究领域文献信息全面了解。
1 数据来源与数据处理
1.1 数据来源
以Web of Science(WoS)核心合集为数据来源,时间跨度为2003—2016年,以(“DNA barcoding” or “DNA bar coding” or “DNA barcode” or “DNA barcodes”) plant為Topic进行精确检索,下载文献信息保存为纯文本文档,为后续分析做准备。
1.2 数据处理
1.2.1 Web of Science文献处理 通过在线http://ip-science.thomsonreuters.com/thanks/histcite/ 网站,获得Histcite软件,同时把Web of Science获得的文献信息整合为一个完整的txt文档,导入Histcite软件,基于Web of Science创建引文报告和分析检索结果以及Histcite统计,分析的内容主要包括:每年出版的文献量、被引频次、核心作者、出版的期刊、主要研究机构和国家/地区等(Shadbolt et al., 2013; Bharathi, 2013)。
1.2.2 Histcite文献处理 以LCS作为节点,再用其中的Graph maker制作引文编年图,探究文献间的引用关系及获得重要文献。
1.2.3 研究热点的可视化分析 将分析后获得的文献信息导入Citespace软件中,对该领域研究热点、研究前沿和突现点进行可视化分析。
2 结果与分析
用植物DNA条形码为主题在Web of Science 核心合集上进行检索,共获得检索结果为1 097条,这些论文的被引频次总计为27 125次,去除自引频次18 922次。
2.1 发文量和引文量分析
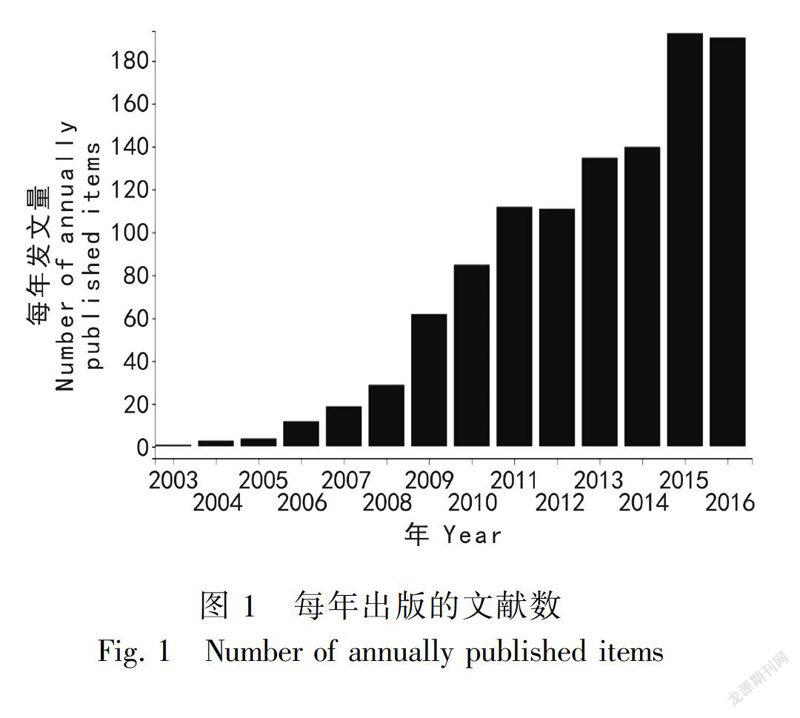
基于Web of Science 核心合集,时间段位2003—2016年,以DNA条形码为关键词进行检索,用Web of Science在线工具进行引文报告分析,通过创建引文报告获得每年文献量和被引频次(图1,图2)。
2.2 论文被引数量分析
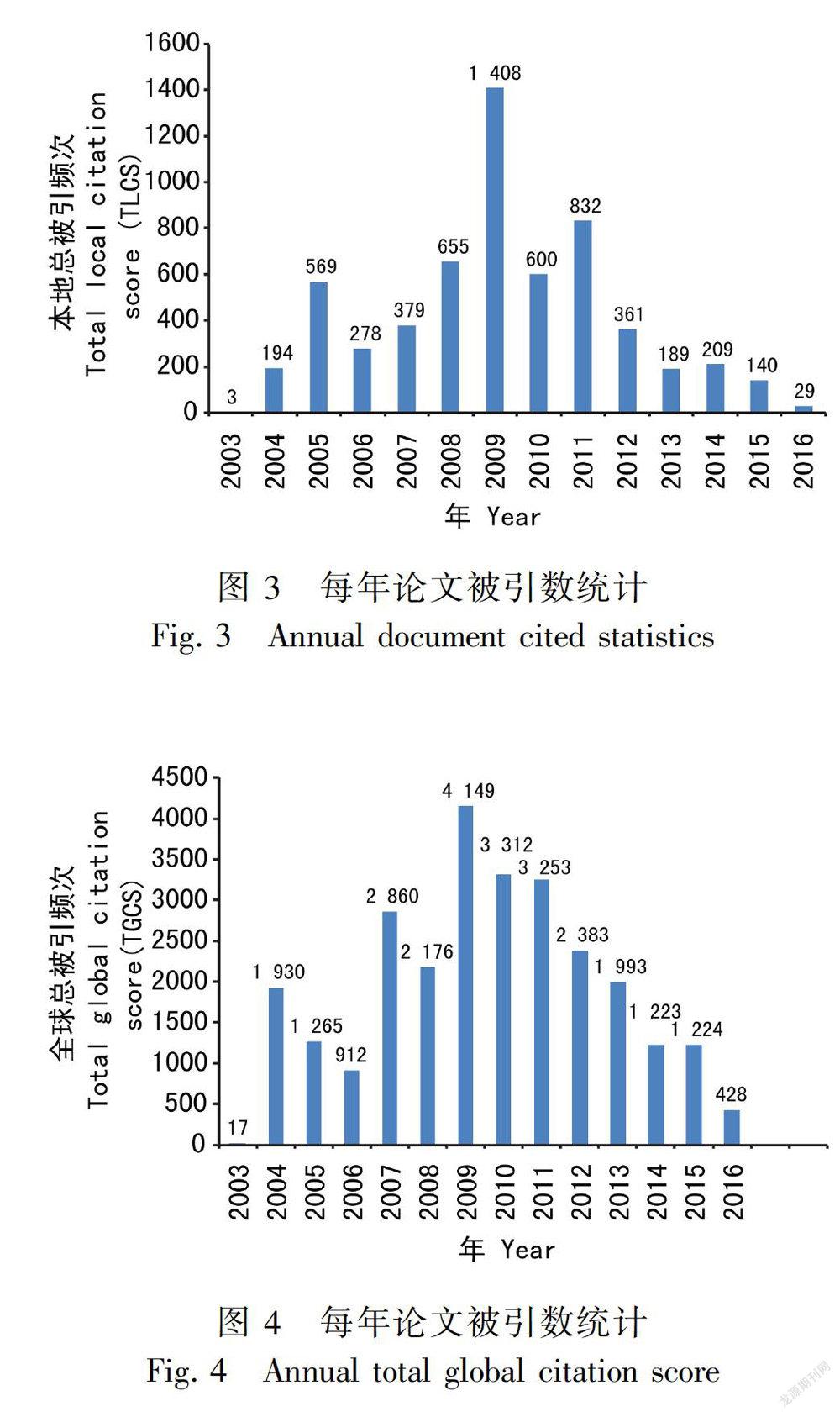
对获得原始文档的适当修改,导入Histcite软件,进行LCS(Local Citation Score )和GCS(Global Citation Score)指标分析(图3,图4)。LCS是指论文在本地数据集(WoS中输入关键词后导出的所有文献)中被引用次数,LGS值越高表示该论文在该研究领域内的重要性高,GCS值指论文在整个WoS数据中的总被引次数,但施引论文不一定是该领域内的论文。
图3和图4为2003—2016年每年TLCS(Total Local Citation Score)和TGCS(Total Global Citation Score)的统计图。从图3和图4可以看出,TLCS和TGCS都在2007—2012年这几段达到较高水平,而后半段2013年开始都呈下降趋势,这种趋势与每年的发文量和论文引用数量不一致。TLCS和TGCS在2009年都达到了高峰期,2009年发的文献对该领域研究具有重要的作用和很大的影响力,其中CBOL Plant Working Group、Hollingsworth ML等合作发表的论文“AdNA barcode for land plants”和“Selecting barcoding loci for plants:evaluation of seven candidate loci with species-level sampling in threedivergent groups of land plants”(Hollingsworth et al.,2009),前者对现有的7条候选DNA条形码进行对比,推荐rbcL + matK组合作为陆生植物DNA条形码的标准,后者是对陆生植物DNA条形码提出了7条候选序列(atpF-atpH spacer, matK gene, rbcL gene, rpoB gene, rpoC1 gene, psbK-psbI spacer, and trnH-psbA spacer),两篇论文对后面陆生植物DNA条形码的研究起到了指导性作用。从后面的文献被引用频次的可视化分析得出,依据TLS值获得排名前30的论文,其中2009年附近文献就有13篇,可见2009年是植物DNA条形码的研究的高峰期和热点区。
2.3 核心作者分析
把获得文献信息导入Histcite软件进行分析,获得2003—2016年发表有关植物DNA条形码英文论文的作者有4 023位,筛选出发文量前10的作者(表1),其中有5名为中国学者,说明中国在这一领域研究文献贡献率很大,但从文献的被引用频次上和影响力上,与国外学者有一定差距,核心作者中是美国Smithsonian Institution的 Kress WJ的TLCS最高,其次是加拿大University of Guelph 的Newmaster SG,中国中医科学院中药研究所陈士林教授在中药材DNA条形码研究具有很高的成就,所编写《中国药典中药材DNA条形码标准序列》是该行业指导性书籍,发文量第二是中国医学科学院药用植物研究所的宋经元教授,在中药DNA条形码鉴定研究中起到领头羊的作用。
2.4 国家(地区)和研究机构分析
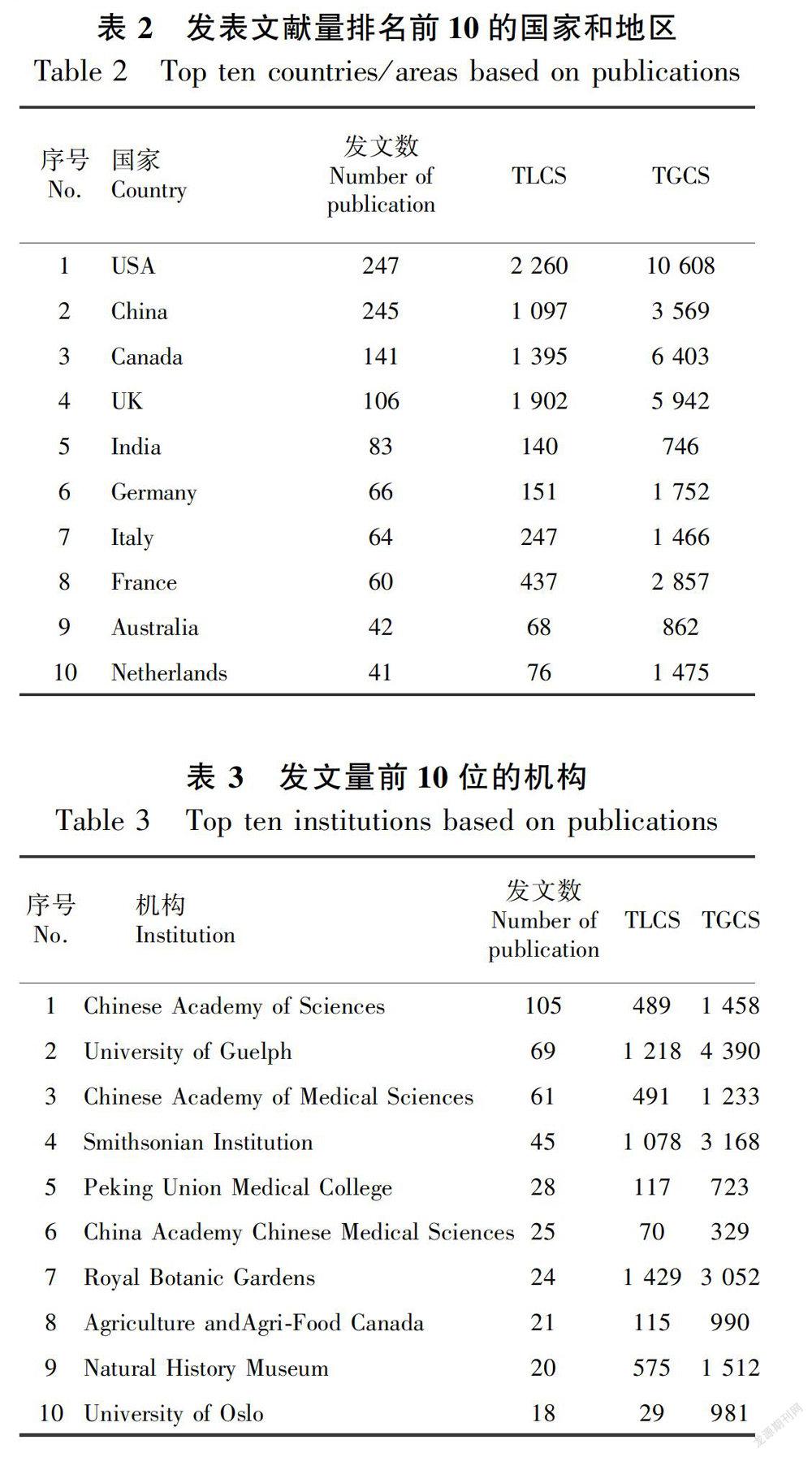
对国家(地区)和研究机构文献量进行统计,排名前10见表2和表3。从表2可以看出,发文量前五的国家都是在DNA条形码研究领域进行比较早或者具有植物研究传统国家,美国和中国发文量最多,且数量相差不大,与其他国家(地区)相比优势很明显,但从文献的影响力和被引用频次来看,中国与美国、加拿大和英国都有差距,可能跟我们国家科技发展相对滞后或文化有关,但是从前10名国家(地区)上看,该领域的研究主要集中在科技相对发达的国家和地区,中国对该领域的研究也有很多成绩和贡献。
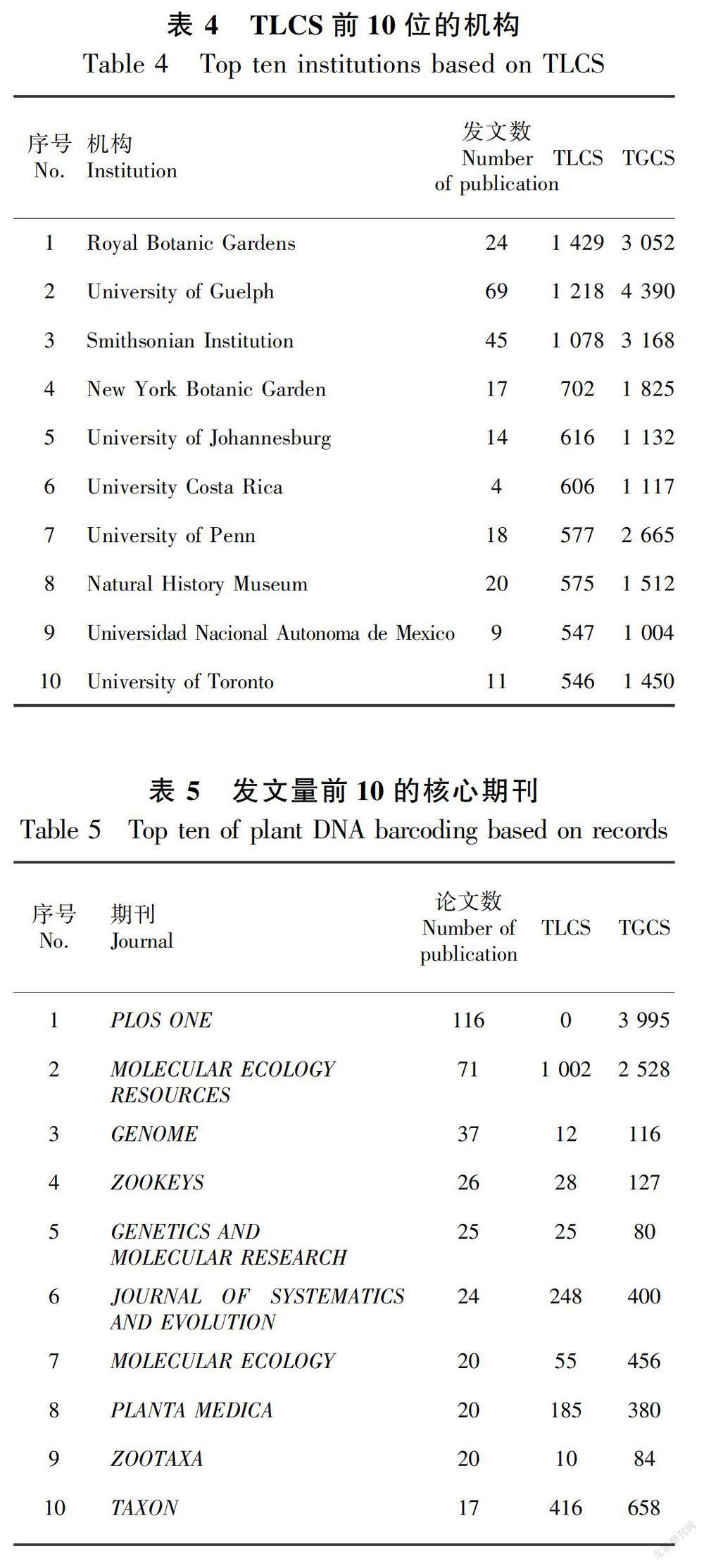
从表3的研究机构前10名可以看出,与核心作者和国家相似的结果,进行植物DNA条形码研究主要机构还是集中在3个国家,中国、加拿大、美国,中国科学院发文量最多。以TLCS为指标进行排序(表4),前3名为英国Royal Bot Gardens,加拿大的University of Guelph和美国的Smithsonian Institution,前10名研究机构没有中国研究机构。因此,从发文量来看, 前10名中有四个中国研究机构,说明我国有很多学者在进行植物DNA研究,并且也获得不错的成果。
2.5 核心期刊
对于学术期刊的发文量与引文量统计不仅可以让我们知道不同期刊在该领域的影响力,也能为学者们选择目标期刊投稿与参考文献研究提供有價值的参考依据。对相关的期刊进行分析,总共有338个期刊发表于植物DNA条形码相关论文,以发文量进行排名,前10名期刊见表5,发表在PLOS ONE期刊上的文献量最大,其次是MOLECULAR ECOLOGY RESOURCES和GENOME,核心期刊中大部分都是与分子和系统进化相关,码引文编年图(图5)。由图5可知,从上到下是年份(2004—2011),箭头指向的文献是被引用的文献,方框内所标数字是该节点文献在所分析文献中的序号,文献引用频次越高方框越大,就是该领域比较重要的文章。图5展示植物DNA条形码研究不同文献之间引证的关系链,文献之间关系紧密,其中序号为4、5、8、28、48、110和326是被引用频次较多的文献,这些文献对该领域的研究具有重要作用。文献序号5和序号110方框最大,被引用的频次最多,文献序号5是Kress et al.(2005)利用DNA条形码技术鉴定开花植物,该论文是较早使用DNA条形码技术应用于植物研究,序号110 是CBOL Plant Working Group 合作所写关于DNA条形码技术规范具有指导性的一篇论文。从年份我们看出,2009年文献TLCS排名前30最多一年,是研究的热点和高峰期,2009年对于植物DNA条形码候选序列是一个寻找和验证的时期,对寻找适合的DNA条形码起到了奠定作用,而到了2011年对植物DNA条形码具有很大发展,多了ITS序列(序号326)(Li et al., 2011)和对现在候选序列的改善(文献序号325)(Hollingsworth, 2011)。
我们还可看出,被引频次排在前3位的均是发达国家研究机构,可见DNA条形码研究的高质量和影响力大的论文还是集中在发达的西方国家研究机构。植物DNA条形码候选序列文章一经发表,就引起了相关学者的广泛关注,说明DNA条形码技术的规范化和对候选序列的优选是最前沿课题。
2.7 热点分析
对于一个学科的范式可视化研究主要可分为基于文献的共引分析(Co-citation)和基于词或词组的共词分析(Co-word),相较之下共引分析更广泛应用于系统揭示学科结构、学科的发展规律和发展趋势(伍若梅和孔悦凡,2010)。植物DNA条形码研究领域是一个交叉学科,学科环境内不同子领域之间较难有共现的高频关键词,共词法难以反映领域间知识的输入输出。综合考虑,本研究选择共引法来分析植物DNA条形码的学科发展的热点与前沿。利用Citespace软件,对所得文献数据进行共引分析,时区选择(Timespan)为2003—2016年,时间跨度(Slice Length)为1 a,节点类型选择“Cited reference”,而筛选阈值Thresholds(c;cc;ccv)(c为节点出现频次、cc为共同出现频次、ccv为共现率)被设定为(2,2,5;3,2,10;3,3,15)。得出图6和图7两种形式的聚类分析图,而每个聚类代表着该领域的一个研究前沿。经过参数筛选,图谱中共出现了255个点(每个点代表一篇文献),441条连线,其模块值(Modularity Q)为0.710 8,轮廓值(Mean Silhouette)为0.563 3,表明形成的聚类结构显著、置信度高且有意义。此外,采用LLR算法(对数似然率算法)从施引文献的关键词(K,Keyword list)中提取名词性术语对聚类进行命名(Cluster Labeling),命名结果统计如表7。
从图6可以看出,出现大的13个关键词:DNA barcoding、DNA barcode、land plant、identification、sequence、taxonomy、diversity、species identification、evolution、phylogeny、rbcl、plant和region, 从而把植物DNA条形码研究领域分为13个方面的聚类研究前沿,关键词字体越大表示研究的文献越多,从中得出,DNA条形码在物种的鉴定、分类、进化、系统发生、生物多样性和陆生植物的应用等具有重要的作用,也是该领域研究的前沿和热点。
表7显示了不同的聚类,Cluster ID 为聚类后的编号,聚类的规模越大(也就是聚类中包含的成员数量越多),则编号越小,Size代表的是聚类中所含有的成员数量,Silhouette为衡量真个聚类成员同质性性指标,该数值越大,则代表该聚类成员的相似性越高,Mean year 代表的事该聚类中文献的平均年份,判断聚类中引用文献的远近,表7只列出聚类LLR(Likelihood Rate)前10名。
综合分析图6和图7及表7,同时参考聚类中的施引文献,可以大致了解到每个聚类所代表的研究前沿。表7中共总结了前10大植物DNA条形码的研究前沿,主要的聚类如下:
聚类0主要关注的是用植物DNA条形码的方法对一些难于鉴定的样品进行鉴定,如对食草动物(家畜和野生动物)的排泄来鉴定动物的饮食成分,代表论文“UniversaldNA-Based Methods for Assessing thediet of Grazing Livestock and Wildlife from Feces”(Pegard et al., 2009)。聚类1主要关注的是核糖体DNA(ITS序列),在植物多样性上的应用,代表论文“Confirming the Genetic Identity ofdendrobium fimbriatum Using an Amplification Refractory Mutation System (ARMS)”(Lu et al., 2010)。聚类2 利用DNA条形码对不同物种进行快速的鉴定方法,如在真菌鉴定中的应用,代表论文“Current state and perspectives of fungaldNA barcoding and rapid identification procedures”(Begerow et al., 2010)。聚类3dNA条形码在不同地区药用植物的应用,如在土耳其山区对当地药用植物的应用,代表论文“DNA barcoding study on sideritis trojana bornm. an endemic medicinal plant of ida mountain, turkey”(Tezcan et al., 2010)。聚类4 主要关注DNA条形码在植物进化、 生物信息学和资源上的应用,代表论文“DNA barcoding in plants:Evolution and applications of in silico approaches and resources”(Bhargava & Sharma, 2013)。聚类5主要关注的是DNA条形码在各个科分类鉴定中的应用,如“DNA barcoding of Orchidaceae in Korea”(Kim et al., 2014)。聚类6 利用核DNA和叶绿体DNA的方法对物种进行鉴定,如“Species identification of Alnus(Betulaceae) using nrDNA and cpDNA genetic markers”(Ren et al., 2010)。
从图8可以看出,2003—2016年这段时间植物DNA 条形码在某一时段的研究突破点或转折点,突现的引文节点用红色表示。23个主要的关键词中得出最大的节点是2008—2011年的鉴定(identify)关键词,最小为2007—2009年的生命(life)关键词。DNA条形码利用标准的基因片段对物种进行快速鉴定是研究的前沿,也是DNA条形码的主要作用和价值,结合前面的每年论文被引数统计和被引频次的可视化分析,可知2008—2011年是DNA条形码领域研究的高峰期和热点区,对该领域的具有巨大的影响和贡献。
3 讨论
本研究通过Web of Science数据库对DNA条形码进行检索,基于文献计量学方法,从每年出版的文献量、被引频次、核心作者、出版的期刊、主要研究机构和国家/地区、研究热点进行了较全面的计量分析,并对被引频次、研究前沿进行了可视化分析。
(1)在国家/地区分布方面,美国、中国、加拿大论文产出数量最多,但从文献的影响力和被引用频次看,中国与美国、加拿大和英国都有一定差距。(2)在研究机构方面,优秀的植物DNA条形码研究机构集中在中国和美国,在发文量前3名的機构有两个是中国研究机构,但在影响力上(TLCS),中国的研究机构没有排在前10名,中国科研机构在提高自身论文水平的同时,还应注意加强与美国和其他国家优秀科研机构的交流与合作,提高自身论文水平。(3)在论文作者方面,发文量、总被引频次,主要集中在美国、中国、英国和加拿大,发文量前10的作者(表1),其中有5名为中国学者,说明中国在这一领域研究文献贡献率很大,但影响力与欧美国家的学者还有一定距离。(4)在论文期刊分布上,优秀期刊主要集中在美国、英国、加拿大等欧美国家,没有中国的英文期刊,通过对被引频次的可视化分析,可以看出DNA条形码候选序列筛选和技术应用是该领域最大关注点。(5)DNA条形码研究热点集中于DNA条形码在陆生植物、物种鉴定、分类、生物多样性、候选序列筛选、系统进化上的应用等方面。
近十多年来,中国DNA条形码研究论文数量一直处于上升态势,发文量居世界第二,但论文影响力有限。中国科学院、中国中医科学院、中国医学科学院与北京协和医学院是中国优秀的DNA条码科机构,陈士林和宋经元的文献较多,质量较高。在SCI收录的期 刊 中,中国没有dNA条形码研究领域的优秀期刊,在影响力较高10篇(LCS)的论文中有中国科学院昆明植物研究所李德珠、高连明等中国植物条形码研究团队(China Plant BOL Group)合作的论文“Comparative analysis of a largedataset indicates that internal transcribed spacer (ITS) should be incorporated into the core barcode for seed plants”(Li et al., 2011)。
我国开展DNA条形码的研究虽然起步较晚,特别是植物dNA 条形码的研究到 2008 年才真正开始,但发展十分迅猛,发表了系列的相关文章,并参与了国际植物核心DNA 条形码的评估与推荐,相信不久的将来我国在国际生命条形码研究中会发挥更大的作用。从我国国务院发布的《中医药发展战略规划纲要(2016—2030年)》,DNA条形码在中药的应用将会不断增多,目前已经获得了不少的成果:《中药DNA条形码分子鉴定》、《中国药典中药材DNA条形码标准序列》、中药材DNA条形码鉴定系统(http://www.tcmbarcode.cn/china/),dNA条形码将为中药的鉴定、中药走出国门被世界接纳奠定基础, 中国植物DNA条形码研究也是蒸蒸日上,中国科学院昆明植物研究所和中国科学院华南植物园共同完成的“中国植物DNA条形码研究”科技研究已获得不错的成果。
植物DNA条形码2017年文献动态及发展前景,在前面的数据分析中没有涉及到2017年的文献数据,Web of Science 核心合集中对2017年植物DNA条形码进行文献检索分析,共获得208个记录,该领域研究蓬勃发展,文献量均高于其他年份,中国和美国发文量占了将近50%(103篇),主要的发文期刊和作者与前面的结果一致,主要文献为“PlantdNA barcodes:Applications today and in the future”(Kress,2017)。2017年延续了前期的研究,并重点开展了以下几个方面研究:群落系统发育和物种编目,功能形状和物种的编目,物种之间的互相影响;识别未知的物种,物种的边界和生物多样性的发现,DNA条形码的取证;商业产品、濒危物种、草药的原材料、民族植物学,物种和栖息地的保护。同时某些在特定的植物(Codonopsis)群中,仍然可以看到各种各样的DNA条形码标记测试的研究报告(Wang et al., 2017)。
自十多年前DNA条码被引入到植物群落中,DNA条码在植物基础研究和应用研究中都得到了广泛的应用。植物系统学家尚未普遍接受DNA条码作为识别物种的核心工具的一个主要原因是,在大多数分类群中,没有一个标记能够完全区分物种。相比之下,生态学家更愿意寻找新的、独特的DNA条码应用,以解决他们的一些基本研究问题,因为总的来说,他们在由多种植物谱系组成的系统中工作,这些系统可以通过DNA条码位点的组合进行唯一的识别。展望未来,植物DNA条码将通过两种关键的方式为植物群落服务:(1)建立一个更全面、通用的全球植物DNA条码库。实现为世界上所有植物提供DNA条码的通用库的目标仍然是遥远的未来,但是一旦实现,基础研究和应用研究都将受益匪浅。(2)开发新的标记并采用新的测序技术,如DNA宏条形码(metabarcoding 或 eDNA)(Taberlet et al.,2012),随着测序技术的提高和费用的降低,DNA条形码向基因组学方向发展 (Coissac et al.,2016)。
在此次分析中,存在以下几方面的不足:(1)用Histcite软件以LGS Count为条件,选用LCS绘制引文编年图,有时候一些新的文章因为年代近,被引用次数暂时还不多,所以它们在图中节点中被引用次数不会很多,也存在一定的缺陷,有些近期的论文很重要,但是没有表现出来。(2)基于Web of Sciences数据库涵盖了多种世界范围内最有影响力的高质量英文期刊,但其侧重于收录母语为英语地区的期刊,来源期刊地域分布的不平衡性对分析结果有一定的影响。
参考文献:
BEGROWd, NILSSON H, UNTERSEHER M, et al., 2010. Current state and perspectives of fungaldNA barcoding and rapid identification procedures [J]. Appl Microbiol Biotechnol, 87(1):99-108.
BHARATHIdG, 2013. Methods employed in the web of science and scopusdatabases to effect changes in the ranking of the journals [J]. Curr Sci Ind, 105(3):300-308.
BHARGAVA M, SHARMA A, 2013.dNA barcoding in plants:Evolution and applications of in silico, approaches and resources [J]. Mol Phlyogenet Evol, 67(3):631.
BLAXTER M, 2004. The promise ofdNA taxonomy [J]. Philosophical Transac Royal Soc B Biol Sci, 359(1444):669-679.
CHASE MW, COWAN RS, HOLLINGSWORTH PM, et al., 2007. A proposal for astandardised protocol to barcode all land plants [J]. Taxon, 56(2):295-299.
COISSAC E, HOLLINGSWORTH PM, LAVERGNE S, et al., 2016. From barcodes to genomes:Extending the concept ofdNA barcoding [J]. Mol Ecol, 25(7):1423-1428.
GARFIELD E, 2009. From the science of science to scientometrics visualizing the history of science with Histcite software [J]. J Informetr, 3(3):173-179.
GU HT, WANG J, 2013. Forecast of thedevelopment tendency fordiscipline based on two models——Take bibliometrics as an example [J]. J Mod Inf, 33(2):162-165. [顧洪涛, 王筠, 2013. 基于两种模型的学科发展趋势预测——以文献计量学为例 [J]. 现代情报, 33(2):162-165.]
HEBERT PDN, CYWINSKA A, BALL S, 2003. Biological identification throughdNA barcodes [J]. Proceed Roy Soc B-Biol Sci, 270(1512):313-321.
HOLLINGSWORTH PM, 2011. Refining thedNA barcode for land plants [J]. P Natl Acad Sci USA, 108(49):19451.
HOLLINGSWORTH ML, ANDRA A, FORREST LL, et al., 2009. Selecting barcoding loci for plants:Evaluation of seven candidate loci with species-level sampling in threedivergent groups of land plants [J]. Mol Ecol Resour, 9(2):439.
KERSS WJ, WURDACK KJ, ZIMMER EA, et al., 2005. Use ofdNA barcodes to identify flowering plants [J]. Proc Natl Acad Sci USA, 102(23):8369-8374.
KRESS WJ, 2017. PlantdNA barcodes:Applications today and in the future [J]. J Syst Evol, 55(4):1-17.
KIM HM, OH SH, BHANDARI GS, et al., 2014.dNA barcoding of Orchidaceae in Korea [J]. Mol Ecol Resour, 14(3):499-507.
LAHAYE R, BANK MVD,BOGARINd, et al., 2008.dNA barcoding the floras of biodiversity hotspots [J]. Proc Natl Acad Sci USA, 105(8):2923-2928.
LIdZ, GAO LM, LI HT, et al., 2011. Comparative analysis of a largedataset indicates that internal transcribed spacer (ITS) should be incorporated into the core barcode for seed plants [J]. Proc Natl Acad Sci USA, 108(49):19641-19646.
LIU J, MOLLER M, GAO L M, et al., 2011.dNA barcoding for thediscrimination of Eurasian yews (Taxus L. Taxaceae) and thediscovery of cryptic species [J]. Mol Ecol Resour, 11(1):89.
LU S,dING X, MA Y, et al., 2010. Confirming the genetic Identity of Dendrobium fimbriatum using an Amplification Refractory Mutation System (ARMS) [J]. Plant Mol Biol Rep, 28(4):712-716.
MOHAMMED AB, MOHD SF, SHAMSIR MS, et al., 2017. Review:DNA barcoding and chromatography fingerprints for the authentication of botanicals in herbal medicinal products [J] Evid-Based Compl Alter Med, (1):1-28.
PEGARD A, MIGUEL C, VALENTINI A, et al., 2009. UniversaldNA-Based methods for assessing thediet of grazing livestock and wildlife from feces [J]. J Agric Food Chem, 57(13):5700.
REN BQ, XIANG XG, CHEN ZD, 2010. Species identification of Alnus (Betulaceae) using nrDNA and cpDNA genetic markers [J]. Mol Ecol Resour, 10(4):594.
SHADBOLT N, HALL W, HENDLER JA, et al., 2013. Introduction:Web of Science:A new frontier [J]. Philosoph Transac Royal Soc A:Mathem Physical & Engineer Sci, 371(1987):1-6.
SUN XH, LU WR, 2012. Research trends of rice science based on Web of Science [J]. Chin J Rice Sci, 26(5):606-614. [孫秀焕, 路文如, 2012. 基于Web of Science的水稻研究态势分析 [J]. 中国水稻科学, 26(5):606-614.]
TABERLET P, COISSAC E, POMPANON F, et al., 2012. Towards next generation biodiversity assessment usingdNA metabarcoding [J]. Mol Ecol, 21(8):2045-2050.
TEZCAN M, VLACHONASIS K, AKI C, 2010.dNA barcoding study on Sideritis trojana Bornm. an endemic medicinal plant of Ida Mountain, Turkey [J]. Fresen Environ Bull, 19(7):1352-1355.
VALENTINI A, POMPANON F, TABERKET P, 2009.dNA barcoding for ecologists [J]. Trend Ecol Evol, 24(2):110-117.
WANGdY, WANG Q, WANG YL, et al., 2017. Evaluation ofdNA barcodes in Codonopsis (Campanulaceae) and in some large angiosperm plant genera [J]. PLoS ONE 12:e0170286.
WU RM, KONG YF, 2010. A comparison on Co-word analysis and Co-citation analysis [J]. Infdoc Serv, (1):26-29. [伍若梅, 孔悅凡, 2010. 共词分析与共引分析方法的比较研究 [J]. 情报资料工作, (1):26-29.]
YANG H, WANG XP, GAN WZ, et al., 2013. Analysis on literaturedevelopment trend of international tea polyphenols research based on the Web of Science [J]. J Tea Sci, (6):541-549. [杨华, 王小萍, 干文芝, 等, 2013. 基于Web of Science的国际茶多酚类研究文献发展态势分析 [J]. 茶叶科学,(6):541-549.]
ZHAO RY, XU LM, 2010. The knowledge map of the evolution and research frontiers of the bibliometrics [J]. J Lib Sci Chin, 36(5):60-68. [赵蓉英, 许丽敏, 2010. 文献计量学发展演进与研究前沿的知识图谱探析 [J]. 中国图书馆学报, 36(5):60-68.]
