基于自然群体随机交配的单个印迹QTL定位的初步研究
2019-09-10郑珂晖黎哲镇叶景山周富杰温永仙
郑珂晖 黎哲镇 叶景山 周富杰 温永仙



摘要:[目的]探索基于自然群体随机交配的单个印迹QTL的定位方法,分析影响定位准确性的关键因素。[方法]若印迹QTL决定的某一性状为数量性状,假设该性状与标记之间的关系存在线性关系,可以采用最小二乘法进行印迹QTL定位和遗传参数的估计。利用计算机模拟单点模拟标记、水稻真实自然群体标记进行印迹QTL定位,比较在不同最小等位基因频率(Minor allele frequency,MAF)、不同遗传率、不同随机交配轮数下的统计功效与参数估计精度,印迹QTL的显著性采用F检验和t检验。[结果]通过模拟研究,证明该试验设计对于检测单个印迹QTL是有效的,在MAF大于5%时,印迹遗传率大于10%时,定位与遗传参数估计趋于无偏。[结论]采用自然群体随机交配产生作图群体,可以用来进行单个印迹QTL的定位,定位的结果较好,是一种有效的试验设计,为下一步进行多个印迹QTL奠定了基础。
关键词:自然群体;关联分析;F检验;印迹QTL;随机交配
中图分类号:Q348文献标志码:A 文章编号:1008-0384(2019)12136407
0 引言
[研究意义]自然群体往往不是严格的遗传群体,如:种质资源、野生种等,它一般不用通过先构建亲本再进行交配产生F2代以后再用自交、回交等方法来产生。相对于结构群体,它的遗传背景一般并不清晰,往往采用关联分析来进行QTL定位。全基因组关联分析”’主要基于基因的连锁不平衡,用以判断表型性状与遗传标记之间的关联性。在全基因组上对逐点扫描遗传标记,依次判断每个标记与相关表型性状的关联水平。关联分析方法出现以后,有力地推动了数量遗传学的发展,对于水稻、玉米等作物以及猪、牛等家畜的育种改良起了重要的推动作用。印迹效应是一种表观遗传效应,指的是由于亲本产生的配子根据其来源父本或母本,使得配子中某些基因呈现特异的表达或沉默,造成子代体细胞中某些等位基因表达活性不同的现象。印迹在某些植物的胚乳形成、哺乳动物的胚胎发育、人类的一些罕见疾病的形成中起着重要的作用。[前人研究进展]由于全基因组数据往往十分庞大,关联分析对计算的要求较高。为了简化计算,早期研究人员进行关联分析时往往将只考虑加性效应,而忽略显性与印迹等效应。近些年来,有部分研究人员将印迹效应引入到全基因组关联分析中。Marcos等采用SNP作为遗传标记,采用回归方法来定位与猪背膘、日增重等性状相关联的SNP,并估计加性、显性和印迹3种遗传效应值。Hu等采用GWAS技术分析了印迹基因组对小鼠BMI指数的影响,研究表明印迹可以世代传播,对小鼠的BMI指数具有一定的影响。Steven等通过研究发现:基因组印迹对在小鼠对于锥虫的抗性具有重要的作用,并在小鼠染色体上定位到了与锥虫抗性相关的3个印迹QTL。Wen等利用永久F2群体来进行印迹QTL定位,可以无偏估计印迹QTL位置和加性、显性和印迹3种遗传效应。[本研究切入点]自然群体的构建需要花费大量的人力与物力,若将自然群体随机交配产生后代,则相对可以用较低廉的价格获得遗传信息丰富的群体。目前,基于自然群体随机交配的方法来定位印迹QTL目前尚未见到报道。由于基因组印迹具有亲本特异的特点,所以,在进行印迹QTL定位时,需要知道某个株系的父本或母本。直接采用自然群体不能够直接进行印迹QTL的定位,但可以将自然群体作为初始群体,进行随机交配,产生F2群体,可以用较小的初始群体来获得一个大的F2群体,该群体包含的遗传重组事件较为丰富,可以用作印跡QTL定位。[拟解决的关键问题]建立性状与加性、显性、印迹关系的遗传模型、给出单个印迹QTL定位与印迹QTL显著性检验的方法。
1试验设计
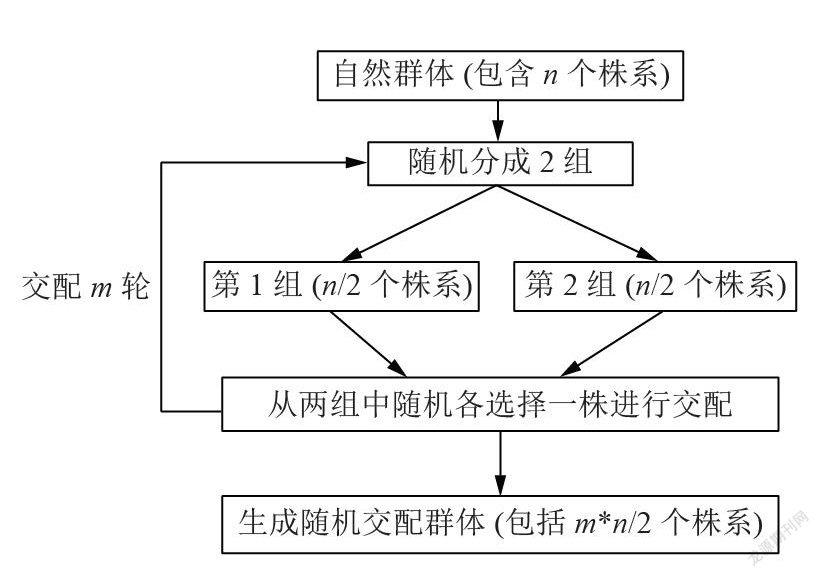
假设有1个已知标记基因型的自然群体,将自然群体植株间进行随机交配产生随机交配群体,采用随机区组设计,调查二倍体植株性状表型值,生成随机交配群体如图1所示。
2 遗传模型与定位方法
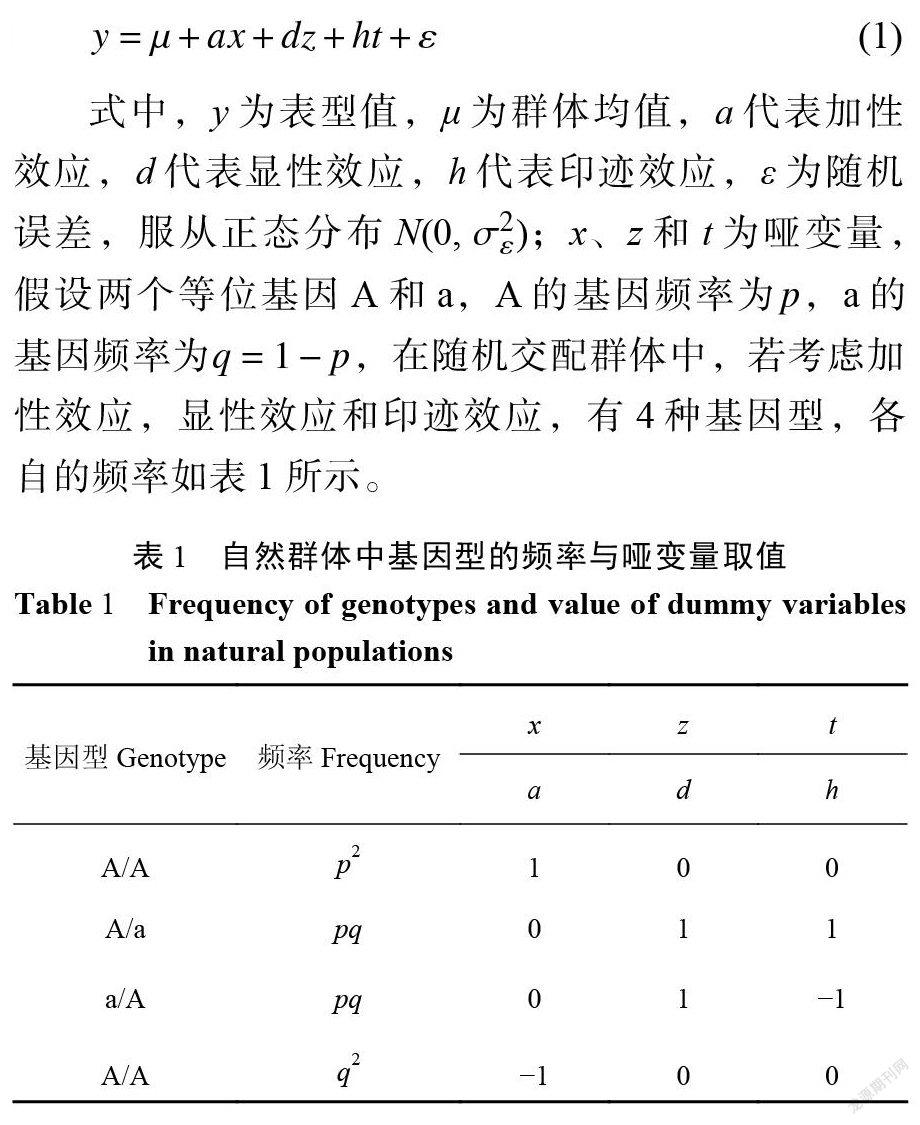
假设基因组上某个位置上存在与表型性状关联的位点,这个位点控制某个性状,那么我们可以定义下列遗传模型:
我们可以推导出,它的理论均值为:
等位基因替代效应的平均效应(the average effectof allele substitution)可以通过式4计算得到:
3 单标记印迹QTL定位方法
假设有一个自然群体进行随机交配后,产生了个体数目为n的群体,为此我们建立了以下线性模型
为了简单起见,本研究采取单标记关联分析,采用最小二乘法回归分析,对基因组上的标记进行逐点扫描,对于式9,我们提出假设H:a=d=h=0,其备择假设H:a,d和h不全为零。若仅检验印迹效应,我们提出假设Ho:h=0,其备择假设H:h≠0,我们采用F检验来检测标记的显著性,因为是单个标记的检验,直接用0.05或0.01下的F临界值进行判断。由于这里有3种遗传效应,那么显然的,如果:
对于给点的显著性水平,可以构建统计量,该统计量服从F(P,n-p-1)的F分布,则有式10。
式中,RSS代表随机残差平方和,ESS为回归平方和,n为样本数,p为变量个数,此处取值为3。如果方程总体通过了显著性检验以后,再逐个对x、z、t做t检验,检验各变量是否显著。F统计量与t统计量计算简单,使用计算机运算速度较快,可以逐点计算全基因组上每个位点的F值,并对每个位点的3种效应估计值进行t检验,将F最大(超过临界值,且通过t检验)的点作为可能的印迹QTL位点。
4 模拟研究
为了验证该方法的可行性,我们采用计算机来模拟验证,由于本文仅是基于自然群体随机交配的进行印迹QTL定位方法的初步研究,目的在探讨其可行性,所以进行2个模拟:第一是采用单点模拟,假设已知QTL位置,仅考察其参数估计的准确度和精度,及其是否达到显著性;第二是采用实际的水稻的基因型数据进行模拟,验证其可行性。
4.1单点模拟研究
由于自然群体有别于结构群体,自然群体的遗传结构是不清晰的,在群体中有大量的进化事件掺杂其中,群体结构较为复杂。在群体中,等位基因A,a的基因频率往往不是1:1,甚至有可能是很极端的,如可能在0.05以下,就是罕见变异。那么自然群体中在不同基因频率下,基于自然群体随机交配产生的群体,我们所建立的遗传模型和提出相应地印迹QTL检测方法与效应估计方法是否还能有效,我们通过模拟来进行检验。在这里,仅考察所建立的遗传模型在不同的MAF下相应地参数估计和显著检验方法是否可行。
我们假定仅有单点标记,在遗传模型中
y=u+ax+dz+ht+ε i=1,2,…,n (11)
进行参数估计,在此基础上,首先我们对方程进行F检验,看其是否存在QTL(a,d和h不全为零),在此基础上进一步对印迹效应做显著性检验(h≠0)。
根据式9可以计算出印迹效应的大小如式12所示。
再计算环境方差,最终产生表型值,这样就可以考虑遗传率的不同,随机交配次数不同的功效,参数估计等。
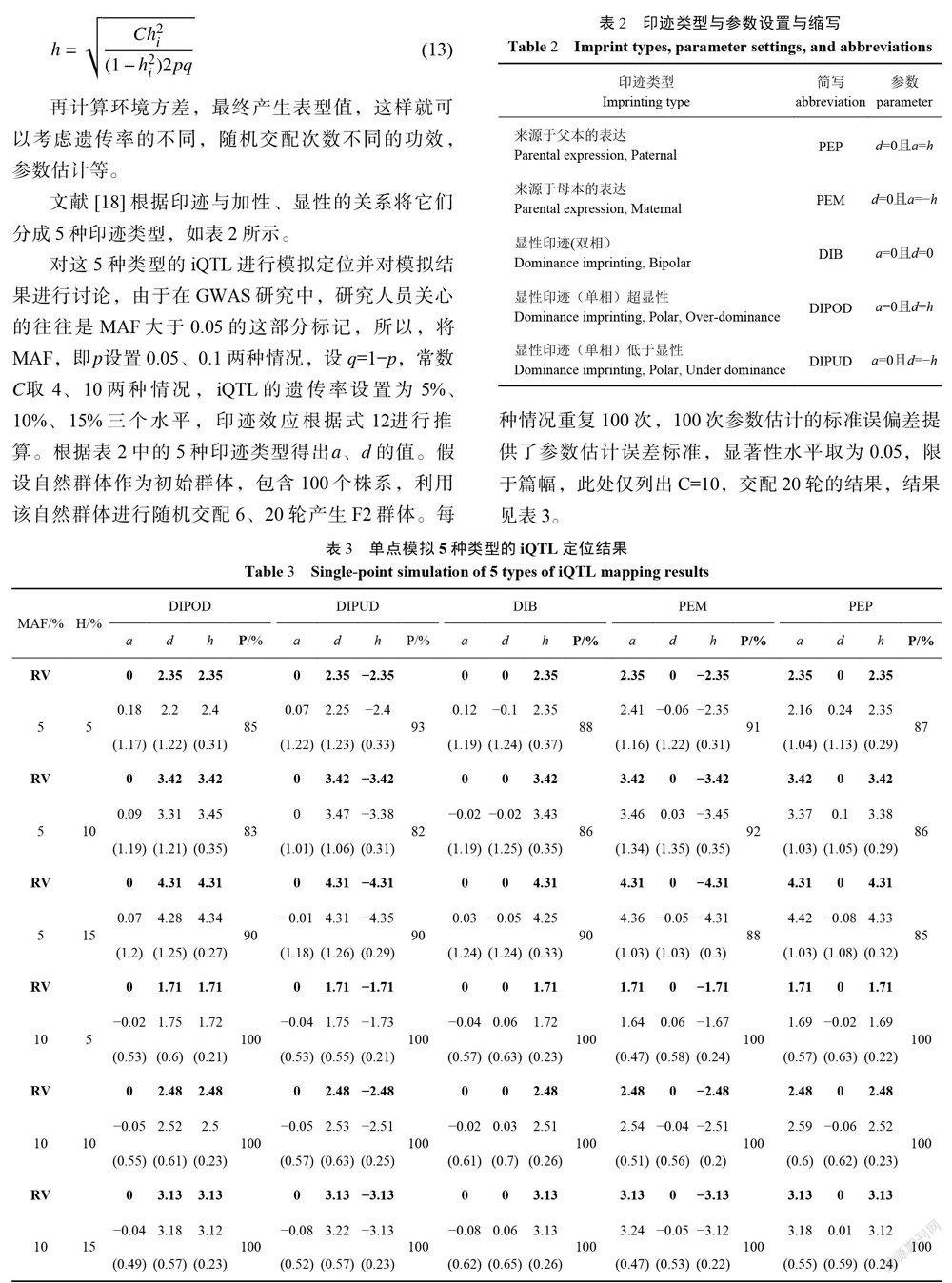
文献[18]根据印迹与加性、显性的关系将它们分成5种印迹类型,如表2所示。
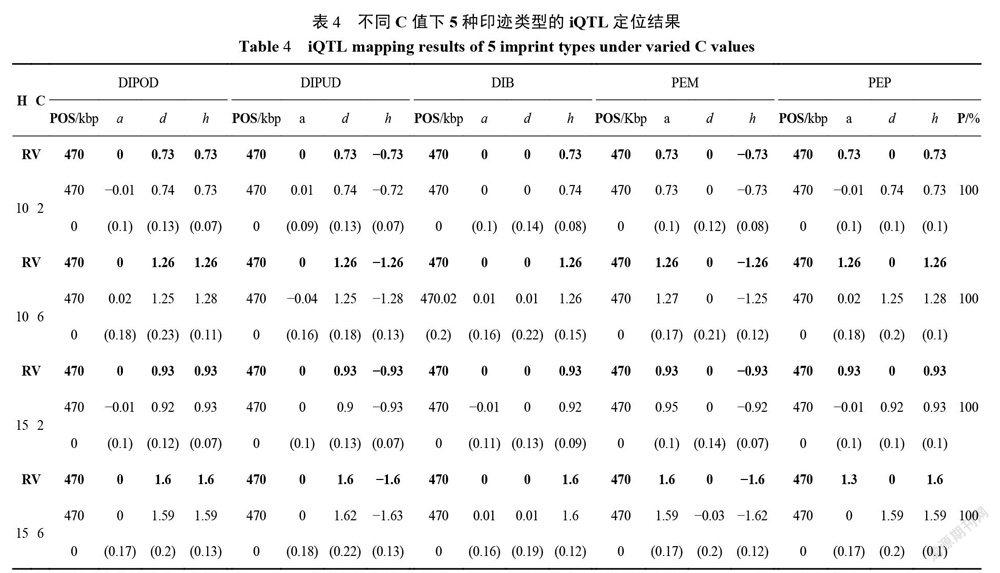
对这5种类型的iQTL进行模拟定位并对模拟结果进行讨论,由于在GWAS研究中,研究人员关心的往往是MAF大于0.05的这部分标记,所以,将MAF,即p设置0.05、0.1两种情况,设q=1-p,常数C取4、10两种情况,iQTL的遗传率设置为5%、10%、15%三个水平,印迹效应根据式12进行推算。根据表2中的5种印迹类型得出a、d的值。假设自然群体作为初始群体,包含100个株系,利用该自然群体进行随机交配6、20轮产生F2群体。每种情况重复100次,100次参数估计的标准误偏差提供了参数估计误差标准,显著性水平取为0.05,限于篇幅,此处仅列出C=10,交配20轮的结果,结果见表3。
结果表明:在MAF较小,为0.05时,即使iQTL的遗传率达到15%,其检测功效和参数估计的标准误都较高,当MAF达到0.1以上时,其检测功效就较高了;随着MAF的增大,遗传率的增加,其估计的标准误逐渐减小。在和遗传率相同的条件下,随着C值的增加,其参数估计的标准误也增加,其原因还有待于进一步的研究。即使在MAF高于0.05时,当遗传率为5%时,效应值估计有一定的误差,检测功效也较低,当遗传率在10%以上的时候,效应估计就较为精准。所以可以看出基因频率和遗传率是影响iQTL定位效果的因素。此外,随着交配次数的增加,iQTL的检测功效随之增加,参数估计的标准误随之减少,从5种iQTL来看,PEP、PEM、DIPOD、DIPUD在MAF、遗传率、C值相同的情况下,其iQTL检测效果相似,然而DIB的iQTL检测效果略差些,这说明加性效应和显性效应影响了iQTL定位。
综上所述,如果某个性状的表型值由一个iQTL确定,那么不论它是上述五种类型中的哪种类型,只要在MAF大于0.1且遗传率大于10%时,其检测功效均较好,说明我们的提出的方法在自然群体随机交配产生的F2群体上定位单个iQTL是有效的。
4.2 基于水稻基因型数据单个位点的模拟研究
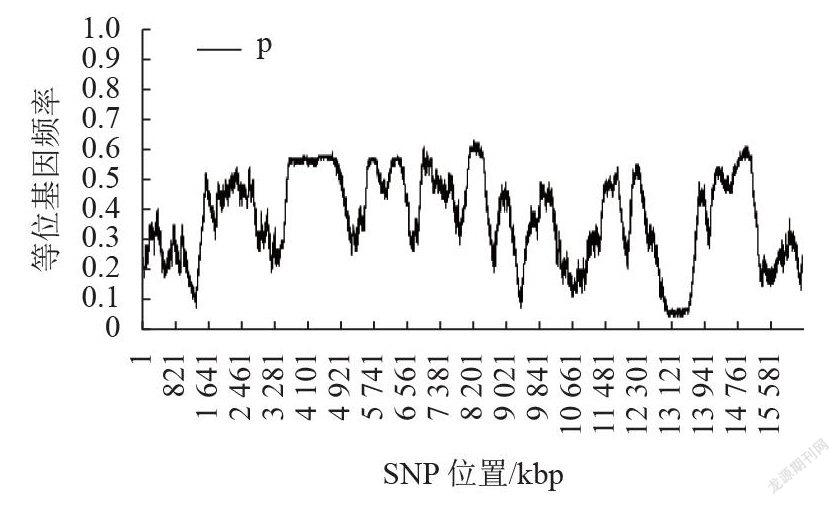
为了验证该方法在实际中的实施情况,我们采用韩斌院士课题组发布的水稻自然群体的数据集,该数据集收集了960多种水稻种质资源。为了简化研究的问题,对原始数据进行了预处理,把其中粳稻的254个株系取出,并将MAF小于0.05的SNP位点去掉,并去除了所有的缺失位点,最终得到了一个包含16382个SNP,在本模拟中,假定这些SNP是等间距分布在一条染色体上,每个SNP间的距离为1kb,254个株系的数据集作为原始群体。原始群体采用随机交配的方式生成随机交配群体,在进行模拟前,统计各个位点的SNP频率。从图2可知,在自然群体中,每个位置上的SNP的MAF并不是均衡的。
假定一个位于470kb位置的这个SNP与某个表型性状关联,群体均值为u=10。模拟前,先计算该位点的SNP频率p和g,印迹效应根据公式12进行推算(C值取2,4,6三种组合)。根据表2中的5种印迹类型得出a,d的值,根据前面的模拟结果,iQTL的遗传力水平设置为10%和15%,利用自然群体进行3、5、7次随机交配,产生随机交配群体。每种模拟情况重复100次,100次参数估计的标准误偏差提供了参数估计误差标准,阈值可用Bonferroni校正,显著性水平为0.05。此处,限于篇幅,仅列出C值取2、6,交配轮数为7次的结果,见表4,交配以后得到的随机交配群体中470kb处的SNP的频率p为0.54,则q为0.46,接近于1:1。由于在模拟时选择的iQTL位置上的MAF接近于0.5,从真实数据的模拟结果来看:当遗传率为10%、15%时,5种类型的iQTL定位都具有较好的结果,iQTL所在位置的估计较为准确,各参数估计的准确度和精度达到较好的效果,而且检测功效也较高,在15%时所有类型的iQTL检测功效均为100%。结果表明,随着交配次数的增加,参数估计的精度也得到提高,这符合我们的构想,当样本量增加时,其估计的准确度和精度得到提高;对于常数C值,从表可以看出,随着C值的增加,其参数估计的精度逐渐下降,其原因还有待于进一步研究;从5种类型的印迹QTL来看,在相同的条件下,DIB的检测功效较差,而其余4种DIPOD,DIPUD,PEM,PEP检测的功效相近,由此可见:加性效应和显性效应对iQTL的检测具有一定的影响。
5 讨论
我们提出了基于自然群体随机交配产生的F2群体进行iQTL定位的思想,构建其初步的定位方法,通过单点模拟,全基因组的单点模拟说明基于自然群体随机交配产生的群体进行iQTL定位是可行的,当然这里没有考虑其他因素(如群体结构等)的干扰。模拟表明,在不考虑其他因素的影响下,该iQTL定位方法的检测功效,以及参数估计的准确度和精度都较好。
虽然自然群体的群体遗传结构不清晰,但在GWAS时代,测序成本不断降低,甚至可以直接对个体进行测序。所以,基因型频率可以看成已知的,基于自然群体随机交配产生的群体,只要交配次数足够,就可以产生较大的样本;从初步研究模拟也可以看出,随着交配次数的增加,其检测功效,参数估计的精度也增加。
在全基因组关联分析(GWAS)中,是以长期重组后保留下来的基因或位点间连锁不平衡为基础,在获得群体表型数据和基因型数据后,采用统计方法检测标记与性状之间关联的分析方法,一般以自然群体或种质资源为研究材料,遗传结构较為复杂。在进行GWAS时,要求遗传背景一致或者相似的群体,但在未知群体结构的关联分析研究中,由不同亚群引起的MAF的差异会造成假关联。通常采用基因组控制、基于家系的关联分析检验和主成分分析等方法控制。一般而言:基于自然群体随机交配产生的群体会产生结构分层,所以在这种情况下,必须要控制因为结构分层造成的假关联,在后续的工作中,可以利用混合线性模型,传递不平衡分析等其他方法来进行印迹QTL定位方法的研究。
