一种微波快速提取可用于qPCR的酵母基因组DNA的方法
2019-09-10孙宇飞方宇婷邓冬梅
孙宇飞 方宇婷 邓冬梅


摘 要:研究微波快速提取可用于qPCR分析的酵母基因组DNA的方法.在微波作用下分别采取干法和湿法制备酵母基因组DNA样品提取物与试剂盒法制备的提取物进行比较,通过紫外分光法、琼脂糖凝胶电泳、常规PCR及qPCR等方法检测提取物浓度与纯度.结果表明微波法提取物可用于常规PCR分析,其中微波干法提取物用于qPCR分析可获得准确的检测结果.
关键词:微波;qPCR;基因拷贝数;酵母基因组DNA
中图分类号:Q78;Q503 DOI:10.16375/j.cnki.cn45-1395/t.2019.02.011
0 引言
毕赤酵母是目前广泛使用的一种重组蛋白表达宿主,具有生长速度快、培养方便、培养基廉价且适合表达真核生物的重组蛋白等优点,已经在科学研究和生产实践中得到广泛应用[1-3].然而在毕赤酵母工程菌株构建过程中,有部分重组子细胞内质粒与基因组DNA发生多次整合,导致这些重组子携带了多拷贝外源基因.多拷贝外源基因对于毕赤酵母细胞表达外源蛋白具有影响,通常可使外源蛋白表达量增加[4],但也有部分情况会使外源蛋白表达量降低[5].另外,多拷贝重组子面临遗传稳定性的风险,后代容易发生丢失高量表达的性状,影响工程菌株在工业生产中的应用.而且多拷贝现象也会影响对外源基因在毕赤酵母宿主上表达量的正确评估:如果未进行拷贝数鉴定,将无从判断重组子的高表达量是由于自身表达调控因素引起的还是由于具有多基因拷贝数引起.因此在使用毕赤酵母作为外源蛋白表达宿主的研究中,对重组毕赤酵母菌株基因组DNA内外源基因拷贝数的鉴定显得非常必要.
定量PCR(Quantitative PCR,qPCR)方法是一种可准确鉴定核酸样品中目的片段数量的方法,可用于毕赤酵母重组子的外源基因拷贝数鉴定[6-7].然而qPCR分析对样品纯度有一定要求,而常规高纯度样品制备需要价格昂贵的基因组提取试剂盒.微波法制备分子生物学样品已有较多实例[8-10],具有简便快捷的特点,但用于qPCR样品的制备仍未见报道.本文利用微波炉开发了一种简便、廉价的快速制备可用于qPCR分析的酵母基因组DNA样品的方法,使毕赤酵母的外源基因拷贝数鉴定时间缩短且费用降低.
1 材料與方法
1.1 菌株、仪器及试剂
毕赤酵母(Pichia pastoris GS115)重组菌株KC-5,整合双拷贝外源CALB基因,本实验室构建.Nanodrop1000微量核酸蛋白测定仪(Thermo),GeneAmp9700 PCR扩增仪(ABI),7500 Real-time PCR扩增仪(ABI),凝胶电泳成像系统(BioRad),Media微波炉(额定微波功率700 W),Centrifuge 5418离心机(Eppendorf).Dream Taq PCR试剂盒(Fermentas),DNA Marker(Takara),SYBR Premix EX Taq II qPCR试剂盒(Takara),PCR八联管(Axygen),EZNA Yeast DNA试剂盒(Omega).YPD培养基(1% Yeast Extract,2% Peptone,2% Dextrose,固体平板加入2%琼脂粉),其余为国产分析纯试剂.
1.2 微波辅助提取基因组DNA
将菌株KC-5在YPD平板上划线培养3 d(30 oC).用无菌牙签刮取直径约5 mm的单菌落到0.5 mL无菌离心管中.
1)微波干法:将样品置于微波炉以中低火处理(额定微波功率30%)5 min,处理时炉中同时放入盛有50 mL水的三角瓶.处理完毕在离心管内加入20 μL TE缓冲液(10 mmol/L Tris-HCl,1 mmol/L EDTA,pH=8.0),手指剧烈弹动离心管1 min,4 000×g离心1 min,上清液即为基因组DNA样品.
2)微波湿法:在离心管中先加入20 μL TE缓冲液,混匀后再同样微波处理5 min.手指剧烈弹动离心管1 min,4 000×g离心1 min,取上清液备用.
取2 μL样品用微量核酸蛋白测定仪检测.
1.3 试剂盒法提取基因组DNA
在50 mL 离心管(含5 mL YPD液体培养基)中接种KC-5单菌落,摇床培养24 h(250 r/min,30 oC).将菌液调至OD600=1,取1 mL菌液经4 000×g离心5 min,收集菌体按照EZNA Yeast DNA试剂盒说明书操作提取酵母基因组DNA.用100 μL试剂盒自带EB缓冲液洗脱,取2 μL样品用微量核酸蛋白测定仪检测.
取5 μL 试剂盒法、微波干法和微波湿法的基因组DNA提取液进行琼脂糖凝胶(1%,m/v)电泳分析.
1.4 PCR检验基因组DNA
取微波法和试剂盒法提取的基因组DNA溶液用TE缓冲液分别稀释至20 ng/μL用作模板.配制50 μL PCR反应体系扩增1 392 bp的毕赤酵母his4基因部分片段(GenBank No. U14126.1),反应体系为Dream Taq预混液25 μL,ddH2O 19 μL,PUhis4(5´-CAGCATCAGGGAAGTGG-3´)2 μL,PDhis4(5´-TTGGCAGAAAGAGTTGT-3´)2 μL,DNA模板2 μL.空白对照组不加DNA模板.扩增程序为94 oC预变性5 min,接着30个循环的扩增反应(94 oC 30 s,55 oC 30 s,72 oC 1.5 min),最后再72 oC延伸7 min.
取5 μL PCR产物进行琼脂糖凝胶(1%,m/v)电泳分析.
1.5 荧光定量PCR(qPCR)检验基因组DNA
将试剂盒法提取的基因组DNA用ddH2O稀释10倍作为模板,配制25 μL qPCR反应体系分别扩增CALB基因和参考基因G片段,而微波干法提取的基因组DNA则直接用为模板.首先配制7倍体积的公共溶液(SYBR Premix EX Taq II溶液 78 μL, ddH2O 63 μL,ROX Reference Dye II 3.1 μL,基因组DNA模板6.2 μL),混匀后分装两管,每管75 μL.其中一管加入引物PRTC1(5′-CCAAACCCATCCTTCTCG-3′)和PRTC2(5′-GGCGTTGACCATGTACTCC-3′)各1.5 μL,另一管加入引物PRTG1(5′-GTCGGGACACGCCTGAAACT-3′)和PRTG2(5′-CCACCTTTTGGACCCTATTGAC-3′)各1.5 μL,混匀后分别以25 μL/管分装到八联管的3个小管中进行扩增.扩增程序为95 oC预变性30 s,40个循环的扩增反应(95 oC变性5 s,60 oC延伸34 s同时收集荧光数据). 平行进行3次实验,用参考基因的△CT法计算CALB基因拷贝数[11].
2 结果与分析
2.1 酵母基因组DNA紫外吸收检测
在波长220 ~350 nm的紫外光范围内扫描样品DNA的吸收峰曲线,结果见图1,样品DNA浓度见表1. 3种方法都能够获得具有核酸特征吸收峰(260 nm)的样品,但样品的浓度和纯度差异显著.根据260 nm吸收值计算提取物核酸浓度,试剂盒法提取物的浓度最低, 微波干法提取物的浓度最高, 微波湿法提取物的浓度居中.这似乎提示微波炉法提取物的核酸含量高、效果好.然而结合260/280及260/230的紫外光吸收比值进行分析,可发现试剂盒法提取物的核酸样品纯度很高(260/280=1.8),而微波法提取物则因含有较多杂质使核酸样品纯度降低.这些杂质(如RNA、DNA片段、核苷酸等)的增色效应使微波法提取物260/280值提高,也导致基因组DNA表观浓度增大.另外胞内释放的碳水化合物、多肽等杂质降低了微波法提取物的260/230值.微波干法和微波湿法提取物中的杂质主要成分也有所区别,总体而言,微波干法提取物中核酸类杂质较多而湿法提取物中碳水化合物、多肽类杂质较多.
2.2 酵母基因组DNA电泳
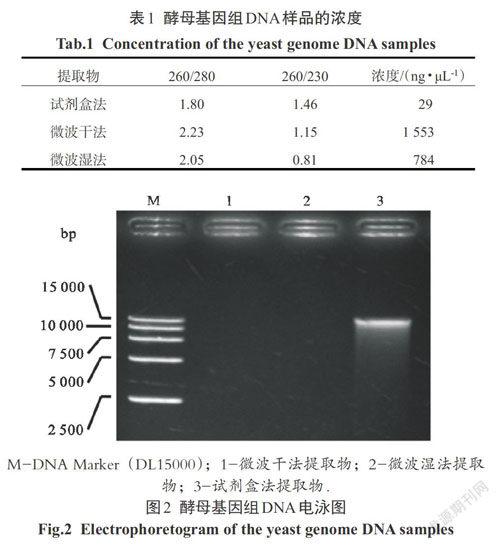
为了检验3种方法提取物中基因组DNA的直观浓度,各取5 μL提取液进行琼脂糖凝胶电泳分析.试剂盒法提取物在图2泳道3中显示具有一条约15 kb大小的较清晰条带,条带弥散少,说明试剂盒法获得的基因组DNA质量较好.而两种微波法的提取物在图2中都没有条带,说明提取物中基因组DNA实际浓度很低以至于所加样液中基因组DNA量达不到溴化乙锭染料显色阈值.结合紫外吸收曲線的结果进行分析,说明微波法提取物的真实核酸浓度比试剂盒法提取物浓度(29 ng/μL)低,其表观浓度高应当是样品中杂质增色效应导致.
2.3 常规PCR扩增his4基因
为了检验微波炉法提取物是否可用于常规PCR扩增,选取毕赤酵母GS115菌株的his4基因作为目的基因进行PCR扩增分析.图3(a)显示试剂盒法提取物和两种微波法提取物都可用作模板,扩增出大小与预期相符的his4基因片段(1 392 bp)且无明显拖带和弥散现象,说明这3种方法提取的DNA都可用于常规PCR扩增反应.然而与试剂盒法提取物相比,微波法提取物的PCR产物条带较淡,说明其样液中基因组DNA浓度较小.根据3种提取物表观浓度数据(表1)统一配制成“29”ng/μL样品溶液并进行PCR扩增检验(图3(b)),发现三者仍都能扩增出1 392 bp的his4基因条带,然而各条带的浓淡差异显著,进一步说明微波法提取物的基因组DNA浓度低于试剂盒法提取物.此外图3(b)中微波干法提取物的PCR产物条带最淡,这是因为所用PCR模板溶液稀释倍数较大(53倍),如用提取物原液为模板,则其PCR产物条带与微波湿法差异并不显著.总之,微波干法和湿法提取物都能用于常规PCR扩增,效果并无明显差异.
2.4 荧光定量PCR(qPCR)分析
因荧光定量PCR分析对样品的纯度和浓度有一定要求,为了保证分析结果的稳定与准确,使用糖类、多肽等杂质相对较少的微波干法提取物作为PCR模板进行qPCR分析,同时以试剂盒法提取物为参照检验两者分析结果的差异.另外为了检验此样品的定量分析效果,选择具有两拷贝CALB脂肪酶基因的重组毕赤酵母菌株KC-5进行qPCR分析.实验结果显示,无论是以试剂盒提取的样品(图4(a))还是微波法提取的样品(图4(b))作为模板进行qPCR分析,都获得令人满意的稳定而准确的CALB拷贝数数据.微波法提取物qPCR分析结果显示KC-5的CALB基因拷贝数为1.92(±0.145),而试剂盒提取物qPCR分析结果显示KC-5的CALB基因拷贝数为2.01(±0.088).虽然微波法提取物的CALB基因CT值达到26.9,比试剂盒法提取物的CALB基因CT值大了8.2,即其提取物中基因组DNA浓度仅为试剂盒法提取物的2-8.2(0.34 %,约10 pg/μL),但这并不影响微波法提取物用于qPCR分析的结果准确性与精确性.
3 讨论
对酵母细胞进行分子生物学分析时首先需要获得其基因组DNA,但结构坚韧的酵母细胞壁给破壁提取基因组DNA及后续PCR检测带来阻碍.如果直接使用酵母菌液或菌苔作为模板进行菌落PCR扩增,细胞破壁的过程主要依靠PCR预变性和变性阶段的热处理,不能保证提取效果使PCR检测的结果不稳定[12].因此酵母细胞的破壁成为酵母菌落PCR的关键.目前常用的破壁方法主要有煮沸法、煮沸冻融法、酶法、超声波法、玻璃珠研磨法、微波法等[12-15].煮沸法设备试剂要求简单,但效果同样不稳定.煮沸冻融法利用高低温差及胞内水分结晶膨胀破壁,效果相对较好但处理时间长、设备贵.酶法采用水解酶类(如蜗牛酶、溶菌酶)温和破壁,对DNA损伤小但处理耗时且破壁不完全.超声波法利用振荡引起的机械剪切力和水力空化效应能较好的达到细胞破壁效果,但对设备的要求高,而且超声处理产生的自由基对核酸等生物分子有损伤.玻璃珠研磨法成本低,但操作过程耗时较长,也有专用玻璃珠振荡破壁仪可快速破壁但成本昂贵.微波法利用微波对酵母细胞内部偶极分子(如液泡中的水分)高频往复运动,产生的内摩擦热而使胞内水分温度升高汽化从而达到胀破细胞壁的目的.该方法破壁快速简便,且只需廉价微波炉即可进行,因此在生物大分子提取中得以应用.有研究报道对长期浸泡于福尔马林的样品采用微波法也能较好的提取DNA, DNA含量可达(2.16±0.95 )μg/μL[16].本研究比较了微波辅助的干法和湿法提取效果.微波干法以酵母菌落为处理对象,微波对酵母细胞的作用更直接而强烈,但胞内核酸酶可能会在基因组DNA释放出来时破坏DNA的结构;微波湿法以TE缓冲液悬浮的酵母菌液为处理对象,TE缓冲液可以在基因组DNA释放时抑制胞内核酸酶的作用从而保护DNA,期待获得更完整的基因组DNA分子样品.
根据核酸分子在260 nm处的特征吸收峰计算核酸浓度是分子生物学常用技术之一.该方法简便快捷,然而对样品的纯度有较高要求.根据样品260/280及260/230数据可定性分析样品中杂质的主要类型.本研究中,微波干法提取物的260/280值偏大,提示杂质主要为RNA、寡核苷酸、核苷酸类产生增色效应的分子.由于微波能够使偶极分子加快振动产生大量热能,因此微波对有极性的DNA分子也有一定作用.微波作用的时间、强度(功率)直接影响到DNA分子的完整性,低强度的微波辐射就能使大鼠DNA损伤[17],还会降低DNA样品的可扩增性[18].在作用时间相同的情况下,干法提取时微波传递给DNA分子的能量比湿法提取的大,因为湿法加入的TE缓冲液消耗了部分微波的能量.另外微波干法提取过程中胞内核酸酶有可能对DNA分子造成伤害,也会增加样品的紫外吸收.因此干法提取物中DNA的损伤程度应更大,受损的DNA分子由于增色效应导致表观浓度增加.湿法提取物的260/280值较小,但260/230值较大,说明含有较多蛋白、多糖类杂质.其来源应当是高温的TE缓冲液将细胞壁的蛋白和多糖类物质提取出来成为杂质.微波干法和湿法都可提取出酵母基因组DNA,但干法提取物中DNA损失较大而蛋白、多糖杂质较少;湿法提取物中DNA较完整但蛋白、多糖类杂质较多.为了获得高质量的DNA样品,可联合其他技术共同处理.如采用SDS、溶菌酶、剧烈振荡和微波共同作用可对难处理的放线菌细胞进行破壁,提取较为满意的基因组DNA样品[19].
使用试剂盒法提取物进行qPCR分析效果好,但是样品平均提取时间长达5 h,且需要专用细胞壁破壁仪器及较昂贵的试剂盒.根据核算,试剂盒法每个样品提取成本约需20元,与此相比微波法制备微量基因组DNA样品所耗时间仅10 min,仅需准备廉价的微波炉以及几乎可忽略的耗费.因此微波法提取的酵母基因组DNA非常适合用于通过qPCR法大量快速鉴定酵母重组子的基因拷贝数等应用.
微波作用形式对提取效果也有影响.孢子具有极坚固的外壁,其DNA的提取难度较高.将炭疽孢子样品放在铝箔膜构成的“蝴蝶结”小穴结构中,微波被引导聚焦到孢子上,20 s即可完全提取所有孢子的DNA[20].因此了解微波作用的机制才能更好的开发微波辅助提取基因组DNA的新方法.微波除了可用于辅助提取基因组DNA外还具有其他生物学过程辅助效果,如可帮助加快DNA限制性酶切、连接、活化等酶法操作DNA的过程[21],只要20~50 s处理即可达到标准处理效果,极大地节约了处理时间.随着微波作用机理的不断深入探讨,微波辅助处理生物样品必将获得更广泛的应用.
4 结论
使用微波干法和微波湿法制备的酵母提取物具有核酸紫外吸收特性,经PCR扩增分析证实其含有酵母基因组DNA,可作为模板用于常规PCR扩增分析.微波干法提取物用于qPCR分析具备一定准确性和精确性,可满足大量筛选酵母重组子基因拷贝数的要求.
参考文献
[1] AHMAD M,HIRZ M,PICHLER H,et al. Protein expression in Pichia pastoris:recent achievements and perspectives for heterologous protein production[J]. Applied Microbiology and Biotechnology,2014,98(12):5301-5317.
[2] LOOSER V,BRUHLMANN B,BUMBAK F,et al. Cultivation strategies to enhance productivity of Pichia pastoris:a review [J].Biotechnology Advances,2015,33(6 Pt 2):1177-1193.
[3] 邓冬梅,潘淑兰,劳振华,等. 脂肪酶Lip2在蚕丝表面交联固定及其催化性质[J]. 广西科技大学学报,2018,29(2): 84-90.
[4] 雷清,陈勇,刘晓. 恶性疟原虫pfs25多拷贝基因在毕赤酵母中的表达分析[J]. 生物技术,2012,22(3):23-27.
[5] 黄义德,吴金芸,徐轶彦,等. 人骨形态发生蛋白4多拷贝表达盒的构建及其表达[J]. 福建师范大学学报(自然科学版), 2011,27(6):66-70.
[6] NYBO K. qPCR:single copy targets[J]. Biotechniques,2013,54(1):22-23.
[7] FLETCHER SJ. qPCR for quantification of transgene expression and determination of transgene copy number[J]. Methods in Molecular Biology,2014,1145:213-237.
[8] 潘力,崔翠,王斌. 一種用于PCR扩增的丝状真菌DNA快速提取方法[J]. 微生物学通报,2010,37(3):450-453.
[9] ORSINI M,ROMANO-SPICA V. A microwave-based method for nucleic acid isolation from environmental samples[J]. Letters in Applied Microbiology,2001,33:17-20.
[10] JOSHI L T, MALI B L, GEDDES C D, et al. Extraction and sensitive detection of toxins A and B from the human pathogen Clostridium difficile in 40 seconds using microwave-accelerated metal-enhanced fluorescence[J]. PLoS One,2014,9(8): e104334.
[11] SUN Y F,LIN Y,ZHANG J H,et al. Double Candida antarctica lipase B co-display on Pichia pastoris cell surface based on a self-processing foot-and-mouth disease virus 2A peptide [J]. Applied Microbiology and Biotechnology,2012,96(6):1539-1550.
[12] 劇海,梁东春,郭刚, 等. 用于PCR实验的毕赤酵母基因组DNA制备方法的比较[J]. 天津医药,2003,31(5):270-272.
[13] 罗中钦,程琳,张茜,等. 丝状真菌PCR 模板DNA 的快速制备方法[J]. 生物技术通报,2015,31(9):79-83.
[14] 王志纯. 超声波温育处理提取水稻种子DNA方法探讨[J]. 福建农林大学学报(自然科学版),2015,44(5):449-455.
[15] REBECKA von P,LARS von P,CHRISTOPHE D,et al. A high-throughput DNA extraction method for barley seed[J]. Euphytica,2003,130(2):255-260.
[16] WU S S,QIAN X,YU X C,et al. Microwave heating of long-term formalin-fixed surgical pathology specimens improves quality of extracted DNA[J].Applied immunohistochemistry & molecular morphology,2012,20(5):512-517.
[17] MEGHA K,DESHMUKH P S,BANERJEE B D,et al. Low intensity microwave radiation induced oxidative stress, inflammatory response and DNA damage in rat brain[J]. Neurotoxicology,2015,51158-51165.
[18] IMAIZUMI K,TANIQUCHI K,OQAWA Y. An evaluation of the effect of microwave irradiation on bone decalcification aimed to DNA extraction[J].LeQ Medical (Tokyo),2013,15(5):272-277.
[19] JIANG Y X,WU J G,YU K Q,et al. Integrated lysis procedures reduce extraction biases of microbial DNA from mangrove sediment [J]. Journal of Bioscience and Bioengineering,2011,111(2):153-157.
[20] ASLAN K, PREVITE M J, ZHANG Y X,et al. Extraction and detection of DNA from Bacillus anthracis spores and the vegetative cells within 1 min [J]. Analytical Chemistry,2008,80(11):4125-4132.
[21] DAS R H,AHIRWAR R,KUMAR S,et al. Microwave-mediated enzymatic modifications of DNA[J].Analytical Biochemistry,2015,471:26-28.
A rapid yeast genomic DNA extracting method assisted by microwave using in qPCR test
SUN Yufei1,2, FANG Yuting1,2, DENG Dongmei1,2
(1.School of Biological and Chemical Engineering, Guangxi University of Science and Technology, Liuzhou 545006, China; 2.Guangxi Key Laboratory of Green Processing of Sugar Resources (Guangxi University of
Science and Technology) ,Liuzhou 545006, China )
Abstract: A rapid yeast genomic DNA extracting method assisted by microwave using in qPCR test was studied. The yeast genomic DNA was prepared by the microwave-assisted dry method and the wet method respectively. Then the samples were tested by ultraviolet spectrometry, agarose gel electrophoresis, general PCR amplification and quantitative PCR assay successively, comparing with the sample prepared by a commercial reagent kit. Results show that both extracts from the microwave-assisted dry method and the wet method were able to use for general PCR amplification. Moreover, a considerable accurate and precise result can be achieved when the extract from dry method was used in the qPCR assay.
Key words: microwave; quantitative PCR; gene copy number; yeast genomic DNA
(責任编辑:黎 娅)
