HPV阳性口咽癌患者预后与T细胞浸润和新抗原负荷相关性分析
2019-08-26卢涣滋王迪侃王智
卢涣滋,王迪侃,王智
HPV阳性口咽癌患者预后与T细胞浸润和新抗原负荷相关性分析
卢涣滋,王迪侃,王智
中山大学光华口腔医学院·附属口腔医院,广东省口腔医学重点实验室, 广州 510080
口咽癌是头颈鳞癌中重要的癌种之一,与人类乳头瘤状病毒(human papillomavirus, HPV)感染有关,其发病率逐年上升。但在临床治疗过程发现,HPV阳性口咽癌患者整体预后好于HPV阴性患者。目前,导致这一现象发生的分子机制尚不明确。本研究利用TCGA数据库,对HPV阳性和阴性患者的免疫细胞浸润和效应功能以及新抗原负荷进行了生物信息学分析。结果发现,HPV阳性患者的总体生存率比HPV阴性患者显著提高。对肿瘤组织中的免疫细胞相对丰度进行分析显示,HPV阳性患者的CD8+T细胞较HPV阴性患者显著提高,其效应分子IFN-γ和Granzyme B表达量显著升高。同时对新抗原数目进行分析发现,HPV阳性患者肿瘤组织中新抗原较HPV阴性患者低。本研究结果为HPV相关口咽癌的治疗提供了一定的基础理论支撑。
口咽癌;人类乳头瘤状病毒;生物信息学分析;T细胞浸润;新抗原负荷
头颈部鳞状细胞癌(head and neck squamous cell carcinoma, HNSC)是全球范围内发病率排名第7的癌症,每年新增病例达60万[1]。头颈鳞癌包括口腔癌、口咽癌、喉癌等,尽管目前治疗理念已从手术治疗发展为以手术为主,放化疗及靶向药物治疗为辅助的综合治疗模式[2],但其5年总体生存率并无显著提高(50%)[3]。口咽癌是头颈鳞癌中的一种重要亚类,相对于其他头颈肿瘤,口咽癌发病率呈现逐年增加的趋势[4]。同时,相比于口腔癌,其预后更差[5];且因其位置关系,术后常导致患者出现言语及进食功能障碍,严重影响生活质量[6]。
口咽癌的致病因素包括烟、酒、槟榔,以及人类乳头瘤状病毒(human papillomavirus, HPV)等。其中,HPV16型(p16)是导致口咽癌的主要病毒[7]。据报道,在美国,HPV感染已经成为最主要的口咽癌致病因素,带来严重的公共卫生问题[8]。有趣的是,当研究HPV阳性(HPV+)和HPV阴性(HPV–)口咽癌患者的预后时发现,HPV+患者的总生存率显著高于HPV–患者[9]。导致HPV+口咽癌患者预后较好的原因,目前仍未完全阐明。有研究表明,HPV+口咽癌患者肿瘤细胞中基因突变率较低,所以对放化疗敏感性高[10];另一项研究表明,HPV+头颈鳞癌中免疫细胞,特别是T细胞的浸润更加丰富,是一种“热肿瘤”,因此对放化疗及免疫治疗等较敏感[11]。然而,目前对HPV相关口咽癌的免疫微环境及其与肿瘤突变的关系的研究仍较缺乏。
本研究将从免疫细胞浸润角度,利用生物信息学分析方法,阐述HPV+和HPV–口咽癌患者中的CD8+T细胞浸润程度差异,并分析其效应分子的表达差异,同时分析HPV+和HPV–肿瘤的基因突变负荷,为更深入理解HPV相关口咽癌提供一个新的方向,同时为HPV+口咽癌的治疗方案的优化提供一定的基础理论支撑。
1 材料与方法
1.1 口咽癌患者队列的获取
口咽癌患者队列来自癌症基因组图谱(The Cancer Genome Atlas, TCGA)中的HNSC患者,TCGA是由美国国立癌症研究院和美国国立人类基因组研究院合作开展的肿瘤基因组数据库计划,旨在通过高通量测序和多维度分析方法来深入了解多种肿瘤在发生发展过程中基因水平、转录水平及表观遗传水平等层面的变化情况[12]。其中头颈鳞癌患者基本信息、3级转录组测序结果和肿瘤突变数据从UCSC Xena上下载(https://xenabrowser.net/datapages/)。口咽癌患者纳入标准为:肿瘤解剖部位为舌根(base of tongue),扁桃体(tonsil)和口咽部(oropharynx),排除标准为接受术前新辅助化疗的患者。患者的HPV状态由人类乳头状瘤病毒p16阳性或阴性进行鉴定,根据HPV状态,将口咽癌患者分为HPV+组及HPV–组。
1.2 免疫细胞构成比计算
将口咽癌患者的3级FPKM标准化的Illumina HiSeq RNA表达数据(版本:09-14-2017)使用CIBERSORT反卷积算法[13]估计每个肿瘤组织中22种免疫细胞类型的相对比例,以评估肿瘤内免疫细胞的构成比,这22种免疫细胞的构成比是通过在线计算得到的(https://cibersort.stanford.edu/)。将CIBERSORT运算结果中>0.05的样本排除并将剩余样本进行进一步分析。
1.3 RNA表达量的计算和比较
将口咽癌患者的基本临床数据(版本:11-27- 2017)和3级FPKM标准化的Illumina HiSeq RNA表达数据(版本:09-14-2017)在Excel表格中进行合并。CD8+T细胞浸润程度由CD8A和CD8B的表达量平均值定义,T细胞的效应功能由(干扰素γ,IFN-γ)、(肿瘤坏死因子α,TNF-α)、(白细胞介素2,IL-2)、(穿孔素,Perforin)、(颗粒酶A,Granzyme A)和(颗粒酶B,Granzyme B)的表达量来反映,使用GraphPad Prism v7.0 (Graphpad Software,Inc.,美国)绘制直方图对HPV+组和HPV–组基因表达水平进行比较,使用双尾非参数Mann-Whitney U检验计算值,<0.05被认为差异具有统计学意义。
1.4 基因集富集分析(gene set enrichment analysis, GSEA)
采用GSEA软件对HPV+和HPV−患者队列的转录组进行基因集富集分析(软件地址:http://software.broadinstitute.org/gsea/index.jsp),使用C7(免疫相关基因集)作为分析中的参照基因集。结果中标准化富集分数(normalized enrichment score, NES)>1,<0.05,同时错误发现率(false discovery rate, FDR)< 0.25被认为具有统计学意义。
1.5 肿瘤新抗原负荷(tumor neoantigen burden, TNB)分析
头颈肿瘤新抗原数据下载自TSNAdb v4.0 (http://biopharm.zju.edu.cn/tsnadb/),TSNAdb是基于TCGA肿瘤数据库的肿瘤特异性新抗原数据库[14],其中的新抗原类型均为主要组织相容性复合体I型(major histocompatibility complex class Ⅰ, MHC-Ⅰ)。TCGA头颈肿瘤患者基因突变数据下载自UCSC Xena,采用的算法为mutect2。利用Excel对表格中每个患者的新抗原频次进行统计分析,得到TNB值,并计算HPV+和HPV–组的TNB的差异,使用双尾非参数Mann-Whitney U检验计算值,<0.05被认为差异具有统计学意义。同时对HPV+和HPV–口咽癌患者中出现频数最高的新抗原、基因和对应蛋白的突变位点、产生的新表位肽及其频率进行统计分析。
1.6 生存分析及统计分析
采用SPSS25.0软件进行统计分析。对于HPV+组和HPV−组中的连续变量和等级变量采用均值±SEM表示,采用Mann-Whitney检验评估两组间的差异;对于分类变量,使用卡方检验来评估各种危险因素之间的关联程度。采用Kaplan-Meire法对各种危险因素进行单因素预后分析,用Log-rank检验计算风险比(Hazard ratio, HR)及值;单因素预后分析中有统计学意义的危险因素进行多因素Cox回归分析。采用GraphPad Prism v7.0绘制生存曲线,并比较HPV+组和HPV–组的总体生存率(overall survival, OS)和无病生存率(Disease-free survival, DFS)的差异,用Log-rank检验计算值,<0.05被认为具有统计学意义。
2 结果与分析
2.1 HPV+和HPV–口咽癌患者的临床病理学特征和接受治疗的差异
TCGA数据库中头颈鳞癌患者共有612例,其中口咽癌患者共80例,HPV+33例,HPV–47例;HPV+组中女性3例,男性30例;HPV–组中女性9例,男性38例,如表1所示,两组间男女比例无统计学差异(>0.05);两组患者的年龄、吸烟史、饮酒史均无统计学差异(>0.05);此外,两组患者肿瘤部位、T分期、N分期、AJCC分期和淋巴血管侵犯均无显著差异(>0.05)。
为比较HPV+和HPV−组中接受不同治疗方案的患者的差异,根据术后是否接受放疗、化疗、放化疗、靶向治疗将口咽癌患者分为“手术+术后放疗手术+无术后放疗”、“手术+术后化疗手术+无术后化疗”、“手术+术后放化疗单纯手术”和“手术+术后靶向治疗手术+无术后靶向治疗”4种方案。通过卡方检验发现,HPV+和HPV–患者的接受4种方案的病例数及比例总体上无显著差异(表2,>0.05)。

表1 TCGA数据库中HPV+和HPV–口咽癌患者的临床资料和病理学资料
2.2 HPV+和HPV–口咽癌患者预后分析
为确定HPV是否对口咽癌患者的预后是否具有显著影响,对HPV+和HPV–口咽癌患者队列进行生存分析。结果显示,HPV+组5年OS显著高于HPV–组(89.8%. 39.8%,<0.05) (图1A),5年DFS同样稍高于HPV–组,但结果无统计学差异(55.2%32.2%,>0.05) (图1B)。将患者年龄、性别、吸烟史、饮酒史、肿瘤部位(舌根、扁桃体、口咽)、T分期、N分期、AJCC分期、淋巴血管侵犯状况、HPV状态作为危险因素进行单因素及多因素预后分析,结果显示在单因素分析中,年龄>60岁(HR=7.906,<0.001)为患者总体生存率的危险因素,而HPV+(HR=0.302,<0.05)为患者总体生存率的保护因素(表3)。然而,当进行多因素Cox回归分析时,仅年龄为独立危险因素(HR=7.190,<0.001),而HPV+在对患者总体生存率的作用上无显著差异(HR=0.352,>0.05) (表2)。以上结果表明HPV+口咽癌患者总体生存率显著优于HPV–患者。

表2 TCGA数据库中HPV+和HPV–口咽癌患者的接受不同治疗方式的人数
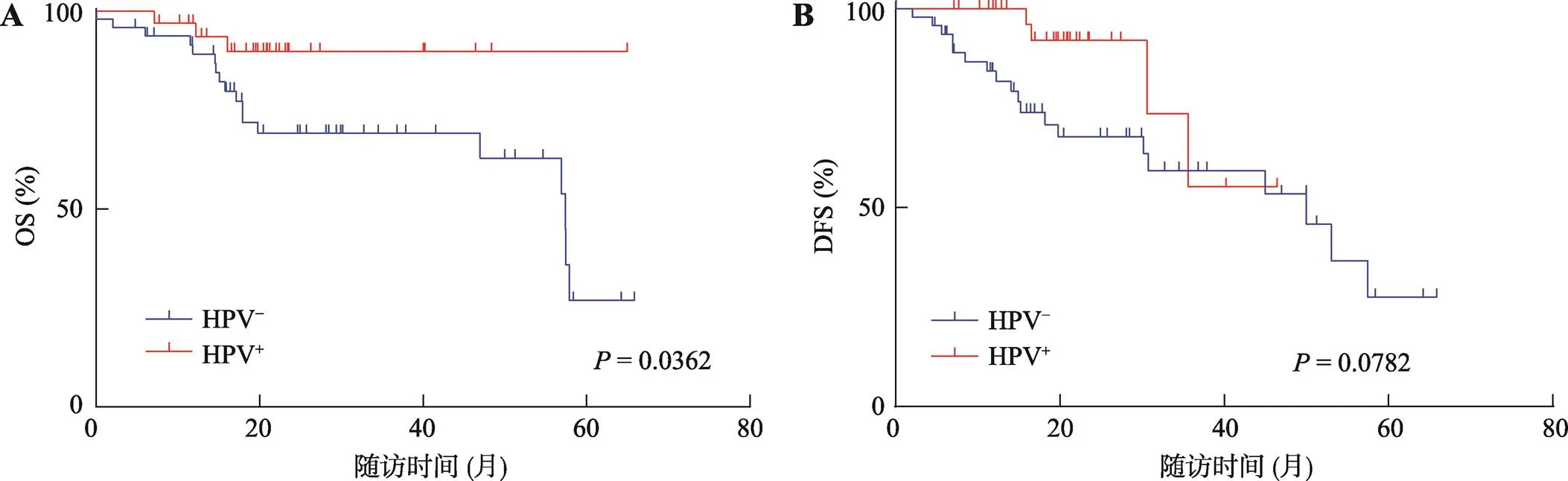
图1 TCGA数据库中HPV+与HPV–口咽癌患者的生存分析
A:总体生存率(overall survival, OS);B:无病生存率(disease-free survival, DFS)。

表3 TCGA数据库口咽癌患者队列的单因素和多因素预后分析
*表示<0.05;***表示<0.001。
2.3 HPV+和HPV–口咽癌患者免疫细胞浸润情况
为进一步探讨HPV+口咽癌患者和HPV–口咽癌患者在肿瘤微环境中免疫细胞浸润情况的差异,采用CIBERSORT算法对TCGA数据库中两组患者的肿瘤样本转录组数据用反卷积算法进行分析,获得22种免疫细胞在肿瘤样本中的相对丰度,然后去除CIBERSORT分析结果中>0.05的样本,剩余67个样本做进一步分析(HPV+:42个,HPV–:25个)。由图2A可知,HPV+组的CD8+T细胞相对丰度显著高于HPV–组(<0.05),同时滤泡辅助T细胞也高于HPV–组(<0.001),但静止CD4+记忆T细胞丰度较低(<0.01);另外,HPV+组的激活肥大细胞和中性粒细胞比例均显著低于HPV–组(<0.05) (图2B)。该结果提示CD8+T细胞在HPV+组的浸润程度显著高于HPV–组。此外,HPV+组中具有肿瘤杀伤作用的浆细胞、激活CD4+记忆T细胞、激活NK细胞的比例均稍高于HPV–组,但无统计学差异。进一步检测HPV+组和HPV–组转录组数据中CD8 (CD8A和CD8B的均值)的表达情况,结果显示HPV+组的CD8表达水平显著高于HPV–组(<0.05) (图2C)。因此推测,CD8+T细胞可能在HPV+患者的肿瘤微环境中起关键作用。
2.4 HPV+口咽癌患者T细胞效应分子的表达情况
为进一步探究CD8+T细胞在HPV+口咽癌患者中的作用,对HPV+患者中的T细胞效应分子的表达情况进行了分析,结果发现在HPV+组中显著上调(<0.01) (图3A),同时也显著上调(<0.05) (图3B)。此外,、、和在HPV+组中均稍高于HPV–组,但无统计学差异(>0.05) (图3,C~F)。对HPV+和HPV–患者的转录组进行GSEA分析,发现相对于T细胞激活和效应功能相关基因集显著富集到HPV+组中(CD8+T细胞激活基因集:NES=1.55,=0.01, FDR=0.12;CD8+T细胞效应功能基因集:NES=1.56,=0.04,FDR=0.12) (图3G)。因此,在HPV+口咽癌患者的肿瘤微环境中,CD8+T细胞杀伤功能显著增强,主要通过分泌IFN-γ和Granzyme B来促进细胞杀伤功能。
2.5 HPV+口咽癌患者肿瘤新抗原负荷较少
因为CD8+T细胞的产生需要MHC-I类限制性抗原的呈递和激活,因此对CD8+T细胞浸润是否与肿瘤组织中MHC-I类新抗原的负荷(tumor neoatigen burden, TNB)相关进行了探究。对HPV+和HPV–口咽癌患者肿瘤细胞新抗原频数进行分析,结果发现HPV+组的TNB显著低于HPV–组(23.2± 6.2431.33±3.36<0.01,图4)。进一步比较两组之间MHC-I限制性新抗原之间的差异,分析发现HPV+组中新抗原负荷最大的基因为(表4),HPV–组中新抗原负荷最大的基因分别为和(表5),两组中均显示为最常见的产生新抗原的突变位点;同时,编码肌联蛋白的基因TTN也同时出现于两组中,但没有共同突变位点和新抗原表位。值得注意的是,HPV+组中突变频数为0,而HPV–组中突变频数为5,其中和为产生新抗原的突变位点。

图2 HPV+和HPV–口咽癌患者的肿瘤浸润免疫细胞相对丰度和CD8 mRNA表达量
A:采用CIBERSORT算法计算HPV+和HPV–口咽癌中naive B细胞、记忆B细胞、浆细胞、CD8+T细胞、naive CD4+T细胞、静止CD4+T细胞、激活CD4+T细胞、滤泡辅助T细胞、调节性T细胞、静止NK细胞、激活NK细胞的相对丰度;B:采用CIBERSORT算法计算HPV+和HPV–口咽癌中γδT细胞、单核细胞、巨噬细胞M0、巨噬细胞M1、巨噬细胞M2、静止树突状细胞、激活树突状细胞、静止肥大细胞、激活肥大细胞、嗜酸性粒细胞、中性粒细胞的相对丰度;C:HPV+和HPV–肿瘤组织中的表达量(CD8A和CD8B表达量的FPKM均值)。n.s.表示无统计学意义;*表示<0.05;**表示<0.01;***表示<0.001。
3 讨论
本研究从HPV相关口咽癌的肿瘤免疫微环境浸润角度,探讨了HPV相关口咽癌的预后及其与CD8+T细胞的关系。通过比较分析发现,HPV+口 咽癌患者虽然在性别、年龄、肿瘤分级上无显著差异,但整体预后较HPV–患者好。进一步研究发现,HPV+口咽癌患者的CD8+T细胞丰度显著高于HPV–患者,同时通过比较CD8+T的效应因子的表达情况,结果表明HPV+组的CD8+T细胞中IFN-γ和Granzyme B的mRNA表达量显著提高,而且其他效应分子(IL-2、TNF-α、Perforin和Granzyme A)也较HPV–组稍升高。再进一步通过GSEA基因集富集分析,发现CD8+T细胞激活和效应功能的相关基因集富集于HPV+组中,证明了HPV+口咽癌中T细胞效应功能较为活跃。最后,通过对新抗原的检测,发现HPV+口咽癌患者的新抗原频数显著低于HPV–患者。

图3 HPV+和HPV–口咽癌患者中CD8+ T细胞效应分子mRNA表达量和GSEA分析结果
A:HPV+和HPV–口咽癌的IFNG的mRNA表达量;B:HPV+和HPV–口咽癌的GZMB的mRNA表达量;C:HPV+和HPV–口咽癌的TNF的mRNA表达量;D:HPV+和HPV–口咽癌的PRF1的mRNA表达量;E:HPV+和HPV–口咽癌的GZMA的mRNA表达量;F:HPV+和HPV–口咽癌的IL2的mRNA表达量;G:HPV+和HPV–肿瘤转录组的GSEA分析结果。n.s.表示无统计学意义;*表示<0.05;**表示<0.01。
根据免疫细胞浸润程度的高低,可将肿瘤分为“热肿瘤”(hot tumor)和“冷肿瘤”(cold tumor)[15]。这种分类方法最早见于结直肠癌患者,根据免疫细胞浸润程度,研究者将结直肠癌分为高免疫浸润和低免疫浸润,结果发现高免疫浸润的结直肠癌的预后显著高于低免疫浸润患者,且其预后的判定价值比TNM分期、淋巴血管侵犯和肿瘤分化程度更高[16]。另一项研究表明,高免疫浸润的结直肠癌中CD8+T细胞丰度更高,且其分泌的细胞因子IFN-γ、Granzyme A、Granzyme B和Perforin等均较高,这些因子可介导肿瘤细胞的裂解和凋亡[17]。本研究与既往结直肠癌研究结果一致,即HPV+口咽癌患者免疫浸润程度较HPV–患者更高,且T细胞效应因子分泌比HPV–患者高,表明CD8+T细胞参与了肿瘤杀伤作用。因此,提高肿瘤微环境中CD8+T细胞的丰度,可能有助于抗肿瘤免疫反应。有研究通过注射肿瘤疫苗或者过继性T细胞转输等方法,提高肿瘤微环境中CD8+T细胞丰度,并显著提高了抗肿瘤治疗的效果[18,19]。

图4 HPV+和HPV–口咽癌患者的肿瘤新抗原负荷的差异
**表示<0.01。
肿瘤新抗原是由肿瘤特异性体细胞非同义突变产生的表位,在MHC上表达。因其具有肿瘤细胞的高表达频率、正常细胞的低表达频率、高亲和力免疫原性、低耐受性等特点,目前已成为个性化癌症免疫治疗的有希望的靶点[20]。既往研究表明,肿瘤中新抗原的产生与预后较好相关,因为新抗原的增加与免疫细胞特异性识别和浸润相关[21,22]。但本研究结果显示,虽然HPV–患者免疫浸润较低,但HPV–患者肿瘤中新抗原显著高于HPV+患者,似乎与既往在其他肿瘤中的研究结果相悖。Rosenthal等[23]研究表明,肺非小细胞癌细胞可以不断产生新抗原,以适应免疫微环境的选择压力并促使免疫逃逸。本研究发现,突变仅发生在HPV–患者中,HPV+患者未见突变及新抗原产生。是一个非常重要的抑癌基因,其基因突变或高甲基化状态可导致肿瘤细胞增殖不受控制,并与头颈鳞癌患者存活率降低显著相关[24,25]。此外,Guo等[26]研究显示,突变可以诱导抑制性免疫微环境,促进肿瘤免疫逃逸。另外,相比于HPV–口咽癌,HPV+口咽癌中和突变率较高,而有研究表明头颈肿瘤细胞中和突变与免疫细胞浸润增多相关[27,28]。
此外,HPV+患者肿瘤细胞表达外源性p16病毒抗原E6和E7[29],因此更容易产生病毒抗原特异性T细胞,因此免疫浸润程度较高,同时对肿瘤免疫治疗响应程度更高[30]。Ramous等[31]通过收集HPV+口咽癌患者的外周血在体外用HPV特异性抗原E6和E7对T细胞进行免疫,发现有62%的口咽癌患者的外周血T细胞可以响应刺激并大量增殖,表明HPV相关口咽癌患者体内存在HPV抗原特异性T细胞;此外,Masterson等[32]发现,外周血可检测到E6/E7抗原特异性CD8+T细胞的患者对放疗及化疗更加敏感,其无病生存率较无抗原特异性T细胞的患者更高。因此,HPV相关抗原E6/E7可能促进CD8+T细胞的识别和浸润,并介导肿瘤杀伤作用。关于HPV相关口咽癌的免疫浸润和新抗原负荷的关系,仍需进一步研究。

表4 HPV+口咽癌中频率最高的新抗原及其突变位点和MHC-Ⅰ限制性表位肽序列
下划线:氨基酸突变位点。

表5 HPV–口咽癌中频率最高的新抗原及其突变位点和MHC-Ⅰ限制性表位肽序列
下划线:氨基酸突变位点。
本研究表明HPV+患者口咽癌患者中免疫细胞,特别是CD8+T细胞浸润程度以及效应功能显著高于HPV–患者,同时新抗原负荷显著低于HPV–患者,这可能是导致HPV+患者预后较好的原因。本研究为口咽癌患者的临床治疗提供了一种新的思路,即提高免疫细胞浸润,可以提高口咽癌患者治疗效果。
[1] Ferris RL, Blumenschein G Jr, Fayette J, Guigay J, Colevas AD, Licitra L, Harrington K, Kasper S, Vokes EE, Even C, Worden F, Saba NF, Iglesias Docampo LC, Haddad R, Rordorf T, Kiyota N, Tahara M, Monga M, Lynch M, Geese WJ, Kopit J, Shaw JW, Gillison ML. Nivolumab for recurrent squamous-cell carcinoma of the head and neck., 2016, 375(19): 1856–1867.
[2] Shang W, Zheng JW. Interpretation of the new TNM classification and the NCCN guidelines for cancers of the oral cavity and oropharynx., 2018, 16(06): 533–546.尚伟, 郑家伟. 口腔及口咽癌新版TNM分期与NCCN诊治指南部分解读. 中国口腔颌面外科杂志, 2018, 16(06): 533–546.
[3] Amornphimoltham P, Rechache K, Thompson J, Masedunskas A, Leelahavanichkul K, Patel V, Molinolo A, Gutkind JS, Weigert R. Rab25 regulates invasion and metastasis in head and neck cancer., 2013, 19(6): 1375–1388.
[4] Chaturvedi AK, Engels EA, Pfeiffer RM, Hernandez BY, Xiao W, Kim E, Jiang B, Goodman MT, Sibug-Saber M, Cozen W, Liu L, Lynch CF, Wentzensen N, Jordan RC, Altekruse S, Anderson WF, Rosenberg PS, Gillison ML. Human papillomavirus and rising oropharyngeal cancer incidence in the united states., 2011, 29(32): 4294–4301.
[5] Chi AC, Day TA, Neville BW. Oral cavity and oropharyngeal squamous cell carcinoma--an update.-, 2015, 65(5): 401–421.
[6] Clarke P, Radford K, Coffey M, Stewart M. Speech and swallow rehabilitation in head and neck cancer: united kingdom national multidisciplinary guidelines., 2016, 130(S2): S176–S180.
[7] Fischer CA, Kampmann M, Zlobec I, Green E, Tornillo L, Lugli A, Wolfensberger M, Terracciano LM. P16 expression in oropharyngeal cancer: its impact on staging and prognosis compared with the conventional clinical staging parameters., 2010, 21(10): 1961–1966.
[8] Marur S, D'souza G, Westra WH, Forastiere AA. HPV- associated head and neck cancer: a virus-related cancer epidemic., 2010, 11(8): 781–789.
[9] Fakhry C, Westra WH, Li S, Cmelak A, Ridge JA, Pinto H, Forastiere A, Gillison ML. Improved survival of patients with human papillomavirus-positive head and neck squamous cell carcinoma in a prospective clinical trial., 2008, 100(4): 261–269.
[10] Lechner M, Frampton GM, Fenton T, Feber A, Palmer G, Jay A, Pillay N, Forster M, Cronin MT, Lipson D, Miller VA, Brennan TA, Henderson S, Vaz F, O'flynn P, Kalavrezos N, Yelensky R, Beck S, Stephens PJ, Boshoff C. Targeted next-generation sequencing of head and neck squamous cell carcinoma identifies novel genetic alterations in HPV+and HPV- tumors., 2013, 5(5): 49.
[11] Welters MJP, Ma W, Santegoets SJAM, Goedemans R, Ehsan I, Jordanova ES, van Ham VJ, van Unen V, Koning F, van Egmond SI, Charoentong P, Trajanoski Z, van der Velden LA, van der Burg SH. Intratumoral HPV16-Specific T cells constitute a type I-Oriented tumor microenvironment to improve survival in HPV16-Driven oropharyngeal cancer., 2018, 24(3): 634–647.
[12] Li X, Li MW, Zhang YN, Xu HM. Common cancer genetic analysis methods and application study based on TCGA database., 2019, 41(03): 234–242.李鑫, 李梦玮, 张依楠, 徐寒梅. 常用肿瘤基因分析方法及基于TCGA数据库的分析应用. 遗传, 2019, 41(03): 234–242.
[13] Newman AM, Liu CL, Green MR, Gentles AJ, Feng W, Xu Y, Hoang CD, Diehn M, Alizadeh AA. Robust enumeration of cell subsets from tissue expression profiles., 2015, 12(5): 453–457.
[14] Wu J, Zhao W, Zhou B, Su Z, Gu X, Zhou Z, Chen S. TSNAdb: a database for Tumor-specific neoantigens from immunogenomics data analysis., 2018, 16(4): 276–282.
[15] Galon J, Bruni D. Approaches to treat immune hot, altered and cold tumours with combination immunotherapies., 2019, 18(3): 197–218.
[16] Mlecnik B, Tosolini M, Kirilovsky A, Berger A, Bindea G, Meatchi T, Bruneval P, Trajanoski Z, Fridman WH, Pagès F, Galon J. Histopathologic-based prognostic factors of colorectal cancers are associated with the state of the local immune reaction., 2011, 29(6): 610–618.
[17] Narayanan S, Kawaguchi T, Yan L, Peng X, Qi Q, Takabe K. Cytolytic activity score to assess anticancer immunity in colorectal cancer., 2018, 25(8): 2323–2331.
[18] Brown CE, Alizadeh D, Starr R, Weng L, Wagner JR, Naranjo A, Ostberg JR, Blanchard MS, Kilpatrick J, Simpson J, Kurien A, Priceman SJ, Wang X, Harshbarger TL, D'apuzzo M, Ressler JA, Jensen MC, Barish ME, Chen M, Portnow J, Forman SJ, Badie B. Regression of glioblastoma after chimeric antigen receptor T-Cell therapy., 2016, 375(26): 2561–2569.
[19] Ott PA, Hu ZT, Keskin DB, Shukla SA, Sun J, Bozym DJ, Zhang WD, Luoma A, Giobbie-Hurder A, Peter L, Chen C, Olive O, Carter TA, Li SQ, Lieb DJ, Eisenhaure T, Gjini E, Stevens J, Lane WJ, Javeri I, Nellaiappan K, Salazar AM, Daley H, Seaman M, Buchbinder EI, Yoon CH, Harden M, Lennon N, Gabriel S, Rodig SJ, Barouch DH, Aster JC, Getz G, Wucherpfennig K, Neuberg D, Ritz J, Lander ES, Fritsch EF, Hacohen N, Wu CJ. An immunogenic personal neoantigen vaccine for patients with melanoma., 2017, 547(7662): 217–221.
[20] Schumacher TN, Schreiber RD. Neoantigens in cancer immunotherapy., 2015, 348(6230): 69–74.
[21] Le DT, Uram JN, Wang H, Bartlett BR, Kemberling H, Eyring AD, Skora AD, Luber BS, Azad NS, Laheru D, Biedrzycki B, Donehower RC, Zaheer A, Fisher GA, Crocenzi TS, Lee JJ, Duffy SM, Goldberg RM, De La Chapelle A, Koshiji M, Bhaijee F, Huebner T, Hruban RH, Wood LD, Cuka N, Pardoll DM, Papadopoulos N, Kinzler KW, Zhou S, Cornish TC, Taube JM, Anders RA, Eshleman JR, Vogelstein B, Diaz LA Jr. PD-1 Blockade in Tumors with Mismatch-Repair Deficiency., 2015, 372(26): 2509–2520.
[22] Rizvi NA, Hellmann MD, Snyder A, Kvistborg P, Makarov V, Havel JJ, Lee W, Yuan J, Wong P, Ho TS, Miller ML, Rekhtman N, Moreira AL, Ibrahim F, Bruggeman C, Gasmi B, Zappasodi R, Maeda Y, Sander C, Garon EB, Merghoub T, Wolchok JD, Schumacher TN, Chan TA. Cancer immunology. Mutational landscape determines sensitivity to PD-1 blockade in non-small cell lung cancer., 2015, 348(6230): 124–128.
[23] Rosenthal R, Cadieux EL, Salgado R, Bakir MA, Moore DA, Hiley CT, Lund T, Tanić M, Reading JL, Joshi K, Henry JY, Ghorani E, Wilson GA, Birkbak NJ, Jamal- Hanjani M, Veeriah S, Szallasi Z, Loi S, Hellmann MD, Feber A, Chain B, Herrero J, Quezada SA, Demeulemeester J, Van Loo P, Beck S, Mcgranahan N, Swanton C, TRACERx consortium. Neoantigen-directed immune escape in lung cancer evolution., 2019, 567(7749): 479–485.
[24] Cancer Genome Atlas Network. Comprehensive genomic characterization of head and neck squamous cell carcinomas., 2015, 517(7536): 576–582.
[25] Chen Q, Li SW, Jia YC, Wang L. Blueberry anthocyanins induce G2/M cell cycle arrest and apoptosis of oral cancer KB cells through down-regulation methylation of p53., 2014, 36(06): 566–573.陈琦, 李少伟, 贾宇臣, 王利. 蓝莓花青素通过下调p53基因DNA甲基化抑制口腔癌KB细胞增殖及诱导细胞凋亡. 遗传, 2014, 36(06): 566–573.
[26] Guo G, Marrero L, Rodriguez P, Del Valle L, Ochoa A, Cui Y. Trp53 inactivation in the tumor microenvironment promotes tumor progression by expanding the immunosuppressive lymphoid-like stromal network., 2013, 73(6): 1668–1675.
[27] Lu X, Jiang L, Zhang L, Zhu Y, Hu W, Wang J, Ruan X, Xu Z, Meng X, Gao J, Su X, Yan F. Immune Signature- Based subtypes of cervical squamous cell carcinoma tightly associated with human papillomavirus Type 16 expression, molecular features, and clinical outcome., 2019, 21(6): 591–601.
[28] Siemers NO, Holloway JL, Chang H, Chasalow SD, Ross- Macdonald PB, Voliva CF, Szustakowski JD. Genome-wideassociation analysis identifies genetic correlates of immune infiltrates in solid tumors., 2017, 12(7): e0179726.
[29] Cui GX, Gao XL, Liang XH. Invasion and metastasis mechanism of human papillomavirus in head and neck squamous cell carcinomas., 2018, 36(05): 544–551.崔广学, 高晓磊, 梁新华. 人乳头瘤病毒相关头颈部鳞状细胞癌侵袭转移的分子机制. 华西口腔医学杂志, 2018, 36(05): 544–551.
[30] Nelson HH, Pawlita M, Michaud DS, Mcclean M, Langevin SM, Eliot MN, Kelsey KT. Immune response to HPV16 E6 and E7 proteins and patient outcomes in head and neck cancer., 2017, 3(2): 178–185.
[31] Ramos CA, Narala N, Vyas GM, Leen AM, Gerdemann U, Sturgis EM, Anderson ML, Savoldo B, Heslop HE, Brenner MK, Rooney CM. Human papillomavirus type 16 E6/E7-specific cytotoxic T lymphocytes for adoptive immunotherapy of HPV-associated malignancies., 2013, 36(1): 66–76.
[32] Masterson L, Lechner M, Loewenbein S, Mohammed H, Davies-Husband C, Fenton T, Sudhoff H, Jani P, Goon P, Sterling J. CD8+T cell response to human papillomavirus 16 E7 is able to predict survival outcome in oropharyngeal cancer., 2016, 67: 141–151.
Correlation analysis of the prognosis of HPV positive oropharyngeal cancer patients with T cell infiltration and neoantigen load
Huanzi Lu, Dikan Wang, Zhi Wang
Oropharyngeal cancer is one type of head and neck squamous cell carcinoma that is commonly associated with human papillomavirus (HPV) infection. Its incidence is increasing year by year. However, in the clinical treatment, it is found that the overall prognosis of HPV positive oropharyngeal cancer patients is better than that of HPV negative patients. But till now, the underlying mechanism that leads to this phenomenon has not been fully elucidated. This research analyzed the immune cell infiltration and function, as well as neoantigen loads between HPV positive and negative patients by bioinformatic analysis using the TCGA database, and found that the overall survival rate of HPV positive patients was significantly higher than those in the HPV negative group. Analysis of the relative abundance of immune cells in tumor tissues showed that CD8+T cells in HPV positive patients were significantly increased compared to those in HPV negative patients, and the expression levels of effector molecules, like IFN-γ and Granzyme B, were significantly upregulated. Meanwhile, the analysis of tumor neoantigen load (TNB) showed that the TNB of HPV positive patients was lower than that of the HPV negative group. This study provides some basic theoretical support for the treating HPV-related oropharyngeal cancer.
oropharyngeal cancer; human papillomavirus; bioinformatic analysis; T cell infiltration; tumor neoantigen load
2019-05-13;
2019-07-23
国家自然科学基金项目(编号:81772896,81630025,81602383)和广州市科技计划项目(编号:2017004020102)资助[Supported by the National Natural Science Foundation of China (Nos. 81772896, 81630025, 81602383) and the Science and Technology Planning Project of Guangzhou City of China (No. 2017004020102)]
卢涣滋,博士研究生,研究方向:头颈肿瘤免疫。E-mail: luhuanzi@mail2.sysu.edu.cn
王智,博士,教授,博士生导师,研究方向:头颈肿瘤免疫。E-mail: wangzh75@mail.sysu.edu.cn
10.16288/j.yczz.19-135
2019/8/6 11:24:34
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20190806.1124.002.html
(责任编委: 岑山)
