基于SSSL文库的水稻设计育种平台
2019-08-26张桂权
张桂权
基于SSSL文库的水稻设计育种平台
张桂权
华南农业大学农学院,广东省植物分子育种重点实验室,亚热带农业生物资源保护与利用国家重点实验室,广州 510642
“设计育种”是21世纪初提出的新概念,是指有目的地利用作物基因组中有利等位基因来设计并培育人们所需要的品种。在过去的20年中,本实验室制定了“三步走”的策略开展水稻设计育种研究,构建了基于染色体单片段代换系(single-segment substitution line, SSSL)文库的水稻设计育种平台,并在水稻设计育种上取得了初步的成效。基于SSSL平台开展水稻设计育种,能有效地利用SSSL文库的基因资源,设计并培育出各种各样的水稻新元件、新品系和新品种。本文详细介绍了水稻SSSL文库的构建以及利用该文库开展水稻设计育种的探索,以期为水稻乃至作物的设计育种提供参考。
设计育种;分子育种;单片段代换系;基因聚合;水稻
自20世纪80年代以来,由于DNA标记的出现,基因组研究取得了快速的发展。21世纪初,先后有多个复杂真核生物完成了全基因组测序,标志着基因组研究已进入后基因组时代。后基因组时代的到来,使人们对生物的遗传改良,特别是植物育种的发展,产生了前所未有的想象空间。“设计育种”(breeding by design)正是在这个时候提出的新概念[1]。
水稻是世界上重要的粮食作物,也是基因组研究的模式植物。因此,水稻在基因组研究和育种方面一直受到高度重视。半个多世纪以来,我国在水稻遗传育种方面取得了瞩目的成就[2~4],为开展水稻的设计育种研究提供了必要的条件,促使人们探索实施水稻设计育种的可能方案[5,6]。在过去的20年中,本实验室制定了“三步走”的策略开展水稻设计育种研究,构建了基于染色体单片段代换系(single- segment substitution line, SSSL)文库的水稻设计育种平台,并在水稻设计育种上取得了初步的成效。本文详细介绍了基于SSSL文库的水稻设计育种平台,以期为作物设计育种提供案例。
1 设计育种的概念和策略
如何提高育种的可预见性和可操作性,一直是植物育种需要解决的问题。传统的植物育种主要是凭借育种家们的经验,通过表型选择来实现,因此具有很大的不确定性,育种目标往往难以实现。全基因组测序的完成,预示着生物整个基因组的基因是可知的、可检测的,因此是可利用的。为此,人们设想怎样才能有效地利用全基因组基因开展育种,怎样才能把分散在不同个体中的优良基因集中到一个个体中,从而培育出所需要的优良品种?这个设想就是设计育种。实现设计育种包括3个主要环节:(1)对基因组的基因进行定位,明确基因在基因组中的位置;(2)分析主要基因座的等位基因变异,筛选出优良的等位基因;(3)利用基因组的优良等位基因开展设计育种,把分散在不同个体中的优良基因聚合在一起,从而实现设计育种的目标[1]。
早在1998年,本实验室开始在理论上探索设计育种的可行性,在实践上摸索设计育种的实施方案,制定并实施了水稻设计育种的“三步走”策略。第一步,构建水稻SSSL文库,把水稻育种上能利用的基因最大限度地收集到文库中来;第二步,对SSSL代换片段中的基因进行分析,广泛获取育种上有用的基因信息,为设计育种提供依据;第三步,利用SSSL文库中的优良基因开展设计育种,设计并培育出各种各样的水稻新元件、新品系和新品种。
2 水稻SSSL文库的构建
稻属()共有24个种,包括2个栽培种和22个野生种[7]。其中,AA基因组有8个种,包括2个栽培种—亚洲栽培稻()和非洲栽培稻(),和6个野生种—尼瓦拉野生稻()、普通野生稻()、短舌野生稻()、南方野生稻()、展颖野生稻()和长雄蕊野生稻()[8]。两个栽培稻种中,除非洲部分种植非洲栽培稻外,全世界主要种植亚洲栽培稻,东北亚、东南亚和南亚地区是亚洲栽培稻的主产区。亚洲栽培稻携带AA基因组,育种上能通过有性杂交利用的遗传资源主要是稻属AA基因组的8个种。
自1998年始,本实验室以自主培育的籼稻品种“华粳籼74 (HJX74)”为受体,以稻属AA基因组的8个种为供体构建水稻SSSL文库。在构建SSSL中,以HJX74为母本,分别与AA基因组8个种的40多个供体杂交,并利用HJX74回交4~7代。在分离世代中,每个组合选择约400个均匀分布于全基因组的多态性标记检测代换片段和遗传背景。当基因组中只检测到一个来自供体的代换片段时,让其自交纯合,最终成为只携带来自供体一个代换片段、遗传背景与HJX74一致的SSSL (图1)。
到目前为止,本实验室已获得了除长雄蕊野生稻以外的稻属AA基因组7个种为供体的SSSL共2360份[9~13]。整个SSSL文库包括3个子库:第1子库为亚洲栽培稻库。该子库以HJX74为受体,以不同来源、不同类型的亚洲栽培稻的28个材料为供体,共获得1610份SSSL;第2子库为非洲栽培稻库。该子库以HJX74为受体,以非洲栽培稻的5个材料为供体,共获得190份SSSL;第3子库为野生稻库。该子库以HJX74为受体,以野生稻种为供体,目前已获得以5个野生稻种的10个材料为供体的SSSL共560份(表1)。这些SSSL的代换片段平均长度约为19 cM,整个文库的代换片段总长度约为44 000 cM,大约是水稻基因组大小的29倍。由此可见,该SSSL文库包含了稻属AA基因组丰富的基因资源,实现了稻属AA基因组基因资源的大规模收集和活化,为育种上有效地利用这些基因资源奠定了基础。
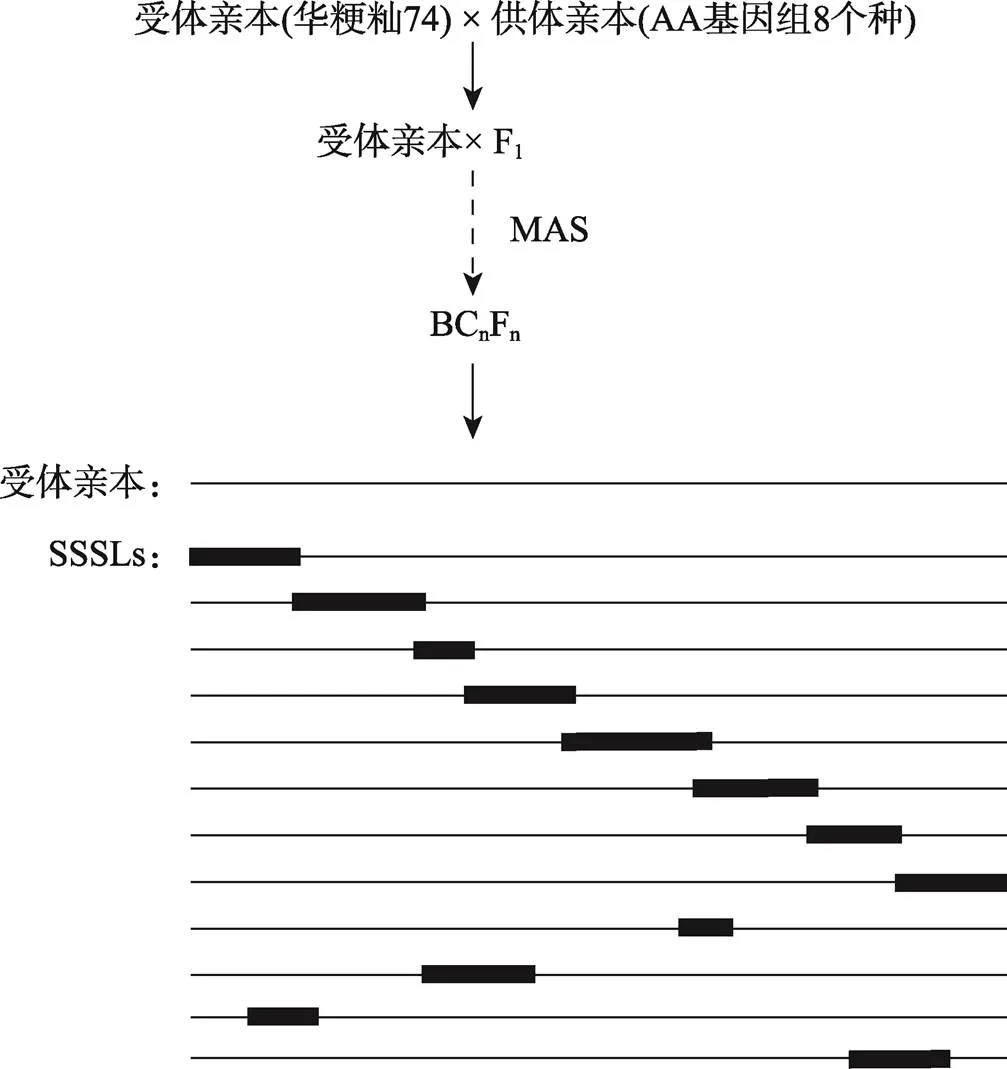
图1 水稻染色体单片段代换系(SSSL)的构建
细线条表示基因组,粗线条表示来自供体的代换片段。每个SSSL携带来自供体的单一代换片段。MAS:marker-assisted selection。
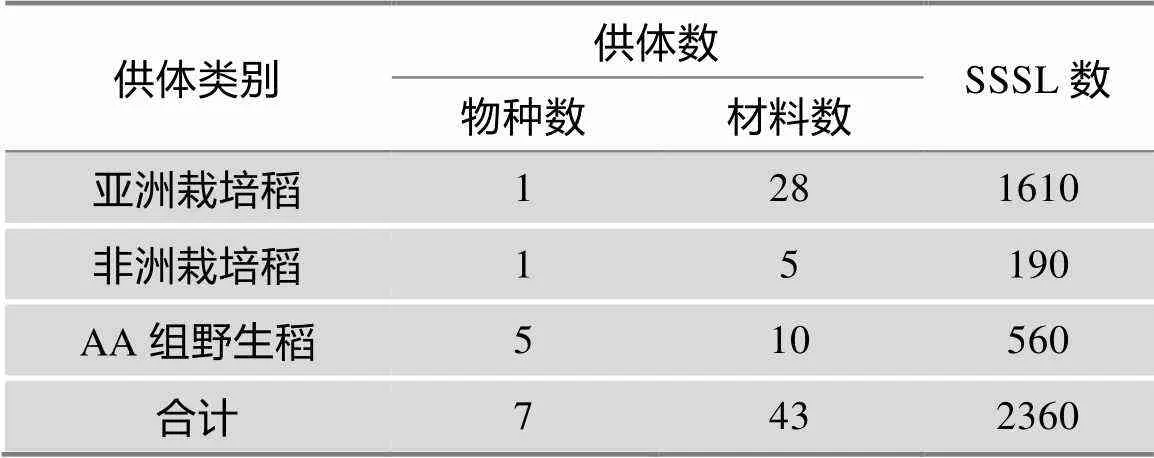
表1 文库中包含的染色体单片段代换系(SSSL)的数目及其供体来源
3 SSSL代换片段的基因分析
与受体亲本相比,SSSL只携带一个来自供体的染色体片段,其遗传背景与受体亲本相似。因此,利用SSSL对代换片段上的基因进行分析,能排除遗传背景的干扰。生物的复杂性状由多个数量性状基因座(quantitative trait locus, QTL)控制,SSSL的代换片段通常只携带控制复杂性状的某一QTL,实现了对复杂性状的遗传剖析(genetic dissection),因此SSSL是复杂性状QTL分析的理想材料。
根据研究和利用的需要,利用已获得的SSSL对一些重要性状的基因(QTL)进行了鉴定和定位[14~22],发掘了一些重要基因座的等位基因变异[23~25],对基因与基因之间及基因与环境之间的互作进行了分析[26~31],同时还对一些重要基因进行了克隆及功能分析[32~35]。通过研究,获得了一些SSSL代换片段上的基因信息,为利用这些基因开展设计育种提供了依据。
例如,从已构建的SSSL文库中筛选出代换片段上携带基因的SSSL,对基因进行了等位基因变异的分析。利用16份来自不同供体亲本的SSSL,通过检测稻米直链淀粉含量(apparent amylose content, AAC)、颗粒结合淀粉合成酶(granule-bound starch synthase, GBSS)活性、基因序列以及基因RT-PCR表达量,从中鉴定出5个等位基因。每个等位基因控制的直链淀粉含量均具有显著差异,包括稻米中直链淀粉含量的各种类型。明确了各等位基因DNA序列的功能变异点,及其转录和翻译产物的差异(图2)[24]。这些结果为利用基因开展设计育种,培育具有不同直链淀粉含量的水稻新品系和新品种提供了设计依据及可利用的基因资源和育种材料。
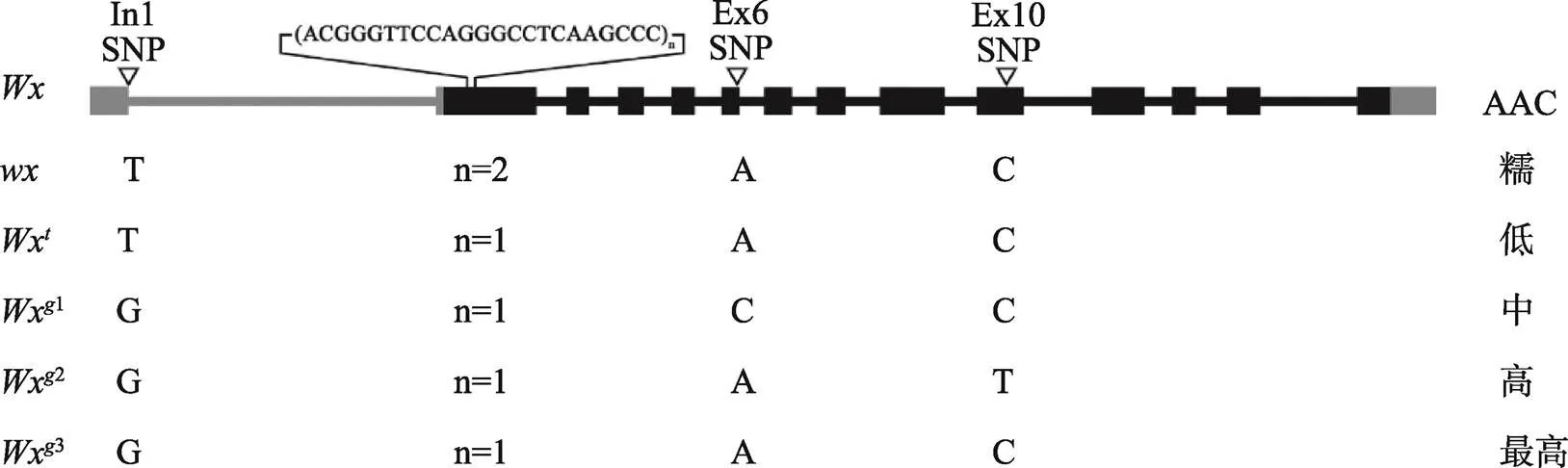
图2 利用染色体单片段代换系(SSSL)分析水稻Wx基因的等位基因变异
根据文献[24]修改绘制。AAC:直链淀粉含量(apparent amylose content)。
为了更好地对SSSL代换片段上的基因进行分析,本实验室对亚洲栽培稻子库的受体和供体共29个材料进行了全基因组重测序。每个材料的测序深度约20倍,各测序材料获得了覆盖基因组90%左右的序列。同时,利用这些序列分析了亲本间SNP和InDel在全基因组的分布,为SSSL代换片段的基因型分析提供了序列数据。这样,在SSSL平台上,一方面利用SSSL进行表型分析,另一方面利用全基因组序列进行基因型分析,将表型与基因型紧密地联系在一起,构建了水稻全基因组的基因分析平台,为水稻全基因组基因的发掘、检测和利用提供了技术支持。
4 基于SSSL文库的设计育种
开展设计育种,首先是根据需要制定育种目标,然后根据育种目标设计基因型,再根据设计方案把目标基因聚合在一起,培育出符合设计要求的新品系或新品种。SSSL文库的构建和利用,为设计育种方案的实施提供了可操作的平台。利用SSSL文库中已知的基因信息,围绕设计目标制定基因组合方案,然后把代换片段上的目标基因聚合在一起,从而使设计育种的目标得以实现(图3)。
本实验室在构建SSSL文库和对文库的基因进行分析的同时,也在探索利用SSSL文库开展设计育种的效果。例如,在培育SSSL的过程中,发现了1份紫色种皮的SSSL,其携带来自黑米亲本联鉴33第4染色体的一个代换片段,该片段上包含紫色种皮基因。对该SSSL进行品比和生产实验表明,该 SSSL除了表现为紫色种皮性状外,其余性状与受体亲本HJX74差异不显著,产量相当。由于该“黑米”SSSL具有较高的利用价值,于2005年通过了广东省的品种审定,定名为“华小黑1号”(粤审稻2005015)。华小黑1号的审定和推广应用,表明以该SSSL文库为平台开展设计育种能培育出生产上可利用的水稻新品种。
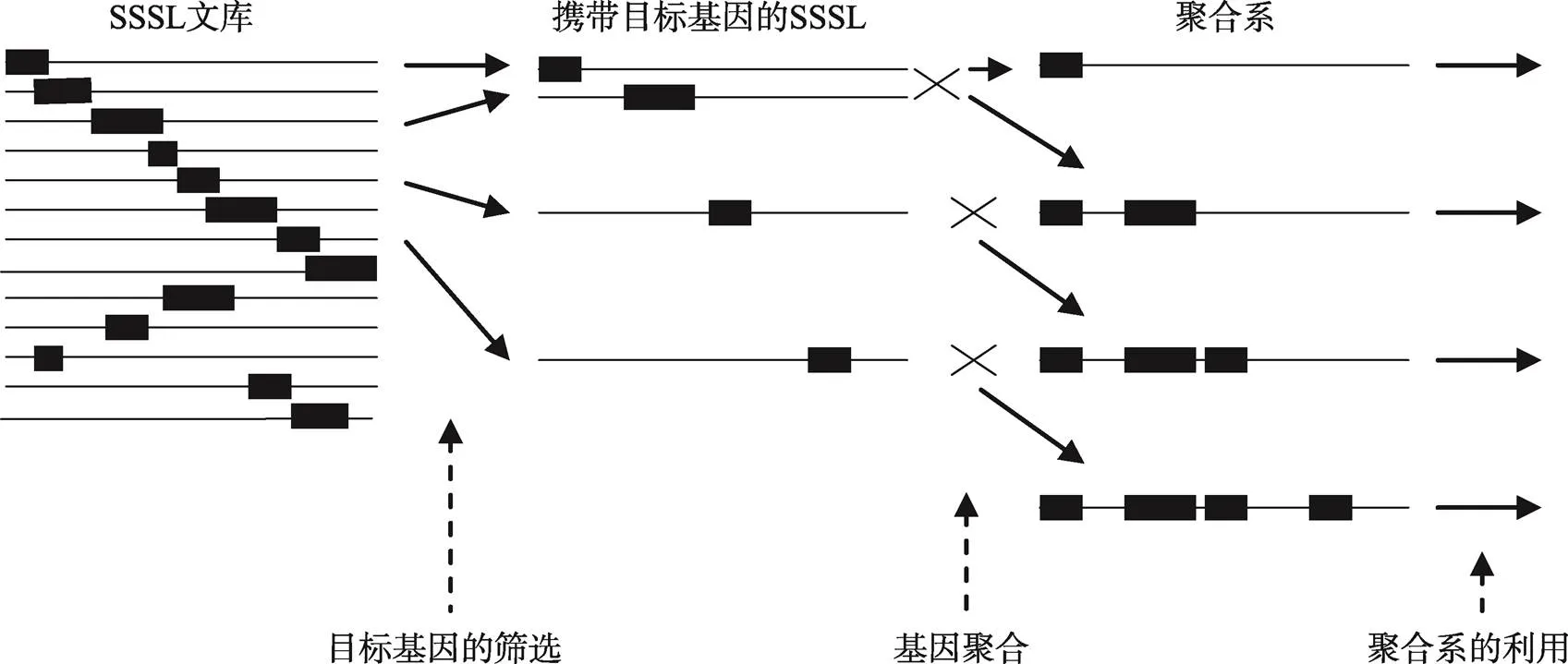
图3 基于染色体单片段代换系(SSSL)文库的水稻设计育种平台
此后,围绕更复杂的育种目标开展了进一步的设计育种。由于HJX74的稻米品质没有达到国家优质稻米标准,希望通过改良HJX74的米质,设计并培育出符合国家优质稻米标准的新品种。通过对稻米品质指标的对比分析,从SSSL文库中筛选出3个SSSL,预期将这3个SSSL携带的4个目标基因聚合在一起,可以培育出符合国家优质稻米标准的新品种。这3个SSSL中,一个携带来自Lemont第6染色体含有和基因的代换片段,另外2个分别携带来自中4188第3染色体含有基因的代换片段和第8染色体含有基因的代换片段。将这3个片段上的4个基因聚合在一起,育成了1个三片段聚合系。该三片段聚合系随后参加了广东省的水稻新品种区域试验。结果表明,该品种的米质达到国家优质稻米2级标准,产量与对照品种相当。该品种定名为“华标1号”,于2009年通过了广东省的品种审定(粤审稻2009033)。
在常规(纯合)水稻品种的设计育种取得成效后,本实验室把设计育种的注意力转移到杂交水稻“三系”(不育系、保持系和恢复系)的设计育种上。与常规品种相比,杂交水稻“三系”的设计育种需要解决细胞质雄性不育性(cytoplasmic male sterility, CMS)及其育性恢复的问题。首先,选择杂交水稻育种上广泛利用的不同类型的不育系,包括野败型(WA)的珍汕97A和博白A、矮败型(DA)的协青早A以及夜公型(YA)的华农A,为不育细胞质和核不育基因和的供体,分别与HJX74杂交,并用HJX74连续多代回交,育成了HJX74具有3种不育细胞质的同核异质不育系。HJX74不育系与HJX74-SSSL文库结合在一起,构建了基于HJX74-SSSL文库的水稻CMS不育系和保持系的设计育种平台。利用该平台对HJX74不育系进行有目的的改良,通过聚合目标基因,育成了早熟优质的不育系H131A[36]。与此同时,利用SSSL对和的等位基因进行了分析,在这两个基因座上各鉴定出4个具有不同恢复力的等位基因[25]。从中选择恢复力最强的-和-等位基因用于改良HJX74的恢复力,育成了具有强恢复力的HJX74恢复系。HJX74恢复系与HJX74-SSSL文库结合在一起,构建了基于HJX74- SSSL文库的水稻CMS恢复系的设计育种平台。利用该平台对HJX74恢复系进行有目的的改良,通过聚合目标基因,育成了优质恢复系H121R,以及优质抗稻瘟病恢复系H131R[37]。实践证明,通过对HJX74-SSSL文库有目的的拓展,构建了基于HJX74-SSSL文库的杂交水稻“三系”的设计育种平台。利用该平台开展杂交水稻“三系”的设计育种,可以设计并培育出各种各样的“三系”新品系。
5 展望
5.1 设计育种是一个长期探索的过程
研究表明,利用SSSL文库作为设计育种平台,无论是开展常规水稻育种,还是开展杂交水稻“三系”育种都是可行的。当然,水稻育种是一项复杂的工作。大多数的水稻重要性状为复杂性状,因此要全面开展水稻的设计育种,首先需要对复杂性状进行元件设计和培育。近年来,本实验室在开展上述水稻设计育种探索的同时,也对一些复杂性状的设计和培育进行了探索,包括粒形、稻米垩白度、饭粒延伸性、柱头外露率等。不但从SSSL中鉴定和定位了这些复杂性状的QTL,还通过聚合这些QTL实现了设计目标,表明这些复杂性状是可以设计和培育的,这为复杂性状的设计育种积累了经验。
下一步本实验室还将探索通过把这些复杂元件组装来设计和生产复杂产品的过程。例如,将粒形、稻米垩白度、饭粒延伸性等复杂元件组装在一起,培育特优质的水稻新品种;将不育系与柱头外露率等复杂元件组装在一起,培育具有高柱头外露率的不育系;然后再将具有高柱头外露率的不育系与特优质性状组装在一起,培育具有高柱头外露率、特优质的不育系。水稻育种是永无止境的,水稻设计育种的探索和追求也是永无止境的。
5.2 HJX74-SSSL设计育种平台的应用前景
本实验室构建的HJX74-SSSL设计育种平台,充分收集了水稻育种可利用的基因资源,实现了基因资源的收集、发掘和利用的一体化,因此可以充分利用现有的基因资源开展设计育种。实践表明,利用HJX74-SSSL设计育种平台可以解决常规水稻育种和杂交水稻“三系”育种的各种实际问题,能够设计并培育出各种各样的水稻新元件、新品系和新品种。
HJX74-SSSL设计育种平台是一个开放的平台,不仅可以利用文库中的基因资源,还可以结合其他技术拓展有用基因的利用。例如,可以利用各种诱变技术(包括基因编辑技术)诱导基因组的基因变异,也可以利用转基因技术将外源基因引入到基因组中来,从而进一步丰富育种上可利用的基因资源。同时,还可以利用信息技术和大数据技术,提高设计育种的效率和水平,实现智能化育种。这样,HJX74- SSSL设计育种平台可以通过利用各种先进技术,进一步拓展基因利用空间,提高基因利用水平,在设计育种上发挥更大的作用。
设计育种作为一个新概念、新设想、新技术、新产品还有许多问题需要探索解决,不可能一蹴而就,必然是长期的探索过程。随着新技术的不断出现和利用,设计育种无疑具有更大的拓展空间、巨大的发展潜力和广阔的应用前景。
[1] Peleman JD, van der Voort JR. Breeding by design., 2003, 8(7): 330–334.
[2] Birchler JA. An overview of rice genetics research in China., 2018, 45(11): 563–564.
[3] Bai S, Yu H, Wang B, Li J. Retrospective and perspective of rice breeding in China., 2018, 45(11): 603–612.
[4] Wu B, Hu W, Xing Y. The history and prospect of rice genetic breeding in China., 2018, 40(10): 841–857.吴比, 胡伟, 邢永忠. 中国水稻遗传育种历程与展望. 遗传, 2018, 40(10): 841–857.
[5] Qian Q, Guo L, Smith SM, Li J. Breeding high-yield superior quality hybrid super rice by rational design., 2016, 3(3): 283–294.
[6] Zeng D, Tian Z, Rao Y, Dong G, Yang Y, Huang L, Leng Y, Xu J, Sun C, Zhang G, Hu J, Zhu L, Gao Z, Hu X, Guo L, Xiong G, Wang Y, Li J, Qian Q. Rational design of high-yield and superior-quality rice., 2017, 3: 17031.
[7] Lu BR, Sharma SD, Shastry SVS. Taxonomy of the genus(Poaceae): historical perspective and current status., 1999, 24: 4–8.
[8] Khush GS. Origin, dispersal, cultivation and variation of rice., 1997, 35(1–2): 25–34.
[9] Zhang GQ, Zeng RZ, Zhang ZM, Ding XH, Li WT, Liu GF, He FH, Tulukdar A, Huang CF, Xi ZY, Qin LJ, Shi JQ, Zhao FM, Feng MJ, Shan ZL, Chen L, Guo XQ, Zhu HT, Lu YG. The construction of a library of single segment substitution lines in rice (L.)., 2004, 21: 85–87.
[10] He FH, Xi ZY, Zeng RZ, Talukdar A, Zhang GQ. Developing single segment substitution lines (SSSLs) in rice (L.) using advanced backcrosses and MAS., 2005, 32(8): 825–831.何风华, 席章营, 曾瑞珍, Akshay Talukdar, 张桂权. 利用高代回交和分子标记辅助选择建立水稻单片段代换系. 遗传学报, 2005, 32(8): 825–831.
[11] Xi ZY, He FH, Zeng RZ, Zhang ZM, Ding XH, Li WT, Zhang GQ. Development of a wide population of chromosome single-segment substitution lines in the genetic background of an elite cultivar of rice (L.)., 2006, 49(5): 476–484.
[12] He N, Wu RX, Pan XP, Peng LP, Sun K, Zou T, Zhu HT, Zeng RZ, Liu ZQ, Liu GF, Wang SK, Zhang GQ, Fu XL. Development and trait evaluation of chromosome single-segment substitution lines ofin the background of., 2017, 213(12): 281.
[13] Zhao HW, Sun LL, Xiong TY, Wang ZQ, Liao Y, Zou T, Zheng MM, Zhang Z, Pan XP, He N, Zhang GQ, Zhu HT, Liu ZQ, He P, Fu XL. Genetic characterization of the chromosome single-segment substitution lines ofandand identification of QTLs for yield-related traits., 2019, 39: 51.
[14] Zhao FM, Zhu HT, Ding XH, Zeng RZ, Zhang ZM, Li WT Zhang GQ. Detection of QTLs for important agronomic traits and analysis of their stabilities using SSSLs in rice., 2007, 40(3): 769–778.赵芳明, 朱海涛, 丁效华, 曾瑞珍, 张泽民, 李文涛, 张桂权.基于SSSL的水稻重要性状QTL的鉴定及稳定性分析. 中国农业科学, 2007, 40(3): 447–456.
[15] Liu GF, Zhu HT, Liu S, Zeng RZ, Zhang Z, Li W, Ding X, Zhao F, Zhang G. Unconditional and conditional QTL mapping for the developmental behavior of tiller number in rice (L.)., 2010, 138(8): 885– 893.
[16] Zhang Y, Yang J, Shan Z, Chen S, Qiao W, Zhu X, Xie Q, Zhu H, Zhang Z, Zeng R, Ding X, Zhang G. Substitution mapping of QTLs for blast resistance with SSSLs in rice (L.)., 2012, 184(1): 141–150.
[17] Naeem M, Freed S, Zhang GQ. Molecular genetic studies of heading date geneby using single segment substitution lines in., 2013, 15: 631–639.
[18] Zhu Y, Zuo S, Chen Z, Chen X, Li G, Zhang Y, Zhang G, Pan X. Identification of two major rice sheath blight resistance QTLs,HJX74andHJX74, in field trials using chromosome segment substitution lines., 2014, 98(8): 1112–1121.
[19] Yang TF, Zhang SH, Zhao JL, Liu Q, Huang ZH, Mao XX, Dong JF, Wang XF, Zhang GQ, Liu B. Identification and pyramiding of QTLs for cold tolerance at the bud bursting and the seedling stages by use of single segment substitution lines in rice (L.)., 2016, 36: 96.
[20] Zhou Y, Xie Y, Cai J, Liu C, Zhu H, Jiang R, Zhong Y, Zhang G, Tan B, Liu G, Fu X, Liu Z, Wang S, Zhang G, Zeng R. Substitution mapping of QTLs controlling seed dormancy using single segment substitution lines derived from multiple cultivated rice donors in seven cropping seasons., 2017, 130(6): 1191–1205.
[21] Okpala NE, Duan L, Shen G, Zhang G, Qi X. Comparisons of cooking and eating qualities of tworice cultivars., 2017, 5: 180.
[22] Zhu H, Li Y, Liang J, Luan X, Xu P, Wang S, Zhang G, Liu GF. Analysis of QTLs on heading date based on single segment substitution lines in rice (L.)., 2018, 8(1): 13232.
[23] Zeng RZ, Zhang ZM, He FH, Xi ZY, Talukdar A, Shi JQ, Qin LJ, Huang CF, Zhang GQ. Identification of multiple alleles at thelocus and development of single segment substitution lines for the alleles in rice., 2006, 13(1): 9–14.
[24] Teng B, Zeng RZ, Wang YC, Liu ZQ, Zhang ZM, Zhu HT, Ding XH, Li WT, Zhang GQ. Detection of allelic variation at thelocus with single-segment substitution lines in rice (L.)., 2012, 30(1): 583–595.
[25] Cai J, Liao Q, Dai Z, Zhu H, Zeng R, Zhang Z, Zhang G. Allelic differentiations and effects of theandgenes on fertility restoration in rice with wild abortive cytoplasmic male sterility., 2013, 57(2): 274–280.
[26] Liu G, Zhang Z, Zhu H, Zhao F, Ding X, Zeng R, Li W, Zhang G. Detection of QTLs with additive effects and additive-by-environment interaction effects on panicle number in rice (L.) with single-segment substitution lines., 2008, 116(7): 923– 931.
[27] Liu G, Zhu H, Zhang G, Li L, Ye GY. Dynamic analysis of QTLs on tiller number in rice (L.) with single segment substitution lines., 2012, 125(1): 143–153.
[28] Chen J, Li X, Cheng C, Wang Y, Qin M, Zhu H, Zeng R, Fu X, Liu Z, Zhang G. Characterization of epistatic interaction of QTLsandcontrolling heading date in rice., 2014, 4: 4263.
[29] Qin M, Zhao X, Ru J, Zhang G, Ye GY. Bigenic epistasis between QTLs for heading date in rice analyzed using single segment substitution lines., 2015, 178: 16–25.
[30] Zhao F, Zhu H, Zeng R, Zhang G, Xu SZ. Detection of additive and additive × environment interaction effects of QTLs for yield-component traits of rice using single- segment substitution lines (SSSLs)., 2016, 135(4): 452–458.
[31] Yang Z, Jin L, Zhu H, Wang S, Zhang G, Liu G. Analysis of epistasis among QTLs on heading date based on single segment substitution lines in rice., 2018, 8(1): 3059.
[32] Wang SK, Wu K, Yuan Q, Liu X, Liu Z, Lin X, Zeng R, Zhu H, Dong G, Qian Q, Zhang G, Fu X. Control of grain size, shape and quality byin rice., 2012, 44(5): 950–954.
[33] Wang S, Li S, Liu Q, Wu K, Zhang J, Wang S, Wang Y, Chen X, Zhang Y, Gao C, Wang F, Huang H, Fu X. Theregulatory module determines grain shape and simultaneously improves rice yield and grain quality., 2015, 47(8): 949–954.
[34] Fang C, Li L, He R, Wang D, Wang M, Hu Q, Ma Q, Qin K, Feng X, Zhang G, Fu X, Liu Z. Identification ofcausing both interspecific hybrid male sterility and environment-conditioned male sterility in rice., 2019, 12(1): 10.
[35] Sui F, Zhao D, Zhu H, Gong Y, Tang Z, Huang XY, Zhang G, Zhao FJ. Map-based cloning of a new total loss-of- function allele ofcauses high cadmium accumulation in rice grain., 2019, 70(10): 2857–2871.
[36] Dai Z, Lu Q, Luan X, Cai J, Zhu H, Liu Z, Zeng R, Zhang Z, Wang S, Zheng L, Li J, Zhang G. Development of a platform for breeding by design of CMS lines based on an SSSL library in rice (L.)., 2015, 205(1): 63–72.
[37] Dai Z, Lu Q, Luan X, Ouyang L, Guo J, Liang J, Zhu H, Wang W, Wang S, Zeng R, Liu Z, Zhang Z, Zhu X, Zhang G. Development of a platform for breeding by design of CMS restorer lines based on an SSSL library in rice (L.)., 2016, 66(5): 768–775.
The platform of breeding by design based on the SSSL library in rice
Guiquan Zhang
Breeding by design is a new concept proposed in the beginning of the century. It refers to the breeding of varieties by crop design utilizing favorable alleles dispersed in different genetic resources in a genome. In the past 20 years, we have proposed a "three-step" strategy to carry out the research on breeding by design in rice. Firstly, we constructed a library of chromosomal single-segment substitution lines (SSSLs) by using of Huajingxian74 (HJX74), an elite() variety from South China as the recipient and 43 accessions of seven species of rice AA genome as donors. The genes in the substituted segments of SSSLs were then detected. Breeding by design was conducted by selecting the favorable genes from the SSSL library. Our practice indicates that the SSSL library is a powerful platform for breeding by design and various "traits", "lines" and "varieties" of rice can be designed and bred by utilizing abundant genes in the SSSL library. Here, we introduce the platform of the HJX74-SSSL library and our work of breeding by design on the platform. It will provide a case study for crop design.
breeding by design; molecular breeding; single-segment substitution line (SSSL); gene pyramiding; rice
2019-04-16;
2019-07-04
国家自然科学基金项目(编号:91735304,91435207)和广州市科技计划项目(编号:201607020040)资助[Supported by the National Natural Science Foundation of China (Nos. 91735304, 91435207) and the Project of Science and Technology of Guangzhou (No. 201607020040)]
张桂权,博士,教授,研究方向:水稻分子育种。E-mail: gqzhang@scau.edu.cn
10.16288/j.yczz.19-105
2019/7/15 10:15:14
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20190715.1014.002.html
(责任编委: 储成才)
