不同择伐方式下云南松群体遗传多样性SSR分析
2019-08-08许玉兰汪梦婷蔡年辉王亚楠
许玉兰,汪梦婷,蔡年辉,王亚楠
(1.西南林业大学 云南省高校林木遗传改良与繁育重点实验室,云南 昆明 650224;2.西南林业大学 西南山地森林资源保育与利用教育部重点实验室,云南 昆明 650224)
【研究意义】遗传多样性是生物多样性的重要方面,也是森林生态系统可持续经营策略制定与评价中的重要参数[1],对保护资源和合理利用有重要意义[2],多采用形态、细胞学、生化和分子标记进行评价,其中分子标记是基于DNA多态性而建立起来的标记形式,以SSR标记即微卫星标记的应用较为广泛[3-4],该方法在云南松群体遗传多样性的评价中也得到应用[5]。云南松是中国西南地区重要的造林树种,但是,云南松林多为纯林,其林分稳定性和生产力下降[6]。云南松纯林中的许多个体表现为低矮、弯曲扭曲等,导致林分质量下降和遗传品质退化[7],势必会影响云南松林分应有效益的发挥。因此,开展择伐对提高林分质量具有重要意义。【前人研究进展】已有的研究多集中在林分结构和林木生长方面,研究表明,抚育间伐可促进林木的胸径生长和树高生长[8-10]。【本研究切入点】择伐因保留种质不同,可能对林分的遗传结构造成影响,分析不同择伐条件下遗传多样性变化对于确定合理的抚育管理措施具有重要实践意义。【拟解决的关键问题】采用SSR分子标记分析技术,通过对比留优去劣、无人为择伐、伐优留劣3种不同择伐方式的云南松群体遗传多样性变化,及群体遗传变异特征,揭示它们的遗传多样性水平、遗传关系以及遗传分化。以期为云南松的经营管理提供指导。

表1 云南松采样群体的地理位置[13]
1 材料与方法
1.1 材料
样地选择在云南省双江县境内,按“留优去劣”、“无人为择伐”和“伐优留劣”不同择伐方式各设置1个样地(30 m×30 m)[11]。2015年11月进行调查和采样,采集样地中每株云南松树冠中上部位两年生针叶,用自封袋保存并进行单株编号,带回实验室备用,每个样地的植株数100~200株[11-13]。后述分析中,以每一种择伐方式的样地作为一个群体。样本采集信息详见表1。
1.2 DNA提取与SSR-PCR扩增
云南松针叶DNA的提取采用4×CTAB提取法[14-15]。SSR引物信息、SSR扩增反应体系与扩增程序、扩增产物检测同前期研究[12-13,16],15对引物分别为PtTX2123[17]、PtTX2146[17]、PtTX3118[18]、PtTX3127[18]、Pr001[19]、PyMR05[20]、PyMR06[20]、PyMR08[20]、PyTr06[21]、PyTr09[21]、PyTr19[21]、PyTr20[21]、PyTr26[21]、PyTr28[21]、PyTr31[21]。
1.3 数据分析
数据的收集与分析同前期研究[5,11,13],采用GeneMarker V2.2.0收集数据、采用Convert l.3.1对数据进行转换、采用GenALEx 6.4和Arlequin 3.0进行遗传多样性及遗传分化的分析。根据遗传距离,采用NTSYSpc 2.10s软件中的算术平均数非加权成组配对法,构建UPGMA聚类图。

表2 不同位点扩增统计

表3 云南松群体的遗传多样性
2 结果与分析
2.1 不同位点扩增分析
如表2所示,15对引物共检测74个等位基因,平均每个位点4.911个等位基因。有效等位基因数波动于1.004(PyMR05)~4.611(PtTX2146),平均每个位点2.121个有效等位基因。Shannon’s信息指数为0.012(PyMR05)~1.765(PtTX2146)。观测杂合度为0.004~0.833、期望杂合度为0.004~0.782,平均分别为0.285和0.423,各位点的扩增效果有所不同,以位点PtTX2146扩增的多态性较高。
F统计量分析(表2)可知,FIS在位点PyMR05和PyMR06表现为负值,其余位点均表现为正值,平均值为0.368(>0),存在一定程度的偏离Hardy-Weinberg平衡,且在群体内个体间表现为杂合子缺失;FIT在PyMR05和PyMR06位点中表现为负值,其余位点均表现为正值,平均值为0.375(>0),同样存在一定程度的偏离Hardy-Weinberg平衡,并表现为杂合子缺失。群体间遗传分化系数FST为0.001(PyTr28)~0.037(Pr001),平均值为0.012,即群体间存在1.2 %的遗传变异,而98.8 %的遗传变异存在于群体内,说明云南松变异主要来源于群体内。基因流在不同位点的变化为6.495~173.409,位点间差异较大,各位点间平均基因流为35.316,表明它们之间的基因流动频繁。
2.2 云南松群体的遗传多样性分析
对云南松群体的遗传多样性参数进行估算(表3),等位基因数为4.600~5.267,平均为4.911;有效等位基因数2.061~2.195,平均为2.121,其中留优去劣群体(P1)的等位基因数和有效等位基因数最高,分别为5.267和2.195,伐优留劣群体(P3)的等位基因数和有效等位基因数最低,分别为4.600和2.061。各群体间的等位基因数和有效等位基因数差异较小,均表现较高水平。观测杂合度波动于0.272~0.305,期望杂合度波动于0.412~0.440,平均值分别为0.285和0.423,各群体间差异较小,均表现为期望杂合度稍高于观测杂合度,表明3个群体的杂合子缺失。3个群体Shannon’s信息指数的变化范围为0.791~0.871,平均为0.819,各群体的Shannon’s信息指数差异很小(0.080),表现为:P1>P3>P2。在3个群体中,多态性位点百分率为93.33 %~100.00 %,P1为100.00 %。3个群体检测到的私有基因波动为3~10个,留优去劣群体(P1)最多,伐优留劣群体(P3)最少。各群体的固定指数(F)介于0.299~0422,平均为0.374。整体来看,3个群体的遗传多样性都非常接近,且其多态性位点百分率较高,遗传多样性丰富。综合比较各个遗传参数值的大小,伐优留劣群体(P3)的遗传多样性较低,留优去劣群体(P1)的遗传多样性较高,但它们之间的差异均较小,即云南松群体遗传多样性对不同择伐方式的响应不明显。
2.3 云南松群体的遗传分化
对云南松成对群体间的遗传分化系数进行估算(表4)。结果表明,FST值介于0.008~0.013,平均值为0.010,FST值在无人为择伐群体(P2)和伐优留劣群体(P3)之间最大、在留优去劣群体(P1)和其它群体之间较小。比较各成对群体间的FST值均小于0.05,说明群体间的遗传分化很小[22]。相应的,成对群体间的基因流介于19.701~32.288,平均为27.627,各成对群体间的基因流远大于1,各成对群体间的基因流强度大、频率高,削弱群体间遗传分化。

表4 云南松群体的分化系数和基因流
注:对角线以下为FST;对角线以上为Nm。群体信息详见表1。
Note:FSTvalues below diagonal,Nm values above diagonal. Populations are described in table 1.

表5 云南松群体间和群体内分子方差分析
为进一步探明云南松群体的遗传变异的分布情况,采用Arlequin3.0软件对云南松群体进行分子方差分析(AMOVA)(表5)。变异分量百分比表明云南松所有群体总体的遗传变异主要分布于群体内(98.45 %),而群体间的遗传变异只占1.55 %,揭示了云南松群体内的变异是其变异的主要来源。
2.4 云南松群体的遗传距离及其聚类
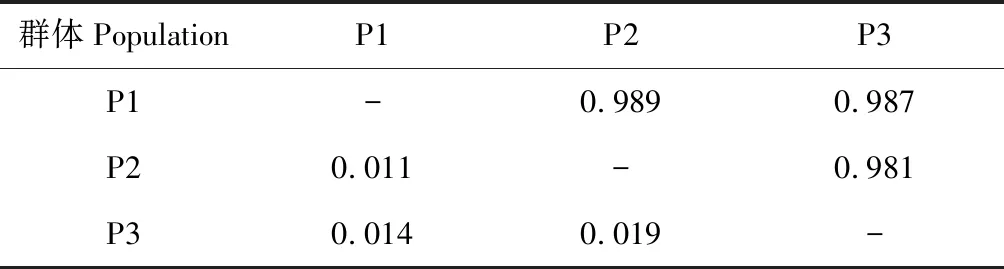
为进一步比较云南松群体间的遗传关系,用GenALEx6.4软件计算云南松成对群体间的遗传一致度(I)和遗传距离(D)(表6)。各成对群体间的遗传一致度为0.981~0.989,平均0.985,遗传一致度的最小值在无人为择伐群体(P2)和伐优留劣群体(P3)之间,最大值在留优去劣群体(P1)和无人为择伐群体(P2)之间。但是,总体来看,各成对群体间的遗传一致度和遗传距离差异都非常小,整体表现为群体间遗传一致度高,遗传距离低。
基于遗传距离进行算术平均数非加权成组配对法(UPGMA)聚类分析(图1)。当阈值约为0.016时,可把3个云南松群体分为2个类群,即留优去劣群体(P1)和无人为择伐群体(P2)聚为一类,伐优留劣群体(P3)为一个类。当阈值约为0.011时,留优去劣群体(P1)和无人为择伐群体(P2)又各为一个类。整体来看,各群体间的遗传距离小,说明各群体在择伐方式上无明显的遗传分化。
表6 云南松群体间遗传一致度和遗传距离
Table 6 Genetic identity and genetic distance among populations ofPinusyunnanensis
群体PopulationP1P2P3P1-0.9890.987P20.011-0.981P30.0140.019-
注:对角线以下为遗传距离;对角线以上为遗传一致度。
Note: Genetic distance is listed below diagonal and genetic identity is listed and above diagonal.
3 讨 论
运用的15对SSR引物表达出较为丰富的遗传多态性,在检测的3个群体中,等位基因数和有效等位基因数平均分别为4.911和2.121,观测杂合度和期望杂合度波动于0.272~0.305和0.412~0.440,平均值分别为0.285和0.423,此值远大于Vendramin等[23]所提出的遗传衰退临界值(H﹤0.05)。3个群体中,Shannon’s信息指数的变化为0.791~0.871,平均为0.819。多态位点百分率为93.33 %~100.00 %,平均为95.56 %,总体来看,云南松群体的遗传多样性丰富。比较留优去劣群体、无人为择伐群体和伐优留劣群体的各项遗传参数,它们无明显变化。因此,云南松遗传多样性与择伐方式间无明显的关联性,以留优去劣择伐方式的云南松群体的遗传多样性稍高。
通过F统计量分析可知,FIS平均值为0.368,FIT平均值为0.375,说明无论是在整体水平上还是群体内个体间,云南松群体都表现为杂合子缺失现象;FST平均值为0.012,表明云南松群体遗传变异主要来源于群体内(98.8 %),群体间的遗传分化很小。分子方差分析(Molecular AMOVA)也表明,云南松群体的遗传变异主要存在群体内(94 %),群体间的遗传变异很小(1 %),与前期研究报道相似[5]。云南松成对群体的基因流平均为27.627,远远大于1,云南松的分布范围十分广泛,生态环境复杂多样,但是云南松是典型的风媒传粉植物,花粉具有气囊,借助气流可以远距离传播,它的种子有种翅,传播距离远,从而加强了群体间的基因交流、削弱群体间的遗传分化。

图1 基于遗传距离的不同择伐方式云南松群体的UPGMA聚类图Fig.1 UPGMA cluster dendrogram of Pinus yunnanensis populations sampled from different logging modes based on genetic distance
不同择伐方式的云南松群体遗传分化不明显,但是留优去劣择伐方式的云南松群体的遗传多样性高于其余2个群体,在聚类图中也区别于伐劣留优群体。其遗传分化不明显一部分原因可能是由于云南松生物特性导致的基因互渗引起的。综上分析,无人为择伐群体与伐优留劣群体遗传距离稍远,在个别遗传参数上伐优留劣群体大于无人为择伐群体。因此,择伐在一定程度上会诱导遗传多样性的变化,前人研究报道表明,择伐后可调整林分结构[8-9],激发土壤微生物活性,加速土壤养分循环,促进林木生长[10]。同时云南松群体生境的异质性是云南松群体遗传变异丰富的重要原因,要重视对云南松群体周围生境的保护,同时避免伐优留劣逆向选择的方式,进而保护云南松群体遗传多样性。当然,云南松林的间伐还受间伐强度、间伐始期、次数等的影响[8-9],对其遗传多样性的影响也是长期的,可进一步开展相应研究。
4 结 论
不同择伐方式的云南松群体遗传多样性丰富,各群体间的遗传分化低,基因流较高,云南松群体的变异主要来源于群体内,不同择伐方式对云南松群体遗传多样性的影响不明显,但是留优去劣择伐方式的云南松群体的遗传多样性稍高于其余2个群体。因此,生产中可对林分适度择伐,且保留优质木。研究结果可为云南松群体遗传多样性保护与利用的改良策略的制定提供科学依据。
