血清circRNA与甲状腺乳头状癌的相关性研究
2019-08-05杨雯雯杨镇玮依地热斯艾山
白 超, 杨雯雯, 罗 军, 田 野, 张 磊, 杨镇玮, 王 亮, 依地热斯·艾山
(新疆医科大学1第一临床医学院血管甲状腺外科, 乌鲁木齐 830054; 2第二附属医院内分泌科, 乌鲁木齐 830028)
甲状腺结节是头颈部常见的多发病,近年来有高发的态势,人群中甲状腺结节的检出率也随之升高,检出结节中恶性的占比为10%~15%[1-2]。在恶性结节中,甲状腺微小乳头状癌明显增多。甲状腺微小乳头状癌(papillary thyroid mircocarcinoma,PTMC)被定义为甲状腺乳头状癌病变最大处的直径≤1 cm,是最“懒惰”的甲状腺恶性肿瘤[3-4]。这类PTMC患者多数由于甲状腺结节的生长位置较深而且结节体积较小,不适合做穿刺活检。虽然甲状腺影像报告和数据系统(Thyroid imaging reporting and data system,简称为TI-RADS)[5]被广泛采纳,该系统更加客观的判断甲状腺结节的性质,但临床中仍有一部分结节难以判断其良、恶性。
环状RNA(circular RNA,circRNA)是20世纪70年代在植物类病毒中发现的一类非编码的RNA,其单链和共价封闭的RNA,缺少3′游离帽子端或5′多聚尾巴端[6-7]。哺乳动物的细胞中广泛存在着环状RNA分布[8],环状RNA在细胞中稳定存在[9],而且具有miRNA海绵的功能,即吸附miRNA[10]。近1~2年来circRNA与恶性肿瘤发生和发展的相关性已经成为学术界研究热点,例如:circRNA与膀胱癌的发生、发展的相关性研究[11],circRNA与乳腺癌早期诊断的相关性研究[12],circRNA与结肠癌的发生、发展的相关性研究[13],circRNA与抑肝癌细胞增殖的相关性研究[14]等。虽然circRNA与人类各种恶性肿瘤之间的关系已经得到了一定的评价和建立,但是circRNA在PTMC中的诊断价值和生物学功能的作用研究较少,Vea等[15]研究表明circRNA在红细胞、白细胞及血清中均有大量的表达。
本研究比较PTMC不伴颈部淋巴结转移的患者和结节性甲状腺肿(nodular goiter,NG)患者血清中circRNA表达的差异,探讨circRNA在PTMC发生、发展中的作用,寻找可能的甲状腺癌血清标记物,帮助临床医师在术前对甲状腺结节良、恶性进行初步鉴别,并以此为基础,为甲状腺结节患者制定下一步治疗方案,以此提高甲状腺结节的诊断和治疗水平。
1 资料与方法
1.1 研究对象选择2018年11-12月在新疆医科大学第一附属医院甲状腺外科行甲状腺切除术,并经病理证实的结节性甲状腺肿患者(NG组)和PTMC患者(PTMC组)各13例,均为女性。收集研究对象的一般资料。术前收集患者晨起空腹上肢静脉血5 mL,离心得血清,将其储存在-80℃的冰箱内,解冻后可使用。2组各选取3名患者进行circRNA芯片检测(NG组3例患者年龄分别为40、52和60岁;PTMC组3例患者年龄分别为40、49和53岁),后再各选取3例对circRNA芯片检测结果进行验证,2组各剩余7例,对血清样本进行circRNA扩大样本验证。本研究经新疆医科大学第一附属医院伦理委员会审核并批准(伦理审批号为:20181029-17)。手术前告知患者其权利和义务,并签署书面知情同意书。纳入标准:(1)年龄>18岁;(2)甲状腺结节直径≤1 cm;(3)均无甲状腺恶性肿瘤家族史;(4)均无头颈部放射史;(5)均无甲状腺疾病病史,未行甲状腺手术史。排除标准:(1)术前已经使用补充甲状腺素药物或者抗甲状腺素药物;(2)肝肾功能异常的患者;(3)妊娠期患者。
1.2 方法
1.2.1 circRNA表达谱芯片检测 circRNA表达谱芯片检测委托上海康成生物有限公司进行。
1.2.1.1 实验步骤 (1)血清总RNA的提取(提取方法参阅Trizol-LS Invitrogen说明书),包括冷冻血清解冻、血清匀浆、两相分离、RNA沉淀、RNA清洗及重新溶解RNA沉淀过程;(2)RNA质量检测;(3)RNA浓度测定,使用NanoDrop-1000超微量分光光度仪测定RNA的浓度和纯度;(4)总RNA的质量检测;(5)样品标记;(6)标记扩增后的cRNA并质检;(7)芯片预杂交;(8)配制杂交液;(9)芯片杂交;(10)洗片;(11)芯片扫描。
1.2.1.2 实验主要试剂、仪器和设备 TRIZOL-LS试剂(购自Invitrogen Life technologies),Cy3标记链霉亲和素(购自sher Scientific公司),CyDyev Fluors(CF)荧光染料(购自Healthcare公司),RNA扩增试剂盒(恒温扩增法)(购自kiinina公司),DEPC(购自gma公司),琼脂糖(Agarose)(购自owest公司),无RNA酶的水(购自vitrogen life technologies公司)。Agilent扫描仪(购自安捷伦科技有限公司Agilent),NanoDrop-1000超微量分光光度仪(购自Thermo Scientific公司),计算机图像分析系统(购自Leica Qwi 3),基因芯片杂交仪(购自Leica公司)。
1.2.2 circRNA芯片数据的生物信息学分析 circRNA芯片数据的生物信息学分析委托上海康成生物有限公司进行。
1.2.2.1 差异基因(Differentially Expressed Genes,DEGs)筛选 将原始数据导入生物通路可视化软件,采用基因表达差异显著性分析方法,P<0.05为差异有统计学意义。通过R语言的limma语言包对芯片原始数据进行统一的标准化处理。
1.2.2.2 聚类分析 采用Pusamen和Cluser 2.2软件,将数据导入其中,进行聚类分析,上述软件将算得的结果以聚类图的形式进行输出。
1.2.2.3 基因本体(Gene Ontology)分析 采用DAVID分析软件,将PTMC组和NG组2组间存在差异表达的环状RNA数据导入DAVID,DAVID软件对导入的数据进行分析后,将自动计算出分类的每个种类、每个种类所包含的基因、P值等内容,再根据P值的大小依次将结果进行输出。
1.2.2.4 信号通路分析 将“1.2.2.3”基因本体分析步骤中获得的数据导入Pathway Studio 5.0软件和DAVID分析软件,在软件中选择信号通路分析的功能,软件会自动输出与基因相关的信号通路,并计算出P值的大小。
1.2.2.5 circRNA/miRNA结合位点分析 使用美国Arraystar公司的miRNA结合位点预测软件为前面部分得出的每个差异倍数在1.5倍以上的circRNA预测5个具有较高匹配值的miRNA结合位点,形成circRNA-miRNA关系网络图。
1.2.3 RT-PCR验证 RT-PCR验证委托乌鲁木齐市格林金诺生物科技有限公司进行,采用RT-PCR实验原理进行验证。
1.2.3.1 选择验证的circRNA 基于PTMC组和NG组基因芯片的检测,根据2组circRNA差异表达的Fold change倍数和P值,选取了3个上调的circRNA和3个下调的circRNA进行RT-PCR验证。
1.2.3.2 聚合酶链式反应PCR (1)引物合成:逆转录PCR所使用的引物由上海生工公司Primer Premier 5.0软件设计、合成,以管家基因GAPDH为内参(表1)。(2)Real time-PCR反应;(3)PCR扩增。

表1 RT-PCR所用引物
1.2.3.3 数据读取 打开Agilent数据处理软件Feature Extraction进行芯片扫描数据的读取。
1.2.3.4 数据分析 采用Aglieng提供的锌芯片扫描仪扫描芯片,对低强度信号值进行过滤。借助截尾均数(Trimmed Mean,TM)对芯片的初始信号值进行统一的标准化处理。
2 结果
2.1 circRNA表达谱芯片检测
2.1.1 血清提取总RNA质检结果和cRNA荧光染料的标记效率
2.1.1.1 RNA浓度和纯度测定 所有样品的A260/A280的比值为1.12~1.57,样品浓度为189.14~288.03 ng/μL(表2)。
2.1.1.2 cRNA特异性标记活性 对1 μg cRNA采用Cy3染料进行标记,特异性标记活性即为每1 μg cRNA中含有多少pmol的染料,放射性比度=(pmol per μL dye)/(μg per μL cRNA)。如果出现染料呈双色,则收益率是<825 ng和放射性比度<8 pmol Cy3或Cy5每μg cRNA不进行杂交步骤。重复cRNA准备。如果染料为一种颜色,收益率是<1.65 μg和放射性比度<9 pmol Cy3或Cy5每μg cRNA不进行杂交步骤。重复cRNA准备(表3)。
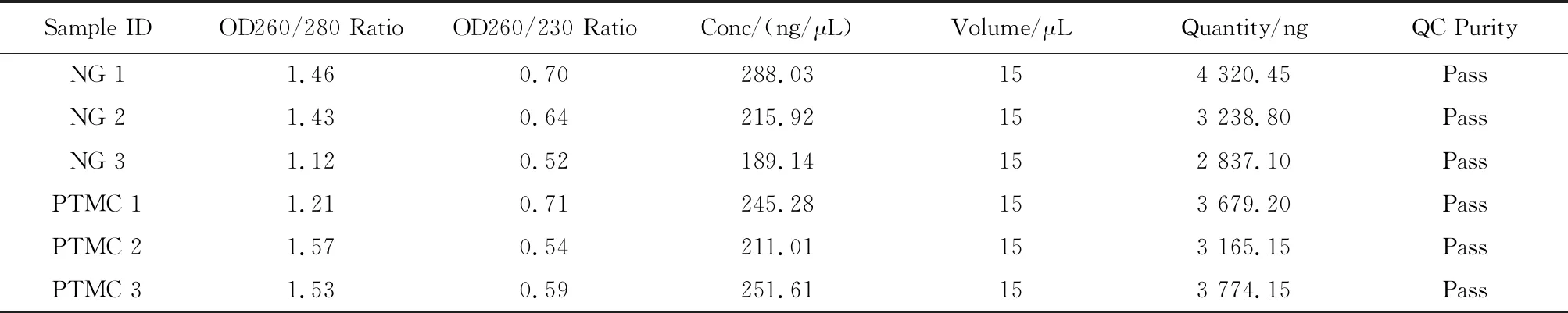
表2 利用NanoDrop ND-1000进行RNA定量和质量保证
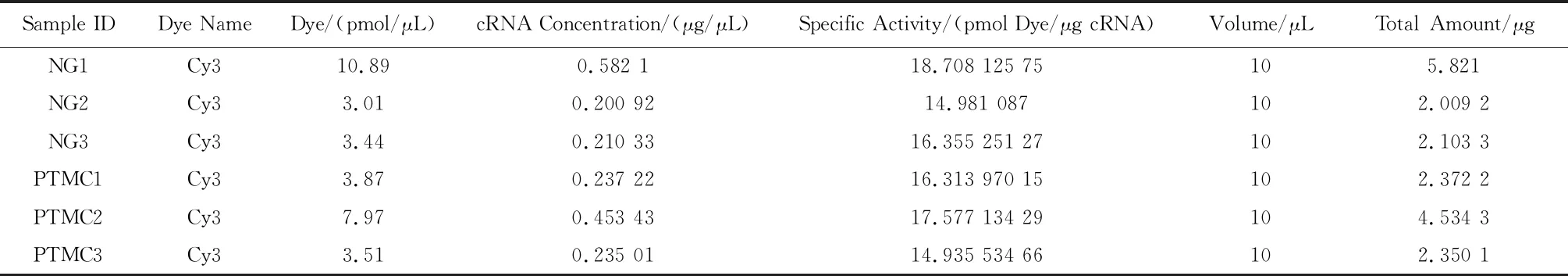
表3 cRNA的标记效率
2.1.2 circRNA芯片数据分析
2.1.2.1 所有表达的circRNA circRNA芯片检测到12 900个circRNA在NG、PTMC血清中表达,检测6个血清样本中这些circRNA的表达水平的一致性,进一步分析其表达差异。芯片检测到的部分circRNA信息,包括原始circRNA信号值,标准化后的信号值,基因表达水平标识,基因序列、类型等见表4。
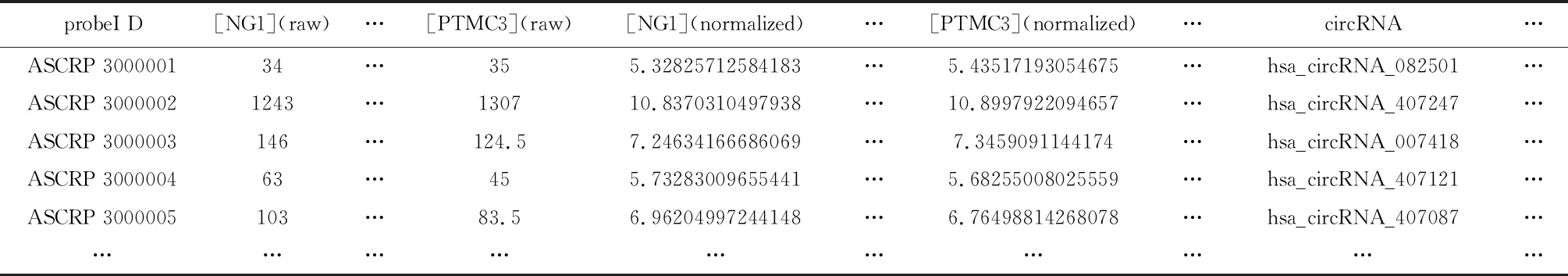
表4 xls.格式的circRNA表达配置文件片段
注:probe ID:探针序列号;circRNA:circular RNA序列号;NG1~PTMC3(raw):各个标本的原始信号强度;NG1~PTMC3(normalized):各个标本的原始信号强度标准化后取对数值(log2 transformed)。
2.1.2.2 circRNA数据的质量评估 箱形图是将数据的分布采用四方形的盒子形式加以形象地展示,可以从视觉的效果上快速简便地观察一组数据的分布情况,包括集中趋势和离散趋势(图1)。
2.2 circRNA芯片数据的生物信息学分析
2.2.1 PTMC血清中差异表达circRNA的筛选 筛选差异基因标准是P<0.05,组间差异倍数>1.5,结果得到差异表达的circRNA有20个,其中表达上调的circRNA有14个,分别是hsa_circRNA_406821,hsa_circRNA_406395,hsa_circRNA_003416,hsa_circRNA_407160,hsa_circRNA_087192,hsa_circRNA_001359,hsa_circRNA_000639,hsa_circRNA_104982,hsa_circRNA_100015,hsa_circRNA_104640,hsa_circRNA_104172,hsa_circRNA_001131,hsa_circRNA_407191,hsa_circRNA_081199。下调的circRNA有6个,分别是hsa_circRNA_043614,hsa_circRNA_104755,hsa_circRNA_103037,hsa_circRNA_101871,hsa_circRNA_405481,hsa_circRNA_103944。
采用火山图直观显示NG组与PTMC组中血清学差异表达的circRNA。图中红色方格代表差异基因,两条绿色垂直线对应差异倍数为1.5,绿色水平线对应P=0.05(图2)。
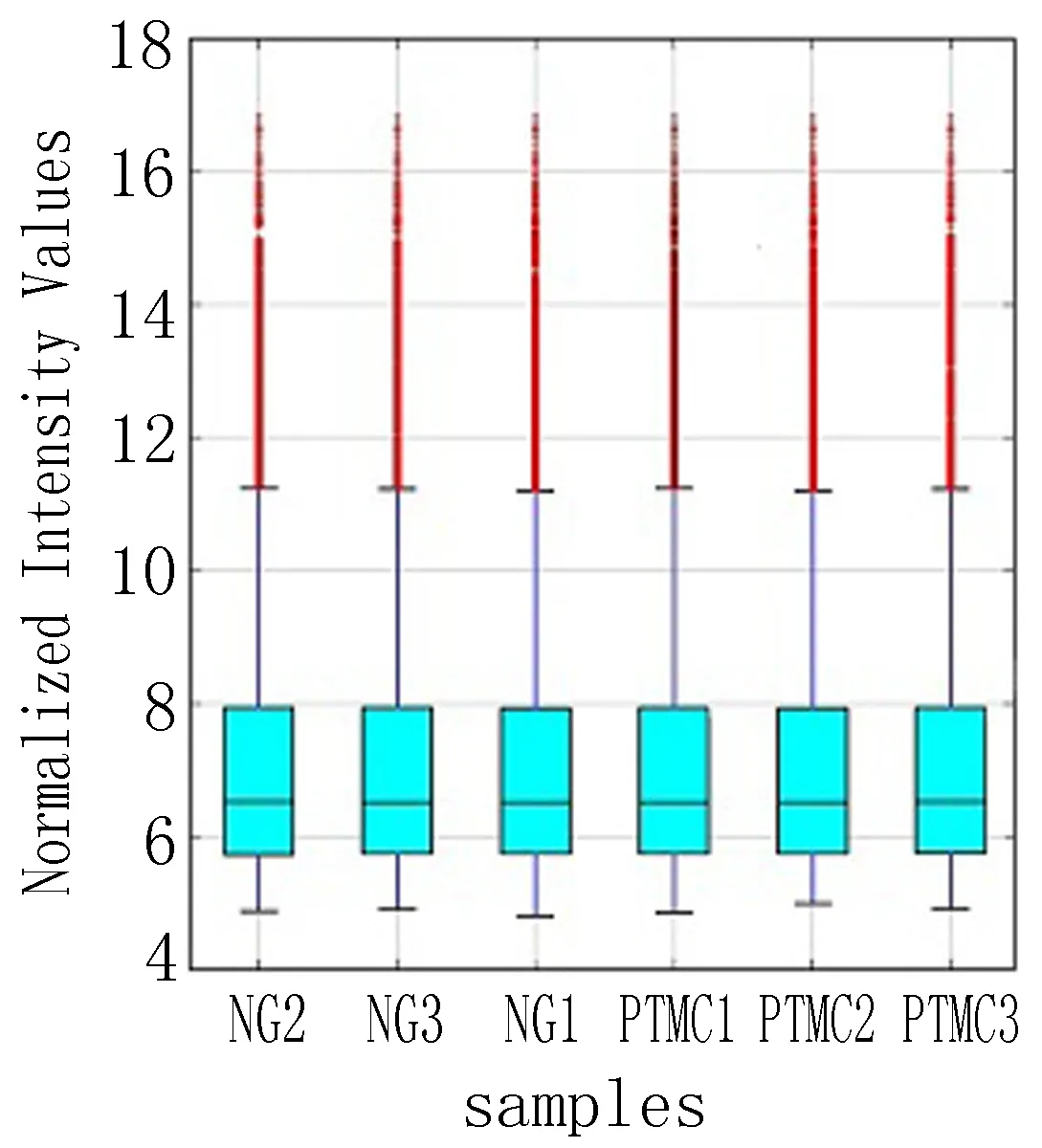
图1 NG 1~3及PTMC 1~3 circRNA分布箱型图
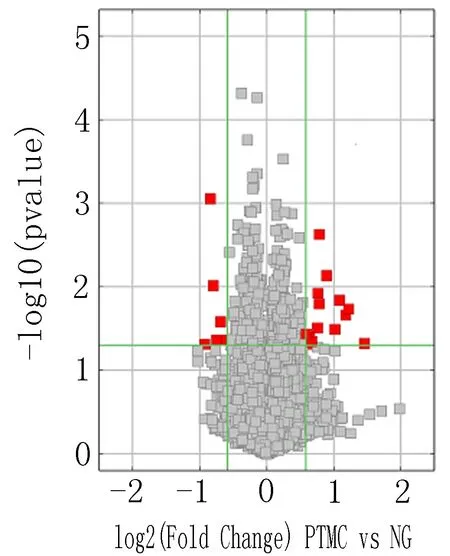
图2 NG1~3及PTMC1~3 circRNA表达火山图
2.2.2 差异表达circRNA的亲本基因的本体(Go)分析 PTMC组中差异表达的20个circRNA,其中上调的14个circRNA的亲本基因包括:ARMC2,ECT2,TMSB4X,CNTLN,TMEM2,SELL,SETBP1,TXLNG,GNB1,CSPP1,FIG4,ACTR1B,AL161626.1,MGC720;下调的6个circRNA的亲本基因包括:KRT14,UBAP2,EDEM2,ZFHX3,TERF2,SEC24A。PTMC中差异表达上调circRNA的亲本基因本体分析,分别选择排名前10的生物学过程、细胞组分和分子功能中Enrichment score(图3)。
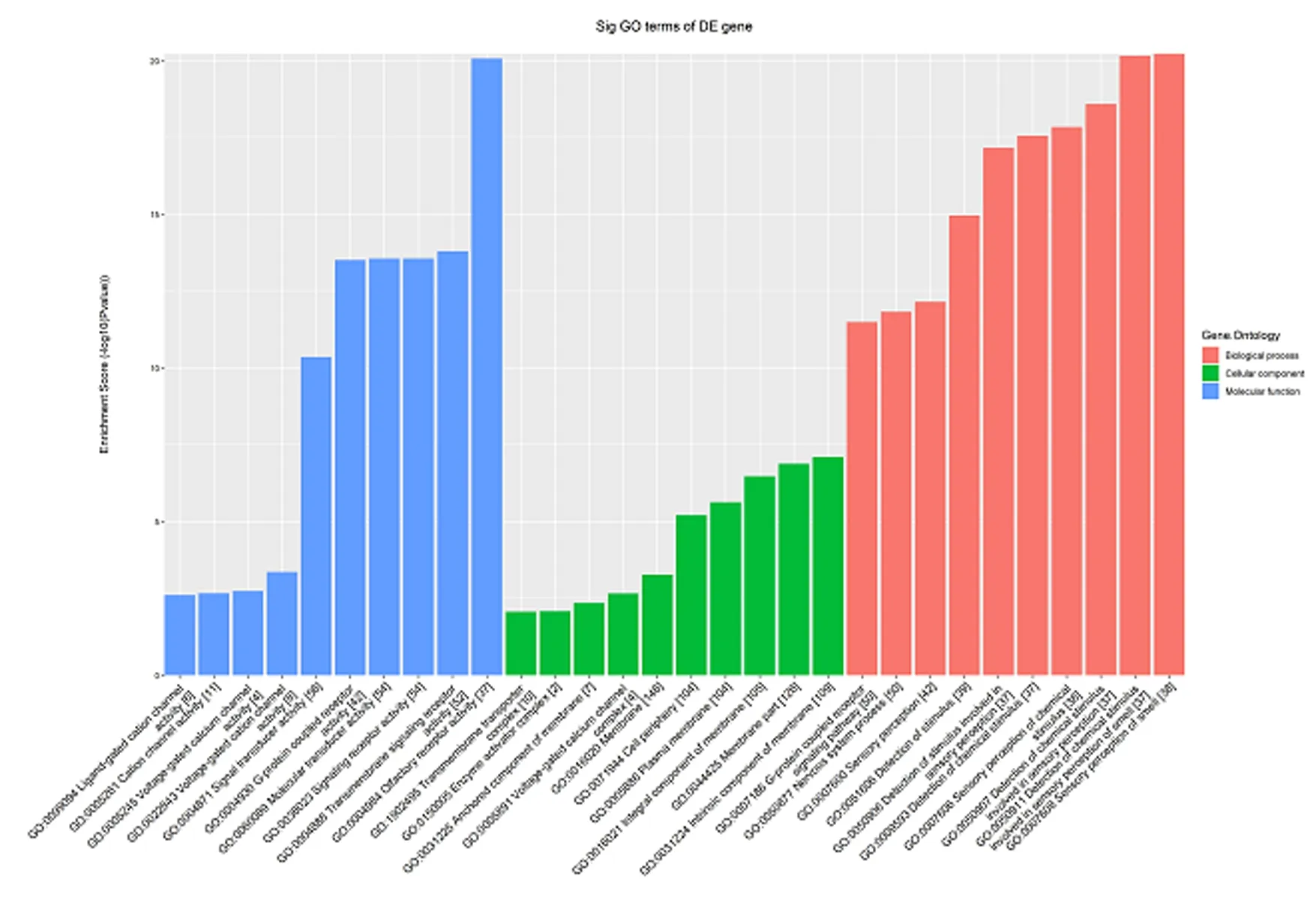
图3 NG与PTMC差异表达的circRNA亲本基因的Go富集分析
2.2.3 circRNA/miRNA的结合位点分析 对每个差异表达的circRNA预测5个具有高匹配值的miRNA结合位点,每个circRNA/miRNA结合注释。挑选上调和下调各3个circRNA进行罗列(表5)。
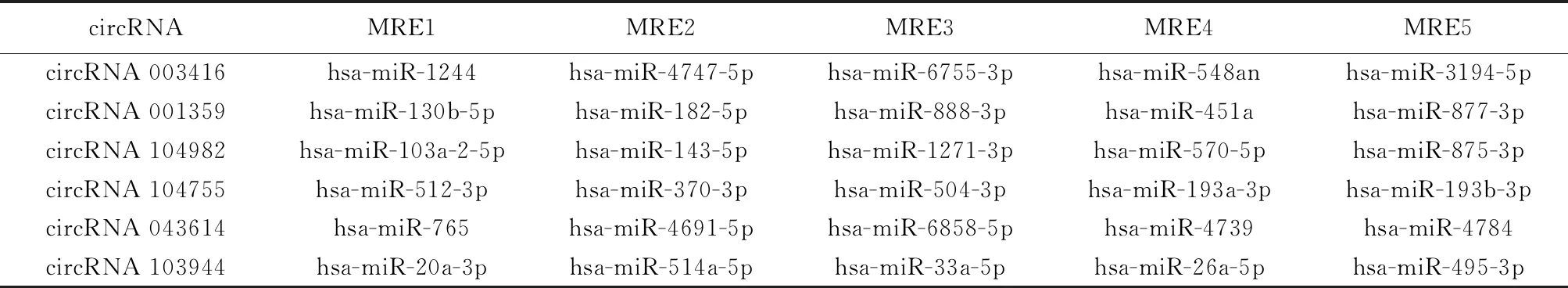
表5 与PTMC、NG上/下调circRNA高匹配的miRNA
2.2.4 circRNA-miRNA-mRNA网络图 circRNA-miRNA-mRNA网络图中黄色圆点代表circRNA,红色圆点代表miRNA,蓝色圆点代表miRNA靶信使RNA(Messenger RNA,mRNA),具体见图4。
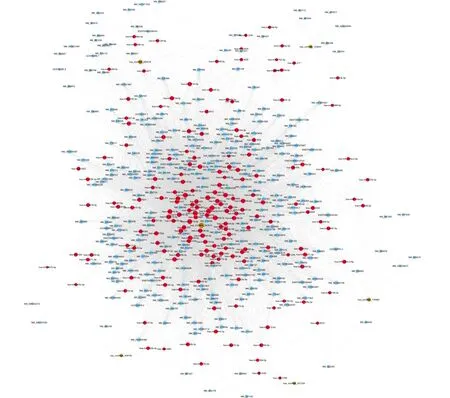
图4 circRNA-miRNA-mRNA网络图
2.2.5 Pathway分析 通过KEGG-Pathway分析,明确NG患者与PTMC患者血清中的差异表达circRNA相关的基因参与了Olfactory transduction通路(37个基因)(图5)。
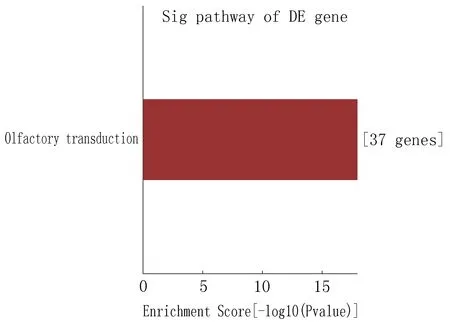
图5 Pathway分析
2.3 RT-PCR验证
2.3.1 RT-PCR结果 与NG组相比较,PTMC组hsa_circRNA_001359表达水平上调4.6倍、hsa_circRNA_003416表达水平上调3.82倍、hsa_circRNA_104982表达水平上调2.95倍,上述基因的表达水平显著上升(P<0.01)。与NG组相比较,PTMC组hsa_circRNA_043614表达水平下调2.56倍、hsa_circRNA_103944表达水平下调1.75倍、hsa_circRNA_104755表达水平下调2.27倍,上述基因的表达水平显著下降(P<0.01)(表6)。
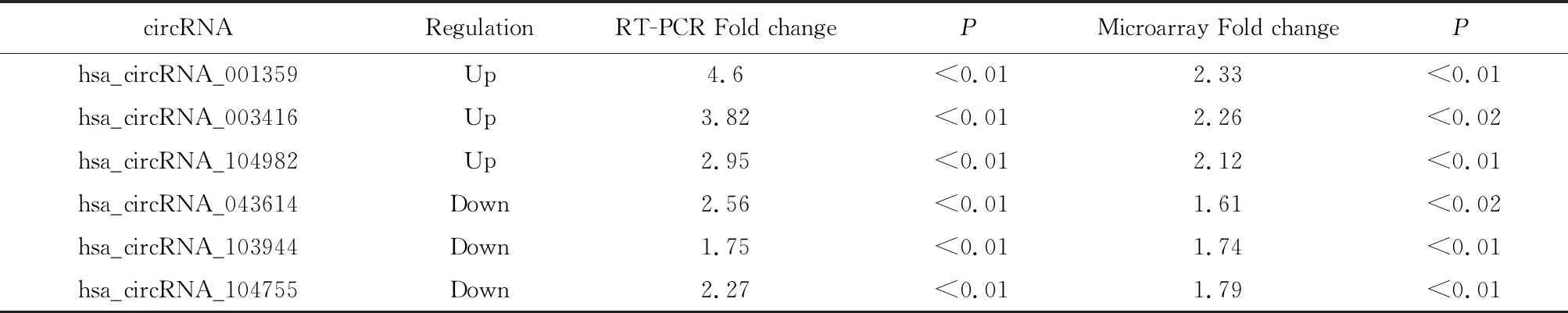
表6 RT-PCR验证结果与芯片结果比较
注:circRNA:circular RNA序列号;Regulation:“up”表示表达上调,“down”表示表达下调。
2.3.2 扩大样本进行RT-PCR验证 经RT-PR芯片结果验证,挑选倍数最高且P值小的circRNA001359,取NG组7例与PTMC组7例血清标本进行RNA纯度的检测,质检通过。经RT-PCR检测NG与PTMC,并进行对比,表明hsa_circRNA_001359表达水平显著上调5.8倍(P<0.01)。
3 讨论
基因芯片检测恶性肿瘤的标记物是目前基因检测较为常用的方法。Zhang等[16]对非小细胞癌组织和癌旁组织进行circRNA芯片分析检测,结果表明hsa_circ_0014130是显著上调的,并分析了该circRNA与非小细胞癌组织患者临床特征的关系,提示前者可能参与了非小细胞癌组织肿瘤的发生与发展,该研究结果提示hsa_circ_0014130可能作为一种新的非小细胞癌组织诊断生物学标志物。Zheng等[17]收集原发性胆管炎患者的血浆,同期收集健康人群的血浆,进行circRNA芯片检测,结果提示circRNA的表达异常可能在原发性胆管炎发病的机制中发挥着重要的作用,研究结果表明hsa_circ_402458有望成为原发性胆管炎的候选生物学标志物。Xu等[18]对肺部鳞状细胞癌进行研究,收集肺鳞状细胞癌的癌组织和癌旁组织,对两者进行circRNA芯片检测分析,生物学数据分析和后期的验证,研究表明circRNA在肺鳞状细胞癌中存在差异表达,并且与肺鳞状细胞癌的癌变密切相关。该研究提示hsa_circRNA_103827和hsa_circRNA_000122可能可以作为该疾病的潜在预后生物学标志物和治疗的靶点。但应用血清标本进行芯片检测明确PTMC性质的研究尚未查阅到。
本研究使用12 900个circRNA探针对6例血清标本中circRNA的表达水平一致性及表达差异进行分析。对被检测的6例血清标本,以箱式图的形式进行质量评估,结果显示2组中circRNA的表达水平均值为4~6,表明一致性和可比性较好。随后研究采用火山图直观地显示2组中circRNA表达水平的变异度。
本研究结果显示,NG和PTMC血清中circRNA表达显著上调的有 14个和下调的有6个。挑选出上调circRNA 3个,分别是:hsa_circRNA_001359、hsa_circRNA_003416和hsa_circRNA_104982,下调circRNA 3个,分别是:hsa_circRNA_043614,hsa_circRNA_103944和hsa_circRNA_104755。考虑以上circRNA表达的差异可能是调控PTMC的发生与发展,但上述的circRNA是通过何种途径影响PTMC的发生与发展,还需要进一步的研究。釆用生物信息学方法分析得出差异性circRNA线性基因本体分析,包括生物学过程、细胞组分及分子功能。对存在1.5倍以上差异的circRNA采用美国Arraystar公司的miRNA结合位点预测软件预测5个与CircRNA具有高匹配值的miRNA结合位点。本研究还预测了circRNA的潜在miRNA靶点,以KEGG数据库对差异表达circRNA预测结合的与PTMC相关的miRNA可能调控的靶基因进行生物信号通路分析,构建circRNA-miRNA-mRNA网络图,探讨circRNA表达异常在PMTC中发挥的可能作用。该网络阐明了circRNA、miRNA和mRNA之间潜在的ceRNA关系。
经RT-PCR芯片检测的验证,表明hsa_circRNA_001359、hsa_circRNA_003416、hsa_circRNA_104982、hsa_circRNA_043614、hsa_circRNA_103944、hsa_circRNA_104755的验证和芯片检测的调控趋势是一致的。
经查阅大量的文献得出,hsa_circRNA_001359高配位的hsa-miR-130b-5p,hsa-miR-182-5p和hsa-miR-451a均与甲状腺乳头状癌的发生和发展密切相关。
根据本研究结果结合文献分析,选取hsa_circRNA_001359作为PTMC诊断的靶点,并经RT-PCR方法进行扩大样本量的验证。结果表明hsa_circRNA_001359可能可以作为诊断女性、甲状腺结节患者术前诊断PTMC性质的生物学标记物。
