基于神经网络的药物实体与关系联合抽取
2019-07-15曹明宇杨志豪林鸿飞
曹明宇 杨志豪 罗 凌 林鸿飞 王 健
(大连理工大学计算机科学与技术学院 辽宁大连 116024)
近年来,伴随着生物医学研究的发展,生物医学文本的数量也迅速增长.生物医学文本中包含着丰富实体关系信息,利用文本挖掘技术自动抽取这些信息,对生物医学研究具有促进作用.其中,药物-药物交互(drug-drug interaction, DDI)关系抽取近年来受到了研究者们广泛关注,根据海量医学文本中抽取出的DDI,可以推测药物间隐含的相互作用及不良反应,对药物的研究具有重要的意义.
为了促进DDI关系抽取研究发展,2013年的国际语义评测(semantic evaluation exercises, SemEval)中包含了DDI关系抽取任务(DDI Extraction 2013, DDI2013)[1].DDI2013将DDI抽取具体分为了2个子任务:1)药物实体识别;2)DDI关系抽取.目前的研究基本都是将这2个任务分别地单独研究,然后再通过串行流水线(pipeline)的方法来抽取DDI.即首先通过命名实体识别(named entity recognition, NER)提取出文本中的药物实体,再对每个候选实体对进行关系分类(relation classification, RC).对于实体识别子任务,传统的条件随机场(conditional random field, CRF)方法具有良好的性能,该方法也在DDI2013的药物实体识别任务上取得了第一的成绩[2].随着神经网络的发展,深度学习在NER上表现出了更好的性能,其中BiLSTM-CRF模型结合了循环神经网络和CRF的模型的优点,广泛地应用于序列标注任务[3].在生物医学领域,Habibi等人[4]使用BiLSTM-CRF模型在5类生物医学实体总共33个数据集上进行了实验并取得了不错的效果.
在关系分类子任务上,Chowdhury等人[5]使用结合浅层语言核函数及最短路径树核函数的方法,在DDI2013评测关系抽取任务上取得了第一的成绩;Zhao等人[6]使用卷积神经网络(convolutional neural network, CNN)模型,对词特征及位置特征进行学习;Zhang等人[7]提出了分层的循环神经网络(recurrent neural network, RNN)模型,底层的RNN用来分别地学习句子的词特征、位置特征及句法依存特征,顶层的RNN对这3种特征学习,同时引入了attention机制来加强对关键词表示的信息的学习,在DDI2013语料集上达到了最先进的性能.尽管2个子任务的研究已经分别取得了不错的进展,但是要获得DDI关系抽取的结果,就需要先利用NER模型进行实体识别,然后对识别出来的结果使用RC模型进行关系分类.这种流水线的方式主要存在2个问题:1)分离训练的NER模型和RC模型忽略了这2个子任务间的相互影响,无法利用其交互信息来提升模型性能;2)RC的结果依赖于NER的结果,NER的错误会进行传播,导致最后的DDI抽取效果不佳.
不同于流水线方法,联合抽取的方法旨在将2个相关任务一起学习,目前在通用领域(如新闻领域)基于联合抽取的关系抽取研究开始受到研究者们的关注;Miwa等人[8]提出了一种使用表格来同时表示实体和关系的方法,将实体识别和关系抽取转化为填表问题,可以准确表示句子中的所有实体和关系,但是该方法仍然依赖于特征工程及自然语言处理工具包;为了避免复杂的特征工程,Miwa等人[9]提出了一个基于端对端的神经网络的共享参数方法,2个子任务共享部分模型,又各自具有单独的输出,这种模型需要在实体预测的结果上进行关系分类,仍然相当于2个任务分开进行,并产生冗余的信息;为了充分考虑2个子任务间的相互作用,Zheng等人[10]提出了一种同时包含实体和关系信息的标注模式,将实体和关系的联合抽取转化为了端对端的序列标注任务,在通用领域取得了很好的效果.但该方法默认实体只参与1个关系,无法识别重叠关系(重叠关系是指该关系的实体参与不止1个关系,同时也是另一个关系中的实体,存在重叠现象).而在生物医学文本中,同一个实体常常参与多个关系,该方法匹配关系的方式可能会损失大量的关系.
为了缓解上述流水线方法存在的问题和生物医学文本中存在大量实体重叠关系的问题,本文提出了一种基于神经网络的药物实体与关系联合抽取方法.借鉴Zheng等人[10]的工作,将药物实体和关系的联合抽取看作序列标注问题,然后提出了一种改进的标注模式,使用BiLSTM-CRF模型进行标注,最后根据标注结果抽取药物实体和关系.本文主要有2方面贡献:
1) 不同于以往将NER和RC独立研究的工作,本文试图使用联合模型同时抽取药物和药物之间的关系,提出了一种基于神经网络的药物实体与关系联合抽取方法.
2) 针对生物医学实体存在大量重叠关系的特点以及Zheng等人[10]提出的标注方法没有考虑重叠关系的问题,本文改进了原始的标注模式以及匹配策略,有效地缓解了重叠实体关系无法抽取的问题.
本文在DDI 2013语料集进行实验,与传统的流水线方法相比,本文的联合抽取方法取得了更好的表现,药物-药物关系抽取的F-score达到67.3%.
1 药物实体与关系联合抽取方法
在本节中,首先描述了本文使用的标注模式,然后阐述如何根据标签序列产生实体和实体间关系,最后介绍本文使用的输入特征和BiLSTM-CRF模型.
1.1 标注模式
本文将药物实体与关系联合抽取转化为每个词预测标签的序列标注任务.本文借鉴了Zheng等人[10]提出的标注模式,并针对他们标注模式无法识别重叠实体关系的问题,改进了标注模式.此外,本文还增加了实体类别标签来丰富实体包含的信息.本文提出的标签最多由4个部分组成,分别为:实体边界、实体类别、关系类别、实体位置.其中,实体边界标签使用“BIOES”模式,B表示实体头部,I表示实体中间,E表示实体末尾,S表示单一实体,O表示非实体;实体类别和关系类别标签是由语料集预定义的,在本文使用的DDI2013数据集中,分别为4种实体类别(drug,group,brand,drug_n)和4种药物关系(mechanism,effect,advice,int).对于关系类别,本文增加了一个M标签来表示该词所属的实体参与多种不同类型的关系;实体位置表示实体在关系中的位置,由1,2,M定义,1表示该词是关系中的第1个实体,2表示第2个实体,M表示该词存在重叠关系中且分别是不同位置.最后抽取结果能被表示为三元组{实体1,关系类别,实体2}.
图1中的例子解释了本文的标注模式.输入句子包含2个三元组(metopirone,mechanism,aceta-minophen1)和(metopirone,effect,acetaminophen2),其中mechanism和effect是预定义的关系类别.每个词都依据其实体信息和关系信息被标注相应的标签,例如:metopirone属于单个实体,实体类别为drug,参与mechanism和effect这2种关系(重叠关系),且在关系中的位置都是第1个实体,故其标签为S-drug-M-1.Inhibits不属于实体,故其标签为O.
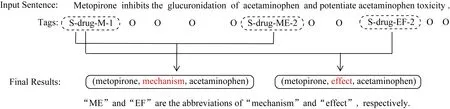
Fig. 1 Tagging sample图1 标注样例
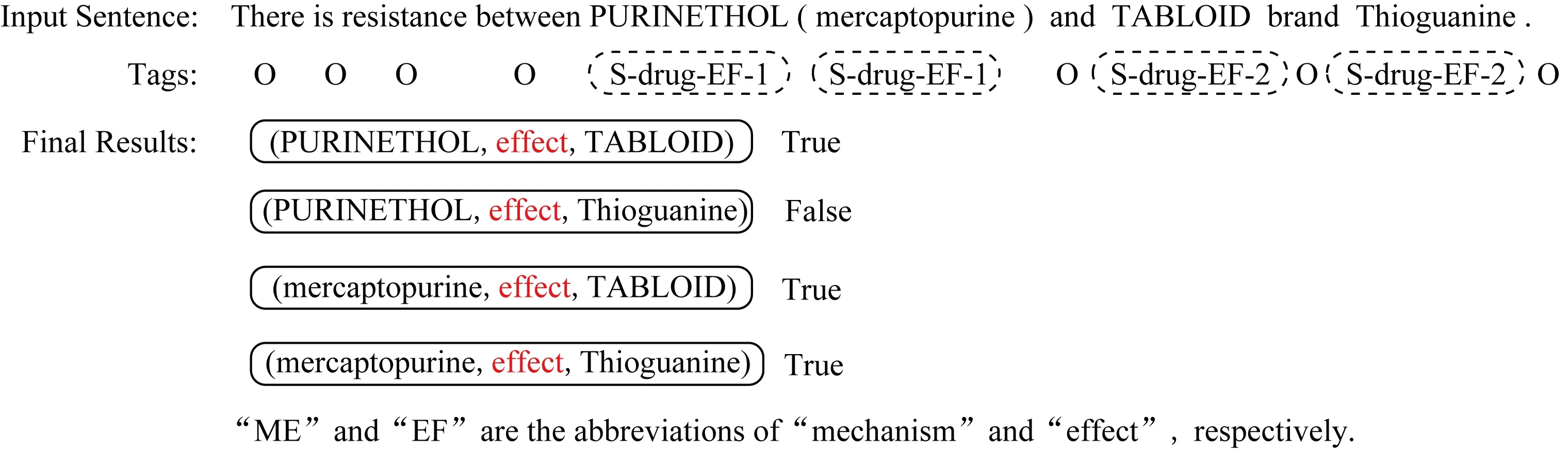
Fig. 2 A relation sample which can’t be represented correctly by our tagging scheme图2 标注模式无法正确表示的关系样例
由于并非所有的词都是实体的组成部分,也并非所有的实体都参与关系,实际的标签格式有3种类型,共计257种标签类别,具体如表1所示.
此外,仍然有少量的关系无法使用本文的标注模式和抽取规则来表示,如图2所示的样例,共有4个关系:(PURINETHOL,effect,TABLOID),(mercaptopurine,effect,TABLOID),(PURINETHOL,effect,Thioguanine),(mercaptopurine,effect,Thioguanine),按照标注的关系产生图中的标签序列.但是在抽取关系时,实体Thioguanine向前查找可匹配实体,与mercaptopurine匹配生成三元组(mercaptopurine,effect,Thioguani ne)便停止抽取,而关系(PURINETHOL,efect,Thioguanine)则由于抽取规则而损失.
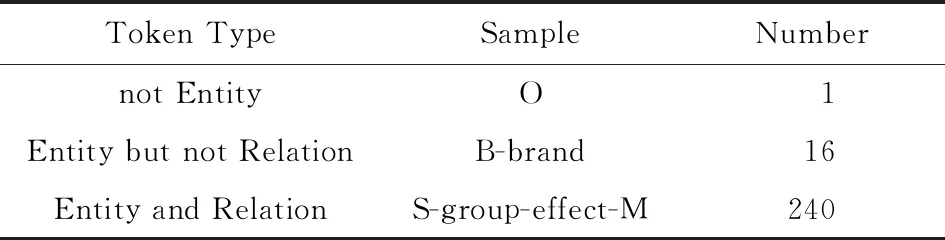
Table 1 Three Tag Formats表1 3种标签格式
1.2 抽取实体与关系
不同于Zheng等人[10]仅使用最近距离匹配原则抽取实体与关系,本文充分考虑了重叠关系提出了新的抽取规则来从已标注的标签序列中抽取实体及关系:
1) 根据词的实体边界和实体类别标签抽取出实体,实体的关系类别和实体位置被定义为实体首词的关系类别和实体位置.
2) 关系的抽取遵循最近距离匹配的原则,对于每个实体,查找与其距离最近的关系类别及实体位置可匹配的实体,组成一个关系三元组.
3) 关系类别为预定义的4种关系类别实体只能与相同关系类别的实体匹配,关系类别为M的实体可与任意关系类别的实体匹配,实体位置为1的实体可与实体位置为2,M的实体匹配,实体位置为2的实体可与实体位置为1,M的实体匹配,实体位置为M的实体可以与实体位置为1,2,M的实体匹配.
4) 查找距离最近的可匹配实体是有方向的,实体位置为1的实体只向后查找,实体位置为2的实体向前查找,实体位置为M的实体同时前后查找.
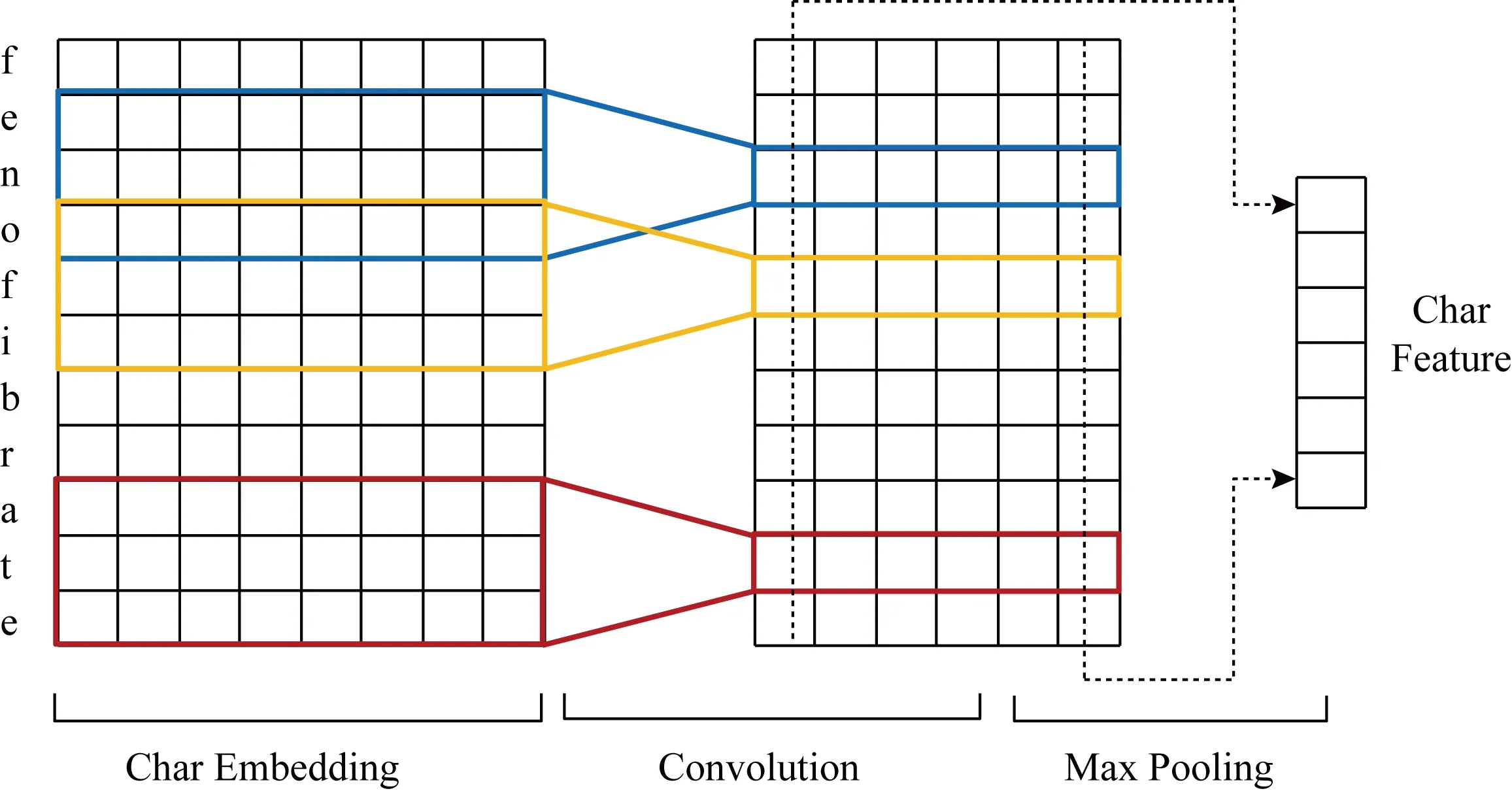
Fig. 3 Process of character-level features图3 字符级特征的获取
例如图1中的样例,首先根据规则1抽取到(metopirone,drug,M,1),(acetaminophen1,drug,ME,2),(acetaminophen2,drug,EF,2)三个实体.对于这3个实体,首先实体metopirone向后查找到实体acetaminophen1,由于2个实体的关系类别分别为M,ME,实体位置分别为1,2,故可以匹配,生成三元组{metopirone,mechanism,acetaminophen1};其次,实体acetaminophen1向前查找到实体metopirone,该三元组已存在不重复抽取;最后,实体acetaminophen2向前查找到实体metopirone,二者的关系类别分别为M,EF,实体位置分别为1,2,生成三元组{metopirone,effect,acetaminophen2}.
1.3 模型输入特征
1) 词向量.目前,在各种自然语言处理任务中,谷歌的word2vec技术[11]被广泛地使用.传统上表示词向量的方法是one-hot编码的词向量表示法,词与词之间相互独立,且向量的长度取决于语料库词数,很容易造成维度灾难.word2vec是一种分布式的低维度、稠密词向量表示,可以充分考虑词的上下文信息,将语义相似的词映射到向量空间的相近位置.为了获得更高质量的词向量,本文使用基于PubMed下载的1 918 662篇MEDLINE摘要训练出的50维的词向量表示作为词的特征.
2) 字符向量.字符向量是词的另一种表示,不同于词向量更关注词的语义特征,字符向量被用来表示词本身的形态特征,比如首字母大写、前缀后缀等.在生物医学文本中,化学物的命名遵循一定的命名规范,所以字符向量更充分地表示化学物实体的形态特征.本文首先将字符随机初始化为固定维度的字符向量,对于一个词,使用其字符向量序列表示该词的字符特征.然后经过卷积和最大池化从字符向量序列中学习到向量,将其与词向量拼接,作为词最终的向量表示.字符级特征的获取过程如图3所示:
1.4 BiLSTM-CRF模型
循环神经网络近年来常用于自然语言处理领域,与传统神经网络相比,它最大的特点是可以接受序列的输入产生序列的输出,每个时刻学习到的信息不仅取决于当前时刻的输入,还依赖上一时刻的输出.但是RNN不具备强大的记忆结构,距离当前时刻越近的时刻,对当前时刻的输出影响越大,较远的时刻对当前的影响十分微弱.这就导致RNN只能学习到离当前时刻较近的信息,不能学习到距离较远的信息.
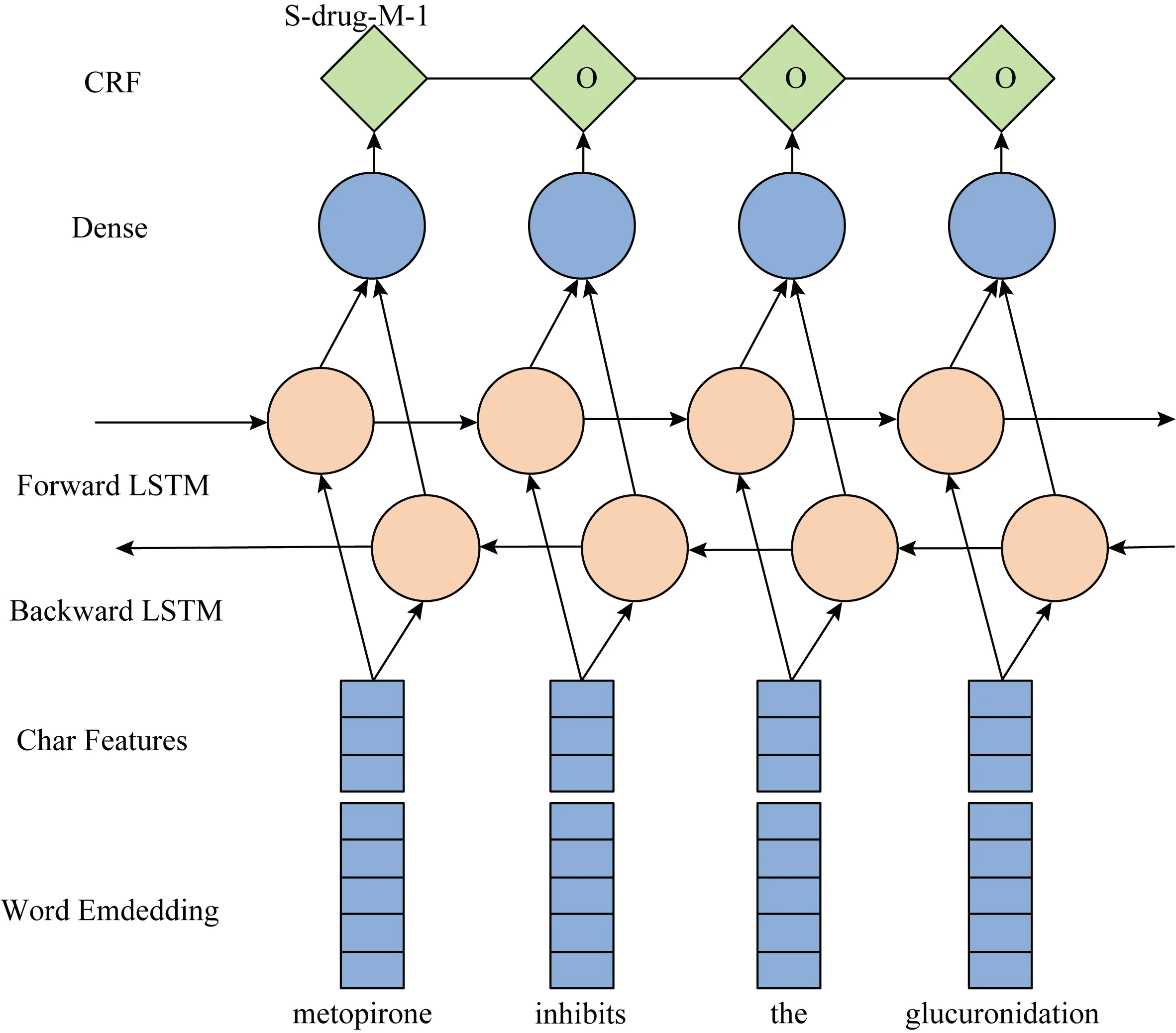
Fig. 4 The BiLSTM-CRF model图4 BiLSTM-CRF模型
长短期记忆模型(long short-term memory, LSTM)[12]是对RNN的一种改进,它使用一种被称为LSTM 记忆细胞的结构来判别哪些信息应该被保留,控制信息从前一时刻到后一时刻的传输.LSTM有效地解决了RNN具有的长期依赖问题,使当前时刻学习到的信息能充分利用之前时刻学习到的有用信息.本文参考Huang等人[13]使用的LSTM神经元,其数学模型为
it=tanh(Wxixt+Whiht-1+Wcict-1+bi),
(1)
ft=tanh(Wxfxt+Whfht-1+Wcfct-1+bf),
(2)
ct=ftct-1+ittanh(Wxcxt+Whcht-1+bc),
(3)
ot=tanh(Wxoxt+Whoht-1+Wcoct+bo),
(4)
ht=ottanh(ct),
(5)
其中,W*是LSTM神经元的参数矩阵;b*是LSTM神经元的偏置项;tanh是激活函数,为神经网络加入非线性信息.
由于LSTM具有方向性,当前时刻只能参考之前时刻的学习到的特征.而在自然语言处理任务中,通常不仅需要参考上文信息,也需要参考下文信息,双向长短时记忆循环模型(bi-directional LSTM,BiLSTM)被提出.BiLSTM由2个相反方向的LSTM组成,它们可以分别从前向和后向学习词的上文特征ht,forward和下文特征ht,backward,再将2者拼接成[ht,forward,ht,backward],常被用于序列标注任务进行词表示的学习.
然而,当前词的标签不仅受到词本身特征和上下文特征的影响,还受到上下文标签的影响.比如,在表示实体的“BIOES”标注模式中,I必须在B之后,E必须在I之后,实体的前后被标注为O标签,这些标签之间是相互依赖的,但基于BiLSTM的序列标注模型不能充分考虑标签之间的依赖关系,每个词标签的预测都是单独的分类任务,可能产生不合规则的标签序列,如B出现在I之后的情况.本文参考Huang等人[13]的方法,在BiLSTM模型之后加入CRF层对标签序列进行全局优化.CRF层可以学习整个句子的标签转移概率,充分地考虑标签间的依赖关系.令x表示输入的词序列,fθi,t表示在BiLSTM模型参数为θ的条件下,为词序列中的第t个词分配第i个标签的分数.本文使用标签转移矩阵Ai,j作为CRF层的参数,表示前一时刻的标签为i时转移到第j个标签的得分.最终,输入词序列x的得到标签序列y的分数为
(6)
用于序列标注的BiLSTM-CRF模型的基本结构如图4所示.首先由将输入词序列映射为50维的词向量和15维的字符向量序列,将词向量与CNN学习到的字符特征拼接,然后经过双向LSTM层进行学习,最后由CRF层来预测全局优化的标签序列.
在整个模型上,本文使用自适应学习率算法RMSprop[14]进行参数的优化,该算法可以为不同参数选择不同的学习速率,在序列标注问题上具有很好的收敛速度及收敛效果.
2 实验分析
2.1 实验设置
本文使用DDI 2013关系抽取语料集进行实验,该语料集标注了4种类别的药物实体(drug,group,brand,drug_n)和4种类别的药物关系(mechanism,effect,advice,int).此外,本文还统计了该语料中存在重叠关系的数量,该语料集的信息如表2所示.从表2可以看到,整个数据集有60%的关系是重叠关系.

Table 2 Information of DDI2013 Corpus表2 DDI2013语料集信息
本文随机地从训练集抽取了10%的样本,在所有实验中作为开发集,用于超参数的调整选择,表3展示了本文模型主要的超参数.此外,本文根据模型在开发集上的结果使用早停机制[15]选择模型的训练迭代次数.本文使用查准率(precision,P)、召回率(recall,R)和F值(F-score,F)对预测结果进行评价.对于药物实体识别的结果,当实体的左右边界和实体类别均正确时,认为其正确.对于药物关系抽取的结果,当其组成实体的左右边界及关系类别均正确时,认为其正确.

Table 3 Hyper-Parameters of the Model表3 模型的超参数
2.2 模型对比实验
本节在使用词向量及字符向量作为输入特征,使用本文改善的标注模式及抽取规则的情况下,对BiLSTM-CRF模型的双向LSTM层数及是否增加单向LSTM层上进行了对比实验,实验结果如表4所示:
Table 4 Performance Comparison of Different Models
表4 不同模型的性能比较
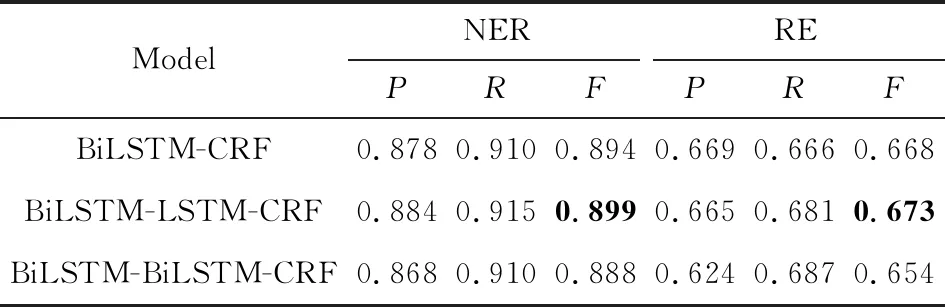
ModelNERREPRFPRFBiLSTM-CRF0.8780.9100.8940.6690.6660.668BiLSTM-LSTM-CRF0.8840.9150.8990.6650.6810.673BiLSTM-BiLSTM-CRF0.8680.9100.8880.6240.6870.654
Note: The bold text is the result of the best model.
由表4可以得到2个结论:
1) 用1个单向LSTM层对BiLSTM层学习到的信息进行解码,可以提高模型的性能.
2) 随着BiLSTM层数的增加,模型未表现出更好的性能.因此在后续实验中,都使用BiLSTM-LSTM-CRF模型.
2.3 标注模式对比实验
本节在使用词向量及字符向量作为输入特征,使用BiLSTM-LSTM-CRF模型的情况下,对比了不同标注模型以及抽取规则的性能.本文使用在Zheng等人[10]提出的标注模式和最近匹配规则作为Baseline,然后逐次在原有基础上加入改进抽取规则、增加实体类别标签及增加重叠关系M标签方法进行对比,结果如表5所示,可以得到3个结论:
1) Zheng等人[10]遵循的实体只参与1个关系的抽取规则在DDI语料集上会造成关系的损失,改进抽取规则后关系抽取可达到18%的F值提升,其中重叠关系的召回率提升了45%,说明改进后的抽取规则可以更充分地抽取重叠的药物关系.
2) 增加实体类别标签后,关系抽取得到了1.1%的F值提升,证明了实体识别子任务对关系抽取子任务具有促进作用,丰富的实体信息表示对关系抽取的性能有帮助.
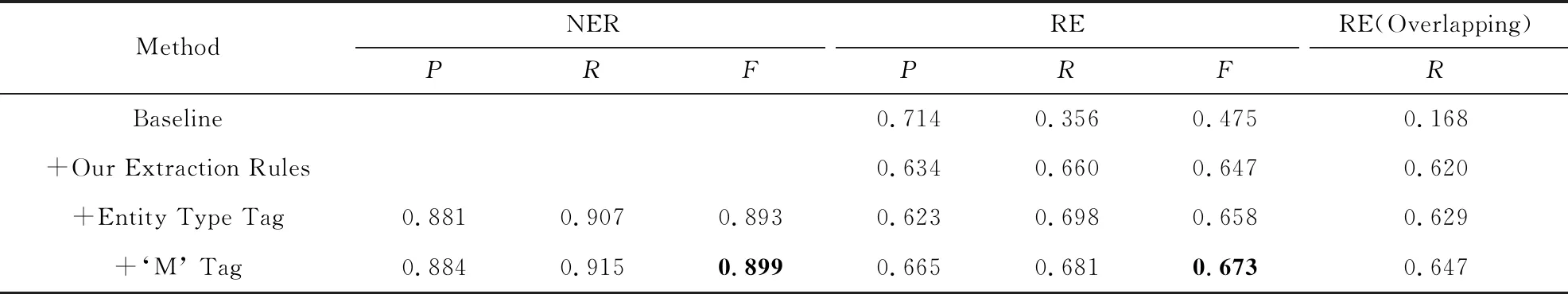
Table 5 Performance Comparison of Tagging Scheme表5 标注模式的性能比较
Note: The bold text is the result of the best tagging scheme.
3) 增加本文提出的M标签后,关系抽取得到1.5%的F值提升,其中重叠关系的召回率提升了3%,说明使用M标签来表示实体参与不同类别关系或实体处于关系的不同位置是有效果的,同时M标签是可以被深度神经网络学习的.
2.4 模型成份性能实验
本节在使用改进后的标注模式及抽取规则的情况下,每次分别对预训练的词向量、字符向量及CRF层等模型的1种成份进行了消减实验,以探究这些成份对于性能的影响,结果如表6所示:
Table 6 Performance Comparison of Model Components
表6 模型成分的性能比较
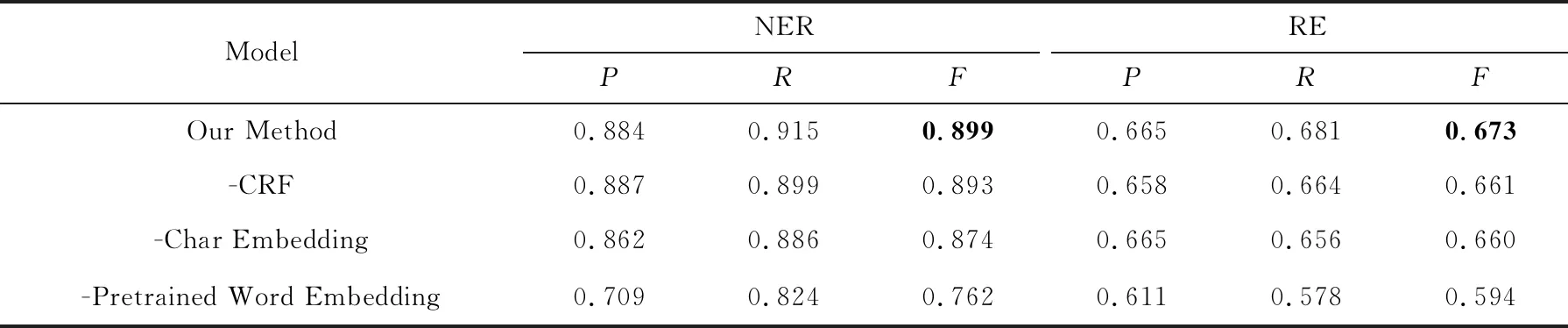
ModelNERREPRFPRFOur Method0.8840.9150.8990.6650.6810.673-CRF0.8870.8990.8930.6580.6640.661-Char Embedding0.8620.8860.8740.6650.6560.660-Pretrained Word Embedding0.7090.8240.7620.6110.5780.594
Note: The bold text is the result of our method.
由表6可以得到3个结论:
1) 使用预训练的词向量,与使用随机初始化的词向量相比,在实体识别和关系抽取上都有更显著的表现,证明了预训练的词向量更有利于表达词本身的语义信息,而随机初始化的词向量尽管可以在训练的过程中被优化,但其参考的仅仅是词语在语料集中的上下文特征.
2) 增加字符向量来表示词的形态特征,与只使用词向量相比,显著提升了药物实体识别的准确度(F值增加2.5%),也为关系抽取的F值带来了提升.从以上2点中可以得到,充分的词特征表示对实体识别和关系抽取的性能都具有促进作用.
3) 增加CRF层对预测的标签序列进行全局优化,与只使用神经网络模型相比,提升了1.2%的关系抽取F值.
2.5 与其他方法比较
传统的在DDI语料集上进行关系抽取的研究都将其看作基于完全正确的标注实体的关系分类任务.而联合抽取方法的关系抽取仅依赖于预测出的实体,直接进行联合抽取和关系分类的性能比较是忽略实体条件的.因此,本文搭建了流水线模型,先进行实体识别,使用其结果生成实体对,再通过关系分类模型进行关系的预测,将联合抽取方法与流水线模型进行比较.对于实体识别子任务,本文使用主流的BiLSTM-CRF模型;对于关系分类子任务,本文使用了BiLSTM模型作为基线系统.进一步,本文还使用Zhang等人[7]的方法来进行关系分类,该方法使用了丰富的句法依存特征,并结合了RNN和CNN模型,在目前DDI关系分类任务上取得了先进水平.此外,本文还与使用Zheng等人[10]提出的联合抽取方法进行对比.从表7的实验结果可以看出:
1) 本文方法在只使用词特征作为输入的条件下,表现出了比使用词特征及位置特征的流水线方法更好的关系抽取性能(F值提升3.4%).
2) 与加入句法依存特征的流水线方法相比,本文方法在更少输入特征的情况下可以达到稍低的性能(F值相差1.9%),可以展望在加入更多输入特征(如句法特征、位置特征)的情况下,本文方法具有进一步研究的意义.
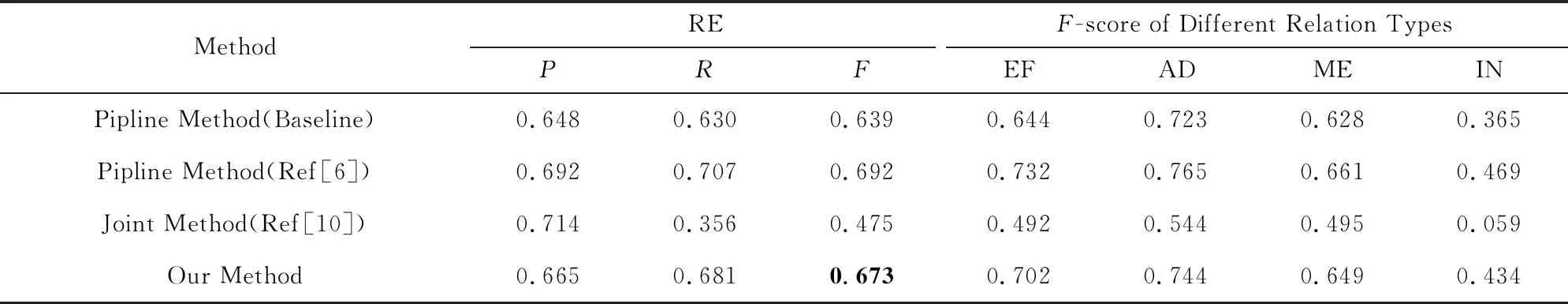
Table 7 Performance Comparison of Different Methods表7 不同方法的性能比较
Note: The bold text is the result of our method.
3) 与Zheng等人[10]的方法相比,本文改进标注模式及匹配规则后的方法更充分考虑了生物医学文本中药物关系的特点,显著提升了关系抽取的性能.
2.6 关系抽取结果对比
本文进一步对流水线Baseline方法、本文方法的关系抽取结果进行对比,将3个典型的样例展示在表8中,每个样例包含3行,第1行是标准答案,第2行是流水线方法抽取的关系,第3行是本文方法抽取的关系.“[]”中为实体,角标为该实体的关系及位置,加粗部分为抽取结果错误的实体.
对于样例1,流水线方法在实体识别子任务上产生了实体边界上的错误,该错误传播给关系分类,即使关系分类正确也得到了错误的关系抽取结果,而联合抽取方法则避免了该错误,证明了联合抽取方法具有减少错误传播的可能性.对于样例2,句子中包含1个实体和5个并列实体存在的重叠关系,流水线方法分别对这5个实体对进行关系分类,结果只抽取出了第1个关系,而联合抽取方法则成功抽取出了5个并列关系,结合2种方法分析原因,联合抽取的方法对5个关系同时进行学习,更充分地考虑了关系之间的依赖性,证明了联合抽取方法不仅考虑实体和关系间的影响,也能学习到关系之间相互影响的可能性.对于样例3,流水线方法和本文方法均未抽取出正确的关系,说明本文方法依然具有进一步研究的空间.

Table 8 Comparisons of Different Methods’ Extraction Results表8 不同方法的关系抽取结果对比
3 结 论
本文提出了一种基于BiLSTM-CRF的药物实体与关系联合抽取方法.具体地,将药物实体及关系的联合抽取转化为端对端的序列标注任务,使用词向量及字符向量作为输入特征,BiLSTM-CRF模型进行标注.针对生物医学文本中大量存在的重叠关系,本文改进了原始的标注模式,增加了M标签来缓解重叠关系的问题,且增加了实体类别标签来更充分利用实体信息.此外还改进了关系抽取规则,相比简单的最近匹配规则,本文的方法能够显著提升重叠关系的召回率.实验表明,与传统的流水线模型相比,本文提出的方法可以更简单地从预测的标签序列中同时抽取出实体及关系,具备与单独的子任务性能相当的实体识别性能及更好的关系抽取性能.在DDI2013药物-药物关系抽取语料集上达到了67.3%的F值.
但是目前本文方法还未能完全覆盖所有的重叠关系情况,未来工作中本文将进行更深入的研究来解决重叠关系的问题.此外,在实体识别和关系分类子任务上,现存的研究已经展示了额外特征(例如词性、句法依存特征)能够有效提升模型性能,探索这些额外特征对联合学习的影响也是未来的研究工作.
