山东省济宁青山羊群体遗传多样性分析
2019-07-02王可刘昭华曹洪防楚惠民崔绪奎
王可 刘昭华 曹洪防 楚惠民 崔绪奎

摘要:为了解近年来山东省对济宁青山羊品种资源保护效果,采用微卫星DNA标记技术,对山东省境内的4个济宁青山羊群体进行了遗传多样性和遗传分化分析。结果显示:24个微卫星位点在4个群体135个个体中共检测到159个等位基因,平均每个位点6.6个;4个群体的PIC在0.6016~0.6653之间,群体的He在0.6750~0.7559之间,但是群体的Ho均低于He,LWQ最低(0.5375),JXQ最高(0.6260);群体分化系数(Fst)为0.0774,基因流(Nm)分析表明每世代4个群体间有效迁移个体数为1.8905;遗传距离和遗传相似性指数分析表明,SXQ、HHQ和JXQ群体间具有较近的亲缘关系,而与LWQ群体间遗传距离相对较远。结果表明:济宁青山羊群体遗传多样性丰富,群体间存在一定的基因交流,遗传分化水平较低,但4个群体都存在不同程度的近交,SXQ和LWQ群体的近交程度较高,所以在实际工作中,应注意扩大群体数量,避免近交衰退。
关键词:济宁青山羊;微卫星;资源保护;遗传多样性;遗传分化
中图分类号:S813.9文献标识号:A文章编号:1001-4942(2019)05-0129-06
济宁青山羊(Jininggreygoat)是山东省优良的地方山羊品种,具有性成熟早、多胎多羔、常年发情、抗病力强、肉质鲜美等优良种质特性[1],2006年被列为国家级畜禽遗传资源保护品种[2]。近年来,随着人们对羊肉需求量的日益增加,养羊效益和保种矛盾逐步凸显,济宁青山羊杂交滥交现象和品种资源消耗严重,纯种数量不断减少,优良种羊流失,品种资源保护形势严峻[3]。这一问题引起相关政府部门足够的重视,对济宁青山羊的育种与改良开展了大量工作,使得济宁青山羊养殖业步入良性发展轨道。经过系统的选种选配、提纯复壮、核心群选育和加强饲养管理,济宁青山羊种群数量、羊群整体质量得到显著提高。为了进一步摸清济宁青山羊品种资源最新动态及其遗传结构和起源分化,本研究应用微卫星DNA标记技术,对济宁青山羊群体遗传多样性和群体间的遗传分化进行分析。
济宁青山羊是在当地生态环境条件下,经过数百年的选育形成的优良地方品种,一直保持着闭锁的繁殖方式,没有受外来品种的影响,是非常宝贵的资源。目前,对济宁青山羊分子遗传方面的研究甚少,且多集中于对其繁殖性能相关候选基因的多态性研究[4-6]等方面,而在其种群和遗传多样性方面的研究相对滞后。所以,对济宁青山羊的种群进行深入研究,掌握其资源现状和发展趋势,对制定合理有效的保种策略,促进济宁青山羊品种资源的保护、开发及利用具有深远的意义。
1材料与方法
1.1样本采集
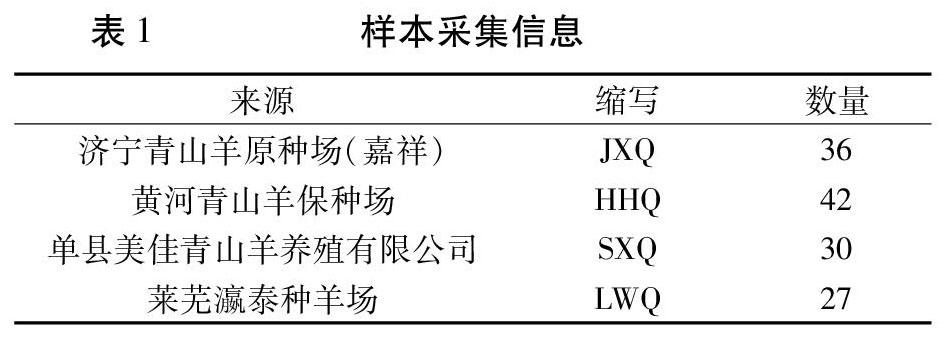
试验样本分别采自山东省4个济宁青山羊种群血样或小块耳组织,共计135份(表1),个体之间无亲缘关系,品种的外貌特征明显。
1.2DNA提取
采用血液/细胞/组织基因组DNA提取试剂盒或常规酚氯仿抽提法提取基因组DNA,-20℃保存备用。
1.3试剂与仪器
试剂:血液/细胞/组织基因组DNA提取试剂盒,购自北京艾德莱生物科技有限公司;PCRMix、DNAMarker等,购自南京诺唯赞生物科技有限公司。
仪器:Eppendorf高速台式离心机、EppendorfPCR扩增仪、电热恒温水槽、BIO-RAD凝胶成像仪及电泳系统、ABI3100-Avant全自动基因序列分析仪。
1.4微卫星引物
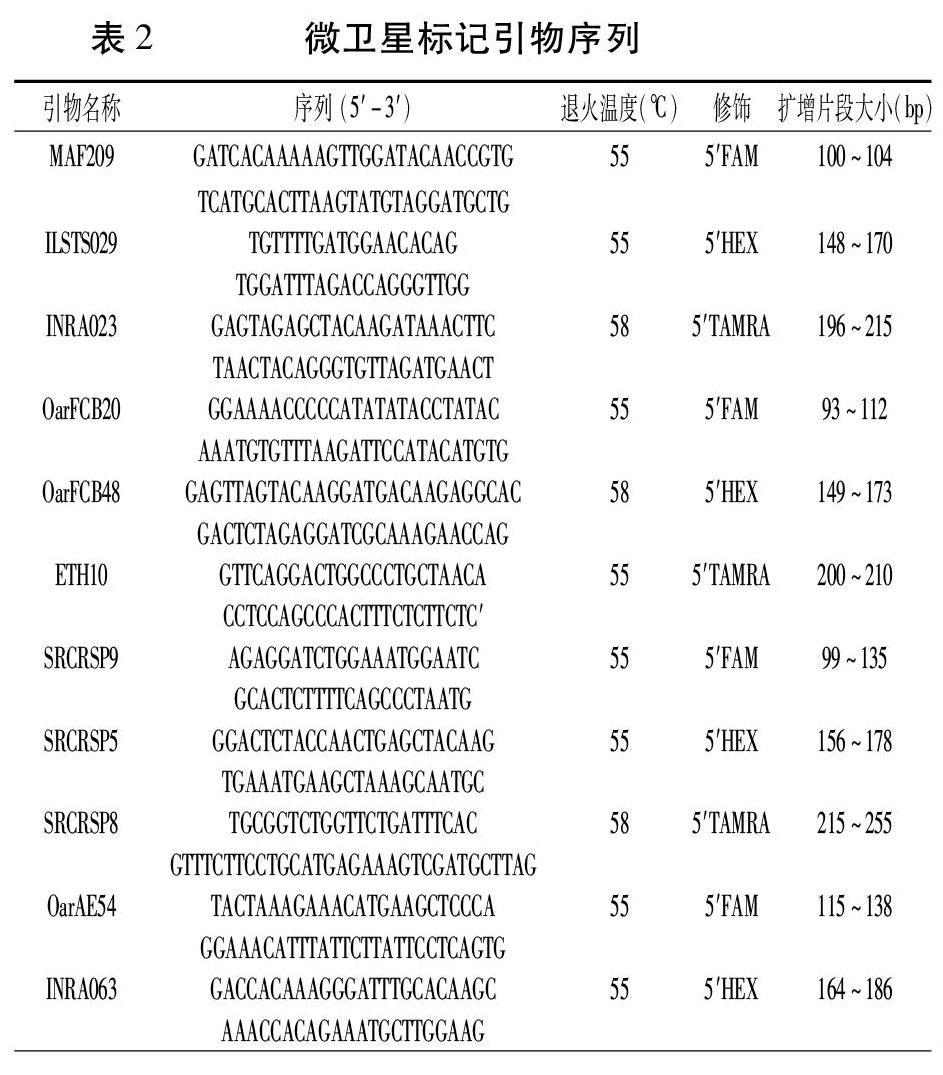
参照联合国粮农组织(FAO)及国际遗传学会(ISAG)的推荐,共筛选出24对荧光标记微卫星引物(表2)。引物由生工生物工程(上海)股份有限公司合成。
1.5PCR扩增
PCR反应体系:PCRMix6.25μL,上下游引物(10mmol/L)各1μL,模板DNA1μL,补水至12.5μL。PCR反应条件:94℃5min;94℃30s,54、55、58℃或65℃30s,72℃30s,30个循环;72℃5min。
1.6微卫星DNA多态性分析
PCR产物、去离子甲酰胺、TAMRA(FAM或HEX)以0.5μL∶10μL∶0.5μL的比例混合,95℃变性5min,采用ABI3100-Avant全自動基因序列分析仪进行分析,用3100DataCollection软件分析微卫星基因型。
1.7数据分析
用POPGEN1.32计算有效等位基因数(Ne)、杂合度及基因流等。多态信息含量(PIC)用PIC分析软件计算。
2结果与分析
2.1微卫星DNA多态性分析
利用ABI3100-Avant全自动基因序列分析仪对135个样本的PCR扩增产物进行分型测定,部分结果见图1和图2。24个微卫星位点的电泳图峰形典型,易于判型,适于后续分析。
2.2群体内遗传变异分析
微卫星位点上的观察等位基因数(Na)和有效等位基因数(Ne)能够反映群体内的变异程度,4个群体24个微卫星位点的PCR产物共检测到159个等位基因,平均每个位点为6.6个,各位点有效等位基因数范围为1.3423~6.4800,平均有效等位基因数3.5282(表3),说明选择的微卫星标记在4个群体中具有多态性,适宜群体间遗传关系的分析。
由表4可知,济宁青山羊具有丰富的遗传多样性。4个群体的PIC在0.6016~0.6653之间,各群体He也较高(0.6750~0.7559),但是群体的Ho均低于He,LWQ最低为0.5375,JXQ最高为0.6260。
2.3群体间遗传关系分析
从整个济宁青山羊种群来看,群体内近交系数(Fis)为0.1099,其中有5个位点的Fis为负值,总群体近交系数(Fit)为0.2138,其中CSRD247基因座的Fit为负值,说明群体存在一定程度的近交,但群体内杂合体较多,近交程度尚不严重。群体间基因分化系数(Fst)为0.0774,说明7.741%的遗传变异是由群体间差异引起的,而92.259%的遗传变异是由各群体内个体间的差异引起的,群体间的遗传分化水平较低。基因流(Nm)分析表明,每世代4个群体间有效迁移个体数为1.8905,说明群体间存在一定的基因交流(表5)。
2.4遗传距离和遗传相似性指数分析
由表6可知,SXQ、HHQ和JXQ群体间遗传距离较近,遗传相似性指数较高,表明3个群体间具有较近的亲缘关系。而LWQ与3个群体间遗传距离相对较远,遗传相似性指数相对较低,因此,群体间亲缘关系相对较远。
2.54个群体之间的聚类分析
由图3可知,SXQ与HHQ群体的遗传距离最小,亲缘关系最近,首先聚在一起;然后两者再与JXQ群体聚合,三者与LWQ群体的遗传距离相对较远。SXQ、HHQ和JXQ位于山东省的济宁、菏泽地区,为济宁青山羊主产区,而LWQ位于边缘产区莱芜,可见地理距离与遗传距离具有较强的正相关性。
3讨论与结论
Barker等[7]提出要满足微卫星遗传标记研究的需要,每个群体的样本数量不能小于25。汤青萍等[8]认为,利用微卫星标记进行遗传多样性检测时,选择的引物应尽量均匀地分布于物种的各染色体上,引物数量应不低于20对,这样结果才较准确。本研究采用的24个位点分布于9条染色体上,分析的个体数量、选用的微卫星标记数量都基本达到了遗传多样性检测的要求,具有一定的可靠性。
本试验检测到的JXQ和HHQ的有效等位基因数略高于SXQ和LWQ的,其中以LWQ的有效等位基因数最低,表明各群体之间的遗传结构存在差异,分析原因一方面可能是由于地理位置差异造成的,另一方面也可能与人工选择有关。采样的群体规模小、数量少,选育过程中存在的人为因素都可对群体的基因平衡产生影响[9]。4个群体的等位基因数均高于有效等位基因数,根据Hines等[10]的研究,这可能与近交有关,导致等位基因分布不均匀。
Botstein等[11]提出多态信息含量(PIC)是用来描述微卫星标记的变异程度和估计群体内的遗传变异程度,当PIC>0.5时为高度多态标记,0.25 遗传分化系数(Fst)可以反映群体近交或群体间遗传分化程度[12]。本研究中Fst为0.0774,说明4个群体间的遗传分化水平低,而且基因流大于1(1.8905),均质化作用强,能够抵制遗传漂变作用,防止群体间遗传分化发生。结果表明济宁青山羊已经在遗传水平上形成了相对独立的体系,依据不同地理位置形成了不同的生态类型。 总之,通过近年来加大对济宁青山羊的保护力度,群体的遗传多样性得到了保留,群体间的基因流较大,遗传分化水平较低。从保种效果来看,JXQ属于国家级保种场,每年都得到政府的经费支持,遗传基础比较好,保留了济宁青山羊最完整、最原始的优良基因,多态信息含量最高。LWQ位于边缘产区,可能由于长期的地理隔离和生理隔离,群体数量不大,群体的同质性加大,遗传多样性丢失,平均杂合度降低。另外,从整个济宁青山羊群体来看,都存在不同程度的近交,JXQ和HHQ的近交程度稍低于SXQ和LWQ,所以在实际保种工作中,应扩大群体数量,尽量避免近交,以维持济宁青山羊群体的遗传多样性。 参考文献: [1]蔡泉.济宁青山羊品种资源调查[J].中国草食动物,2008,28(6):64. [2]许腾.鲁西南地区青山羊种质资源调查与分析[J].中国畜牧兽医,2011,38(1):148-152. [3]王可,蔡中峰,楚惠民,等.山东省济宁青山羊种质资源调查与分析报告[J].江苏农业科学,2013,41(7):215-217. [4]葛仕豪,高立坤,侯衍猛,等.济宁青山羊发情周期内促性腺激素和性激素分泌规律的研究[J].西南农业学报,2007,20(6):1348-1352. [5]梁琛,储明星,张建海,等.FSHβ基因PCR-SSCP多态性及其与济宁青山羊高繁殖力关系的研究[J].遗传,2006,28(9):1071-1077. [6]冯涛,赵有璋,储明星,等.济宁青山羊多羔性候选基因BMP15的研究[J].畜牧兽医学报,2009,40(4):468-475. [7]BarkerJSF.Aglobalprotocolfordetermininggeneticdistancesamongdomesticlivestockbreeds[C]//Proceedingsofthe5thWorldCongressonGeneticAppliedtoLivestockProduction,1994,21:501-508. [8]汤青萍,陈宽维,李慧芳,等.中国斗鸡遗传分化的微卫星标记分析[J].西北农林科技大学学报(自然科学版),2005,33(3):19-23,28. [9]江燕,傅彤,廉红霞,等.不同产区郏县红牛生态差异性分析[J].畜牧獸医学报,2014,45(1):46-55. [10]HinesHC,ZikakisJP,HaenleinGF,etal.Linkagerelationshipsamonglociofpolymorphismsinbloodandmilkofcattle[J].J.DairySci.,1981,64(1):71-76. [11]BotsteinD,WhiteRL,SkolnickM,etal.Constructionofageneticlinkagemapinmanusingrestrictionfragmentlengthpolymorphisms[J].AmericanJournalofHumanGenetic,1980,32(3):314-331. [12]曾检华,曹少先,舒邓群,等.基于微卫星标记的湖羊资源保护效果的评价[J].江苏农业学报,2010,26(6):1283-1287.
