实践十号搭载白色念珠菌的代谢组学分析
2019-07-02赵光显王佳平高建义辛冰牧李勇枝
赵光显,王佳平,刘 宇,高建义,辛冰牧,王 珩,李勇枝
(中国航天员科研训练中心,北京100094)
1 引言
近年来,空间微生物安全问题已成为相关领域的研究热点。 白色念珠菌是一种常见的条件性致病菌,常寄生于人体皮肤表面、口腔、胃肠道以及外生殖系统内[1],当外界环境发生变化时,其致病性往往会发生变化。 无论是在阿波罗号飞船的航天员身上[2],还是在和平号空间站和国际空间站舱内[3],均发现了白色念珠菌的存在。 而在太空飞行环境下人体免疫力会降低[4-5],白色念珠菌的存在对航天员的健康构成了潜在威胁。 虽然近年来有一些研究表明包括白色念珠菌在内的一些致病微生物空间环境下的生长繁殖加快[6-9]、毒性增强[10-11],但其生物学性状变化的深层机制目前仍不十分清楚,尤其在代谢机制方面研究较少,亟待深入研究[12-13]。
代谢组学是近年来继基因组学和蛋白质组学之后新兴的一门学科,主要通过组群分析对生物细胞中全部低分子质量代谢产物进行定性、定量分析,阐释内外因素的作用下产生的代谢物质的动态变化[14]。 代谢组学根据实验目的可分为靶向代谢组学和非靶向代谢组学。 靶向代谢组学是检测特定种类内源性代谢物的实验方法,而非靶向代谢组学是检测整个生物体内源性代谢物的实验方法,其能系统地阐释生物体内内源性代谢物的变化规律。 非靶向代谢组学中的高效液相色谱-飞行时间质谱技术(High Performance Liquid Chromatography Quadrupole-Time of Flight Mass Spectrometry, UPLC-QTOF/MS)具有高灵敏度、高分辨率、高分离率等特点,可对生物代谢产物的分析结构及质量信息能进行精确分析,目前已在生命科学的研究中广泛应用[15]。
本实验通过UPLC-QTOF/MS 分析鉴定实践十号卫星搭载白色念珠菌的显著差异生物代谢物,通过分析特征性代谢物的代谢通路,阐释太空环境可能对白色念珠菌生物学性状的影响。
2 实验材料
2.1 菌株
白色念珠菌[Candida albicans,CMCC(F)98001],购自中国医学细菌微生物菌种保藏管理中心。
2.2 培养基
沙氏液体培养基(OXIOD 公司,CM0147)组成:葡萄糖20 g/L,动物组织胃蛋白酶水解物5 g/L,胰酪胨5 g/L。
2.3 主要试剂
甲醇(Fisher Scientific 公司,A456-4),PBS 溶液(Gibco 公司,C20012500BT),氮气(氮气发生器产生)。
2.4 主要仪器及耗材
超快速液相色谱:Acquity UPLC,Waters 公司;震荡混匀器:Mix-3000,杭州米欧仪器有限公司;四极杆飞行时间质谱:Xevo G2-Si,Waters 公司;台式高速冷冻离心机:Mikro 220R,Hettich 公司;超声波振荡器:KQ-500E,昆山市超声仪器有限公司;反向色谱柱:ACQUITY UPLC CSH C18,Waters 公司;2 mL 进样瓶:5320,DIKMA 公司;1.5 mL离心管:Axygen MCT-150-C,Corning 公司;250 μL 内插管: VDAP-4025-6297E-100, ANPEL公司。
2.5 主要分析软件
Masslynx 4.1 质谱数据采集软件,Waters 公司;Progenesis QI 2.1 代谢组数据处理软件,Nonlinear Dynamics 公司;Simca 14.1 统计分析软件,Umetrics 公司。
3 实验方法
3.1 搭载菌株的培养
将白色念珠菌培养到对数生长期OD600nm=1.0 的菌液20 μL 接种到沙氏葡萄糖半固体培养基,30 ℃恒温培养30 h 后,一部分搭载实践十号返回式科学实验卫星,太空飞行12 d,回收后-20 ℃甘油保存菌种,一部分在地面常规培养设立对照组。
3.2 样本制备
首先,将实践十号卫星搭载的白色念珠菌菌株和地面常规对照组菌株在沙氏葡萄糖平板上30 ℃过夜活化,将活化的两组菌株取少量接种于沙氏液体培养基中30 ℃振荡培养(150 r/min)至对数中期(OD600nm=1.0)时停止培养,4 ℃离心5 min(4000 r/min),弃上清,无菌PBS 溶液冲洗菌液,同条件离心5 min,弃上清,再次用无菌PBS溶液冲洗菌液,再离心,重复冲洗3 次后最后离心收集菌沉淀于1.5 mL 的样品离心管中-80 ℃保存。 其次,将离心管中菌沉淀样本在4 ℃条件下融化30 ~ 60 min,加入300 μL 甲醇,超声振荡提取10 min。 最后,将离心管中液体充分振荡15 s,然后在4 ℃条件下用离心机离心10 min(12000 r/min),离心完毕后取上层溶液100 μL,加入200 μL的内衬管中以待后续检测。
3.3 UPLC-QTOF/MS 检测
UPLC-QTOF/MS 检测首先使用高效液相色谱(Ultra Performance Liquid Chromatography, UPLC)分离菌液样本代谢物;其次运用四极杆飞行时间质谱进行一二级质朴扫描,模式为全信息串联质谱模式(MSE),得出母离子与碎片离子相关质谱信息;然后运用主成分分析(Principal Component Analysis, PCA)及正交偏最小二乘判别分析(Orthogonal Partial Least Squares Discriminant Analysis, OPLS-DA)对2 组样本的差异代谢物进行定性分析,根据一二级质谱得出代谢物的质荷比、保留时间、特征性碎片离子信息匹配公共数据库找出组间的潜在显著差异生物代谢物。
3.3.1 反相色谱分离条件
色谱柱(waters UPLC HSS T3)的规格:1.8 μm*2.1 mm*100 mm。
流动相条件。 A:水,0.1%甲酸;B:乙腈,0.1%甲酸;流速:0.3 mL/min;进样量:1.0 μL;柱温:45 ℃。 具体洗脱程序见表1。

表1 C18 反相色谱离子测定洗脱程序Table 1 The gradient conditions for reversed phase C18 separation for ion
3.3.2 质谱条件
运用四极杆飞行时间质谱进行质谱信息采集,采集使用电喷雾离子源(ESI)正负离子模式,碰撞气、翘气以及辅助气均为氮气,应用Masslynx4.1 软件进行数据采集和处理,质谱检测模式为MSE模式,其通过高低2 种能量交替扫描的方式得到代谢物一二级质谱相关数据。 具体条件见表2。

表2 质谱条件Table 2 Mass spectrometry condition
3.3.3 质量控制
质量控制是评价质谱系统能否得到重复性好且准确性高的差异代谢物的重要环节。 根据保留时间、质量准确度和信号强度这3 个可能影响质量的因素,本实验首先采用5 个空白样本平衡色谱柱,再采用3 个质控样本平衡柱条件。 每间隔3 个样本柱加入1 个质控柱用于监测系统的重复性和稳定性,剔除变异系数超过15%的特征值以确保数据准确可靠。
3.3.4 非靶向代谢组数据处理
无论是正离子还是负离子模式,均用Progenesis QI 软件处理所有采集好的数据并作图(峰提取强度调整为模式1)。 运用Simca 软件对采集的离子数据进行PCA 分析及OPLS-DA 分析。PCA 分析及OPLS-DA 分析的条件如下:
1)PCA 分析。 为了判别搭载组白色念珠菌代谢物与地面对照组是否具有组间差异,本实验首先采用无监督的PCA 建模方法对2 组进行分析。 R2X 代表模型的解释率,Q2代表了模型的可预测变量。 2 种扫描模式的PCA 模型的参数见表3。

表3 2 组比较主成分分析模型参数Table 3 Comparison of PCA model parameters
2)OPLS-DA 分析。 为获得有显著差异的代谢物信息,以探究太空飞行对白色念珠菌引起的代谢变化,从而找到潜在的生物标志物,本实验在进行PCA 分析后,采用有监督的OPLS-DA 分析对代谢数据重新进行建模。 OPLS-DA 分析可减小其他因素干扰,剔除无关分类变量,从而放大组间差异,得到更好的分类效果。 本实验将OPLSDA 模型的变量重要性值(Variable Importance in the Projection,VIP)定为大于1,通过ANOVA 分析(P<0.05)找出相关差异生物标志物。 组间显著差异生物标志物通过以下方式定性:搜索公共数据库(METLIN、LIPID MAPS、PUBCHEM、YMDB以及KEGG)等,比较质谱的质荷比,误差限制0.01 Da,然后根据其保留时间以及二级图谱中特征性碎片离子辅助定性鉴别,并找出其可能的代谢途径。
使用OPLS-DA 模型对2 组样本进行统计分析,R2Y(模型的解释率)及Q2值(模型的预测率)可判别模型的质量好坏。 本实验的模型参数见表4。

表4 2 组比较OPLS-DA 模型参数Table 4 Comparison of OPLS-DA model parameters
表4 说明该模型在正离子状态下有97.5%的可能性解释2 组样本的差异性,负离子状态下有90.1%的可能性解释2 组样本的差异性。 以上参数说明数据的拟合度比较好,可满足OPLS-DA 的建模要求。
4 实验结果
4.1 质控评估结果
PCA 分析结果如图1~2 所示。 图1 为反相色谱正离子PCA 质控得分图,图2 为反相色谱负离子PCA 质控得分图。 图中每一个绿色圆圈代表1 个质控样本(qc),每一个蓝色三角符号为1个空白样本(sample),椭圆内代表95%可信区间,横纵坐标代表不同主成分积分值。 由图1~2 可以看出正负离子模式的质控样本都聚集在了一起,这说明质谱系统的稳定性和准确性很好,可以进行下一步实验研究。
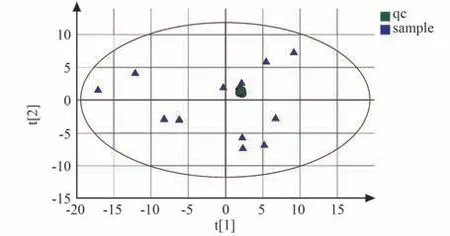
图1 正离子质控评估(PCA 得分图)Fig.1 Quality control sample evaluation of positive ions(PCA scores)

图2 负离子质控评估(PCA 得分图)Fig.2 Quality control sample evaluation of negative ions(PCA scores)
4.2 总离子流图比较
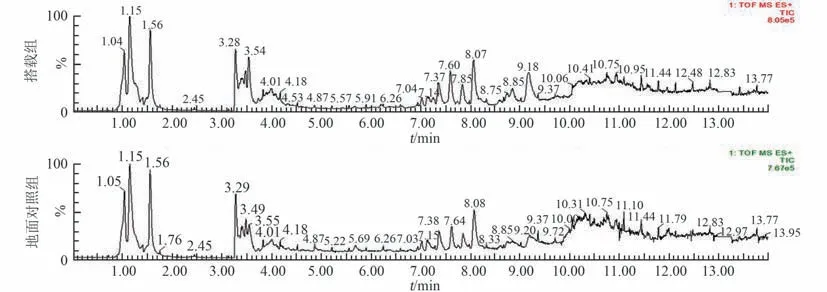
图3 正离子总离子流图Fig.3 Total ion current chromatogram of positive ions

图4 负离子总离子流图Fig.4 Total ion current chromatogram of negative ions
各组反相色谱正离子、负离子的总离子流图如图3~4 所示。 每幅图的上图为搭载组离子流图,下图为地面对照组离子流图。 每幅图横坐标为保留时间,纵坐标为相对峰强度百分比。 其中正离子提峰759 个,负离子提峰有706 个。 从正负离子的总离子流图可以看出,搭载组和地面对照组白色念珠菌存在很多相同的代谢物,但根据代谢物峰值初步判断一些代谢物在量上存在一定差异。
4.3 两组样本差异代谢物显著性分析
4.3.1 PCA 分析结果
2 组样本PCA 得分图如图5~6 所示。 图5代表正离子PCA 得分图,图6 代表负离子PCA得分图,图中每一个绿色圆圈符号代表1 个搭载组样本(space flight),每一个三角符号代表1 个地面对照组样本(control)。 横纵坐标依次分别为第一、第二主成分得分值。 椭圆内代表主成分积分值95%可信区间范围。 从图5~6 可以看出,无论是正离子PCA 得分图,还是负离子PCA 得分图,2 组代谢物均在可信区间内,差异并没有明显分离,但可反映出代谢谱有组间差异。 需进一步进行OPLS-DA 分析。
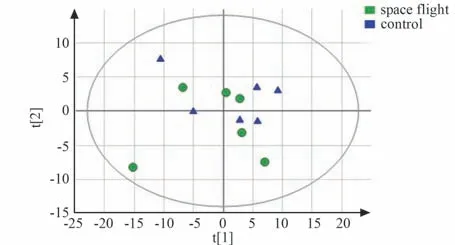
图5 正离子PCA 得分图Fig.5 PCA scores of positive ions
4.3.2 OPLS-DA 分析结果

图6 负离子PCA 得分图Fig.6 PCA scores of negative ions
OPLS-DA 得分图(Scores plot)如图7~8 所示。 图7 代表正离子OPLS-DA 得分图,图8 代表负离子OPLS-DA 得分图,图中每个圆圈符号代表每个搭载组样本,每个三角符号代表每个地面对照组样本。 椭圆内代表OPLS-DA 积分值95%可信区间范围。 横纵坐标表示OPLS-DA 相关积分参数。 从正、负离子OPLS-DA 得分图可以看出搭载组样本和地面对照组样本被显著划分为2 群,表明代谢谱有明显的组间差异。
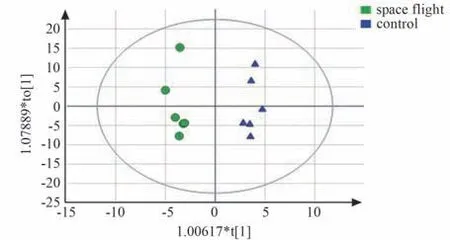
图7 正离子OPLS-DA 得分图Fig.7 OPLS-DA scores of positive ions
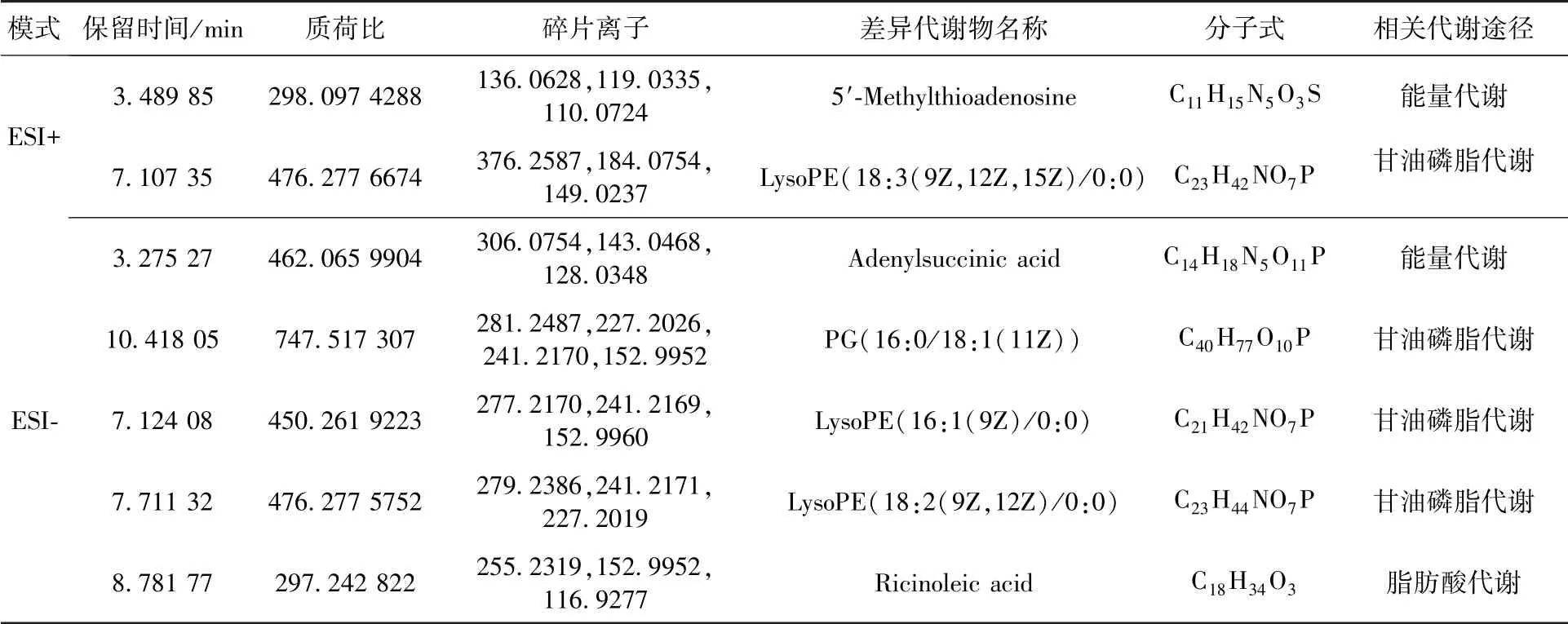
表5 组间显著差异生物标志物及可能的代谢途径Table 5 Significantly different biomarkers and possible metabolic pathways

图8 负离子OPLS-DA 得分图Fig.8 OPLS-DA scores of negative ions
4.4 组间显著差异生物标志物的鉴定
通过对OPLS-DA 模型中VIP>1 的代谢物进行筛选,结合ANOVA 分析(P<0.05)共找出7 种差异生物标志物,其中正离子模式下有2 种,负离子模式下有5 种,根据其一二级质谱信息匹配公共数据库得到的差异生物代谢物名称、分子式及相关代谢途径如表5 所示,由表可以看出这7 种差异生物代谢物与能量代谢、甘油磷脂代谢以及脂肪酸代谢密切相关。 定量分析如图9~10 所示,从差异生物标志物箱形图中可以看出这7 种菌类代谢物的含量发生了明显改变,其中搭载组白色念珠菌与地面对照组相比,含量上升的有3种代谢物(图9),分别是5′-甲硫腺苷(5′-Methylthioadenosine)、腺苷酸代琥珀酸(Adenylsuccinic Acid)和磷脂酰甘油PG(16:0/18:1(11Z))。 含量下降的有4 种代谢物(图10),其种3 种溶血磷脂酰乙醇胺(LysoPE)含量下降,还有一种不饱和脂肪酸蓖麻油酸(Ricinoleic Acid)含量下降。
5 讨论

图9 在搭载组含量上升的3 种生物标志物箱形图Fig.9 Box-plot of 3 biomarkers with increased contents in spaceship-carried group

图10 在搭载组含量下降的4 种生物标志物箱形图Fig.10 Box-plot of 4 biomarkers with decreased contents in spaceship-carried group
本研究中,搭载组白色念珠菌与地面对照组相比,共发现了2 种与能量代谢相关的潜在生物标志物代谢上调,一种是5′-甲硫腺苷,另外一种是腺苷酸代琥珀酸。 5′-甲硫腺苷广泛存在于原核生物、真核生物、植物及高等生物中间,5′-甲硫腺苷中的甲基是由蛋氨酸来提供,剩下的部分分子是由ATP来提供,有研究表明5′-甲硫腺苷与生物的能量代谢与增殖密切相关[16]。 腺苷酸代琥珀酸是AMP和GMP 的生成过程中一种重要的代谢中间物,参与生物能量代谢与三羧酸循环。 搭载组白色念珠菌5′-甲硫腺苷和腺苷酸代琥珀酸代谢水平增加高度提示搭载组能量代谢旺盛,潜在的增殖水平可能提高。 谢琼等[7]用航天器搭载白色念珠菌在轨飞行7 天,发现搭载组比地面对照组存活增长率明显提高;江文俊等[17]也在模拟失重条件下发现白色念珠菌的增殖速率加快。 Crabbe 等[18]在NASA 执行STS-115 任务期间搭载了白色念珠菌,发现在太空搭载环境可促进白色念珠菌的随机出芽,这也间接说明了在太空环境下白色念珠菌的能量代谢水平相对旺盛。
其次,在甘油磷脂代谢中1 种磷脂酰甘油PG(16:0/18:1(11Z))在搭载组显著上升。 磷脂酰甘油(phosphatidylglycerol, PG)存在于生物的细胞膜和线粒体膜中,是合成心磷脂的前体[19],而心磷脂在线粒体内膜中对维持线粒体的能量代谢具有至关重要的作用,它可促进线粒体中氧化呼吸链的电子传递[20]。 PG 本身也有研究表明在线粒体缺陷的酵母细胞中对线粒体功能的修复有重要影响[21]。 搭载组PG 含量的显著上升提示搭载组白色念珠菌细胞线粒体能量代谢相对旺盛,这与之前5′-甲硫腺和腺苷酸代琥珀酸苷含量上升的分析结果相一致,即太空搭载环境提高白色念珠菌的能量代谢水平。
另外,在甘油磷脂代谢中3 种溶血磷脂酰乙醇胺(表5 中LysoPE)的含量略有下降。 从溶血磷脂代谢通路分析推断搭载组3 种溶血磷脂酰乙醇胺含量下降可能与白色念珠菌部分磷脂酶B高表达有关。 有研究表明,在真菌细胞中磷脂酶B 具有溶血磷脂酶-转酰基活性,其溶血磷脂酶活性可水解溶血磷脂,其转酰基活性可将细胞中游离脂肪酸转移到溶血磷脂中生成磷脂[22]。 3 种LysoPE 属于溶血磷脂,可能正是部分磷脂酶B 这种双重高表达活性的综合作用才导致了3 种LysoPE 在搭载组含量下降。 而磷脂酶B 是致病真菌中广泛存在的毒力因子[23],磷脂酶B 表达活性增强,白色念珠菌毒力随之提高[24-25]。 因此3 种LysoPE 含量下降提示搭载组白色念珠菌毒力可能增强。 在模拟微重力条件下用白色念珠菌对小鼠进行毒力实验,发现模拟微重力组白色念珠菌对小鼠的致死能力提高,对巨噬细胞破坏增强[26]。 多个研究表明[27-28]失重环境可促进白色念珠菌由酵母相向菌丝相转变,而白色念珠菌的菌丝相对人体更具侵袭力,毒力更强[29-31]。
最后,本实验发现一种叫蓖麻油酸(Ricinoleic acid)的不饱和脂肪酸含量下降。 目前研究发现蓖麻油酸是念珠菌多功能膜脂肪酸去饱和酶CpFad2 的产物[32],参与念珠菌细胞膜不饱和脂肪酸代谢,但其在细胞中的功能目前还不十分清楚。 搭载组蓖麻油酸含量下降提示搭载组脂肪酸代谢水平下调,相反提示能量代谢相关通路代谢水平可能上调,从而印证搭载组能量代谢水平提高。
6 结论
本研究共发现了7 种差异生物标志物,与对照组相比,搭载组5′-甲硫腺苷、腺苷酸代琥珀酸以及磷脂酰甘油3 种能量代谢物含量上调,1 种不饱和脂肪酸蓖麻油酸含量下调提示太空搭载环境可显著提高白色念珠菌能量代谢水平,其潜在增殖水平可能提高;3 种溶血磷脂酰乙醇胺在搭载组含量下调提示搭载组白色念珠菌磷脂酶B高表达活性,磷脂酶B 是真菌中广泛存在的毒力因子,这提示太空搭载环境可使白色念珠菌的毒性水平增加。
参考文献(References)
[1]Mccullough M J, Ross B C, Reade P C.Candida albicans: a review of its history, taxonomy, epidemiology, virulence attributes, and methods of strain differentiation[J].International Journal of Oral & Maxillofacial Surgery, 1996, 25(2):136-144.
[2]Taylor G R.Recovery of medically important microorganisms from Apollo astronauts[J].Aerosp Med, 1974, 45(8):824-828.
[3]Novikova N, De B P, Poddubko S, et al.Survey of environmental biocontamination on board the International Space Station[J].Research in Microbiology, 2006, 157(1):5-12.
[4]Sonnenfeld G, Butel J S, Shearer W T.Effects of the space flight environment on the immune system[J].Reviews on Environmental Health, 2003, 18(1):1-18.
[5]Crucian B, Stowe R P, Mehta S, et al.Alterations in adaptive immunity persist during long-duration spaceflight[J].2015(1):15013.
[6]Rosenzweig J A, Ohunene A, Kayama T, et al.Spaceflight and modeled microgravity effects on microbial growth and virulence[J].Applied Microbiology & Biotechnology, 2010, 85(4):885-891.
[7]谢琼, 吴元亮, 李勇枝,等.空间环境对13 株微生物生物学特性的影响[J].中华医院感染学杂志, 2004, 4(11):1221-1224.Xie Q, WU Y L,LI Y Z, et al.Effects of space flight on biological characteristics of 13 strains of microorganism[J].Chinese Journal of Nosoconmiology, 2004, 4(11):1221-1224.(in Chinese)
[8]Sugita T, Yamazaki T, Makimura K, et al.Comprehensive analysis of the skin fungal microbiota of astronauts during a half-year stay at the International Space Station[J].Medical Mycology, 2016, 54(3):232-239.
[9]Kim W, Tengra F K, Young Z, et al.Spaceflight promotes biofilm formation by Pseudomonas aeruginosa[J].Plos One,2013, 8(4):e62437.
[10]Knox B P,Blachowicz A, Palmer J M, et al.Characterization of aspergillus fumigatus isolates from air and surfaces of the International Space Station[J].Msphere, 2016, 1(5):e00227-16.
[11]Wilson J W, Ott C M, Quick L, et al.Media ion composition controls regulatory and virulence response of salmonella in spaceflight[J].Plos One, 2008, 3(12):e3923.
[12]Huang B, Li D G, Huang Y, et al.Effects of spaceflight and simulated microgravity on microbial growth and secondary metabolism[J].Military Medical Research, 2018, 5(1):18.
[13]Taylor P W.Impact of space flight on bacterial virulence and antibiotic susceptibility[J].Infection and Drug Resistance,2015, 8:249-262.
[14]Johnson C H, Ivanisevic J,Siuzdak G.Metabolomics:beyond biomarkers and towards mechanisms[J].Nat Rev Mol Cell Biol, 2016, 17(7):451.
[15]Kim M S, Nam M, Hwang G S.Metabolic alterations in two cirsium species identified at distinct phenological stages using UPLC-QTOF/MS[J].Phytochemical Analysis Pca, 2017,29(1):77-86.
[16]Avila M A,Garciatrevijano E R,Lu S C,et al.Methylthioadenosine[J].International Journal of Biochemistry&Cell Biology, 2004, 36(11):2125-2130.
[17]Jiang W,Xu B, Yi Y,et al.Effects of simulated microgravity by RCCS on the biological features of Candida albicans[J].International Journal of Clinical & Experimental Pathology,2014, 7(7):3781-90.
[18]Crabbe A, Nielsenpreiss S M, Woolley C M, et al.Spaceflight enhances cell aggregation and random budding in Candida albicans[J].Plos One, 2013, 8(12):e80677.
[19]Dugail I, Kayser B D, Lhomme M.Specific roles of phosphatidylglycerols in hosts and microbes [J].Biochimie,2017, 141:47-53.
[20]Houtkooper R H, Vaz F M.Cardiolipin, the heart of mitochondrial metabolism[J].Cellular & Molecular Life Sciences Cmls, 2008, 65(16):2493-2506.
[21]Nada K, Mohammad R, Anneflorence B, et al.Interplay of packing and flip-flop in local bilayer deformation.How phosphatidylglycerol could rescue mitochondrial function in a cardiolipin-deficient yeast mutant [J].Biophysical Journal,2014, 107(4):879-890.
[22]Ghannoum M A.Potential role of phospholipases in virulence and fungal pathogenesis[J].Clinical Microbiology Reviews,2000, 13(1):122-143.
[23]Ghannoum M A.Extracellular phospholipases as universal virulence factor in pathogenic fungi[J].Japanese Journal of Medical Mycology, 1998, 39(2):55.
[24]Theiss S, Ishdorj G,Brenot A,et al.Inactivation of the phospholipase B gene PLB5, in wild-type Candida albicans, reduces cell-associated phospholipase A2, activity and attenuates virulence[J].International Journal of Medical Microbiology, 2006, 296(6):405-20.
[25]Kothavade R J, Panthaki M H.Evaluation of phospholipase activity of Candida albicans and its correlation with pathogenicity in mice[J].Journal of Medical Microbiology,1998,47(2):99-102.
[26]王佳平, 王静, 吴元亮,等.回转器模拟微重力对白色念珠菌致病性的影响[J].航天医学与医学工程, 2012, 25(3):162-167.Wang J P, Wang J, Wu Y L, et al.Effects of simulated microgravity with clinostat on pathogenicity of Candida albicans[J].Space Medicine & Medical Engineering,2012,25(3):162-167.(in Chinese)
[27]Altenburg S D, Nielsen-Preiss S M, Hyman L E.Increased filamentous growth of Candida albicans in simulated microgravity[J].Genomics Proteomics & Bioinformatics, 2008, 6(1):42-50.
[28]Microgravity M, Filamentation I, Searles S C, et al.Modeled microgravity increases filamentation, biofilm formation, phenotypic switching,and antimicrobial resistance in Candida albicans[J].Astrobiology, 2011, 11(8):825.
[29]Mayer F L,Wilson D,Hube B.Candida albicans pathogenicity mechanisms[J].Virulence, 2013, 4(2):119.
[30]Mitchell A P.Microbiology: Fungus produces a toxic surprise[J].Nature, 2016, 532(7597):41.
[31]Gow N A R,Van d V F L,Brown A J P,et al.Candida albicans morphogenesis and host defence: discriminating invasion from colonization[J].Nature Reviews Microbiology, 2011,10:1039-1044.
[32]Bucek A, Matouskova P, Sychrova H, et al.Δ12-Fatty acid desaturase from Candida parapsilosis is a multifunctional desaturase producing a range of polyunsaturated and hydroxylated fatty acids[J].Plos One, 2014, 9(3):e93322.
