四倍体棉花GAE基因家族的鉴定及其在棉纤维发育中的表达分析
2019-06-14张松雨王敬敬刘正文张艳杨君马峙英王省芬
张松雨,王敬敬,刘正文,张艳,杨君,马峙英,王省芬
(河北农业大学/ 教育部华北作物种质资源重点实验室/ 河北省作物种质资源重点实验室,河北 保定 071001)
棉花是第一大天然纤维作物,是最重要的纺织原料之一。我国主要种植陆地棉(Gossypium hirsutum)和海岛棉(G.barbadense)两个栽培种,其中陆地棉栽培面积和产量在棉花总产中占主导地位;海岛棉又称“长绒棉”,其纤维长度、比强度、细度等表现优良,是高档棉纺织品的必要原材料[1-3]。
植物细胞壁由纤维素、半纤维素、木质素、果胶和少量蛋白质等组成[4],是由多糖、结构蛋白和各种酶组成的高度复杂的结构。果胶中约60%以上为多聚同型半乳糖醛酸,同聚半乳糖醛酸是由半乳糖醛酸聚合而成的复合体,半乳糖醛酸是合成果胶所必需的前体物质[5];而葡糖醛酸异构酶(UDP-D-glucuronate 4-epimerase,GAE,EC:5.1.3.6)是催化 UDP- 葡萄糖醛酸(UDP-α-D-glucuronic acid,UDP-GlcA)向 UDP- 半乳糖醛酸(UDP-α-D-galacturonic acid,UDP-GalA)转化的关键酶。因此,在植物中含有大量的葡糖醛酸异构酶[6-8]。GAE 属于短链脱氢酶/ 还原酶家族(Short-chain dehydrogenases/reductases,SDR),催化各种依赖于氧化型辅酶I 或辅酶II (Nicotinamide Adenine Dinucleotide Phosphate,NAD(P)+)的反应,包括许多核苷酸糖的相互转换[9-10]。早在1958年,Neufeld 等[11]第一次分离出UDP- 葡糖醛酸差向异构酶。后来从肺炎链球菌中克隆了编码UDP-葡糖醛酸4- 异构酶的基因caplJ[12]。直到2004年,3个课题组几乎同时鉴定出拟南芥GAE 酶并进行了表达水平分析[5,10,13]。拟南芥AtGAEs家族含有6个成员,属于短链脱氢酶/还原酶家族的II 型膜蛋白,被预测均无内含子。该家族所有成员均含有与NAD(P)+结合相关的保守基序GxxGxG[10,14]。AtGAE01 特异性地催化 UDP-GlcA与UDP-GalA 间的转化,且在pH 7.6 时酶活性最高,酶活性受UDP-D-Xyl 强烈抑制。AtGAE01 和AtGAE06 在拟南芥根、叶、花和角果中高效表达,AtGAE05 在叶和角果中适度表达,但是在花或根中不表达[5]。有趣的是,所有AtGAE家族成员均在拟南芥花粉中表达,推测AtGAEs为果胶的合成提供前体物质[10,15]。将AtGAE04 反义基因分别转化拟南芥和烟草,转基因拟南芥形成的愈伤组织较小且时间较晚;转基因烟草多数生长明显受限,表现为植株矮小,生长缓慢,叶片长而细,难以形成完整的植株并最终死亡,只有极少数转基因烟草生长正常,说明反义AtGAE04 基因的确能够干扰GAE家族基因的正常表达,减少了果胶含量,影响了烟草细胞的正常粘连[8,16]。Pang 等[17]证明GhGAE1 在棉花纤维的伸长期表达量显著增高,GhGAE3 在棉纤维快速伸长期高效表达,说明GhGAE对棉纤维的长度有重要影响。
目前,已经公布的陆地棉与海岛棉基因组测序结果为挖掘重要的候选基因提供了可能[18]。本研究基于陆地棉与海岛棉基因组测序数据库,从全基因组水平鉴定GAE基因家族成员,分析其基因结构、蛋白序列保守性及保守位点、理化性质、系统进化等[19]。结合河北农业大学棉花遗传育种课题组已有的棉纤维发育转录组数据,分析GAE基因家族成员在棉纤维发育中的表达规律,这将为今后研究GAE基因家族成员的功能提供理论依据。
1 材料与方法
1.1 GAE基因家族序列搜索
从 TAIR(http://www.arabidopsis.org/)网站搜索拟南芥GAE基因,得到6个基因,再分别在NCBI (https://www.ncbi.nlm.nih.gov/) 中下载其DNA 序列、氨基酸序列。利用已知海岛棉GAE基因的 DNA 序列,在 CottonFGD(https://cottonfgd.org/)搜索并下载其 DNA、氨基酸序列[18],在Pfam(http://pfam.xfam.org/search)网站分析其保守域,得到2个保守域,其ID 分别为 PF16363 和PF0137。借助NCBI 网站分别鉴定筛选出的陆地棉GhGAEs和海岛棉GbGAEs中是否包含完整的 SDR 保守域,再利用 Smart(http://smart.embl-heidelberg.de/)和 NCBI 中 Conserved Domains程序(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)对家族成员进行进一步确认。
1.2 四倍体棉花GAE基因家族染色体定位和基因结构分析
根据GAE家族成员的染色体和位置信息,利用Mapchart 软件绘制基因定位图。将鉴定出的 21个陆地棉GhGAEs基因和 22个海岛棉GbGAEs基因序列通过MEGA 中的Clustal W 进行比对,采用邻近连接法(Neighbor-joining,NJ)构建系统发育树[20]。基因结构图的绘制利用在线网站 GSDS(http://gsds.cbi.pku.edu.cn/)完成。
1.3 四倍体棉花GAEs家族保守域分析
利用 Web Logo (http://weblogo.berkeley.edu/logo.cgi)绘制陆地棉和海岛棉GAE家族氨基酸序列 LOGO[21]图。借助 MEME(http://memesuite.org/tools/meme)分析氨基酸序列,获得保守基序[22]。
1.4 四倍体棉花与拟南芥GAEs的系统进化分析
利用MEGA6 的 Clustal W对21个陆地棉GhGAEs 氨基酸序列、22个海岛棉 GbGAEs 氨基酸序列以及6个拟南芥AtGAEs 氨基酸序列进行比对,采用邻接法构建系统发育树[20]。
1.5 四倍体棉花GAEs理化性质和亚细胞定位
GAE基因家族成员编码产物的氨基酸数量、分子量、等电点等理化性质分析由Prot-Param(http://web.expasy.org/protparam/)进行[23]。通过 Prot Comp9.0(http://www.softberry.com/berry.phtml?topic=protcom ppl&group=programs&subgroup=proloc)、CELLO v.2.5(http://cello.life.nctu.edu.tw/)对蛋白质进行亚细胞定位预测[24]。借助蛋白跨膜分析软件TMHMM(http://www.cbs.dut.dk/services/TMHMM-2.0)检测肽段的跨膜区。
1.6 GAE基因家族在棉纤维发育过程中的表达分析
本实验室前期完成了陆地棉品种邯优405、农大棉13 号、中棉所8 号、农大601 以及海岛棉品种Pima 90-53 和海7124 纤维发育7个时间点0、5、10、15、20、25、30 DPA (开花后天数,Days post-anthesis)的转录组测序,得到不同时间点的RPKM(Reads per kilo bases per million reads)。由于GAEs基因在优质陆地棉品种邯优405 和农大棉13 号、品质一般陆地棉品种中棉所8 号和农大601、优质海岛棉Pima90-53 和海7124 中的表达规律一致,因此本研究在进行基因表达分析时,先分别计算优质陆地棉品种邯优405 和农大棉13 号、品质一般陆地棉品种中棉所8 号和农大 601、优质海岛棉 Pima90-53 和海 7124 的RPKM 平均值,在三类平均值的基础上再进行log2(1+RPKM)处理。表达量热图的绘制借助GPS(http://hemi.biocuckoo.org/)完成。根据纤维发育各时期表达量的不同,将GAE家族进行分类,探究GAE家族成员在不同棉花品种中纤维发育不同时期的表达规律。
1.7 GAE家族成员优势表达基因分析
为了挖掘GAE家族成员中的优势表达基因,本研究将优质陆地棉与品质一般陆地棉、优质海岛棉与优质陆地棉的RPKM 值分别进行比较,探究不同棉花品种在各个时间点GAE表达量的差异。将优势表达基因的阈值定为1.5,即表达量差异在1.5 倍以上,并将优势表达基因标注在进化树中。
2 结果与分析
2.1 四倍体棉花GAE家族全基因组鉴定
借助 Smart和NCBI 中 Conserved Domains程序剔除不含SDR 保守结构域的序列,在CottonFGD 数据库中,从陆地棉和海岛棉最终分别鉴定出21个和22个GAE家族成员,按照它们在染色体上的位置顺序,分别命名为Gh-GAE01-21、GbGAE01-22(表1),其中GhGAE02、GhGAE14、GbGAE11和GbGAE20位于没有组装到染色体的大片段上。从基因在染色体上的分布来看,GAE基因家族成员在8 号染色体上分布最多,在陆地棉中A08和D08 均有 4个GhGAE基因; 在海岛棉中A08 和D08 分别有4个和6个GbGAE基因(图1)。

表1 四倍体棉花GAE 基因家族信息Table 1 Information of GAE gene family in tetraploid cotton
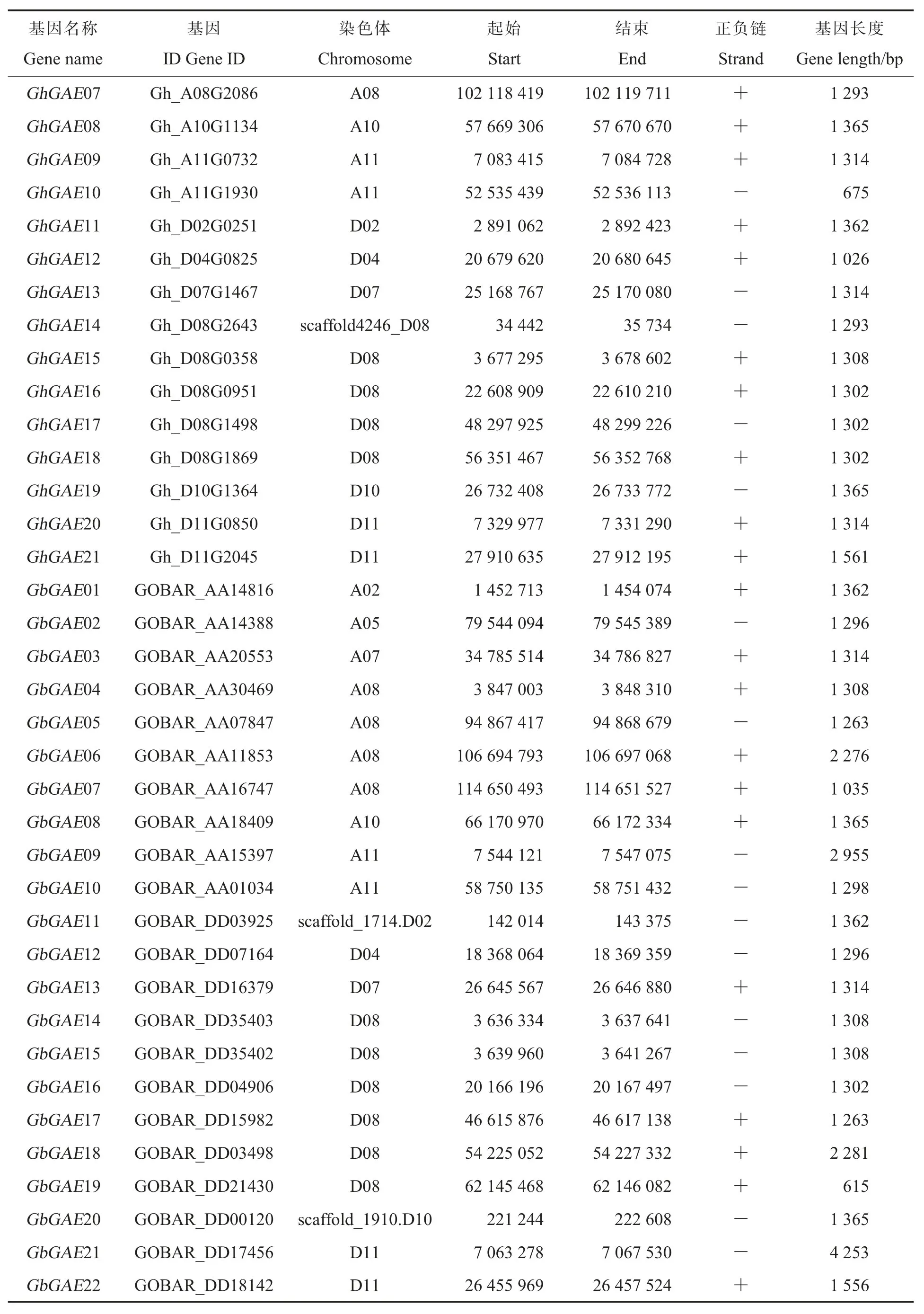
表1 (续)Table 1 (Continued)

图1 四倍体棉花GAE 基因家族的染色体定位Fig.1 Chromosome location of GAE gene family in tetraploid cotton
2.2 GAE家族基因结构分析
将鉴定出的 21个陆地棉GhGAEs和22个海岛棉GbGAEs基因序列构建系统进化树 (图2),可以分为3个亚组,Ⅰ、Ⅱ、Ⅲ亚组分别包含17、8和18个基因。Ⅰ亚组包含 8个GhGAEs和9个GbGAEs,Ⅱ亚组与Ⅲ亚组相同,GhGAEs和GbGAEs各占一半。基因结构分析显示(图2),陆地棉和海岛棉中同一基因位于相邻分支,亲缘关系较近。72%的GAEs基因没有内含子,这与拟南芥GAE 家族成员均不含内含子的情况相似。

图2 四倍体棉花GAE 基因家族进化树和基因结构Fig.2 Phylogenic tree and gene structure of GAE gene family in tetraploid cotton
2.3 四倍体棉花GAE氨基酸序列保守域分析
保守结构域分析表明,GAE家族结构域含有多个保守性的氨基酸残基,如T200、W318、W380、C425 等,部分保守性氨基酸残基组成GAE家族特有的保守基序(图3)。GAE家族存在4个保守基序(表2),第1个是 TDQPASLYAATKKAGEEIAHTYNHIYGLSLTGLRFFTVYGPWGRPDMAYF,整体表示为 TDQPASLYAATKKAGEEI[AT]HTYNHIYGLS[LI]TGLRFFTVYGPWGRPDMAYF(图3 中用细实线标出)。第2个保守基序是 FTHVMHLAAQAGVRYAMZNPGSYVHSNIAGFVNLLEVCKSANPQPAIVWA,整体表示为FTHV[ML]HLAAQAGVRYAM[EQ]NP[GQ]SYV HSNIAG[FL]V[NST]LLEV[CA]K[SA]ANPQPAIVWA(图3 中用粗实线标出)。第3个保守基序是MKLPRNGDVPFTHANISLAQRELGYKPTTDLQTGLKKFVRW,整体表示为[MV]K[LM]P[RG]NGDV[PQ]FTHAN[IV][ST]LAQ[RK][ED][LF]GYKP[TS]TDLQ[TA]GL[KR]KFV[RK]W(图3中用细虚线标出)。第4个保守基序是KGCL GALDTAEKSTGSGGKKKGPAQLRVYNLG,整体表示为KGCL [GA][AS]LDT [AS][EKG]KSTGSGGKK[KR]GPA[QP][LY]R[VI][YF]NLG(图3中用粗虚线标出)。21个GhGAEs和22个Gb-GAEs基因有4个相同的保守基序,存在共同的氨基酸残基,保守性较强;但基因间又存在一定的差异,这些差异可能是导致基因功能发生分化的主要原因。

表2 四倍体棉花GAE 基因家族保守基序Table 2 Conservative motifs of GAE gene family in tetraploid cotton
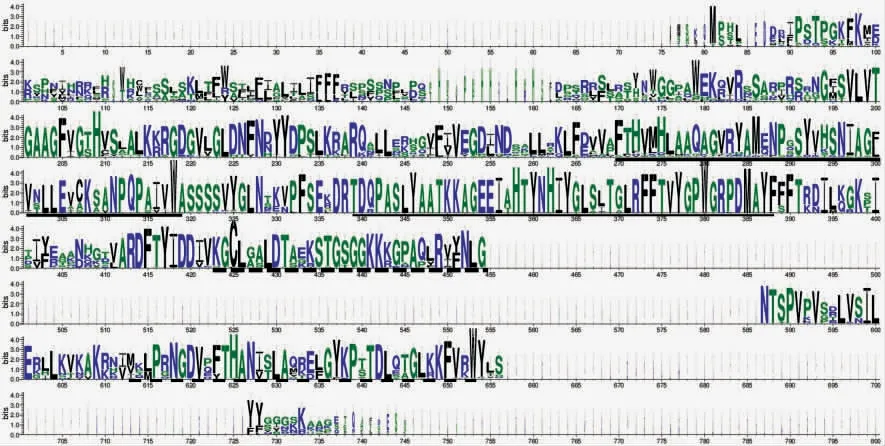
图3 四倍体棉花GAE 家族氨基酸序列保守性分析Fig.3 Conservation analysis of amino acid sequences of 43 GAE genes in tetraploid cotton
2.4 四倍体棉花与拟南芥的GAEs家族系统进化分析
陆地棉、海岛棉、拟南芥的GAEs构建系统进化树 (图 4),结果显示,AtGAE02、AtGAE03、AtGAE04、AtGAE05 位于同一亚组,而AtGAE01和AtGAE06 分别聚类到不同的亚组。前人研究证实,AtGAE01 和AtGAE06 在拟南芥花和角果中均高效表达[9],它们可能为果胶的合成提供前体物质[10,15]。棉纤维原始细胞突起始于开花当天,然后迅速进入伸长,伸长持续时间一般有24~28 d,此阶段决定了纤维细度[25],因此,我们推测与AtGAE01 和AtGAE06 聚类关系较近的陆地棉和海岛棉GAEs可能在棉纤维发育过程中发挥重要作用。

图4 拟南芥和四倍体棉花GAEs 基因家族进化树分析Fig.4 Phylogenic tree of the GAE gene family in Arabidopsis and tetraploid cotton
2.5 GAE基因家族的理化性质和亚细胞定位分析
蛋白理化性质分析表明(表3),陆地棉Gh-GAE家族成员编码的肽链平均由414个氨基酸组成,分子量介于24.87~50.32 kDa,平均45.93 kDa; 海岛棉GbGAEs编码的肽链平均为426个氨基酸,分子量介于22.55~62.53 kDa,平均为47.29 kDa,平均分子量较陆地棉偏大,且分子量分布范围较宽。GhGAEs家族成员理论等电点介于 9.02~10.34,平均值为 9.79;GbGAEs家族成员理论等电点为9.01~9.98,平均值为9.67。
陆地棉GhGAEs蛋白不稳定指数平均值为40.65,其中有 9个GhGAE成员为稳定蛋白(蛋白不稳定指数<40),其余的12个基因为不稳定蛋白(蛋白不稳定指数>40)。GhGAE基因家族成员的脂溶指数AI 都小于100,平均值为83.98,具有较高的脂溶性。在海岛棉中,蛋白不稳定指数平均值为39.60,有10个GbGAE成员为稳定蛋白,其余的12个基因为不稳定蛋白。GbGAEs平均脂溶指数为83.38,与陆地棉GhGAEs家族成员相同,均具有较高的脂溶性; 所有的Gh-GAEs和GbGAEs蛋白序列总平均亲水性(Grandaverage of hydropathy,GRAVY)都是负数,均表现为亲水性。

表3 四倍体棉花GAE 基因家族理化性质及亚细胞定位Table 3 Physicochemical properties and subcellular location of GAE proteins in tetraploid cotton
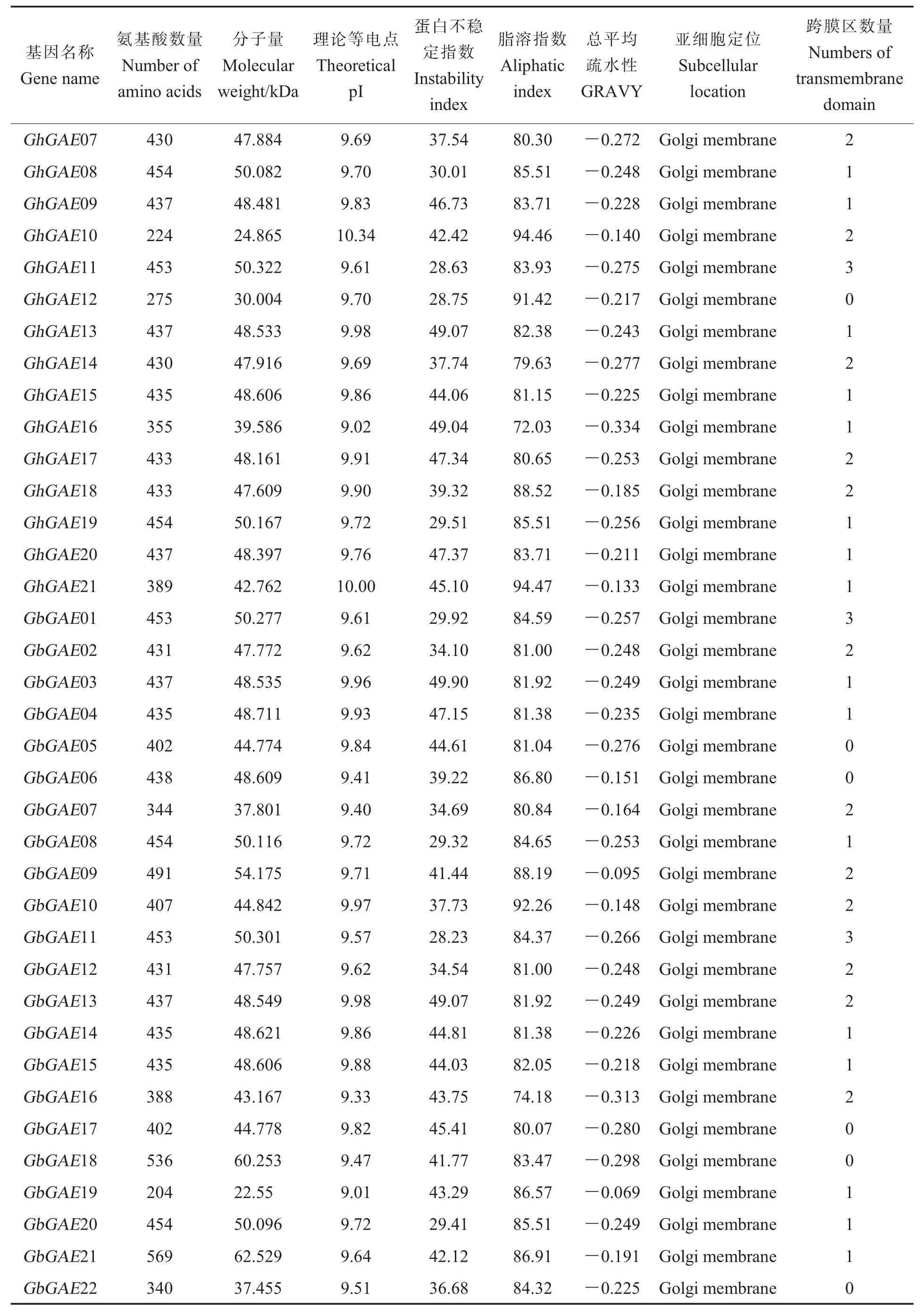
表3 (续)Table 3 (Continued)
对21个陆地棉GhGAEs、22个海岛棉Gb-GAEs家族成员蛋白进行亚细胞定位预测 (表3),显示所有GhGAEs和GbGAEs蛋白均定位在高尔基体膜(Golgi membrane)。蛋白跨膜区分析结果表明,除GhGAE12、GbGAE05、GbGAE06、GbGAE17、GbGAE18、GbGAE22没有检测到跨膜区外,GhGAE01、GhGAE11、GbGAE11 包含 3个跨膜区,其余家族成员均含有1~2个跨膜区。
2.6 GAE基因家族成员在棉纤维发育过程中的表达规律
根据本实验室提供的棉纤维发育转录组数据,对优质陆地棉品种邯优405 和农大棉13 号、品质一般的陆地棉品种中棉所8 号和农大601、优质海岛棉Pima90-53 和海7124 的RPKM 的平均值进行 log2(1+RPKM)处理,可以将GAE家族成员的表达分为:纤维发育起始期高表达、纤维伸长期高表达、次生壁增厚期高表达、全时期低表达 4 类表达模式。在陆地棉中,GhGAE02、Gh GAE03、GhGAE08、GhGAE09 等 8个GhGAEs在纤维起始期(0 DPA)高效表达,推测这些基因主要在纤维发育起始期发挥作用。其中,GhGAE02在纤维发育的整个时期表达量均较高,可能对棉纤维品质有重要影响 (图5 左-A)。在纤维伸长期,GhGAE01、GhGAE05、GhGAE06、GhGAE11等8个基因的表达峰值出现在 5、10、15、20 DPA 的某一时间点,且GhGAE01、GhGAE11 为优势表达基因,推测这类基因可能影响着纤维长度(图5左 -B)。在次生壁增厚期,GhGAE04、GhGAE14的表达峰值出现在25 DPA 或30 DPA,这两个基因可能影响棉纤维的强度(图5 左-C)。全时期低表达模式基因包括GhGAE07、GhGAE16、Gh-GAE17,这3个基因在各个时期的表达量都很低,推测这些基因在棉纤维发育过程中可能作用较小(图5 左 -D)。总体来看,GhGAEs在优质陆地棉与品质一般的陆地棉品种中的表达趋势一致。
在海岛棉中,GbGAE02、GbGAE03、GbGAE08等7个GbGAEs表达峰值出现在0 DPA,随着纤维发育表达量逐渐降低,其中GbGAE02 为优势表达基因,在纤维发育的整个时期表达量均较高,GbGAE02 可能对棉纤维品质的影响更大(图5 右 -A)。在纤维伸长期,GbGAE01、GbGAE06、GbGAE11 等8个基因表达量最高,推测这类基因可能影响着纤维长度,其中GbGAE01、Gb-GAE11 为优势表达基因,这两个基因可能对纤维的伸长作用更大(图5 右-B)。在次生壁增厚期,GbGAE04、GbGAE05、GbGAE19、GbGAE22 的表达峰值出现在25 DPA 或30 DPA,这3个基因可能影响棉纤维的强度 (图5 右 -C)。GbGAE07、GbGAE16、GbGAE17 在各个时期的表达量都很低,推测这些基因在棉纤维发育过程中可能作用较小(图5 右 -D)。
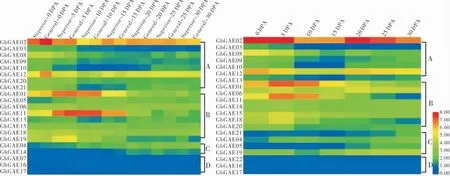
图5 GAE 基因家族表达量热图(左图为陆地棉,右图为海岛棉)Fig.5 The expression heatmaps of GAE gene family (The left is upland cotton,the right is sea island cotton)
GAE家族多数成员如GAE01、GAE02、GAE04 等在陆地棉和海岛棉中表达趋势一致,且在进化树中位于相邻分支,亲缘关系较近。
2.7 棉花纤维发育过程中优势表达的GAE家族成员
将优质陆地棉与品质一般的陆地棉、优质海岛棉与优质陆地棉分别进行比较,在进化树中把GAE家族成员中优势表达的基因用不同颜色的点进行标注(图6 左)。分析优质陆地棉(邯优405和农大棉13 号)与品质一般的陆地棉(中棉所8号和农大601)在纤维发育相同时间点GAE的表达差异 (图6 右),GhGAE10 在纤维伸长期优势表达;GhGAE11、GhGAE19 在次生壁增厚期优势表达;GhGAE08 在纤维伸长和次生壁增厚期均优势表达,推测这些基因在优质陆地棉中的优势表达,可能是引起优质陆地棉品种比品质一般的陆地棉品种纤维更长的重要基因。
分析优质海岛棉 (Pima90-53 和海 7124)与优质陆地棉(邯优405 和农大棉13 号)在纤维发育相同时间点GAE的表达差异,结果显示,GbGAE08、GbGAE10 在 5 DPA、10 DPA、20 DPA 优势表达;GbGAE06 在纤维伸长和次生壁增厚期均优势表达;GbGAE07 在纤维起始和次生壁增厚阶段优势表达,这些基因在海岛棉中表达量显著高于陆地棉,推测可能是引起海岛棉比陆地棉纤维长度、比强度、细度等优良的重要基因。

图6 GAE 家族成员优势表达基因Fig.6 The preferentially expressed genes of GAE family
3 讨论
本研究通过对陆地棉及海岛棉进行全基因组分析,从陆地棉和海岛棉中分别鉴定出21个GhGAE和22个GbGAE基因,均含有 SDR 保守结构域,所有GAE蛋白均定位于高尔基体膜。约72%的GAE家族成员不含有内含子,这与拟南芥AtGAE的结果相似。
本研究根据GAE基因家族的表达模式将其分为4 类,其中,GhGAEs在优质陆地棉与品质一般的陆地棉品种中的表达趋势一致。将优质陆地棉与品质一般的陆地棉、优质海岛棉与优质陆地棉分别进行比较,发现GhGAE08、GhGAE10、GbGAE06、GbGAE08、GbGAE10 在 纤 维 伸 长 期优势表达,伸长期主要进行纤维的极性伸长,推测可能影响棉纤维的长度;GhGAE08、Gh-GAE11、GhGAE19 在次生壁增厚期优势表达,该时期主要是纤维素的沉积,次生壁中的微原纤通过葡萄糖残基在纤维素侧链上的联结使纤维素聚合度增加,导致纤维胞壁加厚[26]。推测这Gh-GAE08、GhGAE11、GhGAE19 在优质陆地棉中的高效表达可能导致优质陆地棉更早地对细胞壁多糖网络进行调整,从而引起棉纤维品质的不同[27-28]。系统进化树结果显示,这些高效表达基因多数与AtGAE01 和AtGAE06 聚类关系较近,而AtGAE01 和AtGAE06 在拟南芥主要器官中均高效表达,它们可能为果胶的合成提供前体物质[10,15],暗示这些基因可能在棉纤维发育过程中发挥重要作用。目前对GAE基因家族在棉纤维发育过程中功能的了解还十分有限。因此,系统研究GAE基因家族可为棉纤维品质分子改良提供重要的候选基因。
4 结论
通过对陆地棉及海岛棉进行全基因组分析,鉴定出21个陆地棉GhGAE基因和22个海岛棉GbGAE基因,分为3个亚组,分布在 12 条染色体上。所有GAE蛋白均定位于高尔基体膜,约72%的GAE家族成员不含有内含子,根据GAEs在陆地棉、海岛棉纤维发育不同时期的表达变化,将其分为起始期高表达、纤维伸长期高表达、次生壁增厚期高表达、全时期低表达4 类模 式 。其中GhGAE01、GhGAE02、GhGAE11、GhGAE12 在陆地棉中高效表达,GbGAE01、Gb-GAE02、GbGAE11、GbGAE12在海岛棉中为高表达基因,推测可能在纤维发育过程中发挥着重要作用。通过对GAE基因进行全基因组的分析,为研究GAE基因家族在棉纤维发育中的功能提供了参考依据。
