33份薄壳山核桃品种种质资源的遗传多样性分析
2019-06-06焦思宇赵珞珞刘晓倩杜德意金继良聂传朋崔亚东
焦思宇,赵珞珞,刘晓倩,杜德意,金继良,聂传朋,崔亚东
(阜阳师范学院 生物与食品工程学院,安徽 阜阳 236037)
美国薄壳山核桃(Carya illinoensis)也叫长山核桃、美国山核桃,属胡桃科山核桃属,是一种优良的干果油料树种,也是木材和绿化环境树种[1]。其原产地是美国,我国于19世纪末20世纪初开始引种栽培。由于薄壳山核桃是品质优良的高档干果,亦是重要的木本油料植物[2],薄壳山核桃在我国表现出了重要的开发利用潜力,目前,安徽、浙江、云南、江苏等多个省份均把薄壳山核桃当作重要植物油料树种进行重点推广[3-5]。安徽省阜阳市林业科学技术推广站从2010年引进、发展薄壳山核桃至今,已建立薄壳山核桃基地6处,引种栽培美国薄壳山核桃面积近2万亩,取得了良好的经济效益和社会效益。但同时,目前存在薄壳山核桃生长管理粗放、树形紊乱、品种良莠不齐,大小年现象严重。所以,加快薄壳山核桃育种进程、丰富优良品种资源、建立丰产栽培技术体系,是实现薄壳山核桃规模化、产业化发展的首要任务。
ISSR(inter-simple sequence repeats),是简单重复序列区间标记技术,综合了RAPD和SSR技术的各种优点,具有信息量大、试验重复性好、多态性高、操作简单、DNA样品用量少、试验成本低[6],标记数量多、不受时空限制、快速稳定、共显性等优点而迅速发展,是一种非常理想的检测物种内遗传变异的分子标记[7],为品种鉴定和遗传多样性评价提供了新的途径[8]。目前薄壳山核桃分子标记方面的研究报道还比较少,Grauke等学者专家用SSR标记对薄壳山核桃进行遗传方面研究[9];李晖等学者专家[10]运用ISSR分子标记技术对安徽省种植的美国薄壳山核桃品种和株系进行遗传研究分析。我国选育的美国山核桃品种主要采用的是表型选择,容易导致不同产区的适生品种选择存在困难[11]。本研究运用ISSR分子标记,对阜阳市林业科学技术推广站引种栽培的薄壳山核桃资源圃内的33份核桃材料进行遗传多样性和亲缘关系分析,期望为新品种的选育和种质资源的开发提供理论依据。
1 材料、方法
1.1 供试材料
实验材料均取自阜阳市林业科学技术推广站薄壳山核桃种植基地。选取33个品种薄壳山核桃样品,各品种具体性状见表1。
1.2 方法
1.2.1 DNA提取
采集薄壳山核桃幼叶作为提取基因组DNA的供试材料,置于密封袋中,做好标记,存放于低温冰箱中保存、备用。DNA提取采用试剂盒提取法,试剂盒购自(宝生物工程(大连)有限公司,型号Code No.9768),提取方法参照试剂盒说明书。

表1 供试ISSR分子标记薄壳山核桃品种信息
1.2.2 ISSR筛选
所使用引物从加拿大哥伦比亚大学公布的100条ISSR序列中选择20条进行引物合成[12](上海生工生物工程公司合成),用1~10号山核桃样品的DNA模板进行PCR扩增,通过电泳分析进行引物筛选。经过10种样品与20种引物PCR的交叉重复实验比较分析,从20条引物中选取出特异性强、稳定性较好、带型清晰、具多态性的8条引物(UBC844、UBC845、UBC846、UBC825、UBC834、UBC835、UBC840 和 UBC811)用 于PCR扩增。
1.2.3 PCR扩增反应、电泳检测
查阅文献资料[13-14],反复对比多个ISSR扩增程序,电泳检测扩增条带的有无和清晰度,选取最合适的反应体系和反应程序。
PCR 反应体系 20 μL:PCR mix 10 μL(上海生工生物工程公司)、primer 1.0 μL(10 μmol·L-1),ddH2O 8 μL,DNA 模板 1 μL(60 ng)。
PCR扩增程序:94℃预变性4 min,(94℃变性 30 s,50~53 ℃退火 45 s,72 ℃延伸 35 s)35 个循环,72℃延伸10 min,最后4℃保存。
ISSR引物扩增的产物于预先加入EB的1.5%琼脂糖凝胶电泳,电泳结束后于凝胶成像系统成像拍照。ISSR为显性标记,扩增产物的统计条带按照清晰可重复为原则,同一引物扩增的多个产物中电泳迁移率一致的条带被称为具有同源性[15],以DNA Marker2000为相对分子量标准,依据PCR扩增产物的电泳结果,在某个相同迁移率位点上有DNA条带的记为“1”,无DNA条带的记为“0”,统计形成以“1”和“0”组成的分子标记表型数据矩阵。
1.2.4 数据统计、分析
用多样性分析软件Popgene 1.32软件[16]对ISSR扩增结果进行遗传分析,在假定种群处于Hardy-Weinberg平衡状态下,分别计算多态位点百分率(PPB)、Shannon信息指数(I)、Nei's基因多样性指数(He)、观察等位基因数(Na)、有效等位基因(Ne)。依照Nei's遗传距离,运用NTSYS-pc 2.1软件[17-21]对各类群进行UPGMA聚类分析。
2 结果
2.1 ISSR-PCR扩增结果
选用8条ISSR引物由33个美国山核桃品种的总基因组DNA为模板进行PCR扩增实验,电泳结束后,一共检测到清晰可重复的DNA片段58条,每个引物平均扩增7.3条,所有片段长度在150~2 000 bp之间。在58个位点中45个位点的片段有多态性,平均多态性位点百分率(PPB)是77.59%见表2。图1为引物ISSR 811扩增后的电泳图谱部分凝胶电泳结果。
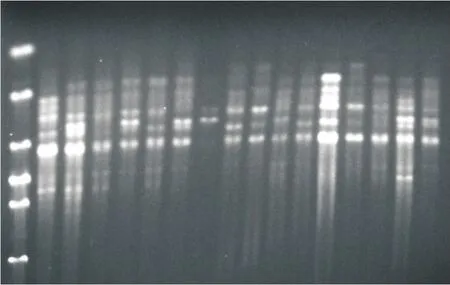
图1 引物ISSR 811部分扩增图(自左至右为样品1~16)
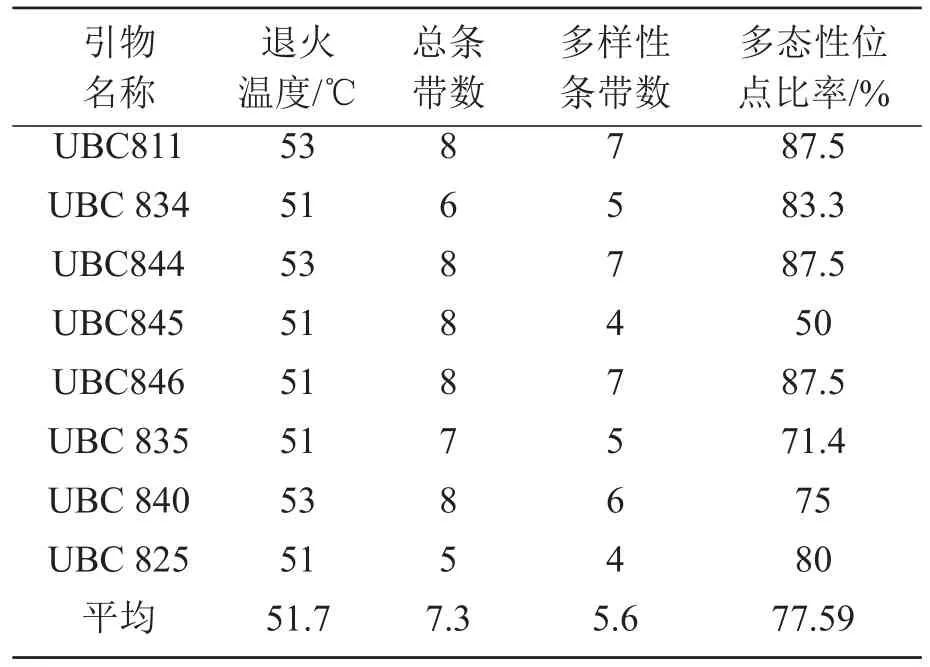
表2 8条ISSR引物扩增产物统计结果
2.2 薄壳山核桃品种遗传多样性水平分析
运用Popgene软件对33个供试材料的薄壳山核桃进行遗传分析,得出结果总结如表3。多态性位点百分率(PPB)为77.59%,有效等位基因数(Ne)为 1.511 3,Shannon 多样性信息指数(I)为0.440 7,Nei's基因多样性指数(H)为 0.298 2。结果表明薄壳山核桃遗传多样性水平比较高。
2.3 山核桃不同品种间的遗传相似系数分析
以33个薄壳山核桃品种和58个位点的谱带数为原始矩阵,对供试材料实验所出现的清晰条带进行赋值,有条带的DNA片段赋值为1,无条带的DNA片段赋值为0,构建ISSR分子标记的“0”、“1”矩阵数据。
使用NTSYS软件进行了DNA电泳条带数据分析,计算出33个供试材料的阜阳地区薄壳山核桃品种的遗传相似系数,运用非加权组平均法(UPGMA)对美国山核桃进行聚类分析得到树状图(见图2)。结果表明,33份阜阳地区引种栽培的薄壳山核桃品种之间遗传多样性丰富,不同种质之间遗传信息差异甚大。
3 讨论
3.1 PCR反应体系优化和退火温度选择
PCR反应体系主要与PCR mix、DNA模板量和引物浓度有关系。PCR反应程序与变性时间,退火温度,退火时间,延伸时间有关系。本文通过试验确定了优化的PCR反应体系和较佳的退火温度,见表2。
3.2 薄壳山核桃品种资源在我国的现况
美国山核桃品种繁多,许多品种在性状上表现的差异性又很小。美国山核桃在我国具有重要的开发和利用市场,可是目前普遍存在品种分类混乱,杂交技术不成熟,干果品质一般,产量效益不稳定等各种问题。所以只有加快薄壳山核桃育种进程、进一步对美国山核桃进行准确分类、探究山核桃遗传多样性、丰富优良品种资源、建立丰产栽培技术体系,才能实现美国山核桃规模化、产业化发展,加快美国山核桃在我国市场的发展。从本研究结果可以看出,通过ISSR分子标记研究薄壳山核桃遗传多样性是可行的,可以为薄壳山核桃的品种鉴定和分类提供准确的参考资料。
3.3 构建ISSR标记图谱对薄壳山核桃品种鉴定的优势
ISSR标记试验重复性好、多态性高、操作简单,并且试验成本低、不受时空限制、快速稳定,是目前非常理想的研究物种遗传多样性的有效手段,对品种鉴定和遗传多样性评价都具有非常重要的指导意义。传统的品种鉴定大多使用形态标记的方法,鉴定周期长,误差大,对于像美国山核桃这种性状差异小的品种鉴定更加困难,因此ISSR分子标记图谱对美国山核桃品种鉴定,优良育种等都具有重要的指导意义。

表3 33种薄壳山核桃遗传多样性

图2 33个薄壳山核桃品种的遗传多样性树状图
4 结论
本实验运用ISSR分子标记技术分析阜阳市林业局地区的部分美国山核桃品种的遗传关系。得到的一系列数据计算出33个美国山核桃品种多态位点百分率达到77.59%,多态性较好;多样性系数为0.298 2,遗传多样性水平较高。接着运用UPGMA法聚类分析,结果分析得出以下结论:33个薄壳山核桃品种在相似系数0.68%的水平上聚为A、B、C、D、E 5大类,D包括的品种最多共11个,E最少包括Gloria Grande品种一种,表明Gloria Grande与其他几种薄壳山核桃遗传距离最远。A类包括1号,2号,3号,5号,15号,20号,21号,24号和28号,其中24号赛甫路斯和28号金华1号相似性非常高;B类包括10号,25号,12号,19号和14号;C类包括4号,7号,17号和32号;D类包括6号,13号,26号,29号,16号,9号,33号,27号,30号,31号,22号,其中13号和26号相似性非常高;E类仅包括一个8号品种。
5 小结
分析表明亚林系列遗传差异性比较大,其中亚林21号和亚林5号遗传距离较近,亚林11号和亚林64号遗传距离较近,安农系列遗传差异性较小,Gloria Grande品种与其他品种遗传距离最远。通过ISSR分子标记来探讨薄壳山核桃遗传多样性是切实可行的,可为薄壳山核桃品种的鉴定与分类提供较准确的参考资料,对薄壳山核桃杂交育种具有重要参考价值。本研究所用材料仅为阜阳地区栽培薄壳山核桃部分品种,不能代表薄壳山核桃的全部遗传背景,有关薄壳山核桃的遗传多样性还需进一步研究。
