猪乳头性状生理学和遗传学研究进展
2019-05-21周李生赵为民涂枫吴云鹤任守文方晓敏
周李生,赵为民,涂枫,吴云鹤,任守文,方晓敏
猪乳头性状生理学和遗传学研究进展
周李生,赵为民,涂枫,吴云鹤,任守文,方晓敏
江苏省农业科学院畜牧研究所,江苏省农业种质资源保护与利用平台,南京 210014
猪乳头性状属于繁殖性状,直接影响母猪哺乳率和仔猪成活率,是种猪遗传改良的重要指标,揭示其遗传机理对提高种猪繁殖性能具有重要意义。猪乳头性状复杂多样,包括乳头数、乳头类型、乳头位置分布和波动不对称性现象等,但其遗传机制尚不清晰。本文对猪乳头及乳腺的形成与发育过程、猪乳头性状调控基因数量性状位点(quantitative trait locus, QTL)定位和候选基因等生理学和遗传学方面的研究进展进行了综述,旨在为今后鉴别影响猪乳头性状的因果突变和主效基因,揭示猪乳头在胚胎发育过程中产生数目、类型和位置差异的复杂遗传机制提供新的视角。
猪;乳头性状;遗传研究
种猪的繁殖性能直接影响养猪生产效益,是品种改良工作中的重要方向,主要包括排卵率、乳头性状、产仔数和断奶活仔数等。其中,乳头性状是衡量母猪哺乳能力的重要指标,具有较多有效乳头数的母猪可以哺乳更多的仔猪,有效乳头数少于产仔数会严重影响仔猪的成活率。同时,乳头数与产仔数也有一定程度的相关性[1],多乳头数和高产仔数的有效结合是实现母猪高繁殖能力的生理基础。
猪乳头数作为具有中等遗传力(约0.3)的数量性状,利用传统育种技术很难在短期内获得较为明显的遗传进展[2]。分子育种技术能够有效克服传统育种的缺点,提高育种效率。然而,猪乳头数在遗传上可能受到多个基因的调控,目前人们对于控制猪乳头数性状的主效基因尚不清晰,也缺乏具有育种价值的分子标记。本文综述了猪乳头在胚胎时期的形成过程、乳头性状表型研究的复杂性以及乳头性状调控基因的遗传定位结果,旨在为今后发掘影响猪乳头性状的因果突变和主效基因,实现猪繁殖性状遗传改良基础理论和技术方法的创新提供参考。
1 猪乳头及乳腺组织在胚胎期的发育
哺乳动物乳头和乳腺在胚胎期的形成和发育过程基本一致,共分为4个阶段[3]:(1)形成乳腺线:在约23天猪胚胎期,由腹部两侧的柱状和多层次外胚层细胞形成乳腺线;(2)形成乳腺原基:在乳腺线形成后的24~36 h内,即26天左右的猪胚胎期,乳腺线逐步特化形成清晰可见的乳腺原基,该过程是决定猪乳头数目的关键发育阶段;(3)形成腺芽:乳腺基板扩增并向下面间质层凹陷为芽状结构形成腺芽;(4)形成原始乳腺导管分支:腺芽上的上皮细胞分化为乳头皮肤,腺芽下的上皮细胞向下分支并伸入到脂肪板中,形成原始乳腺导管分支,组成初级乳腺。至此,乳头及乳腺组织在胚胎期的发育基本完成,它们将在出生后的青春期和妊娠期中进一步发育,最终形成在哺乳期能够产生哺乳功能的组织器官。
乳头及乳腺发育是在众多信号通路共同调控下完成的,目前已知的信号通路包括:(1)影响乳腺线特化和乳腺原基大小的Wnt信号通路[4],引发乳腺线的分化并影响乳腺原基的大小,、和是该过程中的重要候选基因;(2)与Wnt信号通路协同调控乳腺线形成的TBX3信号通路[5,6],基因敲除的胚胎无法正常形成腺芽,且和基因表达也缺失;(3)调控乳腺原基形成的FGF信号通路[7],该通路调控乳腺细胞的形成。对基因敲除小鼠的研究显示,和基因缺失可导致乳腺原基的形成受阻[6];(4)影响乳腺数量和位置的EGFR家族通路[8],该通路中的基因及其受体基因(和)在乳腺线相应的位置细胞中表达,对乳腺的形成具有重要的调控作用;(5)调节胚胎期乳腺腺泡发育的Notch/RBP-J信号通路[9]。对小鼠乳腺祖细胞的研究发现,Notch/RBP-J信号通路中的和基因可调节乳腺泡形成;(6)调节胚胎期乳腺腺芽形成和发育的p190- BRhoGAP/IGF通路[10],该通路中基因通过p190-B和IRS蛋白发挥相应的调控作用。上述研究表明:信号通路及其包含的基因间的相互作用,不仅可调控乳头和乳腺的形成数目与功能学发育状态,也可以调控其形态学发生的位置。然而,多数乳头及乳腺在胚胎时期的发育学研究是利用小鼠等小型模式动物完成,目前在猪等大型哺乳动物中的相关研究尚少。
2 猪乳头性状的复杂性
猪乳头性状复杂多样,主要体现在4个方面:乳头数量在不同品种及同一品种的不同个体间存在变异、乳头类型多样、乳头在腹部的分布具有区域性和不对称性。
2.1 猪乳头数存在变异
根据《中国畜禽遗传资源志-猪志》和已报道的文献公布的数据(表1),发现猪乳头数存在品种间及品种内的差异,并与其繁殖性能相适应。繁殖性能最强的江海型猪乳头数最多,二花脸猪的乳头数可多达22个。大体型的北方猪种乳头数略多于小体型的南方猪种,繁殖力较弱的滇南小耳猪等南方小型猪的乳头数少至10个。二花脸猪、巴马香猪、五指山猪等品种内的乳头数存在较大变异。大白猪等西方商品猪的乳头数多为12或14个,相对固定。这些数据表明:相对于西方商品猪种,中国地方猪种乳头数的遗传多样性更加丰富;中国地方猪种对乳头数的选育程度较弱,品种内乳头数的一致性不强。

表1 中西方部分猪种乳头数分布
2.2 猪乳头存在不同类型
依据猪乳头形态结构的完整程度和生理功能的正常程度,可将其分为正常乳头、翻乳头(又称火山乳头)和副乳头(又称瞎乳头) 3大类型[11]。成年猪的正常乳头凸起完整,乳头体较明显,顶端具有明显开口,具备正常的泌乳和哺乳功能。翻乳头乳头体不明显(不完全翻乳头)或者没有(完全翻乳头),乳头管存在发育缺陷,乳腺与正常乳头的一致,具有泌乳功能,但由于乳头体缺失,导致仔猪吸取乳汁困难,难以实现哺乳功能。副乳头仅在猪体表面上呈现为一个小的凸起,无乳房膨大部,乳腺组织发育不全,基本无泌乳功能。翻乳头和副乳头统称为功能缺陷型乳头,可能是乳头基部结缔组织和乳腺组织在发育的某些阶段受遗传或环境因素的干扰所致。
2.3 猪乳头分布具有区域性
2012年,Nikitin等[12]分析了5个家猪群体(约20 000个体)的乳头表型数据,发现猪乳头在腹部的分布具有一定的规律性。研究发现,猪乳头在腹部的分布可分为10个区域,每个区域包含有1对乳头(图1)。其中,区域1、3、7和8为固定乳头区,即该区域有且仅有1对正常乳头;区域2、4、5、6、9和10为可变乳头区,该区域可能含有1对、1个或者没有乳头。功能缺陷的副乳头大多分布于区域4和5,肛门乳头位于接近尾部的区域9和10,大多功能异常,其他区域的乳头基本为正常乳头。同一区域中2个乳头的乳头类型不一定相同;若某区域仅有1个乳头,则其分布于左侧或右侧的概率基本一致。
上述研究表明:猪乳头数的变化主要受可变乳头区影响,在不同的猪种甚至同一品种的不同个体中,可变乳头区中的乳头数变化直接影响总乳头数。由于乳头数的分布具有上述的严格分区特性,故推测在不同区域中,影响其乳头发育的主效基因及因果位点可能不同。因此在遗传学分析中,建议按照分区原则,准确记录猪乳头性状表型数据,以便后续分析。
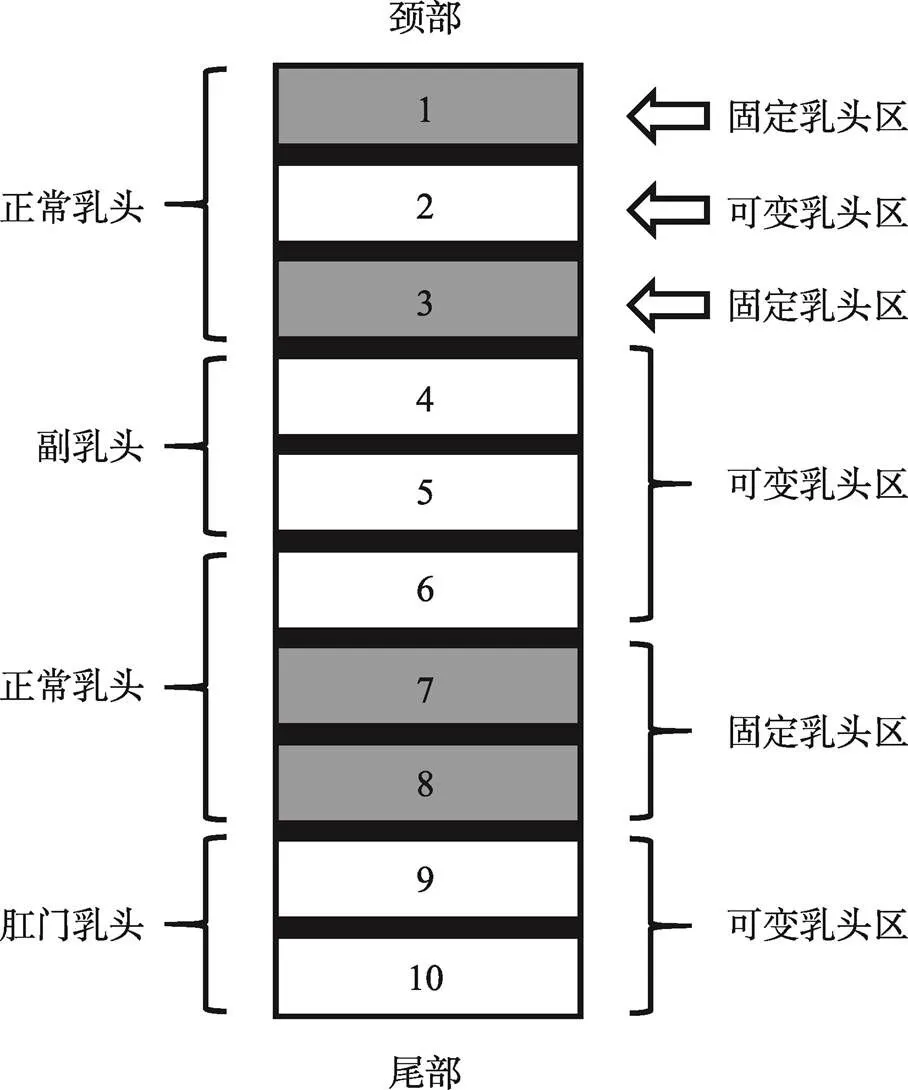
图1 猪乳头在腹部的分布示意图
根据文献[12]修改绘制。
2.4 猪乳头存在波动不对称性现象
大多数情况下,猪左右乳头数及位置分布是对称的(图2A),但也存在左右乳头数不一致和乳头排列不对称的情况,即轴不对称现象[13](图2B)。目前对于该现象的遗传学研究较少,有研究认为是由于非特定的环境因素所导致的随机性“摆动”[14],但该论点尚缺乏有力的证据,需要进一步的深入研究。
3 猪乳头性状的QTL定位
目前,国内外多个研究组对猪乳头性状开展了数量性状位点(quantitative trait locus, QTL)定位研究。据国际猪QTL数据库(http://www.animalgenome. org/cgi-bin/QTLdb/SS/index)最新数据显示,共有35篇文献报道了影响猪乳头性状的QTL。其中,基于低密度微卫星标记构建连锁图谱进行QTL定位的文献为22篇,基于高密度SNP芯片进行全基因组关联分析(genome-wide association study, GWAS)的文献有11篇,基于全基因组重测序基因型填充数据(imputed whole-genome SNPs)进行全基因组关联分析的文献有2篇。

图2 猪乳头左右分布情况
3.1 猪乳头数QTL定位
针对乳头数性状的QTL定位研究较多,如:2000年Rohrer等[15]最先报道利用157个微卫星标记在梅山×大白资源群体SSC1、SSC3和SSC10染色体上定位到影响总乳头数的QTL;Geldermann等[16]通过构建梅山×皮特兰×野猪资源家系,在SSC1、SSC2、SSC5、SSC8、SSC10、SSC12和SSCX染色体上定位到影响总乳头数的QTL;Ding等[17]在二花脸×白色杜洛克资源家系中分别定位到影响总乳头数(SSC1、SSC3、SSC4、SSC5、SSC6、SSC7、SSC8和SSC12)、左乳头数(SSC1、SSC3、SSC4、SSC5、SSC6、SSC7、SSC8和SSC12)和右乳头数(SSC1、SSC3、SSC4、SSC5、SSC7和SSC8)的QTL;2014年,Lopes[18]首次报道利用长白猪高密度基因分型芯片数据,通过GWAS分析在SSC4、SSC6、SSC7和SSC12染色体上检测到与总乳头数显著关联位点;2017年Tan等[19]率先使用全基因组测序基因分型技术(genotyping-by-sequencing, GBS),对2936头杜洛克猪群体进行GWAS分析,分别在SSC1、SSC6、SSC7、SSC10、SSC11、SSC12和SSC14染色体上鉴别到显著影响猪乳头数的关联位点。
值得肯定的是:随着标记密度和群体规模的提高以及分析手段的不断改进,鉴别到影响猪乳头数QTL的数量与准确性也大大提高。然而,上述研究主要集中在梅山/二花脸猪×西方商品猪构建的资源群体和西方商品猪纯种群体,而对中国地方猪纯种群体和含中国地方猪血缘的培育品种的研究则相对较少。
3.2 猪功能缺陷乳头QTL定位研究
关于功能缺陷乳头的遗传定位研究较少:Demeure等[20]利用梅山×大白/长白群体在SSC7染色体上鉴别到影响功能缺陷乳头的QTL;Sato等[21]在梅山×杜洛克资源群体中SSC2和SSC16染色体上检测到影响功能缺陷乳头的QTL;Jonas等[22]在杜洛克×柏林微型猪资源家系中发现,在SSC3、SSC4、SSC6和SSC11染色体上有影响功能缺陷乳头的QTL。上述研究结果表明:不同实验群体所鉴别的功能缺陷乳头QTL定位结果存在较大的差异,表明其遗传机理比较复杂,可能存在多个遗传位点,需深入研究。
4 猪乳头性状候选基因研究
目前,猪乳头性状的候选基因研究主要集中于乳头数和功能缺陷乳头性状。瘦素(protein hormone leptin, LEP)主要由脂肪细胞产生和分泌,主要功能是通过下丘脑产生饱足信号,影响能量摄入来调控体重平衡[23]。然而,Peixoto等[24]对大白×长白×皮特兰F2资源家系的研究发现:基因上的突变C798T与总乳头数(<0.02)、左乳头数(<0.03)也存在显著关联,该基因可作为猪乳头数选育的潜在分子标记。Dragos-Wendrich等[25]对大白×梅山F2资源家系QTL定位结果显示,位于SSC10染色体上的基因是总乳头数的位置候选基因,该基因属于醛酮还原酶家族,可通过催化酮甾体和羟基甾体的还原和氧化,将雄激素、雌激素、孕激素和糖皮质激素相互转化为相对更强的对应物,参与生长发育和生殖等多种生理过程[26]。Martınez-Giner等[27]在含梅山猪血缘的资源群体中,对基因的全长cDNA测序,发现存在2个与乳头数存在显著关联的SNP(<0.03),并且基因在仔猪的乳腺组织中高度表达,提示该基因可能在胚胎发育过程中影响乳头的形成。Jin等[28]在大白×韩国地方猪资源家系中鉴别到SSC7染色体上显著影响猪乳头数的QTL,并确定为该QTL最有可能的位置候选基因,同时检测到该基因上存在2个和总乳头数极显著相关的SNPs,分别为g.13,050A>G(= 6.38E-05)和SNP c.829A>T(= 1.06E-07)。基因是TCF (T-cell specific factor)家族成员之一,在胚胎早期发育过程中调控乳头和乳腺的形态发生。Xu等[29]发现该基因上的2个突变99514A>G和119846C> T在关中黑猪、汉江黑猪、八眉猪和大白猪4个群体中都与总乳头数显著相关(<0.05)。Duijvesteijn等[30]和Rohrer等[31]研究发现,乳头数和脊椎数的遗传机制存在一定的相关性,、、、、和等与脊椎发育相关的基因也是影响乳头数发育的强候选基因。Yang等[32]研究表明,基因上影响脊椎数的因果突变(g.20311_ 20312ins291)与猪乳头数显著关联。
针对功能缺陷乳头,Tetzlaff等[33]在前期QTL定位的基础上[22],发现位置候选基因上的突变C1819T与翻乳头性状显著关联。Chomwisarutkun等[34,35]利用基因表达芯片检测成年猪的正常乳头与翻乳头的上皮组织和间充质组织,分别发现62个和24个显著差异表达基因,它们与细胞增殖、器官与组织发育和核酸修饰等代谢过程紧密相关。
到目前为止,还未鉴别到影响猪乳头性状的主效基因和因果突变,可能有以下4点原因:(1)乳头性状复杂多样,在实际研究中对乳头性状的表型区分不够细化,从而造成阴性结果,影响不同乳头性状的因果突变和主效基因不尽相同;(2)前期研究主要集中在纯种猪群或有限世代的资源家系,群体中连锁不平衡(linkage disequilibrium, LD)程度较高,从而无法对鉴别到的QTL进行精细定位和深入解析;(3)定位获得的强关联位点大部分位于基因组上的非编码区域,可能为调控突变,给精确鉴别因果突变和目的基因带来极大的挑战;(4)未探究影响猪乳头数形成的关键组织、细胞及时间节点,无法合理地利用转录组学、蛋白组学等技术手段,无法通过多维组学分析准确锚定目的基因。因此,未来只有利用合适的实验材料、精准的表型记录和更先进的研究策略,才可能为猪乳头性状的遗传机制解析带来新的突破。
5 结语与展望
猪乳头性状是种猪选育中重要的参考指标之一,从遗传水平上解析猪乳头数变异和乳头缺陷发生的机理,能够更有效地开展优良种猪的选育工作。同时,有研究显示人类中也存在多乳头和乳头缺陷等遗传疾病[36,37]。由于猪在生理结构和代谢机制上与人类高度相似,对猪乳头性状的遗传解析有助于其成为人类乳头疾病的模式动物模型,可以在未来的医学研究中发挥一定的作用。然而,国内外科研人员利用不同的品种开展QTL定位并搜寻其主效基因的结果并不理想,仍需要进一步的探索。
随着测序技术的发展和测序成本的降低,研究人员可以利用全基因组重测序及基因型填充手段,提高检测标记密度,最大程度地利用实验群体中存在的重组事件,并与其他组学数据(转录组学、蛋白质组学、代谢组学等)整合分析,结合跨物种的保守性进化分析及详细的基因组注释信息,以达到迅速鉴别因果突变和主效基因的目的[38]。因此,今后应在科学细致的表型记录基础上,准确把握猪乳头数胚胎发育时期产生数目差异的关键组织与时间节点,并借鉴QTL解析的最新策略,深入挖掘影响猪乳头数的因果位点和主效基因,为选育含有较多乳头数且乳头无缺陷的种猪提供理论依据。
[1] Verardo LL, Silva FF, Lopes MS, Madsen O, Bastiaansen JW, Knol EF, Kelly M, Varona L, Lopes PS, Guimarães SE. Revealing new candidate genes for reproductive traits in pigs: combining bayesian GWAS and functional pathways., 2016, 48: 9.
[2] Felleki M, Lundeheim N. Genetic heteroscedasticity of teat count in pigs., 2015, 132(5): 392– 398.
[3] Robinson GW. Cooperation of signalling pathways in embryonic mammary gland development., 2007, 8(12): 963–972.
[4] Roarty K, Serra R. Wnt5a is required for proper mammary gland development and TGF-β-mediated inhibition of ductal growth., 2007, 134(21): 3929–3939.
[5] Eblaghie MC, Song SJ, Kim JY, Akita K, Tickle C, Jung HS. Interactions between FGF and Wnt signals and Tbx3 gene expression in mammary gland initiation in mouse embryos., 2004, 205(1): 1–13.
[6] Veltmaat JM, Relaix F, Le LT, Kratochwil K, Sala FG, van Veelen W, Rice R, Spencer-Dene B, Mailleux AA, Rice DP. Gli3-mediated somitic Fgf10 expression gradients are required for the induction and patterning of mammary epithelium along the embryonic axes., 2006, 133(12): 2325–2335.
[7] Mailleux AA, Savona-Baron B, Ndiaye D, Savona-Baron C, Itoh N, Kato S, Dickson C, Thiery JP, Bellusci S. Role of FGF10/FGFR2b signaling during mammary gland development in the mouse embryo., 2002, 129(1): 53–60.
[8] Howard B, Panchal H, McCarthy A, Ashworth A. Identification of the scaramanga gene implicates Neuregulin3 in mammary gland specification., 2005, 19(17): 2078–2090.
[9] Buono KD, Robinson GW, Martin C, Shi S, Stanley P, Tanigaki K, Honjo T, Hennighausen L. The canonical Notch/RBP-J signaling pathway controls the balance of cell lineages in mammary epithelium during pregnancy., 2006, 293(2): 565–580.
[10] Heckman BM, Chakravarty G, Vargo-Gogola T, Gonzales- Rimbau M, Hadsell DL, Lee AV, Settleman J, Rosen JM. Crosstalk between the p190-B RhoGAP and IGF signaling pathways is required for embryonic mammary bud development., 2007, 309(1): 137–149.
[11] Yang ZC, Zhou ZX, Li Z, Dong YZ. Anatomical and histological characteristics of in-verted papilla of pig., 1999, 30(6): 519–524.杨志春, 周忠孝, 李照, 董玉珍. 猪翻乳头的解剖组织学特征的研究与探讨. 畜牧兽医学报, 1999, 30(6): 519– 524.
[12] Nikitin SV, Kniazev SP, Ermolaev VI. Model of genetic control of the number and location of nipples in domestic pig., 2012, 48(11): 1128–1140.
[13] Fernández A, Toro M, Rodríguez C, Silió L. Heterosis and epistasis for teat number and fluctuating asymmetry in crosses between Jiaxing and Iberian pigs., 2004, 93(2): 222–227.
[14] Liu WZ, Zhou ZX. Inheritance and influencing factors of papillary traits in pigs., 1994, (4): 33–34.刘文忠, 周忠孝. 猪乳头性状的遗传及其影响因素. 养猪, 1994, (4): 33–34.
[15] Rohrer GA. Identification of quantitative trait loci affecting birth characters and accumulation of backfat and weight in a Meishan-White composite resource population., 2000, 78(10): 2547–2553.
[16] Geldermann H, Müller E, Moser G, Reiner G, Bartenschlager H, Cepica S, Stratil A, Kuryl J, Moran C, Davoli R, Brunsch C. Genome-wide linkage and QTL mapping in porcine F2 families generated from pietrain, meishan and wild boar crosses., 2003, 120(6): 363–393.
[17] Ding NS, Guo YM, Knorr C, Ma JW, Mao HR, Lan LT, Xiao SJ, Ai HS, Haley CS, Brenig B, Huang LS. Genome-wide QTL mapping for three traits related to teat number in a white duroc x erhualian pig resource population., 2009, 10: 6.
[18] Lopes MS, Bastiaansen JW, Harlizius B, Knol EF, Bovenhuis H. A genome-wide association study reveals dominance effects on number of teats in pigs., 2014, 9(8): e105867.
[19] Tan C, Wu Z, Ren J, Huang Z, Liu D, He X, Prakapenka D, Zhang R, Li N, Da Y, Hu X. Genome-wide association study and accuracy of genomic prediction for teat number in Duroc pigs using genotyping-by-sequencing., 2017, 49(1): 35.
[20] Demeure O, Sanchez MP, Riquet J, Iannuccelli N, Demars J, Fève K, Kernaleguen L, Gogué J, Billon Y, Caritez JC, Milan D, Bidanel JP. Exclusion of the swine leukocyte antigens as candidate region and reduction of the position interval for the Sus scrofa chromosome 7 QTL affecting growth and fatness., 2005, 83(9): 1979–1987.
[21] Sato S, Atsuji K, Saito N, Okitsu M, Sato S, Komatsuda A, Mitsuhashi T, Nirasawa K, Hayashi T, Sugimoto Y, Kobayashi E. Identification of quantitative trait loci affecting corpora lutea and number of teats in a meishan x duroc F2 resource population., 2006, 84(11): 2895–2901.
[22] Jonas E, Schreinemachers HJ, Kleinwächter T, Un C, Oltmanns I, Tetzlaff S, Jennen D, Tesfaye D, Ponsuksili S, Murani E, Juengst H, Tholen E, Schellander K, Wimmers K. QTL for the heritable inverted teat defect in pigs., 2008, 19(2): 127–138.
[23] Barb CR, Hausman GJ, Houseknecht KL. Biology of leptin in the pig., 2001, 21(4): 297–317.
[24] de Oliveira Peixoto J, Facioni Guimarães SE, Sávio Lopes P, Menck Soares MA, Vieira Pires A, Gualberto Barbosa MV, de Almeida Torres R, de Almeida E Silva M. Associations of leptin gene polymorphisms with production traits in pigs., 2006, 123(6): 378–383.
[25] Dragos-Wendrich M, Moser G, Bartenschlager H, Reiner G, Geldermann H. Linkage and QTL mapping for Sus scrofa chromosome 10., 2003, 120(s1): 82– 88.
[26] Bauman DR, Steckelbroeck S, Penning TM. The roles of aldo-keto reductases in steroid hormone action., 2004, 17(9): 563–578.
[27] Nonneman DJ, Wise TH, Ford JJ, Kuehn LA, Rohrer GA. Characterization of the aldo-keto reductase 1C gene cluster on pig chromosome 10: possible associations with reproductive traits., 2006, 2: 28.
[28] Jin S, Lee JB, Kang K, Yoo CK, Kim BM, Park HB, Lim HT, Cho IC, Maharani D, Lee JH. The possibility of TBC1D21 as a candidate gene for teat numbers in pigs., 2013, 26(10): 1374–1378.
[29] Xu RX, Wei N, Wang Y, Wang GQ, Yang GS, Pang WJ. Association of novel polymorphisms in lymphoid enhancer binding factor 1 (LEF-1) gene with number of teats in different breeds of pig., 2014, 27(9): 1254–1262.
[30] Duijvesteijn N, Veltmaat JM, Knol EF, Harlizius B. High-resolution association mapping of number of teats in pigs reveals regions controlling vertebral development., 2014, 15: 524.
[31] Rohrer GA, Nonneman DJ. Genetic analysis of teat number in pigs reveals some developmental pathways independent of vertebra number and several loci which only affect a specific side., 2017, 49(1): 4.
[32] Yang J, Huang LS, Yang M, Fan Y, Li L, Fang SM, Deng WJ, Cui LL, Zhang Z, Ai HS, Wu ZF, Gao J, Ren J. Possible introgression of the VRTN mutation increasing vertebral number, carcass length and teat number from Chinese pigs into European pigs., 2016, 6: 19240.
[33] Tetzlaff S, Chomdej S, Jonas E, Ponsuksili S, Murani E, Phatsara C, Schellander K, Wimmers K. Association of parathyroid hormone-like hormone (PTHLH) and its receptor (PTHR1) with the number of functional and inverted teats in pigs.,2009, 126(3): 237–241.
[34] Chomwisarutkun K, Murani E, Ponsuksili S, Wimmers K. Microarray analysis reveals genes and functional networks relevant to the predisposition to inverted teats in pigs., 2011, 90(1): 1–15.
[35] Chomwisarutkun K, Murani E, Brunner R, Ponsuksili S, Wimmers K. QTL region-specific microarrays reveal differential expression of positional candidate genes of signaling pathways associated with the liability for the inverted teat defect., 2012, 44(2): 139–148.
[36] Famá F, Cicciú M, Sindoni A, Scarfó P, Pollicino A, Giacobbe G, Buccheri G, Taranto F, Palella J, Gioffré-Florio M. Prevalence of ectopic breast tissue and tumor: a 20-Year single center experience., 2016, 16(4): e107–e112.
[37] Hiremath B, Subramaniam N, Chandrashekhar N. Giant accessory breast: a rare occurrence reported, with a review of the literature., 2015, 2015: bcr2015210918.
[38] Schaid DJ, Chen W, Larson NB. Larson. From genome- wide associations to candidate causal variants by statistical fine-mapping., 2018, 19(8): 491–504.
Physiology and genetics research progress of teat traits in pigs
Lisheng Zhou, Weimin Zhao, Feng Tu, Yunhe Wu, Shouwen Ren, Xiaomin Fang
The pig teat traits are important indices of genetic improvement in pig breeding, which belong to reproductive traits and can directly affect the sows lactation rate and piglet survival rate. Understanding the genetic mechanism underlying the variation of teat traits is of immense value for the improvement of pig reproductive performance. However, the genetic mechanism underlying teat traits (including teat number, type, location distribution, and fluctuating asymmetry) remains elusive. In this review, we summarize the studies on physiology and genetics of teat traits in pigs, including the development process of the mammary gland, the QTL mapping, and candidate gene researches. This review aims to provide a new perspective for the identification of causal mutations and major genes affecting the teat traits and revealing the complex genetic mechanism of the differences in teat number, type and location distribution during embryonic development in pigs.
pig; teat traits; genetic research
2018-12-12;
2019-04-22
国家生猪现代产业技术体系南京综合试验站项目(编号:CARS-35),江苏省农业重大新品种创制项目(编号:PZCZ201733),江苏省农业科学院科研专项(编号:ZX(18)7006)和江苏省博士后科研资助计划(编号:2018K059B) [Supported by the National Pig Industrial Technology System Nanjing Experimental Station (No. CARS-35), Jiangsu Agricultural Major New Breed Creation Project (No. PZCZ201733), Special Scientific Research Fund of Jiangsu Academy of Agricultural Sciences (No. ZX(18)7006) and Jiangsu Post-doctoral research Funding (No. 2018K059B)]
周李生,博士,助理研究员,研究方向:猪数量性状遗传解析。E-mail: zls123668@qq.com
方晓敏,博士,副研究员,研究方向:猪抗病育种。E-mail: 363927739@qq.com
10.16288/j.yczz.18-336
2019/5/10 15:23:09
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20190510.1522.004.html
(责任编委: 任军)
