青海大通北川河源区鱼类舌状绦虫和双线绦虫的分子鉴定及系统发育关系研究
2019-05-16伊平昌李国平顾冬花赵志刚张成图卢福山
伊平昌 , 李国平 , 顾冬花 , 赵志刚 , 张成图 , 卢福山
(1.青海省大通县畜牧兽医站 , 青海 西宁 810199 ; 2.青海省西宁市畜牧兽医站 ,青海 西宁 810005 ; 3.青海大学农牧学院 , 青海 西宁 810016)
青海大通北川河源区国家级自然保护区位于青海省西宁市大通县境内,湟水河一级支流-北川河源头,海拔2 680~4 622 m,保护区涉及宝库、青林、青山、向化、桦林共5个乡镇,总面积约10.79万公顷,占大通县总面积的34.91%[1-2]。青海大通北川河源区国家级自然保护区特殊的植被和复杂的地理环境,为这里的鱼种提供了丰富的食物来源,使鱼类资源顺利繁衍生息。本试验通过对青海大通北川河源区内鱼类寄生的绦虫进行了分子生物学鉴定,确定为舌状绦虫和双线绦虫,进一步丰富了鱼类动物的生物多样性,为鱼类资源保护和绦虫病防治提供了理论依据和防治资料。
1 材料与方法
1.1 虫体采集 本试验于2016年8月至2017年7月,分别按季节在青海大通北川河源区主要河流段(北纬N37°17′,东经E101°09′)将捕获的367尾鱼类样本进行解剖,用挑虫针钩取扁平长带状、不分节、乳白色“面条样”虫体(见中插彩版图1),用0.9%的生理盐水冲洗干净后,置于70%酒精甘油中保存,备用。

图1 “面条样”虫体标本
a : 舌状绦虫 ; b : 双线绦虫
1.2 引物 所用引物由北京六合华大基因科技股份有限公司合成,见表1。

表1 PCR分子生物学鉴定所用引物
1.3 方法
1.3.1 DNA的提取 剪取各0.5 cm3大小形态的虫体组织块分别放入新的保存管中,用剪刀剪碎组织,利用天根生化科技(北京)有限公司生产的DNA提取试剂盒提取DNA,具体操作步骤按照试剂盒使用说明书进行,DNA置于-20 ℃长期保存。
1.3.2 PCR鉴定 两种虫体的CoxI、ItsI和NadI基因引物参考Zehnder等[3]基因的引物和基于在NCBI上下载绦虫的序列,经Genedoc软件比对后,利用Primer3 Input (Version 0.4.0)软件进行引物设计。为保险起见,将目的片段分两个部分上下引物扩增后,进行PCR分子生物学鉴定。PCR扩增酶为天根生化科技(北京)有限公司的蛋白酶K,扩增反应体系如下:Mastrer Mix 25.0 μL,上、下游引物(浓度10 μmol/L)各2 μL,模板DNA 7 μL,重蒸馏水19 μL至PCR反应体系为50 μL(均购自天根生化科技(北京)有限公司)。为了避免试剂是否存在污染,每组PCR均设立不含模板的空白对照来检测。扩增后PCR产物在2%的琼脂糖凝胶中进行电泳(电压120 V,时间30 min),电泳完成后在凝胶成像仪中观察结果并拍照。目的条带的胶块采用DNA胶回收试剂盒(天根生化科技(北京)有限公司产品)进行胶回收。
1.3.3 序列分析 阳性产物测序由北京六合华大基因科技股份有限公司完成,采用Sanger双脱氧链终止法进行双向测序。测序结果经峰图分析后,在GenBank上用采用BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)方法进行同源序列搜索,应用MEGA 5.10软件和Clustal Omega(http://www.ebi.ac.uk/Tools/msa/clustalo/)在线软件进行分析核苷酸序列和氨基酸序列的同源性。
1.3.4 构建系统发育树 通过NCBI提供的BLAST在线搜索服务软件下载其他舌状绦虫和双线绦虫的CoxI、ItsI和NadI核苷酸基因序列。利用MEGA 5.10软件的系统发育树构建功能对这些基因序列进行分析,选择邻近法(Neighbor-Joining method,NJ)构建系统发育树,并使用自展值(Bootstrap)检验其可靠性,重复次数为2 000,分析不同分离株绦虫间的进化关系。
2 结果
2.1 PCR分子鉴定 利用基因组DNA提取试剂盒提取DNA后,基于CoxI基因进行PCR扩增,扩增出约480 bp的目的片段;基于ItsI基因进行PCR扩增鉴定,扩增出1 500 bp的目的片段;同样基于NadI基因进行PCR扩增鉴定,扩增出500 bp的目的片段;分别得到了3条目的基因片段(与预期的目的条带长度相符),目的条带的胶块进行胶回收、测序。所得序列在GenBank用BLAST进行同源序列搜索,结果表明,两种样品经分子鉴定为舌关绦虫(Ligulaintestinalis)和双线绦虫(Digrammainterupta)。
2.2 基因同源性对比分析 用Clustal Omega在线软件的Clustal 2.1方法,将2种虫体克隆序列(CoxI、ItsI和NadI基因序列)进行研究鉴定,与已上传到GenBank上的舌状绦虫(Ligulaintestinalis)和双线绦虫(Digrammainterupta)核苷酸序列分别进行同源性比较,见表2、表3。结果表明,舌状绦虫的CoxI基因序列与EU241312 (法国)和EU241317(法国)的核苷酸序列同源性分别为97.8%和97.3%,ItsI基因序列与KC900973 (伊朗)和KC900974(伊朗)的核苷酸序列同源性分别为95.1%和99.3%,NadI基因序列与AF524046 (中国)、AF524043 (中国)、AF524044 (中国)和AF524045 (中国)的核苷酸序列同源性分别为84.2%、83.4%、69.9%和69.6%;双线绦虫的CoxI基因序列与EU241317 (法国)、EU241287(法国)、 EU241309 (法国)和EU241281(法国)的核苷酸序列同源性分别为78.3%、78.6%、79.6%和78.6%,ItsI基因序列与KC900973 (伊朗)和KC900974 (伊朗)的核苷酸序列同源性分别为96.0%和98.9%,NadI基因序列与AF524045 (中国)、AF524043 (中国)、AF524044 (中国)和AF524023 (中国)的核苷酸序列同源性分别为84.6%、67.0%、67.6%和99.5%。这两种虫体在GenBan中进行同源性比较分析发现,不同线粒体基因标记显示出的物种差异性较大,且没有1条DNA序列与它们有100%的同源性。本研究鉴定的分离株Ligulaintestinalis和Digrammainterupta与伊朗(KC900974)分离株及中国分离株(AF524023)序列同源性较高,并被发现鉴定。
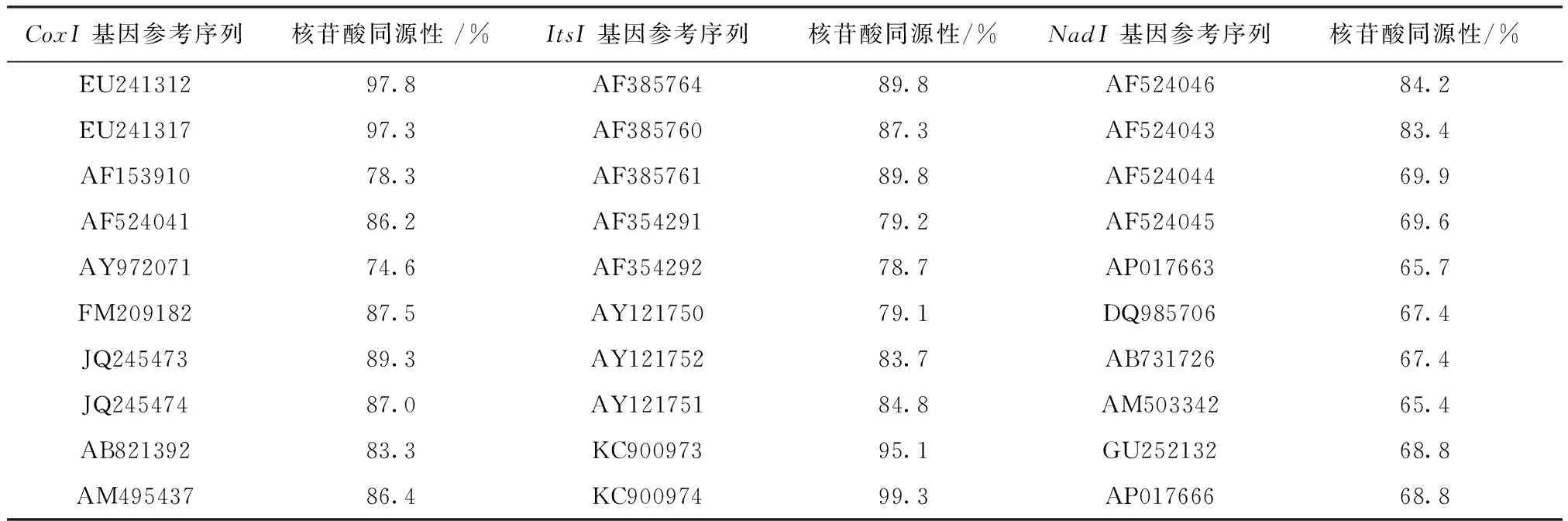
表2 舌状绦虫3种不同基因与其参考序列的核苷酸序列的同源性比较

表3 双线绦虫3种不同基因与其参考序列的核苷酸序列的同源性比较
2.3 系统发育进化分析 选取GenBank已发表的相应序列运用MEGA 5.10中的邻近法(Neighbor-Joining method,NJ)构建两种虫体包含分离株的3种基因(CoxI、ItsI和NadI基因)的相应进化树(图2~7)。结果表明,本研究鉴定的舌状绦虫分离株与伊朗分离株种属亲缘关系较近,与其他绦虫所属分支相隔较远,与李伟平等[4]研究的结果相一致;双线绦虫分离株与我国分离株种属亲缘关系较近,在进化上与其他地区的分离株遗传进化距离较远。从进化树的分支状况发现,舌状绦虫和双线绦虫虽然存在分支跨越,但均聚在一个大的分支上,与其他属种明显分开。
2.3.1 舌状绦虫进化分析
2.3.1.1 基于舌状绦虫CoxI基因对不同种类绦虫遗传距离及系统进化树见图2。根据其系统发育树判断与法国同属一支,和中国本土遗传体系较为接近。
2.3.1.2 基于舌状绦虫ItsI基因对不同种类绦虫遗传距离及系统进化树见图3。根据系统发育树可判断其遗传体系为独立的一支。
2.3.1.3 基于舌状绦虫NadI基因对不同种类绦虫遗传距离及系统进化树见图4。根据系统发育树判断其与中国本土遗传体系同属一支,与日本、韩国遗传体系较为接近。

图2 基于CoxI基因序列以邻近法所构建的系统发育树(NJ树)
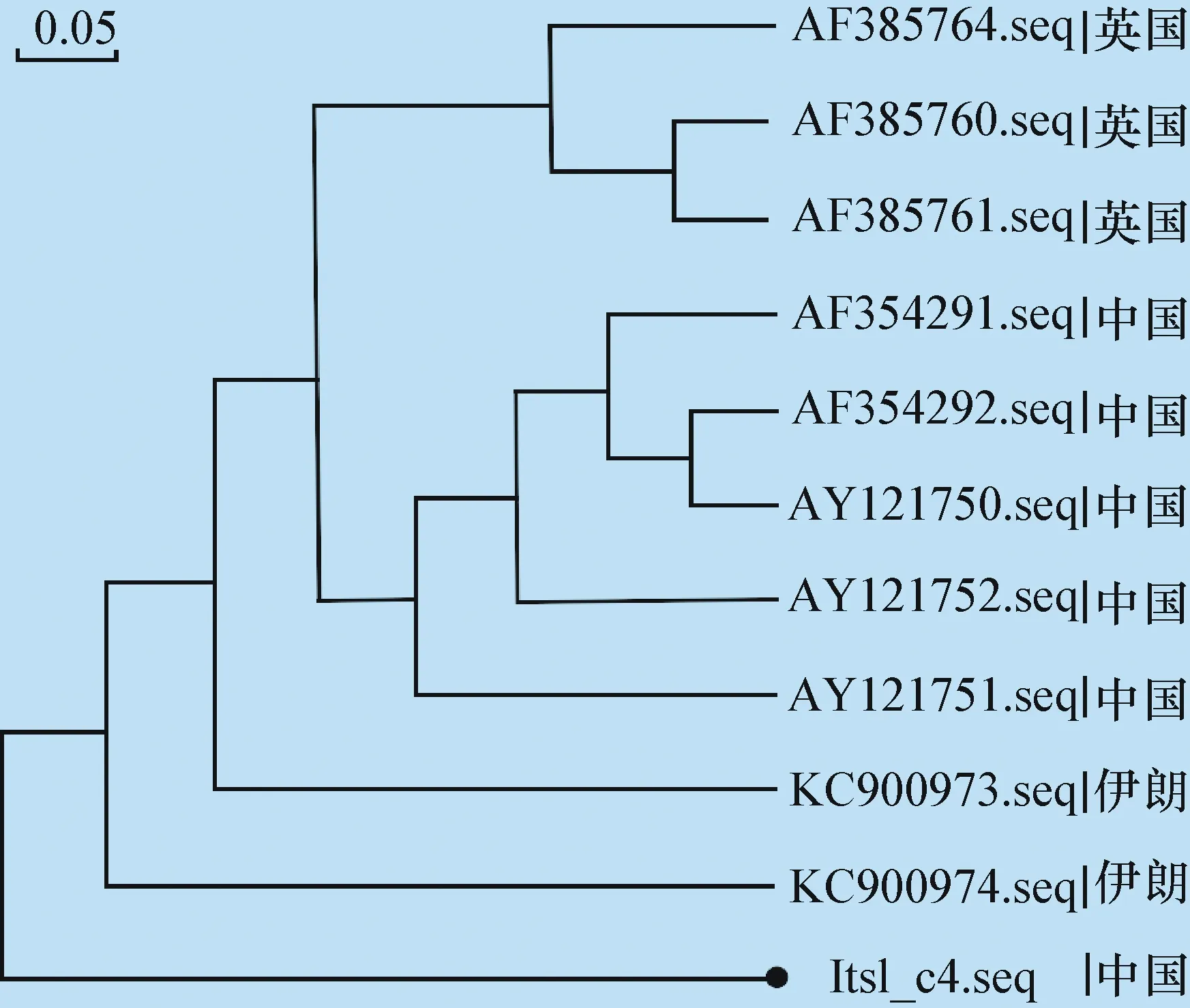
图3 基于ItsI基因序列以邻近法所构建的系统发育树(NJ树)

图4 基于NadI基因序列以邻近法所构建的系统发育树(NJ树)
2.3.2 双线绦虫进化分析
2.3.2.1 基于双线绦虫CoxI基因对不同种类绦虫遗传距离及系统进化树见图5。可判断出与法国同属一支,遗传体系较为接近。

图5 基于CoxI基因序列以邻近法所构建的系统发育树(NJ树)
2.3.2.2 基于双线绦虫ItsI基因对不同种类绦虫遗传距离及系统进化树见图6。可判断出双线绦虫ItsI基因为独立的一支遗传体系。
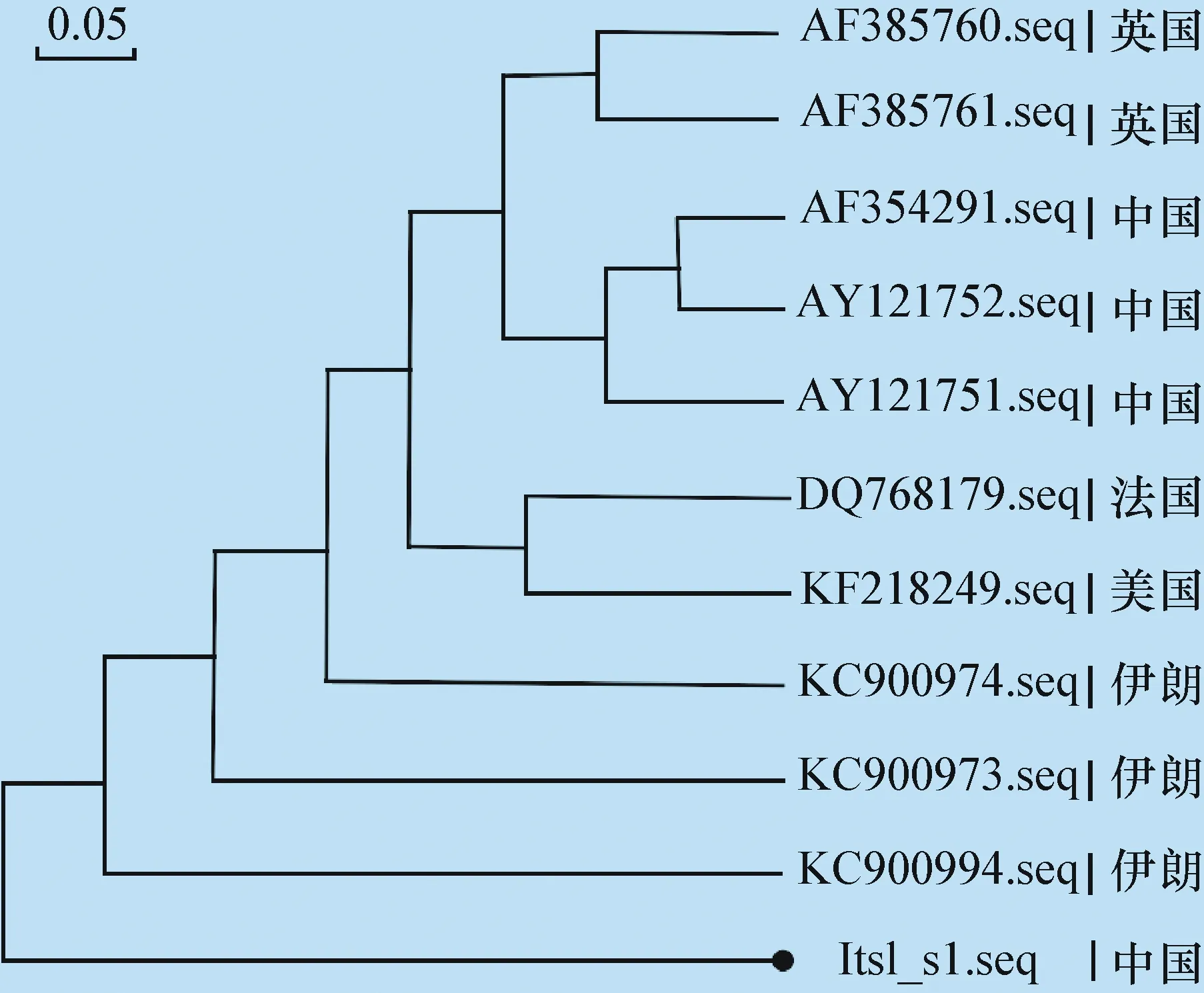
图6 基于ItsI基因序列以邻近法所构建的系统发育树 (NJ树)
2.3.2.3 基于双线绦虫NadI基因对不同种类绦虫遗传距离及系统进化树见图7。可判断出与中国本土的遗传体同属一支,绦虫属遗传体系较为接近。
3 讨论
本研究利用3个线粒体CoxI、ItsI和NadI基因进行了舌状绦虫和双线绦虫的分子鉴定。线粒体基因是区别不同地域绦虫及其幼虫的标识基因,应运最广的为CoxI基因与NadI基因[5],本试验通过对青海大通北川河源区鱼类绦虫种属内对比鉴定分析,发现舌状绦虫的差异性为6%~8%,双线绦虫为8%~10%,两者皆可作为遗传研究的标记。两种虫体序列间分析发现CoxI基因变异程度高于NadI基因,其中舌状绦虫CoxI基因片段中45个变异位点上存在碱基的倒置、转换和缺失,而在双线绦虫的33个变异位点上碱基均缺失。两种虫体种属内对比,发现CoxI基因的差异性达到11%以上,在NCBI-Blast中检索得到的其他绦虫作比对,差异性达6%~8%,与大通地区的地理位置和虫体寄生有关。在ItsI基因种属内对比发现差异性较其他两个基因片段较小,能有效地识别亚种内的物种,在NCBI-Blast中检索得到的其他绦虫比对,发现其同源性很高,而差异性小,在系统发育树上两种虫体的ItsI基因均作为独立的一支。从进化树的分支状况可以发现,舌状绦虫和双线绦虫虽然存在分支跨越,但是均聚在一个大的分支上,与其他属种明显分开。这些结果表明,CoxI和ItsI基因均可用于舌状绦虫和双线绦虫种的系统发育研究。

图7 基于 NadI 基因序列以邻近法所构建的系统发育树 (NJ树)
利用以基因为分子标记进行鉴定研究,丰富了寄生虫的鉴定方法,促进了寄生虫分类的发展[4]。相对于核基因组来说,线粒体基因组具有分子量小、结构简单、基因排列紧密、严格母系遗传、进化速率较快和无组织特异性等特点,特别适合作为遗传学研究的标记[6]。通过 DNA 序列、蛋白质序列、蛋白质结构等构建系统发育进化树,用类似树状分支的图来表示各种(类)生物之间的亲缘关系,通过对生物序列的研究来推测物种的进化历史。
本试验对我国青海大通北川河源区国家级自然保护区河流中鱼类体内感染的舌状绦虫(Ligulaintestinalis)和双线绦虫(Digrammainterupta)的CoxI、NadI和ItsI基因序列进行克隆测序并进行了分子鉴定;同时,运用CoxI、NadI和ItsI3种基因进行了属种之间的分子系统发育初步探究,比较了主要虫种间基因的序列同源性,得到了系统发育树,初步阐明了鉴定种与其他种属之间的系统发育关系。
