适于海岛棉指纹图谱构建的SNP核心位点筛选与评价
2019-05-09李乐晨朱国忠苏秀娟郭旺珍
李乐晨 朱国忠 苏秀娟,2 郭旺珍,*
适于海岛棉指纹图谱构建的SNP核心位点筛选与评价
李乐晨1朱国忠1苏秀娟1,2郭旺珍1,*
1南京农业大学作物遗传与种质创新国家重点实验室/ 杂交棉创制教育部工程研究中心, 江苏南京 210095;2新疆农业大学农学院, 新疆乌鲁木齐 830052
海岛棉具有纤维品质好、抗病性强等优异性状, 不仅为纺织工业提供优质棉纤维原料, 也是陆地棉相关性状改良的重要供体材料。然而, 与陆地棉相比, 开展海岛棉的遗传多样性和基因分型研究相对较少。本研究基于CottonSNP80K芯片对282份不同来源的海岛棉品种/材料进行基因组SNP分型研究, 以选择高效鉴别海岛棉材料的核心位点组合。按照检出率大于95%、具多态性、最小等位频率(MAF)大于0.01、杂合率小于0.05、无冗余位点等条件筛选, 获得2594个高质量SNP位点。对上述位点设置不同数目梯度筛选, 确定最优核心位点数。随着位点个数的增多, 位点组合对海岛棉材料的识别率逐渐增加。当位点数为200时, 识别率为89%; 位点数提高到1500时, 识别率可达99%。进一步增加位点数, 识别率无显著变化。利用中选的1500个SNP位点检测供试材料, 其平均MAF值0.14, 平均杂合率0.007, 平均多态信息含量0.21。SNP位点的聚丙烯凝胶电泳验证表明, SNP-PCR与芯片分型结果一致性达98.3%。本研究提供了适于海岛棉指纹图谱构建、含1500位点的一套核心SNP位点组合, 可用于海岛棉遗传多样性分析和品种身份鉴定。
基因芯片; DNA指纹图谱; SNP; 海岛棉
中国是世界上最大的棉花生产国之一, 植棉历史悠久[1]。棉花属于锦葵科()棉属(), 分为52个种, 包括非洲棉(,2= 26, A1)、亚洲棉(, 2= 26, A2)、陆地棉(, 2= 52, [AD]1)和海岛棉(, 2= 52, [AD]2) 4个栽培种。海岛棉也称长绒棉, 原产于中美洲、南美洲和加勒比地区。20世纪50年代, 海岛棉被引入中国, 在河南、江苏、新疆等地进行实验种植[2]。20世纪80年代后, 海岛棉主要在新疆地区种植。相较于陆地棉, 海岛棉种植面积小, 占世界棉花产量的5%左右[3], 但其纤维优质细长, 以海岛棉织造的衣服有极佳的触感及良好的透气性与吸汗力, 是生产高档或特殊棉纺织品的首选[4]。此外, 海岛棉还具有耐寒、耐旱、抗病和抗虫等其他重要性状[5], 常常被作为改良陆地棉农艺性状和提高抗性的供体亲本。
中国的海岛棉主要种植在新疆塔里木盆地和吐鲁番盆地[6]。自20世纪50年代海岛棉引种成功后, 育种家一直致力于引种、系统选择、杂交育种等新品种培育研究, 选育出胜利1号、军海1号、新海系列等海岛棉品种50余个, 有效服务了海岛棉生产与市场需求, 也积累了较丰富的海岛棉种质资源[7-8]。然而, 在育种实践中, 对海岛棉种质资源遗传多样性研究不多, 针对不同来源的种质资源不能有效区分; 由于少数骨干亲本使用, 新育成品种相对早期品种遗传多样性降低; 也存在“套牌”、乱杂现象, 阻碍了海岛棉产业发展。制定科学、可靠的品种鉴定方法和标准, 开展海岛棉品种鉴定, 具有重要的科学和生产意义。
基于分子标记技术进行棉花品种鉴定及遗传多样性分析, 在陆地棉中已有深入研究[9-10], 而海岛棉品种/材料的基因分型及指纹图谱构建进展相对缓慢。几个代表性报道集中在利用SSR/SRAP标记进行不同来源海岛棉材料遗传多样性分析。潘兆娥等[11]利用SSR分子标记对来自俄罗斯、埃及、美国、阿尔巴尼亚、中国等的56份海岛棉种质进行遗传多样性分析。李金荣等[12]利用SSR标记对14个新疆自育海岛棉品种进行了聚类分析。李武等[13]利用SRAP标记, 对我国引入海岛棉以来培育的36个国内品种及20个国外品种进行遗传多样性分析。上述研究揭示出海岛棉种质资源总体上遗传多样性丰富, 但中国培育的海岛棉品种遗传基础较为狭窄, 品种间平均相似系数较高。在大范围品种鉴定过程中, 低通量的分子标记技术具有较大局限性。
近年来, 海岛棉基因组序列解析及应用取得显著进展。南京农业大学结合海岛棉和陆地棉F2分离群体重测序分析, 构建含4,999,048个SNP位点, 覆盖4042 cM的超高密度SNP和SSR整合海陆遗传图谱, 并成功用于四倍体陆地棉基因组组装及scaffolds方向及顺序确定[14]。基于全基因组测序分析, 溢达集团和华中农业大学分别公布了海岛棉新海21及3-79的基因组框架信息[15-16]。这些研究为进一步筛选适宜于海岛棉不同基因型鉴定的高多态SNP位点, 用于遗传多样性分析和品种身份鉴定奠定基础。
本研究选择不同来源的282份海岛棉品种/材料, 利用实验室自主研发的高密度棉花SNP芯片(CottonSNP80K)[17]开展基因分型研究。通过分析不同SNP位点遗传多样性信息, 筛选出一套高效用于海岛棉品种/材料身份鉴定的SNP位点集合。该研究是我室基于全基因组筛选完成适于陆地棉品种身份鉴定的SNP核心位点组合后[18], 提供的适于另一个栽培四倍体棉种海岛棉身份鉴定的SNP核心位点组合。该研究为开展海岛棉遗传多样性分析、品种确权和保障育种者和消费者权益提供支撑。
1 材料与方法
1.1 试验材料
海岛棉品种/材料共282份(附表1)。其中16份早期国外引进品种和中国自育品种由中国农业科学院棉花研究所国家棉花种质中期库提供, 其余266份由新疆农业大学农学院苏秀娟收集自交保存。
1.2 试验方法
1.2.1 DNA提取和质量检测 取新鲜的棉花嫩芽约0.1 g置1.5 mL离心管中, 采用CTAB法提取基因组DNA[19]。分离出的DNA经1%琼脂糖凝胶电泳, DNA条带明亮且单一, 表明DNA浓度高且没有被降解。同时借助超微量分光光度计(One Drop-OD 1000)进一步检测DNA的质量和浓度, OD260nm/OD280 nm在1.8~2.0之间代表DNA纯度较高, 按照所示DNA浓度将每一样品浓度均一化至100 ng μL–1。
1.2.2 SNP位点筛选 供试材料的SNP检测与数据分析方法同朱国忠等[18]。经CottonSNP80K芯片扫描的原始数据导入GenomeStudio软件(V2011.1, Illumina, Inc.)进行SNP分型。根据SNP位点特性确定筛选条件(位点检出率大于95%, 最小等位基因频率大于0.01, 杂合率小于0.05)获得位点组合。进一步利用位点筛选工具LociScan_V1.0对中选位点进行梯度筛选, 基于R对不同梯度位点之间进行相关性分析, 统计分析评价不同位点组合鉴定效率。LociScan_V1.0位点筛选软件由北京市农林科学院玉米研究中心提供。
1.2.3 SNP结果验证 随机选取12个核心位点进行位点真实性验证。利用WebSNAPER (https://pga. mgh.harvard.edu/cgi-bin/snap3/websnaper3.cgi)进行SNP特异引物设计, 引物由南京金斯瑞生物科技有限公司合成。随机选取芯片检测具有多态性的材料进行引物多态性的初步筛选, 利用筛选获得的多态引物进一步检测10份不同来源的材料, 评价芯片的分型准确率。设计作为内标对照, 检测不同样本模板PCR扩增结果, 确保模板浓度的相对一致性。检测的SNP-PCR反应程序为95℃预变性5 min; 95℃变性30 s, 58℃退火30 s, 72℃延伸30 s, 循环数为28; 72℃延伸10 min。扩增产物经非变性PAGE电泳(恒压200 V, 1.00~1.25 h), 通过银染DNA显带, 记录多态性数据。比较聚丙烯酰胺凝胶电泳检测结果与芯片分型结果, 计算其一致性。
2 结果与分析
2.1 SNP位点特性评估
利用本研究室研发的高密度SNP芯片(Cotton SNP80K)[17]对282份不同来源的海岛棉品种/材料进行基因分型。基于分型结果进行位点检出率、杂合率和多态性评估。CottonSNP80K中包含77,774个有效的SNP位点。通过对282份品种/材料的位点检测结果分析发现, 芯片中完全未检出的SNP位点有225个, 占0.3%; 检出率大于0.95的有72,138个, 占92.8%, 其中检出率为1的SNP位点有36,697个(图1-A)。表明该芯片SNP位点对海岛棉检测效率高, SNP本身也显示很好的稳定性和重复性。SNP位点杂合率评估表明, 90.7%的位点(70,565)杂合率均小于0.05; 有5679个位点的杂合率大于0.5, 占7.3% (图1-B)。这些位点的高杂合率可能是由于异源四倍体棉花A、D亚基因组间部分同源位点干扰造成。在77,774个SNP位点中, 有34,635 (44.5%)个SNP位点具有多态性。其中最小等位基因频率(minor allele frequency, MAF)大于0.01的位点有10,369个, 占30.7% (图1-C)。这可能是由于海岛棉和陆地棉具有独立进化和驯化特征[20], 而该芯片主要侧重于陆地棉种内基因分型开发, 对海岛棉的基因型多态性分型效率比陆地棉低。
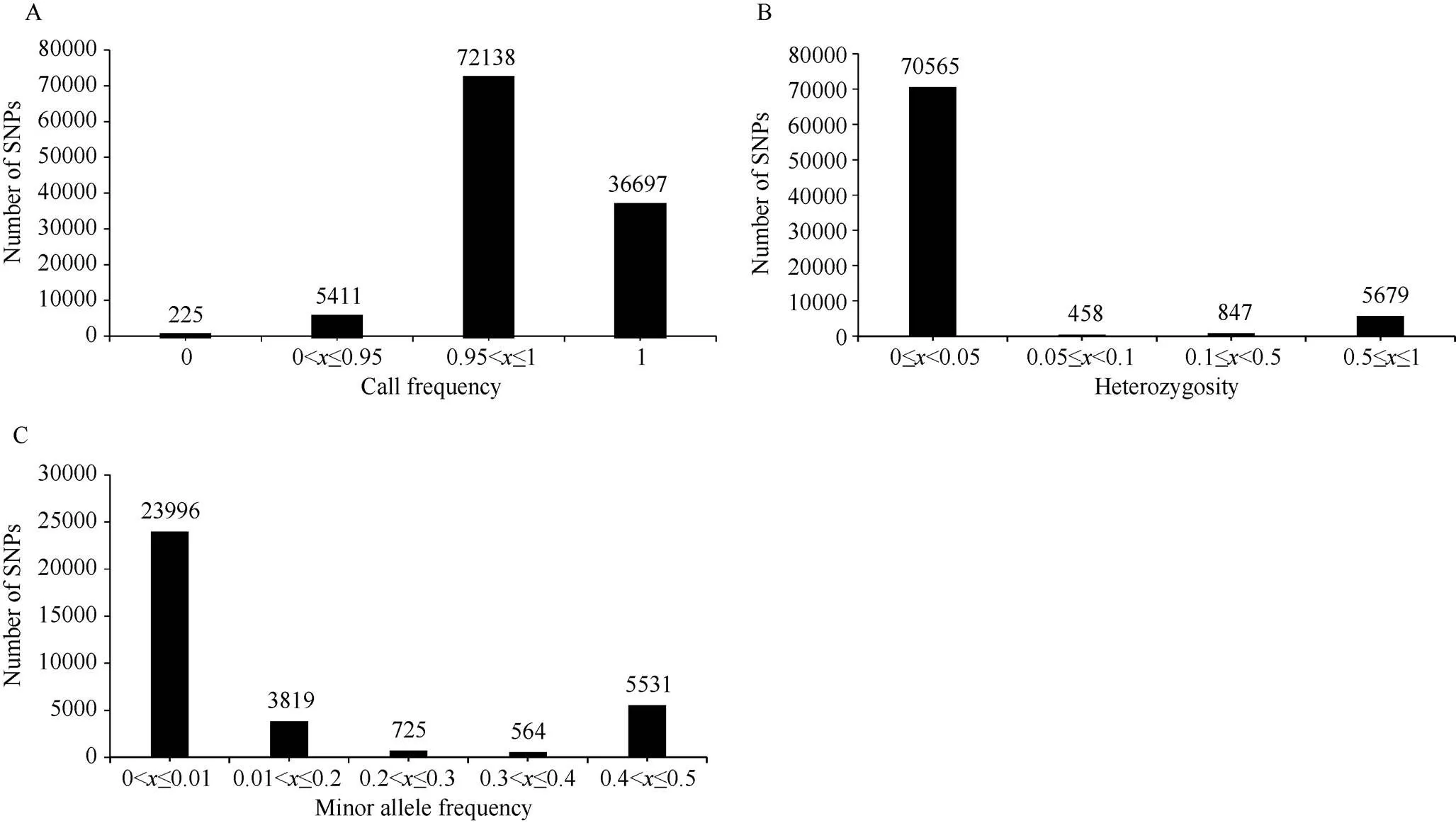
图1 77,774个SNP位点特性评估
横坐标代表被统计的SNP特征参数, 依次为位点检出率(call frequency)、杂合率(heterozygosity)和最小等位基因频率(minor allele frequency), 纵坐标代表SNP的分布数目。
The abscissa represents statistical SNP characteristic parameters, including loci call frequency, heterozygosity, and minor allele frequency. The ordinate represents the number of SNPs.
2.2 核心SNP位点筛选和指纹图谱构建
基于对CottonSNP80K中77,774个SNP位点的特性评估, 确定用于基因型分型的位点筛选条件。(1)筛选检出率大于95%的SNP位点, 共72,138个; (2)筛选MAF大于0.01的位点, 获7498个; (3)筛选杂合率小于0.05的位点, 余3245个; (4)去除基因型相同的位点, 获2594个位点。利用2594个SNP位点组合, 其基因型检出率为99.3%, 其中, 815个位点MAF大于0.2。
用LociScan_V1.0位点筛选工具对2594个位点进行分层筛选。分别设置200、300、400、500、1000、1500、2000、2500共8种不同数量位点, 进行识别效率信息统计。在200~1500位点时, 随着位点个数的增多, 识别效率增加。当位点为200时, 识别率为89%; 当位点为1500时, 识别率为99%。当位点超过1500时, 识别率增长趋于平缓, 都稳定在99%以上(表1)。对8种不同位点组合基于R进行相关性分析(图2), 8种不同位点组合两两之间值都小于0.001, 表明具有极显著的线性相关。其中, 1500、2000和2500位点组合其相互间线性相关性最高。因此, 选择1500位点作为适于海岛棉指纹图谱构建的核心SNP位点组合。
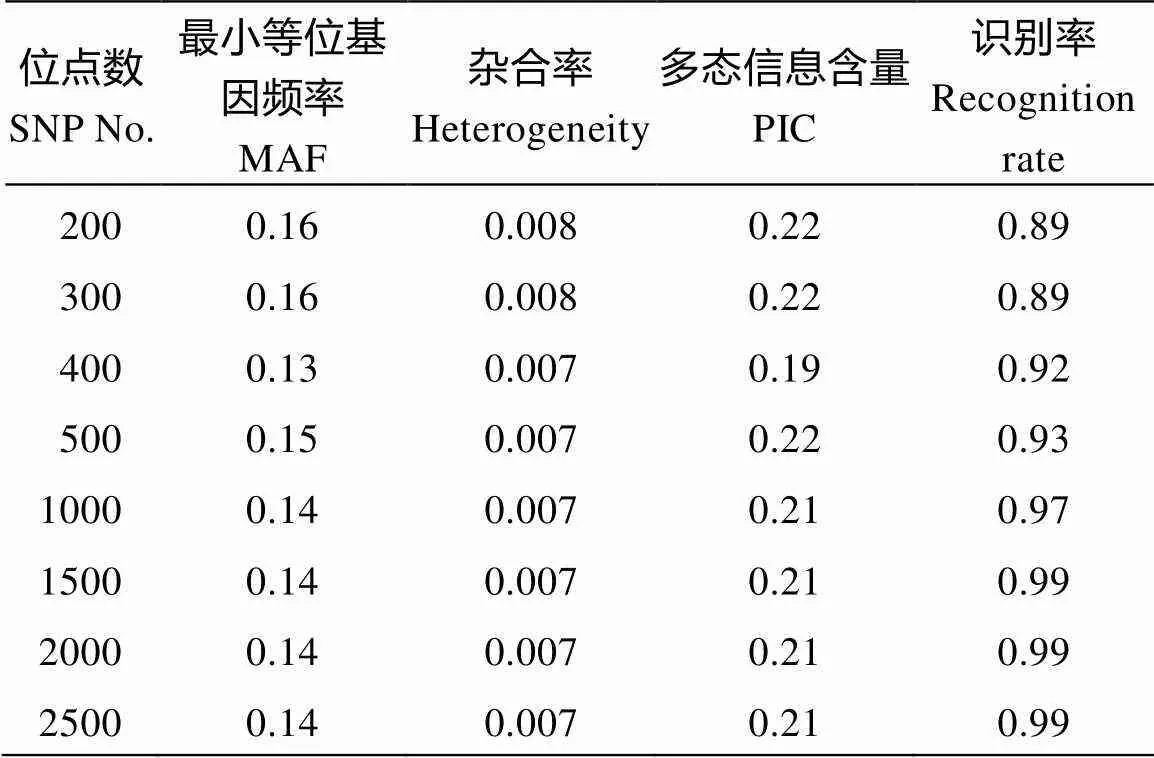
表1 核心位点的分层筛选
利用1500个SNP位点组合检测282份供试材料, 其平均MAF值0.14, 平均杂合率0.007, 平均多态信息含量0.21, 99%以上的海岛棉材料能被有效区分。本研究分别选择3组来自中国农业科学院棉花研究所国家棉花种质中期库和新疆农业大学自交保存的同名材料, 即3-79、Pima 1和新海5号, 进一步评估核心SNP位点组合的分辨力。结果表明, 不同来源的3-79基因型一致性为82.30%, 其17.70%的异质性中, 97.3%由纯合SNP位点差异造成; 不同来源的Pima 1基因型一致性为84.19%, 其15.81%的异质性中, 99.58%由纯合SNP位点差异造成; 不同来源的新海5号基因型一致性为91.67%, 其8.33%的异质性中, 22.13%由杂合SNP位点差异造成。同名海岛棉材料, 在不同单位、不同环境种植保存中, 由于天然突变或异交会导致基因型变化, 通过自交保存, 遗传变异进一步稳定。基于核心SNP位点组合可有效鉴定亲缘关系相近的材料间遗传差异及稳定性。
2.3 核心SNP位点准确性验证
为验证核心SNP位点的准确性, 分别从海岛棉四倍体A、D亚组中各随机挑选6个SNP位点开发特异性SNP引物, 并选择10份海岛棉材料进行基于PCR-PAGE电泳检测SNP位点多态性和芯片分型结果一致性的验证分析。结果显示, 芯片分型结果与SNP-PCR一致性高达98.3% (表2和图3), 进一步证明这些核心SNP位点的可利用性和基于芯片SNP分型的准确性。
3 讨论
海岛棉具有纤维品质好、抗病等优异性状, 是高档和特种棉纺织品的重要原料, 同时, 作为优异的供体材料, 被广泛用于陆地棉纤维品质、抗性等性状改良。南京农业大学以陆地棉遗传标准系TM-1和海岛棉海7124为亲本, 通过回交和分子标记辅助选择构建了一套陆地棉背景的海岛棉染色体片段渐渗系。进一步选育出背景单一、优质、抗病性表现突出的渐渗系材料, 用于改良陆地棉品种的纤维品质和黄萎病抗性[21]。中国最早种植的海岛棉, 是从埃及、美国、前苏联、苏丹、秘鲁等国引种, 进一步系统选育和杂交育种, 自主培育出的[22]。其中, 从“2依3”天然变异株中系选培育的“胜利1号”是中国第一个长绒棉品种[23]。从“9122依”天然变异单株中系选培育的“军海1号”是中国长绒棉生产历史上种植时间最长、推广面积最大的海岛棉品种[23]。“军海1号”也是新疆海岛棉育种的重要基础种质, 后续选育的“新海”系列品种很多均有“军海1号”的系谱, 也导致其遗传相似性较高[23]。为有效区分不同海岛棉品种, 鉴定其纯度和真实性, 本研究选择282份不同来源的海岛棉品种/材料, 利用CottonSNP80K芯片进行全基因组SNP分型分析, 获得一套适于构建海岛棉材料指纹图谱的SNP核心位点组合, 从DNA水平上为海岛棉遗传多样性分析和材料鉴定提供依据。DNA指纹图谱是指通过品种间的差异有效鉴别不同品种的图谱, 具有多位点性、高变异性和稳定的遗传性。利用分子标记技术开展棉花品种鉴定, 在陆地棉上已有系统深入的研究[9]。近年来, 基于基因组序列信息解析, SNP标记被广泛用于品种遗传多样性分析和品种身份鉴定。孙正文等[24]利用CottonSNP63K芯片筛选出393个基因组特异的SNP位点。朱国忠等[18]利用CottonSNP80K芯片, 筛选出含4857个SNP位点, 高效鉴定陆地棉品种身份的核心SNP位点组合。与陆地棉相比, 基于全基因组的海岛棉遗传多样性分析和品种身份鉴定相关研究还较缺乏。潘兆娥等[11]利用SSR标记对56份海岛棉进行遗传多样性分析, 两两之间相似系数位于0.6~0.8之间。吴大鹏等[25]利用SSR标记对4个不同国家20份海岛棉进行遗传多样性分析, 相似系数在0.66~0.94之间。这些研究表明, 育种进程中, 海岛棉遗传基础狭窄, 多数品种之间相似系数偏高, 品种资源的同质化现象日趋严重。此外, 来源于不同单位不同环境种植的同名海岛棉材料, 表现不同程度的异质性。因此, 不同来源的海岛棉材料需要进一步从基因组水平上明确其遗传多样性和稳定性, 为海岛棉种质资源有效利用提供依据。

图2 核心位点分层筛选的相关性分析
对角线提供了不同位点下遗传距离的分布; 下三角形(对角线的左下方)是不同类型SNP位点两两间的散点图, 显示出不同位点条件下的线性相关程度; 上三角形(对角线的右上方)数字表示不同类型SNP位点两两间的相关系数, ***表示在0.001水平显著相关。
The diagonal gives the distribution of genetic distances at different loci. The lower triangle (the lower left of the diagonal) is a scatter plot between the two types among different SNP loci combinations, which shows the degree of linear correlation under different loci combinations conditions. The upper triangle (the upper right of the diagonal) represents the correlation value between the two types among different SNP loci combinations. *** means the significance at the 0.001 level.
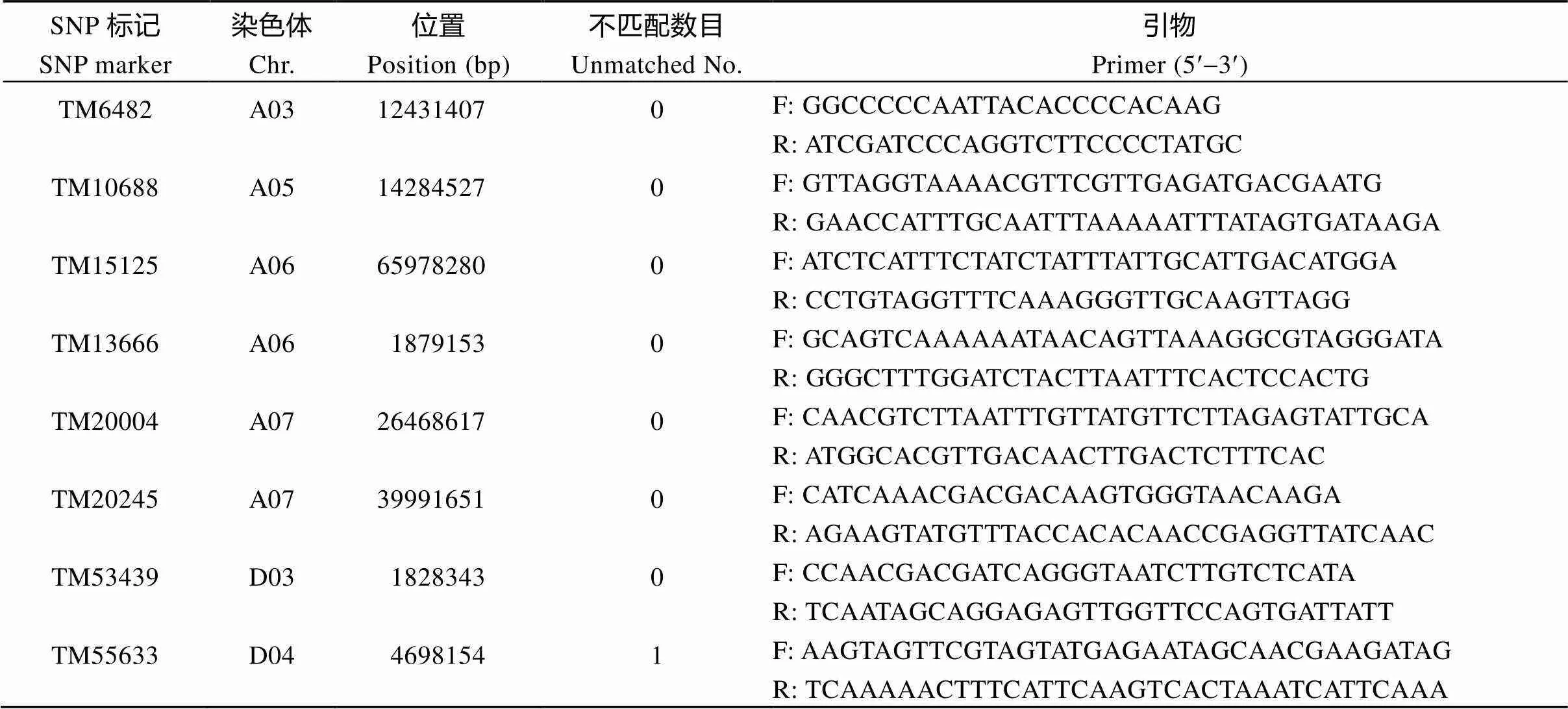
表2 SNP位点信息及验证结果
(续表2)

SNP标记SNP marker染色体Chr.位置Position (bp)不匹配数目Unmatched No.引物Primer (5¢-3¢) TM58458D05527011300F: TTTTTATCTTTAAGGTCGCCGCATGTGACR: ACCGATTGATTTTAAAGTTCCAAAAGAGAAGTTTG TM73511D1064546040F: GGGCACCTATCTTCTCTACACCTAACCCTTGR: AGAGCCAAACAAATGCACCAAATAGGGT TM80347D1354109371F: ATCTATTCCCTTTTTGACCTTCTCGACCAAAR: AAAACAACACTGTTTCAGGTAAAAGCTGACCA TM80357D1354644130F: TGACATGTGTTTGACATATTTGGATATGGATCTGR: AATGAAACCAATTTTAAAAGACCAATGCCTAAAGA Ghhis3D034814901F: CGGTGGTGTGAAGAAGCCTCATR: AATTTCACGAACAAGCCTCTGGAA
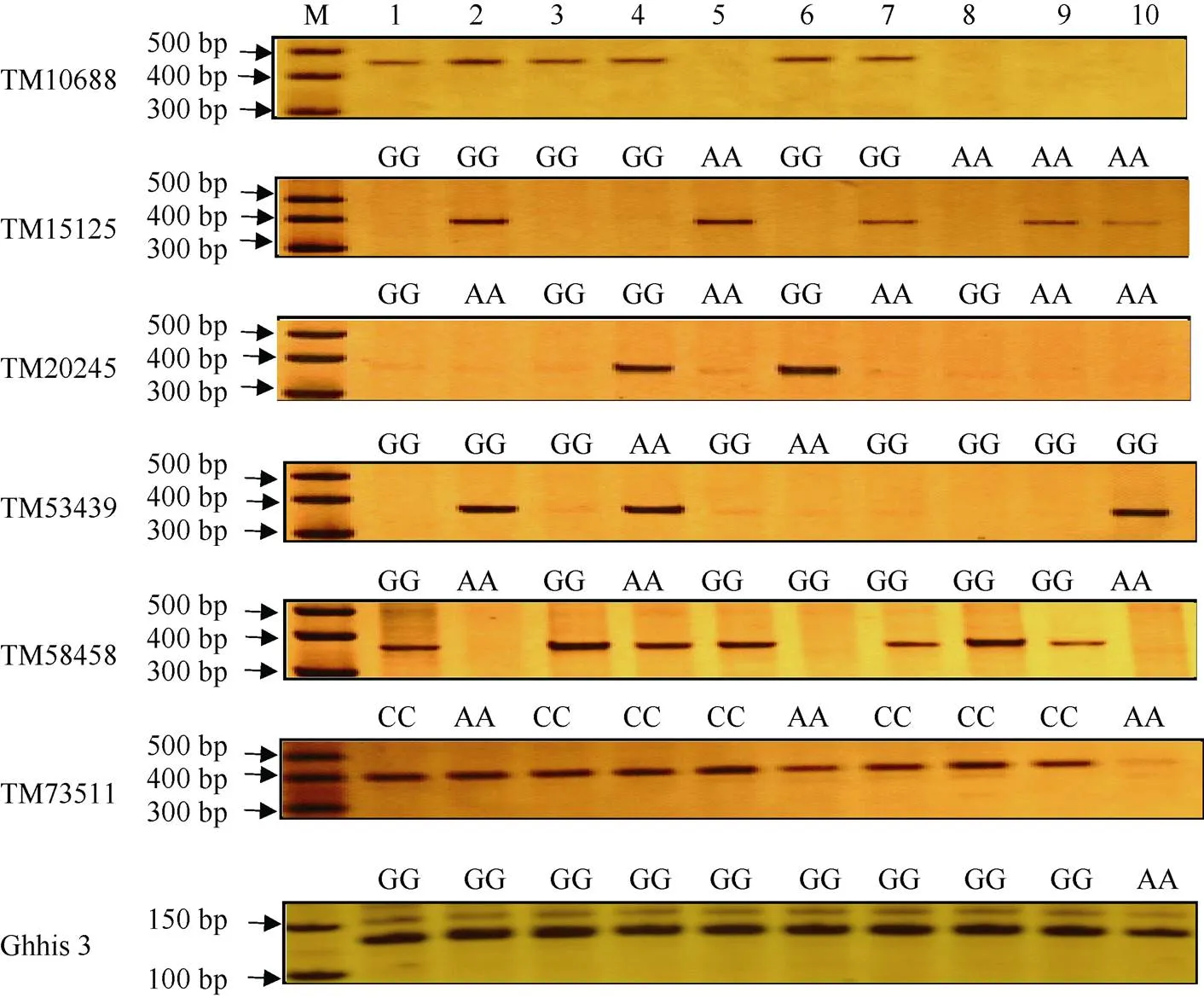
图3 基于SNP-PCR技术验证SNP位点芯片分型结果
M: DNA marker;: 内标对照; 1: 塔海901; 2: AW2046; 3: 农科10; 4: K339; 5: C6020; 6: K146; 7: 农科A16; 8: 司605; 9: 6--20; 10: AW2026。
M: DNA marker;: internal control; 1: Tahai 901; 2: AW2046; 3: Nongke 10; 4: K339; 5: C6020; 6: K146; 7: Nongke A16; 8: Si 605; 9: 6--20; 10: AW2026.
品种鉴定结果的可靠程度主要依赖于分子标记的稳定性和多态性[26], 使用低通量分子标记进行海岛棉品种鉴定, 效率不高。随着高通量测序和芯片技术的发展, 挖掘高质量SNP位点组合为海岛棉品种遗传多样性及身份鉴定提供了有效途径。本研究通过CottonSNP80K芯片筛选获得了2594个高效鉴别海岛棉材料的SNP位点, 且利用其中1500个位点即可对供试海岛棉材料达到99%的识别率。这套SNP核心位点组合是用于海岛棉指纹图谱构建和身份鉴定的首次报道, 将为海岛棉遗传多样性分析、品种身份鉴定和种质资源评价与利用提供技术支撑。
SNP指纹图谱的构建方式也与SNP检测分型技术相关, SNP检测分析方法有很多种。以凝胶电泳检测为基础的检测技术, 包括等位基因特异性PCR[27]和单链构象多态性[28], 具有检测效率高、成本低的优势, 已广泛使用。该技术主要通过在正向引物的3¢端引入错配碱基设计等位基因特异引物, 从而降低变异位点的产物扩增效率。由于模板浓度、扩增循环数、退火温度、错配碱基数目和类型以及不同SNP位点的侧翼序列等因素影响, SNP-PCR扩增会产生部分非特异性扩增产物。Drenkard等[29]通过大量的拟南芥SNP扩增分析, 发现超过1000倍的模板浓度差异和10个PCR循环数差异会对部分非特异等位基因的扩增效率产生较大影响, 同时SNP旁侧序列会影响等位基因特异引物设计。本研究通过控制相同模板浓度和统一PCR程序获得有效区分SNP位点的引物。在相同PCR扩增条件下, 使用内标检测不同模板PCR扩增结果, 以确保模板浓度的相对一致性。此外, 本研究也选择2个SNP位点(TM20245, TM53439)在多态性样本材料中进行测序验证, 证实该SNP多态位点在不同海岛棉材料中存在的真实性。基于上述结果, 尽管部分SNP-PCR引物会产生低扩增效率的PCR产物, 在PCR-PAGE电泳检测时显现较弱的扩增条带, 但在检测条件一致的情况下, 特异性强带和非特异性弱带易于区分, 不影响检测结果的真实性。
随着技术发展, 基于直接测序法、基因芯片技术[30]和竞争性等位基因特异性PCR[31]等检测方法已有效用于高通量的SNP分型研究。本实验核心位点的梯度筛选分析表明, 200个SNP位点就可以达到89%的识别率, 对于亲缘关系较远的海岛棉品种, 可以选用这200个SNP位点集合, 使用KASP或定点测序对其身份鉴定; 对于亲缘关系较近的品种, 需要采用高密度SNP位点集合结合基因芯片技术高通量鉴定, 本研究报道的含1500位点的SNP组合可用于其特异性分析。随着海岛棉基因组测序和重测序研究的进一步开展, 未来可以结合新的SNP数据进一步改进和完善, 形成高效绘制海岛棉指纹图谱的位点组合。
4 结论
基于CottonSNP80K芯片的分型结果, 综合考虑位点多态性、杂合率、准确性、高通量等参数, 筛选出适于海岛棉指纹图谱构建、含1500位点的一套核心SNP位点组合, 可实现海岛棉品种及资源材料的身份鉴定。
附表 请见网络版: 1) 本刊网站http://zwxb.chinacrops.org/; 2) 中国知网http://www.cnki.net/; 3) 万方数据http://c.wanfangdata.com.cn/Periodical-zuowxb. aspx。
[1] Dai J, Dong H. Intensive cotton farming technologies in China: achievements, challenges and counter measures., 2014, 155: 99–110.
[2] 汪志国, 王思明. 美棉在中国的引种与发展. 中国农学通报, 2006, 22: 421–426. Wang Z G, Wang S M. Introduction and development of American cotton in China., 2006, 22: 421–426 (in Chinese with English abstract).
[3] 宋宪亮, 孙学振, 张天真, 王洪刚. 棉花遗传多态性研究进展. 西北植物学报, 2004, 24: 2393–2397. Song X L, Sun X Z, Zhang T Z, Wang H G. Advances on genetic diversity of cotton ()., 2004, 24: 2393–2397 (in Chinese with English abstract).
[4] 姚贺盛, 张亚黎, 易小平, 薛军, 罗毅, 罗宏海, 张旺锋. 海岛棉和陆地棉叶片光合特性、冠层结构及物质生产的差异. 中国农业科学, 2015, 48: 251–261. Yao H S, Zhang Y L, Yi X P, Xu J, Luo Y, Luo H H, Zhang W F. Study on differences in comparative canopy structure characteristics and photosynthetic carbon assimilation of field-grown pima cotton () and upland cotton ()., 2015, 48: 251–261 (in Chinese with English abstract).
[5] Liu N, Tu L L, Tang W X, Gao W H, Lindsey K, Zhang X L. Small RNA and degradome profiling reveals a role for miRNAs and their targets in the developing fibers of., 2014, 80: 331–344.
[6] 孔庆平. 我国海岛棉生产概况及比较优势分析. 中国棉花. 2002, 29(12): 19–23. Kong Q P. Analysis on the situation and comparative advantages of island cotton production in China., 2002, 29 (12): 19–23 (in Chinese).
[7] 刘霞, 白玉林, 王绎衡, 杜斌, 杨勇, 王承强, 李小童. 海岛棉育种现状、未来方向以及生产建议. 中国棉花, 2015, 42(10): 11–13. Liu X, Bai Y L, Wang Y H, Du B, Yang Y, Wang C Q, Li X T. Breed situation, future development direction and production suggest of sea island cotton in China., 2015, 42(10): 11–13 (in Chinese).
[8] 邰红忠, 练文明, 卢金宝. 新疆海岛棉育种现状及存在问题. 中国棉花, 2013, 40(6): 15–17. Tai H Z, Lian W M, Lu J B. Current situation and problems in island cotton breeding in Xinjiang., 2013, 40(6): 15–17 (in Chinese).
[9] Kuang M, Wei S J, Wang Y Q, Zhou D Y, Ma L, Fang D, Yang W H. Development of a core set of SNP markers for the identification of upland cotton cultivars in China., 2016, 15: 954–962.
[10] Sheidai M, Riazifar M, Hoordadian A, Alishah O. Genetic finger printing of salt- and drought-tolerant cotton cultivars () by IRAP-REMAP and SRAP molecular markers., 2018, 14: 12–19.
[11] 潘兆娥, 何守朴, 贾银华, Podolnaya L P, 孙君灵, 王立如, 杜雄明. 引进海岛棉种质的SSR遗传多样性分析. 植物遗传资源学报, 2014, 15: 399–404. Pan Z E, He S P, Jia Y H, Podolnaya L P, Sun J L, Wang L R, Du X M. Genetic diversity analysis of the sea island cotton introduced using SSR markers., 2014, 15: 399–404 (in Chinese with English abstract).
[12] 李金荣, 王小国, 朱永军, 张西英, 张薇. 利用SSR标记对14个海岛棉品种的聚类分析. 新疆农业科学, 2009, 46: 237–241. Li J R, Wang X G, Zhu Y J, Zhang X Y, Zhang W. Cluster analysis on 14 sea-island cotton by SSRs., 2009, 46: 237–241 (in Chinese with English abstract).
[13] 李武, 倪薇, 林忠旭, 张献龙. 海岛棉遗传多样性的SRAP标记分析. 作物学报, 2008, 34: 893–898. Li W, Ni W, Lin Z X, Zhang X L. Genetic diversity analysis of sea-island cotton cultivars using SRAP markers., 2008, 34: 893–898 (in Chinese with English abstract).
[14] Wang S, Chen J D, Zhang W P, Hu Y, Chang L J, Fang L, Wang Q, Lü F N, Wu H T, Si Z F, Chen S Q, Cai C P, Zhu X F, Zhou B L, Guo W Z, Zhang T Z. Sequence-based ultra-dense genetic and physical maps reveal structural variations of allopolyploid cotton genomes., 2015, 16, doi: 10.1186/s13059-015- 0678-1.
[15] Liu X, Zhao B, Zheng H J, Hu Y, Lu G, Yang C Q, Chen J D, Chen J J, Chen D Y, Zhang L, Zhou Y, Wang L J, Guo W Z, Bai Y L, Ruan J X, Shangguan X X, Mao Y B, Shan C M, Jiang J P, Zhu Y Q, Jin L, Kang H, Chen S T, He X L, Wang R, Wang Y Z, Chen J, Wang L J, Yu S T, Wang B Y, Wei J, Song S C, Lu X Y, Gao Z C, Gu W Y, Deng X, Ma D, Wang S, Liang W H, Fang L, Cai C P, Zhu X F, Zhou B L, Chen Z J, Xu S H, Zhang Y G, Wang S Y, Zhang T Z, Zhao G P, Chen X Y.genome sequence provides insight into the evolution of extra-long staple fiber and specialized metabolites., 2015, 5, doi: 10.1038/srep14139.
[16] Yuan D J, Tang Z H, Wang M J, Gao W H, Tu L L, Jin X, Chen L L, He Y H, Zhang L, Zhu L F, Li Y, Liang Q Q, Lin Z X, Yang X Y, Liu N A, Jin S X, Lei Y, Ding Y H, Li G L, Ruan X A, Ruan Y J, Zhang X L. The genome sequence of Sea-Island cotton () provides insights into the allopolyploidization and development of superior spinnable fibres., 2015, 5, doi: 10.1038/srep17662.
[17] Cai C P, Zhu G Z, Zhang T Z, Guo W Z. High-density 80 K SNP array is a powerful tool for genotypingaccessions and genome analysis.s, 2017, 18, doi: 10.1186/s 12864-017-4062-2.
[18] 朱国忠, 张芳, 付洁, 李乐晨, 牛二利, 郭旺珍. 适于陆地棉品种身份鉴定的SNP核心位点筛选与评价. 作物学报, 2018, 44: 1631–1639. Zhu G Z, Zhang F, Fu J, Li L C, Niu E L, Guo W Z. Genome-wide screening and evaluation of SNP core loci for identification of upland cotton varieties., 2018, 44: 1631–1639 (in Chinese with English abstract).
[19] Zhang J, Stewart J M. Economical and rapid method for extracting cotton genomic DNA., 2000, 4: 193–201.
[20] Fang L, Gong H, Hu Y, Liu C X, Zhou B L, Huang T, Wang Y K, Chen S Q, Fang D D, Du X M, Chen H, Chen J D, Wang S, Wang Q, Wan Q, Liu B L, Pan M, Chang L J, Wu H T, Mei G F, Xiang D, Li X H, Cai C P, Zhu X F, Chen Z J, Han B, Chen X Y, Guo W Z, Zhang T Z, Huang X H. Genomic insights into divergence and dual domestication of cultivated allotetraploid cottons., 2017, 18, doi: 10.1186/s13059-017-1167-5
[21] Wang P, Ding Y Z, Lu Q X, Guo W Z, Zhang T Z. Development ofchromosome segment substitution lines in the genetic standard line TM-1 of., 2008, 53: 1512–1517.
[22] 马麒, 宿俊吉, 宁新柱, 李吉莲, 刘萍, 陈红, 林海, 邓福军. 新疆海岛棉种质资源表型性状遗传多样性分析. 新疆农业科学, 2016, 53: 197–206. Ma Q, Su J J, Ning X Z, Li J L, Liu P, Chen H, Lin H, Deng F J. Genetic diversity analysis on phenotypic traits of sea island cotton () germplasm resources in Xinjiang., 2016, 53: 197–206 (in Chinese with English abstract).
[23] 李剑峰. 中国长绒棉研究与生产概况. 江西棉花, 2008, 30(3): 8–11. Li J F. Survey of long-staple cotton research and production of China., 2008, 30(3): 8–11 (in Chinese with English abstract).
[24] 孙正文, 匡猛, 马峙英, 王省芬. 利用CottonSNP63K芯片构建棉花品种的指纹图谱. 中国农业科学, 2017, 50: 4692–4704. Sun Z W, Kuang M, Ma Z Y, Wang S F. Construction of cotton variety fingerprints using CottonSNP63K array., 2017, 50: 4692–4704 (in Chinese with English abstract).
[25] 吴大鹏, 房嫌嫌, 马梦楠, 陈进红, 祝水金. 四个国家海岛棉品种资源的亲缘关系和遗传多态性研究. 棉花学报, 2010, 22: 104–109. Wu D P, Fang X X, Ma M N, Chen J H, Zhu S H. Genetic relationship and diversity of the germplasms inL. from four different countries using SSR markers., 22: 104–109 (in Chinese with English abstract).
[26] Ghislain M, Zhang D, Fajardo D, Huamán Z, Hijmans R J. Marker-assisted sampling of the cultivatedcollection using RAPD markers., 1999, 46: 547–555.
[27] Suzuki Y, Sekiya T, Hayashi K. Allele-specific polymerase chain reaction: a method for amplification and sequence determination of a single component among a mixture of sequence variants., 1991, 192: 82–84.
[28] Orita M, Suzuki Y, Sekiya T, Hayashi K. Rapid and sensitive detection of point mutations and DNA polymorphisms using the polymerase chain reaction., 1989, 5: 874–879.
[29] Drenkard E, Richter B G, Rozen S, Stutius L M, Angell N A, Mindrinos M, Cho R J, Oefner P J, Davis R W, Ausubel F M. A simple procedure for the analysis of single nucleotide polymorphisms facilitates map-based cloning in Arabidopsis., 2000, 124: 1483–1492.
[30] Schmalzing D, Belenky A, Novotny M A, Koutny L, Salas-Solano O, EI-Difrawy S, Adourian A, Matsudaira P, Ehrlich D. Microchip electrophoresis: a method for high-speed SNP detection., 2000, 28: e43.
[31] Semagn K, Babu R, Hearne S, Olsen M. Single nucleotide polymorphism genotyping using Kompetitive Allele Specific PCR (KASP): overview of the technology and its application in crop improvement., 2014, 33: 1–14.
Genome-wide screening and evaluation of SNP core loci for fingerprinting construction of cotton accessions ()
LI Le-Chen1, ZHU Guo-Zhong1, SU Xiu-Juan1,2, and GUO Wang-Zhen1,*
1State Key Laboratory of Crop Genetics & Germplasm Enhancement, Hybrid Cotton R&D Engineering Research Center (the Ministry of Education), Nanjing Agricultural University, Nanjing 210095, Jiangsu, China;2College of Agronomy, Xinjiang Agricultural University, Urumqi 830052, Xinjiang, China
Sea-island cotton (), characterized by its extra-long staple (ELS), strong and fine fibers, and disease resistance, provides important natural fiber for textile industry, also key donor for improving agronomic traits of upland cotton. However, compared to, there are few studies on genetic diversity and genotyping in. To obtain the SNP core loci for fingerprinting construction of sea-island cotton accessions, we performed SNP genotyping within 282 accessions using the CottonSNP80K array. A total of 2594 high-quality SNP loci were obtained based on the selective criteria of call frequency for each locus > 0.95, loci with polymorphism, minor allele frequency (MAF) > 0.01, heterozygosity rate < 0.05, and the removal of same genotype. Further, the number of optimized core loci was screened by gradients analysis. With the number of loci increasing, the discrimination ability of the sea-island cotton accessions increased gradually. When the loci were 200, the recognition rate was 89%, and when the number of the loci increased to 1500, the recognition rate was 99%. When the loci were further increased, no significantly improved recognition rate was detected. Based on the detection using the 1500 core loci combination, the average MAF value was 0.14, the average heterozygosity rate was 0.007, and the average polymorphism information content was 0.21. The polyacrylamide gel electrophoresis for the core SNP loci verified the consistency as high as 98.3% between SNP-PCR and chip genotyping. This study provides a set of core SNP loci suitable for constructing fingerprinting of sea-island cotton accessions, which can be used for genetic diversity analysis and fingerprinting identification of sea-island cotton.
gene array; DNA fingerprint; SNP; sea-island cotton
2018-09-20;
2019-01-12;
2019-02-19.
10.3724/SP.J.1006.2019.84123
郭旺珍, E-mail: moelab@njau.edu.cn
E-mail: 2017101076@njau.edu.cn
本研究由国家重点研发计划项目(2017YFD0102000)和江苏现代作物生产协同创新中心(No. 10)项目资助。
This study was supported by the National Key R&D Program for Crop Breeding (2017YFD0102000) and Jiangsu Collaborative Innovation Center for Modern Crop Production Project (No.10).
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20190218.0903.002.html
