胃癌枢纽基因的筛选和预后分析
2019-05-09孙梦雨邱洁萍吴之涵张倩朱爽秋陈博
孙梦雨,邱洁萍,吴之涵,张倩,朱爽秋,陈博
(1. 安徽医科大学 第一临床医学院,安徽 合肥 230000;2. 安徽医科大学第一附属医院 胃肠外科,安徽 合肥 230000)
胃癌是最常见的胃肠道肿瘤之一,是一种全球性的健康问题。近来,胃癌患者的治疗在很大程度上取决于肿瘤病理分期,病理活检仍然在胃癌的诊断中发挥着重要作用[1]。然而,活检对于潜在胃癌患者没有显著作用,大多数具有特定不适症状的患者其实已发展至中晚期,因此全球胃癌患者的5年生存率仅为10%左右[2]。由于对胃癌进行了广泛筛查,在美国这一比率约为30.4%,而韩国的胃癌患者5年生存率超过65%[1]。
研究表明,大量生化标志物参与肿瘤的发生发展过程并可用于早期筛查[3],但许多现有指标过于敏感,在不同种类的肿瘤发生中均有差异表达。因此,有必要进一步探究胃癌发生发展中新的、特异性高的诊断标志物和治疗靶点。目前,高通量测序已成为生命科学领域研究的重要工具,在癌症早期诊断、癌症分级和预后预测等研究中的使用愈发广泛[4],各种生物信息学数据库如GEO也为癌症基因表达谱的数据挖掘提供了平台[5]。本研究从GEO中下载原始数据,通过比较胃癌样本与正常组织样本的基因表达谱筛选出差异表达基因,对差异基因进行生物信息学分析并结合Kaplan-Meier plotter数据库进行预后分析,进而从分子水平研究胃癌的发生、发展机制,为胃癌的诊断、靶向药物研究及判断预后提供有价值的信息。
1 材料与方法
1.1 基因芯片数据
从美国国立生物技术信息中心(NCBI)的GEO数据库(https://www.ncbi.nlm.nih. gov/geo/)中筛选出3套胃癌数据集(GSE13911、GSE33651、GSE79973),其中数据集GSE13911和GSE79973基于GPL570平台([HG-U133_Plus_2]Affymetrix Human Genome U133 Plus 2.0 Array),数据集GSE33651基于GPL2895平台(GE Healthcare/Amersham Biosciences CodeLink Human Whole Genome Bioarray)。
1.2 差异表达基因的筛选
利用GEO数据库自带的在线分析工具GEO2R(https://www.ncbi.nlm. nih.gov/geo/geo2r/)处理原始数据,将数据分为胃癌组和正常组进行进一步分析。差异基因筛选标准:校正后P值<0.05,|logFC|≥1。对每个数据集进行统计分析,使用在线工具Venn diagram(bioinformatics.psb.ugent.be/webtools/Venn/)确定3组数据的相交部分。
1.3 差异表达基因的基因本体论(gene ontology,GO)分析和通路(KEGG)分析
GO分析是大规模功能富集研究的常用方法,基因功能被分成生物过程(biological process,BP)、分子功能(molecular function,MF)和细胞组分(cellular component,CC)3类。京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)是一个整合了大量关于基因组、疾病、化学物质和药物、生物途径和系统功能信息的数据库,可对基因功能进行系统分析。本研究使用David(https://david.ncifcrf.gov/)数据库进行GO分析和KEGG分析,P<0.05且基因数(gene count)≥10,可认为差异具有统计学意义。
1.4 蛋白互作网络的构建和枢纽基因的筛选
利用STRING(http://string-db.org/)数据库构建差异基因的蛋白互作网络(PPI),互作评分>0.4作为阈值条件。将PPI网络导入Cytoscape(www.cytoscape.org/)进行可视化,计算节点的边(degree),具有较高连接度的节点往往对维护整个网络的稳定更加重要,本研究选取前10位为枢纽基因。
1.5 枢纽基因的预后分析
使用Kaplan-Meier plotter(http://kmplot.com/analysis/)数据库来评估枢纽基因的预后价值。对于每个基因,根据mRNA表达值自动将癌症患者分为高表达和低表达两组进行比较,每个基因所用的探针ID显示在表3中,P<0.05认为差异有统计学意义。
2 结 果
2.1 差异表达基因的筛选
该研究选择了3组基因表达谱(GSE13911、GSE33651、GSE79973)。其中,GSE13911含38个胃癌样本和10个正常组织样本,GSE33651含40个胃癌样本和12个正常组织样本,GSE79973含10个胃癌样本和10个正常组织样本。根据调整后P<0.05和|logFC|≥1的标准,GSE13911中共鉴定出3 288个差异基因,包括1 001个上调基因和2 287个下调基因;GSE33651中共鉴定出2 178个差异基因,包括1 588个上调基因和590个下调基因;GSE79973共鉴定出1 405个差异基因,包括个486上调基因和919个下调基因。绘制韦恩图分析以得到3组数据集差异基因的交集,共135个基因在3组中均有差异表达,其中68个基因明显上调,67个明显下调(图1)。
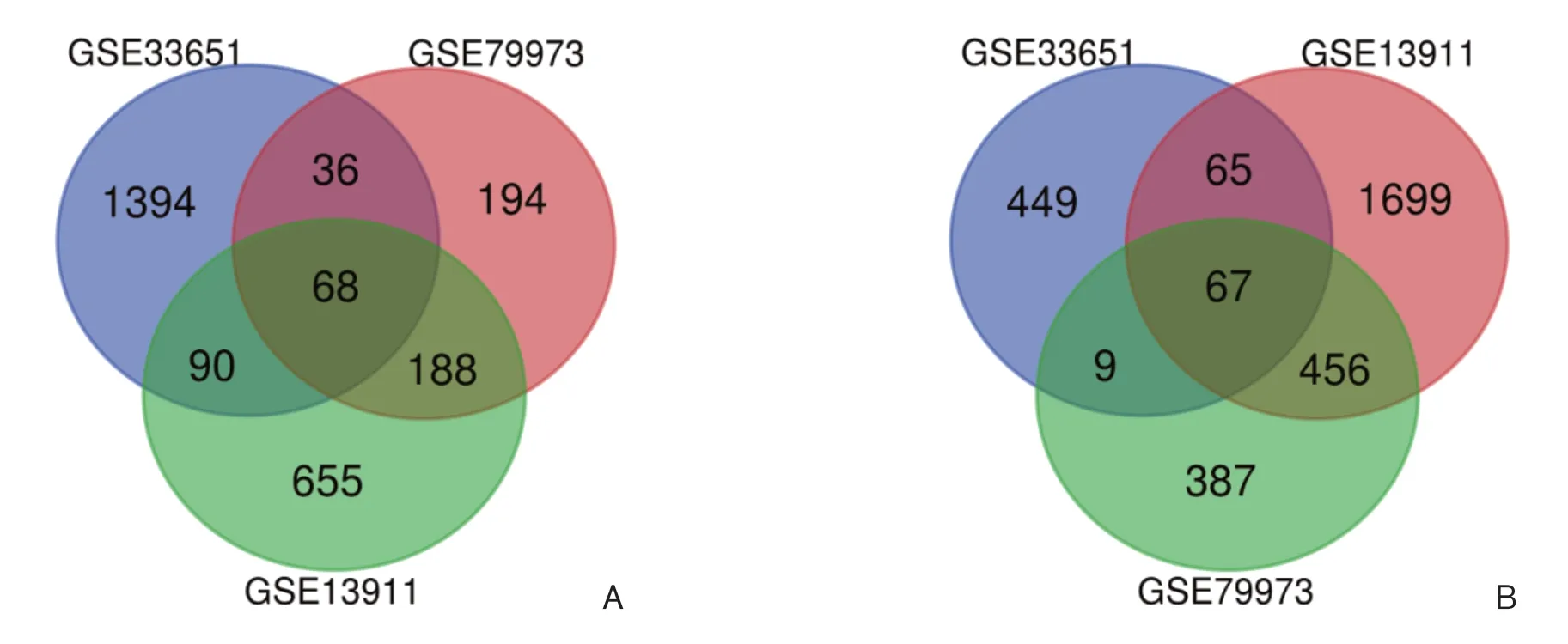
图1 差异表达基因Figure 1 Differentially expressed genes
2.2 差异表达基因的GO和KEGG分析
使用DAVID进行差异基因的GO和KEGG分析。GO分析显示差异基因主要富集的BP:信号转导,细胞外基质组织,细胞黏附,氧化还原过程;主要富集的MF:钙离子结合,相同蛋白结合;主要富集的CC:细胞外外泌体,细胞外空间,细胞外区域,细胞外基质,内质网,细胞表面,内质网腔。KEGG分析显示差异基因主要富集的通路包括:PI3K/Akt信号通路,ECM受体相互作用,黏着斑(表1)。
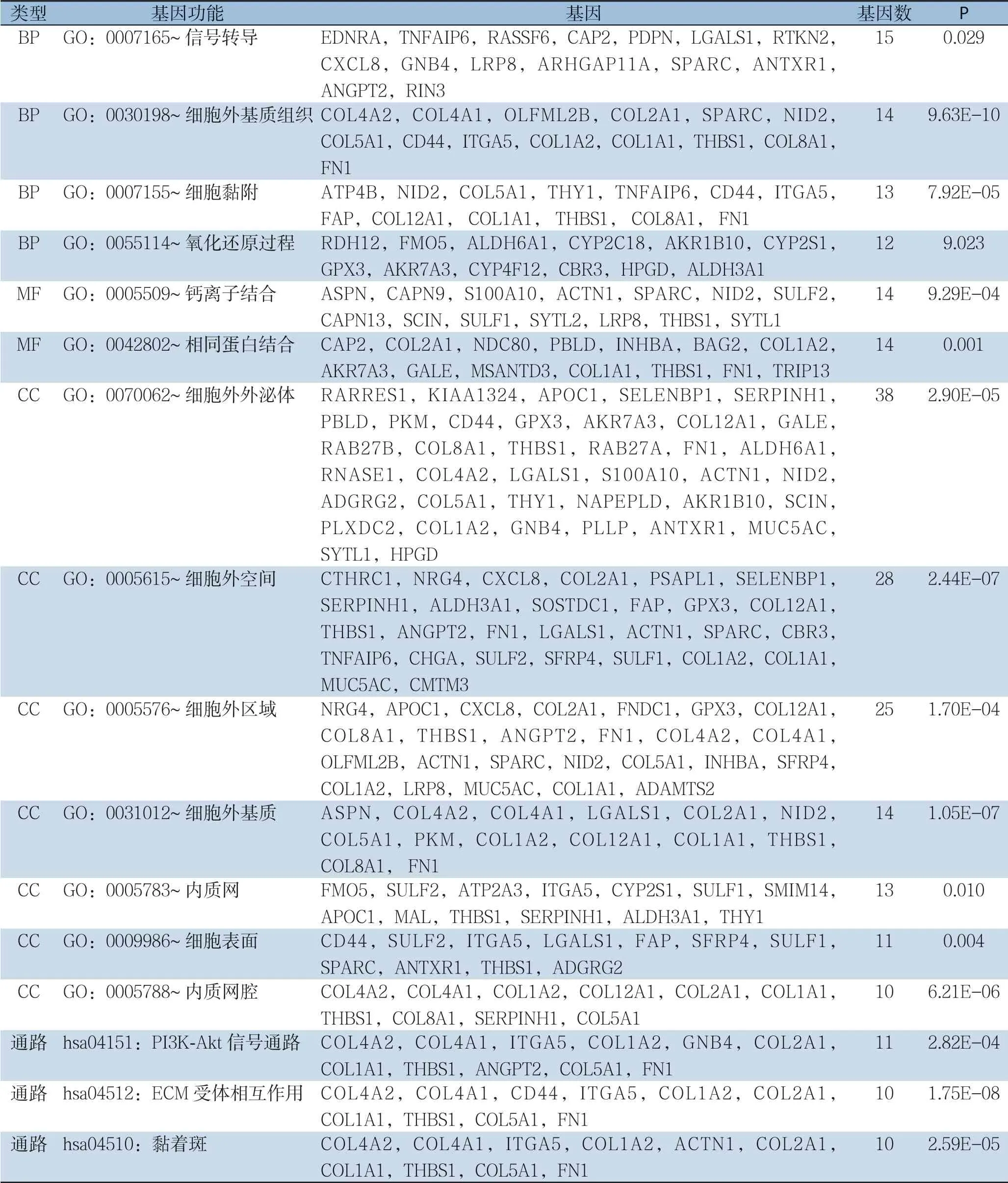
表1 胃癌差异基因的GO和KEGG分析结果Table 1 Results of GO and KEGG analyses of the differentially expressed genes in gastric cancer
2.3 PPI网络构建和枢纽基因鉴定
利用STRING预测差异基因间的相互作用,将数据导入Cytoscape进行可视化,PPI网络共涉及个61节点和170条边(图2)。选取PPI网络中连通度(degree)排序前10的为枢纽基因(表2)。结果表明,最具代表性的基因为COL1A1,连通度为21,其次分别为COL1A2、COL4A1、FN1、THBS1、CD44、COL2A1、COL4A2、CXCL8、COL5A1。
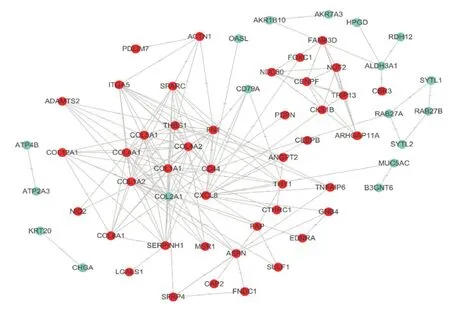
图2 差异表达基因的PPI互作网络(红色:上调,绿色:下调)Figure 2 PPI network of the differentially expressed genes (Red: up-regulated; Green: down-regulated)

表2 枢纽基因名称及连通度Table 2 Names and degrees of the hub genes
2.4 枢纽基因的预后分析
为研究10个枢纽基因的预后价值,本研究使用Kaplan-Meier plotter进行预后分析,共有876例胃癌患者的数据可用于分析总体存活率。其中,除THBS1的上调(HR=0.84,95% CI=0.7~1.01,P=0.065)对胃癌患者总体存活率无明显影响,其余基因的差异表达均影响胃癌患者的总体生存率,9个影响胃癌患者预后的枢纽基因的具体信息见图3。

图3 差异表达基因的预后分析Figure 3 Prognostic analysis of differentially expressed genes
3 讨 论
本研究从GSE13911、GSE33651、GSE79973数据集中共筛选出135个差异表达基因,包括68个上调基因和67个下调基因。此外,GO分析结果表明差异基因主要参与信号转导、钙离子结合、细胞外外泌体等生物学过程。KEGG分析显示差异基因主要富集的通路包括:PI3K/Akt信号通路,ECM受体相互作用,黏着斑。本研究预测了胃癌相关的10个枢纽基因:COL1A1、COL1A2、COL4A1、FN1、THBS1、CD44、COL2A1、COL4A2、CXCL8、COL5A1。
PPI分析结果显示,有6个枢纽基因属于胶原蛋白(collagen,COL)家族,其中COL1A1、COL1A2、COL4A1是连通度排名前三的枢纽基因,这表明胶原蛋白基因与胃癌的侵袭和进展关系最为密切,可能是胃癌的潜在靶点。胃癌发展通常是多步骤的渐进过程,COL1A1在胃癌组织中显著过表达,但从癌前病变到肿瘤阶段其表达量无显著变化[6],这表明COL1A1可能在癌前病变的发生中发挥作用,COL1A1的高表达可能作为胃癌患者的早期诊断标志物,Wang等[7]证实miR-129-5p可以通过选择性降低COL1A1的表达来抑制胃癌细胞的增殖、侵袭和迁移。研究[8-9]表明COL1A2 在结直肠癌和髓母细胞瘤中表达上调,Li等[6]认为COL1A2 的表达与肿瘤大小和侵袭深度有关,其过表达提示不良预后,Ao等[10]证明COL1A2基因的沉默可抑制胃癌细胞的增殖侵袭,同时通过失活PI3k/Akt信号转导途径促进细胞凋亡。COL4A1的突变是多效的,可导致多种肿瘤进展,膀胱癌细胞中过表达的COL4A1通过诱导肿瘤出芽在肿瘤侵袭中起关键作用[11],Jin等[12]研究发现COL4A1高表达有助于乳腺癌细胞的增殖。COL4A1可作为肝内胆管癌预后的生物标志物之一[13],还可通过多种机制如细胞增殖和miRNA介导的转录后修饰驱动胃癌细胞发生曲妥珠单抗耐药[14],这表明靶向COL4A1可能是癌症治疗的潜在方法。COL2A1、COL4A2、COL5A1分子并非特异表达于胃癌,在胃癌研究中相对较少,可作为潜在的靶点进行进一步探索。
一些研究表明,PI3k/Akt途径的许多组分通过扩增、突变和易位比癌症发展中其他途径更易被靶向而导致途径激活[15],纤连蛋白1(FN1)主要参与该途径,在各种恶性肿瘤中表达。FN1参与细胞粘附和迁移过程,有研究[16]表明纤连蛋白的表达可促进胃癌细胞的运动和侵袭,与胃癌进展显著相关。
尽管THBS1在血管生成中的作用已得到充分证实,但其在肿瘤转移中的作用仍有待探索。一项研究[17]表明,THBS1的表达与乳头状甲状腺癌的侵袭程度呈负相关,而其他研究报道THBS1是前列腺癌、结肠癌和乳腺癌细胞迁移和侵袭的有效刺激因子,这表明THBS1在癌细胞侵袭和迁移中的作用产生了混合的结果。有研究[18]表明,与邻近的正常组织相比,THBS1在胃癌组织中显著过表达,Huang等[19]认为肿瘤微环境中的THBS1有调节血管生成、粘附、增殖、侵袭、迁移和免疫的功能,THBS1的多效性取决于环境条件,其与不同受体结合可能对细胞行为和生物过程产生不同甚至相反的影响,THBS1对胃癌进展的影响仍需要进一步研究。
根据癌症干细胞(CSC)理论,癌症干细胞可以驱动肿瘤发生过程,包括癌症的发生、进展、转移和疾病复发[20]。CD44是一种CSC标志物,通过STAT3-cyclin D1途径调节癌症干细胞[21],可在多种肿瘤包括肺癌、结直肠癌、乳腺癌、肝细胞癌、头颈癌和下咽鳞状细胞癌中起预后标志物的作用[22],Wang等[23]研究表明表达CD44的癌细胞易于伪装从而逃脱人体免疫系统的识别和攻击,使肿瘤更易转移。
趋化因子(CXC)在肿瘤生物学中起到重要作用。虽然一些CXC趋化因子通过将免疫细胞吸引到肿瘤组织中或通过抑制肿瘤新血管形成而具有抗肿瘤活性,但其它趋化因子可通过直接刺激生长、增强细胞运动或刺激血管生成来促进肿瘤生长、侵袭和转移[24-26]。已知CXCL8在许多恶性肿瘤中是强血管生成因子,有研究证明人胃癌细胞表达的CXCL8蛋白与肿瘤血管的数量相关,表明CXCL8确是胃癌的血管生成因子[27],可作为胃癌的分子靶标进行研究。
为验证生物信息学分析的结果,本研究使用Kaplan-Meier plotter 数据库检验枢纽基因的预后价值。除THBS1外,所有枢纽基因均与胃癌患者的总体生存率显著相关,此外,预后结果表现出与生物信息学分析基本相同的表达趋势,从而验证了本研究方法的准确性。本研究可为将来胃癌的分子机制、生物标志物及靶向药物的研究提供思路,今后仍需分子生物学实验进一步探讨差异基因在胃癌进展中的具体作用机制。
