腐败干酪中微生物种类的分离与鉴定
2019-04-26杨攀黄丽唐艳冯玲农皓如李玲曾庆坤
杨攀,黄丽,唐艳,冯玲,农皓如,李玲,曾庆坤
(中国农业科学院广西水牛研究所南宁530001)
0 引 言
南方水牛乳主产区气候相对湿热,而水牛饲养模式以规模化与散户并存,机械挤奶与手工挤奶并存,生乳菌落总数偏高,加上干酪生产需要将杀菌温度控制在较低水平。这些因素均不利于产品加工、成熟、贮藏过程中的微生物控制,造成广西区内企业生产的干酪极易发生霉菌、酵母污染。加上成熟干酪需长时间在低温下进行成熟,生产周期长,增加了微生物控制难度,同时也增加了企业前期冷藏存贮设备投入。
霉菌和酵母几乎在任何食品上都可以生长:谷物、水果、蔬菜、坚果、乳制品等[1]。Filtenborg等认为与各食品霉变相关的霉菌只有少量几种,大部分属于青霉、曲霉和镰刀霉。不同的青霉菌会在不同食品冷态储存中出现,P.roqueforti和P.commune经常会导致硬质干酪的腐败[2]。近平滑假丝酵母、黏红酵母、克鲁维酵母汉逊(氏)德巴利酵母菌是酸奶和其他发酵乳制品中主要的腐败菌[3]。
微生物分离和纯化主要指的是在相应技术条件辅助下,从高度混杂、种类众多的细菌群中获取单一菌落的过程。其通过对目标菌株适宜培养条件、培养基进行优化和选择来实现分离、纯化的目的[4]。
随着科学技术的进步,微生物的分析手段也日趋完善,特别是以DNA为基础的分子生物学技术取得较大进展,并广泛应用到食品微生物领域,如实时定量荧光聚合酶链式反应(polymerase chain reaction,PCR)技术[5]、变性梯度凝胶电泳技术(DGGE)[6-7]、末端限制性片段长度多态性分析技术(TRFLP)[8-9]等。近几年来,高通量测序技术快速发展,代表性的测序平台包括Illumina、Roche、Ion Torrent等[10],为微生物群落分析提供了新的手段。高通量测序技术可以检测到样本中传统纯培养不能发现的低丰度细菌种类,从而更加准确、全面地反映样本中的微生物群落结构[11]。
本实验主要是通过传统培养法和高通量测序法研究腐败干酪中微生物的种类和分布情况,为筛选抑制干酪腐败的乳酸菌做基础,对采用天然的防腐剂延长干酪货架期具有一定的意义。
1 材料与方法
1.1 材料与仪器
腐败干酪,孟加拉红培养基(虎红琼脂),马铃薯葡萄糖琼脂(PDA),乳酸酚棉蓝染色液,DNA提取试剂盒,rTaq DNA Polymerase,BSA,Buffer,dNTPS,AxyPrepDNA凝胶回收试剂盒,Tris-HCl,PCR仪:ABI GeneAmp®9700型,小型离心机,电泳仪,恒温水浴锅,QuantiFluorTM-ST蓝色荧光定量系统。
1.2 实验方法
1.2.1 样品前处理
随机抽取霉变干酪,根据不同的霉变颜色分为2组样品,每组样品包含3个样,分别称取霉变干酪2 g放入18 mL无菌水中浸泡15 min,随后不断研磨震荡至样品均匀分散,备用。
选取霉变干酪的表面霉变部分和内部未霉变部分,分别命名为MGL1、MGL2、MGL3、YGL1、YGL2、YGL3,将样品送至上海美吉生物有限公司进行高通量测序。
1.2.2 微生物的分离与纯化
在无菌操作台中,吸取0.5 mL的样品,置于4.5 mL的无菌水中,以10倍稀释法对其进行梯度稀释。依次做6个稀释度10-1、10-2、10-3、10-4、10-5、10-6,吸取100 μL稀释度为 10-3、10-4、10-5、10-6的稀释液分别均匀的涂布于孟加拉红培养基和PDA培养基中,同时用无菌水做平行实验。稍后将平板倒置,在28℃±1℃培养箱中培养3-5天,观察并记录[12]。
根据菌落形态进行分类,用接种针挑取形态学特征不同的菌落,在孟加拉红培养基和PDA培养基进行划线分离,从无明显杂菌开始,28℃±1℃连续培养三代,直至得到单个菌落纯化菌种。
1.2.3 微生物的鉴定与保存
观察菌落在固体培养基上的特征,包括菌落生长速度、大小、形态构造、颜色、质地、高度、表面特征、背面特征、边缘特征以及培养基的颜色变化、气味等。
观察显微镜下菌体的形态。在干净的载玻片上加一滴乳酸酚棉蓝染色液,用接种针从菌落的边缘处取带有孢子的菌丝,放入乳酸酚棉蓝染色液中,再小心地把菌丝散开,然后用盖玻片盖上,注意不要产气,先低倍镜检,必要时再换高倍镜观察。
通过镜检确保菌株一致,用接种针挑取单个菌落,保存菌种于PDA斜面上,于4℃冷藏保存。
1.2.4 总DNA提取方法
使用FastDNA®RSPIN Kit for Soil试剂盒抽提基因组DNA,具体步骤按照说明书进行。
1.2.5 PCR扩增18S rDNA基因序列
扩增18S rDNA基因引物为真菌通用引物,如表1所示。

表1 PCR扩增引物
将上述提取的DNA作为扩增模板,采用20 μL反应体系进行PCR扩增,条件如表2所示,PCR循环参数如表3所示。
1.2.6 样品复杂度分析
采用稀释曲线和菌种丰富度指数(Chao1)、辛普森指数(Simpson)、香农指数(Shannon)等系列统计学分析指数评估样本中微生物群落的物种丰富度和多样性。

表2 PCR扩增体系

表3 PCR循环参数
1.2.7 物种注释与评估
为了得到每个OTU对应的物种分类信息,采用RDP classifier贝叶斯算法,对97%相似水平的OTU代表序列进行分类学分析,并分别在各个分类水平,即:domain(域),kingdom(界),phylum(门),class(纲),order(目),family(科),genus(属),species(种)统计各样本的群落组成。
2 结果与分析
2.1 形态学鉴定
本实验通过严格的实验操作,筛选、分离和纯化霉变干酪,总共得到3株菌,酵母菌1株,青霉菌属2株,编号为A7、A8、A9,其中A7为酵母菌属,A8、A9为青霉菌属。
2.1.1 菌落形态描述
在28±1℃连续培养到3代,得到单个菌落的纯化菌种,培养至第4天拍照记录。如图1所示。
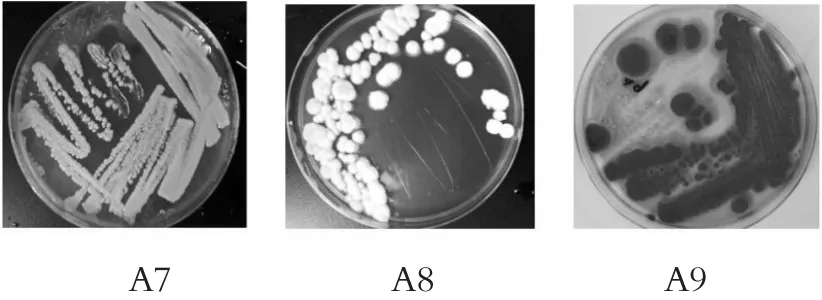
图1 孟加拉红培养基上各菌落图
根据各菌落在不同培养基上的生长状况,进行菌落形态描述,如表4所示。
2.1.2 菌体微观形态
培养至第4天,用接种针挑取少量菌体进行镜检,结果如图2所示。
微观形态描述如表5所示。

表4 菌落形态特征描述

图2 各菌落微观形态图

表5 菌体微观形态描述
2.2 高通量测序
2.2.1DNA提取与PCR扩增结果
利用18S rDNA来鉴定真菌种类,提取到的总DNA以及扩增的电泳图见图1,根据PCR电泳图可知1-5号样品目的条带大小正确,浓度合适,可进行后续实验,而6号样品的目的条带未检测到,无法进行后续测序实验。
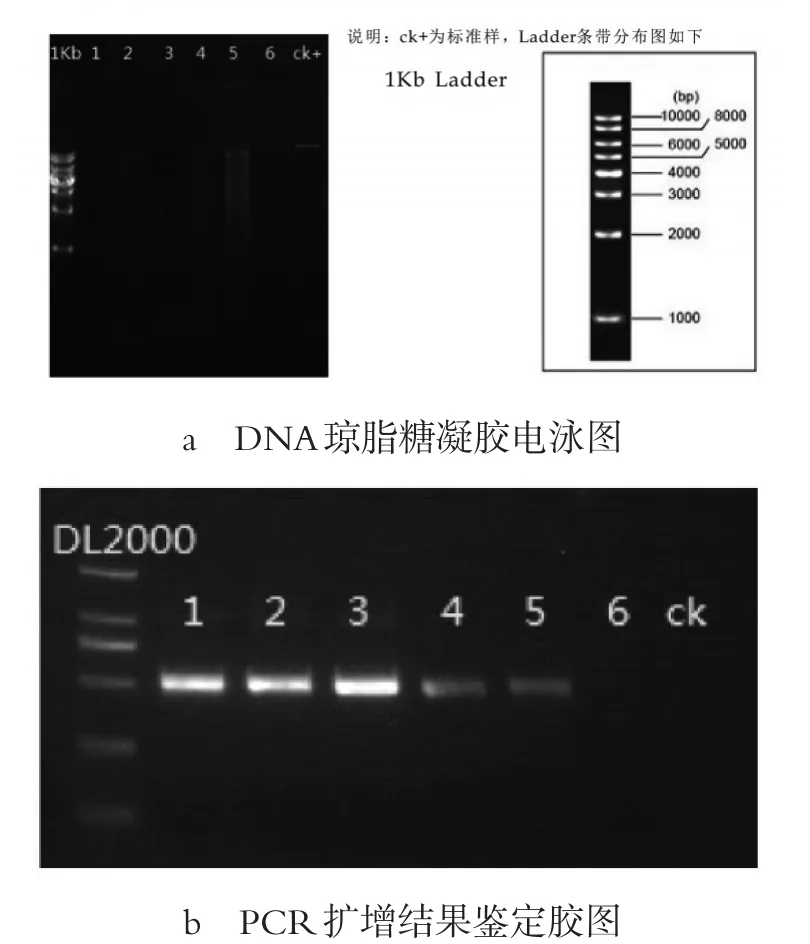
图3 基因组DNA提取与PCR扩增电泳图
2.2.2 样品复杂度分析
2.2.2.1 OTU聚类
经过对所有样品的测序结果进行分析处理,去掉测序质量差的序列后,MGL1-3 3个样品共得到96046条高质量序列,序列平均长度为402.2,YGL1-22个样品共得到73 643条高质量序列,序列平均长度为403.78,每个样品得到的优化序列及序列平均长度见表6。对5个样品的有效数据进行聚类分别得到9个、6个、5个、17个、37个OTU。
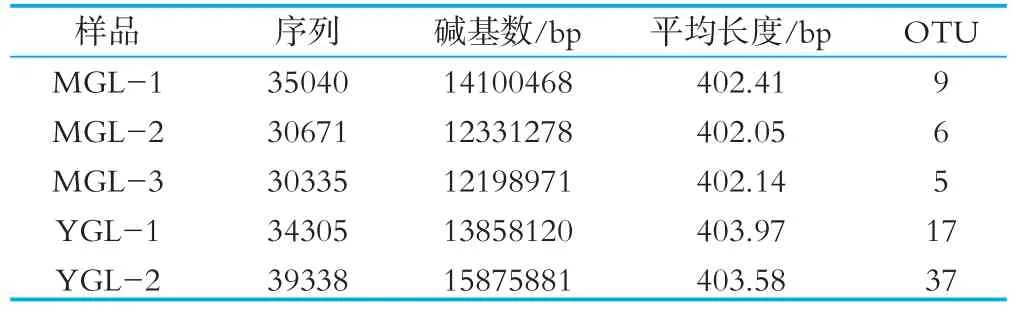
表6 各样品序列统计及OTU聚类表
2.2.2.2 稀释曲线
稀释曲线是从样本中随机抽取一定数量的个体,统计这些个体所代表的物种数目,并以个体数与物种数来构建曲线。它可以用来比较测序数据量不同的样本中物种的丰富度,也可以用来说明样本的测序数据量是否合理。采用对序列进行随机抽样的方法,以抽到的序列数与它们所能代表OTU的数目构建rarefaction curve,当曲线趋向平坦时,说明测序数据量合理,更多的数据量只会产生少量新的OTU,反之则表明继续测序还可能产生较多新的OTU。因此,通过作稀释性曲线,可得出样本的测序深度情况。

图4 样品在相似水平为97%的稀释曲线
从图4可以看出,每个样品在其测序数量下均达到了平稳期,即增加测序数量也没有新的OTU出现,因此,本次测序深度足以检测到样品中真菌类群。
2.2.2.3 样品中真菌类多样性分析
本实验采用 97%水平下的 ace、chao、shannon、simpson和微生物的OTU数量对样品中真菌多样性进行分析,具体指标见表7所示。
Shannon和Simpson均是用来估算样本中微生物多样性指数之一。Shannon值越大,说明群落多样性越高,但该值并非单纯表示样品中存在物种的种类多少,还包含样品中各物种所占比例,即不同物种之间个体分配的均匀性,分布越均匀其多样性越高。simpson在生态学中常用来定量描述一个区域的生物多样性,Simpson指数值越大,说明群落多样性越低。Simpson多样性指数中稀有菌种所起的作用较小,而普遍菌种所起的作用较大。两个指数都是反映样品中菌群丰富度和均匀度的综合指标。
由表5可以看出,样品的测序覆盖率在99%以上,样品YGL-2的Shannon值最大,Simpson指数最小,OTU最大,说明它的微生物种类比较丰富,群落的多样性比较高。样品MGL-3的Shannon值最小,说明它的微生物种类多样性比较低,物种分布不均匀,结合图2可以得知,YGL-2中微生物种类有13种,MGL-3中仅有3种,与多样性指数Shannon和Simpson相一致。

表7 不同样品在97%相似水平下的多样性指数
2.2.3 物种注释
不同分类水平上的物种注释,结果见图2。
由图5可看出,腐败干酪外表霉变部分在目上的分类主要是酵母目和散囊菌目,腐败干酪内部的菌种种类丰富,不仅含有酵母目和散囊菌目,而且包含有真菌银耳目、肉座菌目、锁掷酵母目,真核生物以及其他种类。腐败干酪外表霉变部分在科水平上的分类主要是发菌科、酵母科和耶罗威亚酵母科,腐败干酪内部的菌种,还包含有丝孢酵母科、未分类的肉座菌科、未界定的银耳科和锁掷酵母科以及Cystofilobasidiaceae、Mammalia和Choreotrichia。腐败干酪中霉变部分在属水平上的分类的主要菌群为青霉(发菌科,霉菌)、德巴利(氏)酵母属、耐碱酵母属和未分类的酵母菌属,内部未霉变干酪的菌群种类更丰富,不仅包含有霉变部分的菌群,还有未分类的真核生物、丝孢酵母属、木拉克酵母属、双足囊菌属以及Mammalia和Choreotrichia等。
3 结 论
通过传统的微生物培养法,经过分离、纯化得到3株不同的微生物,通过镜检和微生物形态学分析,得出三株微生物分别属于酵母菌属和青霉菌属。
通过18S rDNA高通量测序法,根据稀释曲线、OTU聚类以及样品的多样性分析来反映腐败干酪中微生物的菌群,得出腐败干酪中微生物菌群主要有青霉菌属、德巴利(氏)酵母属、耐碱酵母属、未分类的酵母菌属、丝孢酵母菌属、木拉克酵母属、双足囊菌属以及未分类的真核生物。
3个霉变干酪样品中50%~80%为青霉菌属,20%~40%为德巴利(氏)酵母属,其余为未分类的酵母菌属。另外3个未霉变部分的微生物菌群比较丰富,包含有丝孢酵母属、木拉克酵母属、双足囊菌属以及Mammalia和Choreotrichia等13种微生物。
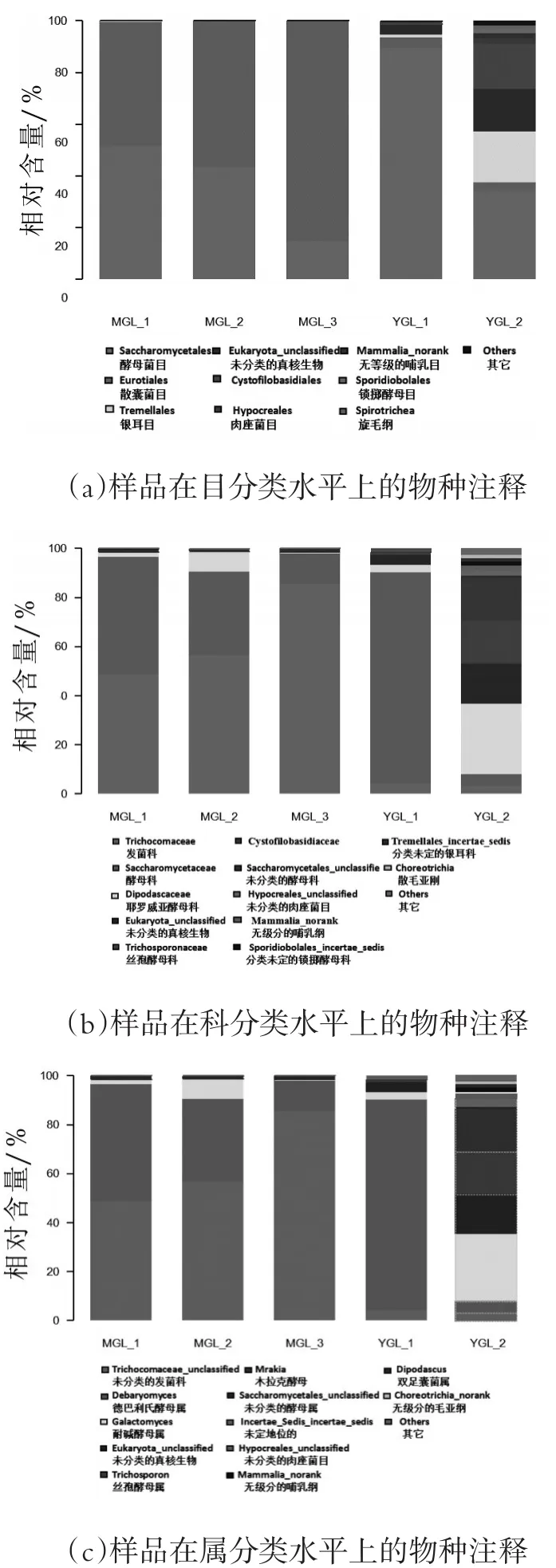
图5 样品在目、科、属水平上的物种注释
[3]LOUREIRO V,QUEROL A.The prevalence and control of spoilage yeasts in foods and beverages[J].Trends in Food Scienceand Technology,1999,10,356-365.
