Alu元件在染色质三维结构层次上的生物信息学分析
2019-03-19何超沈文龙李平张彦曾晶殷作明赵志虎
何超,沈文龙,李平,张彦,曾晶,殷作明,赵志虎
Alu元件在染色质三维结构层次上的生物信息学分析
何超1,2,沈文龙2,李平2,张彦2,曾晶1,殷作明1,赵志虎2
1. 西藏军区总医院,拉萨 850000 2. 军事科学院军事医学研究院生物工程研究所,北京 100071
染色质在细胞核内三维高级结构包括最底层的核小体、核小体组成的“串珠”结构、螺线管纤维结构、染色质/DNA环结构(chromatin/DNA loop)、拓扑结构域(topologically associated domain, TAD)等多层次结构。其中,TAD因在不同的细胞类型中相对稳定且保守,被认为是染色质三维结构的基本单元。Alu元件是一种哺乳动物基因组中占据了较大比例的短散在重复元件,其广泛存在且种类繁多,目前关于Alu元件功能上的研究尚不透彻。本研究对Alu元件与染色质三维结构的关系进行研究,分析了Alu元件在染色质三维结构形成中的作用,并通过染色质三维结构上的距离关系对Alu元件子族的演化流程进行了探索。结果发现,Alu元件参与的染色质间相互作用在高强度的染色质相互作用中的比例随着强度增加而逐渐增高,表明Alu在染色质三维结构构建的过程中发挥了重要的作用。同时,研究还发现Alu元件在染色质上相互作用的强度与进化上的关系存在着一定的正相关性,表现在一维序列进化距离上比较接近的Alu元件在染色质的三维结构上也会彼此相互靠近。
染色质三维结构;Alu元件;生物信息;进化树
短散在元件(short interspersed elements, SINEs)是以散在方式分布于基因组中的较短的重复序列,其单元长度通常在50 bp以下。Alu序列是哺乳动物的短散在元件中所占比例最多的一种重复元件[1]。Alu序列的左右两端各分布着一个不等长的、包含一个RNA聚合酶Ⅲ启动子序列的单体[2],左侧单体比右侧单体短31 bp且只有左侧单体的RNA聚合酶Ⅲ启动子具有活性。研究表明,在Alu元件附近往往富集着长非编码RNA (long non-coding RNAs, lncRNAs),可辅助lncRNAs在细胞核内进行定位[3],而且还能在基因组转录过程中能够充当剪接受体,抑制mRNA的翻译并导致基因组不稳定[4]。此外,还有研究发现Alu元件在基因组中与增强子(enhancer)广泛相互作用[5~7]。
染色质在细胞核内的结构与蛋白质的三维高级结构相似,包括基础的核小体、核小体组成的“串珠”结构、螺线管纤维结构、染色质/DNA环结构(chromatin/DNA loop)、拓扑结构域(topological associated domain, TAD)等多层次结构[8,9]。其中,TAD因在不同的细胞类型中相对稳定且保守,被认为是染色质三维结构的基本单元[10]。目前对于染色质三维结构的研究主要基于近距离连接反应的染色体构象捕获技术、基于图谱技术根据多聚物聚合等原理进行计算机建模、基于CRISPR的DNA片段编辑等的遗传学技术,以及基因光谱反射的显微镜技术。迄今为止,对染色质结构的直接观测受到技术的限制,难以直接在高分辨率下观测染色质的动态变化,因此研究者们采用先将染色质内的相互作用固定下来,通过分子生物学手段进行分析,从而推断生理条件下真实的染色质动态结构。目前对染色质三维结构的研究方法主要包括研究点与点之间距离水平的3C技术[11]、研究一点到基因组范围内其他位点距离水平的4C技术[12]、研究已知范围内多点到多点间相互作用的5C技术[13]、全基因组内染色质相互作用的Hi-C技术[14,15]及进一步衍生出的DNase Hi-C[16]、Micro-C[17]、Capture Hi-C[18]、COLA[19]、C-walk[20]等技术[21,22]。本研究通过生物信息学方法,结合Hi-C实验数据,在染色质三维结构层次上对Alu元件在染色质三维结构形成中发挥的作用进行探索,并根据染色质三维折叠数据对Alu元件的相关功能进行分析。
1 材料与方法
1.1 数据集来源
Hi-C实验数据来源于Rao等[19]所公开的数据集,包括GM12878、IMR90和K562 3个细胞系,均可在最高5 kb分辨率下开展研究。Alu元件数据来自UCSC genome browser repeat masker,包含了Alu元件不同子类在基因组上所在位点的信息[23]。参考基因组选取为人类参考基因组hg19版本。
1.2 数据预处理
目前已经有较多处理Hi-C数据的比较成熟的软件,如GenomicInteractions[24]、HiCPlotter[25]、HiC-Pro[26]等,其中GenomicInteractions可用于分析特殊位点间相互作用网络,然而这一软件并不能整合多组学数据,也不能够进行后续的功能挖掘等分析[24];HiCPlotter侧重于将Hi-C相互作用热图与相应的包括组蛋白修饰等表观遗传特征的结合,但并未关注染色质上特殊序列间的相互作用[25]。利用HiC-Pro软件对原始数据进行预处理,因Hi-C数据是包含相互作用的两个基因组片段的整合序列,通过将原始测序序列比对到基因组上,得到发生相互作用的两个基因组位点。过滤掉自连接或未连接的无用、冗余相互作用对,将剩余相互作用对进行整合汇总,以便后续分析处理。
1.3 Alu元件相互作用频率分布分析
利用开源的R语言及附属软件包对Alu元件不同子类相互作用频率分布进行分析。将不同位点的全部相互作用汇总,设定相互作用上下阈值,超过上限阈值认为其过度连接,低于下线阈值认为其为染色质内随机连接。根据上下阈值进行过滤后,按照相互作用频率强度将所有位点的相互作用分成若干子集。根据Alu元件所在的位置生成随机位点组成随机组,将每一组内的相互作用数据与随机组数据相比较,从而帮助用户判断特殊位点的相互作用在生物学上是否显著,以及是否与染色质三维结构相关。
1.4 基于染色质三维结构的Alu元件演化分析
将Alu元件子族参与的相互作用从全局染色质间与染色质内相互作用中提取出来,并根据Alu元件不同子族进行分类。然后根据不同Alu元件子族间的平均相互作用强度,推算三维结构上的距离关系,并与已知的Alu元件进化树相比较,从而判断这些元件之间是否存在关联。
2 结果与分析
2.1 Alu元件广泛参与染色质三维折叠
首先使用bowtie2软件对数据集进行序列比对,通过比对后得到染色质相互作用序列对。将全部相互作用序列对进行统计,得到不同位点间相互作用频率,根据上下阈值进行过滤,然后按照相互作用频率大小分成若干子集,将每一组内的相互作用数据与随机组数据相比较,从而判断Alu元件的相互作用在生物学上是否显著,以及是否与染色质三维结构相关。为了判断差异是否真实存在,根据相应Alu元件的位置信息,生成了与之对应的随机对照组进行比较[27]。
经计算发现,在不同细胞系中Alu元件所参与的相互作用比例结果显示均具有较高的一致性,Pearson相关系数均在0.997以上,说明Alu元件参与相互作用的比例在不同细胞系中是相对一致的,这些结果提示在不同细胞系中,Alu元件可能对染 色质三维结构的构建、传递和维持发挥着重要的 作用。
2.2 Alu元件参与高强度染色质相互作用
根据UCSC上Repeat Masker数据集中Alu的位点信息,对其在不同强度的染色质相互作用中所占的比例进行了分析,因Hi-C实验数据是若干条序列,其中每条序列为染色质上两段相互靠近的不同位点连接形成,通过序列双端比对即可获取全基因组范围内的染色质相互作用水平[28],将全部相互作用根据上下阈值进行过滤,按照相互作用频率大小分成若干子集,根据Alu元件的位点信息,将Alu元件参与的相互作用从全局相互作用中提取出来,进一步计算其在不同子集中所占的比例。
结果发现,Alu元件在高强度的相互作用中所占比例随着强度增加而逐渐增高,这表明Alu在染色质三维结构构建过程中发挥了重要的作用(图1)。之前的研究也表明,Alu富集的区域会使序列有更高的GC含量,从而也会有更多的CpG岛与甲基化水平,这些使序列和转录调控变得更加灵活,也就更有可能富集着较多的染色质相互作用。同时Alu序列经常在细胞中发挥增强子的作用,这也可能是其广泛参与高强度染色质相互作用的原因[29]。

图1 Alu元件参与的染色质相互作用分布
2.3 从染色质三维结构的角度分析Alu元件的进化过程
本研究从染色质三维结构层面上对Alu元件家族中不同子类之间的分布进行了分析与验证。通过计算不同子类之间的相互作用强度,即染色质三维结构层次上的平均距离,结果见图2。颜色越深表明两种元件在染色质三维结构层面上平均距离越近,可以看出AluJ与AluS子家族呈现出一个局部的热区,而AluY子家族则与其余子家族间相互作用强度都较低。
为排除细胞类型特异性因素的影响,对GM12878、K562和IMR90 3个细胞系中Alu家族所有二级子类所参与的相互作用强度进行了一致性检验。结果发现,3个细胞系的分析结果均具有相对较高的一致性,尤其是GM12878与K562,Pearson相关系数可以达到0.827,说明不同Alu子类参与的相互作用强度水平并不是细胞类型特异的,而是普遍存在且相对一致的。
从上面的研究中可以发现,Alu的3个子家族AluJ、AluS和AluY在相互作用强度上存在着差异,因此对这3个子家族的相互作用强度进行了进一步的观察,并根据三者之间相互作用强度绘制了热图(图3),结果发现AluJ和AluS都是与自身相互作用最强,与相邻的子家族次强,与距离最远的子家族相互作用强度最弱,也就是说,AluJ与AluS、AluS与AluY在染色质三维结构上距离较近,而AluJ与AluY距离较远。这一发现与之前文献中报道的仅仅根据一维核酸序列分析得到的Alu家族进化树(图4)相一致[30,31],AluY自身作用强度较弱,这也许与其较近年代才产生有关,相对于AluJ (21 575个)、AluS (16 536个)而言,AluY的数量较少,只有1731个,分布也较为分散,相互作用强度较弱。因此推测,在进一步的进化过程中,当Alu继续衍生出新的子家族,AluY数量在人类基因组中逐渐增多时,其参与的相互作用的强度也会随之增加。根据相互作用强度做了层次聚类,发现与上面的推测也基本一致(图5)。
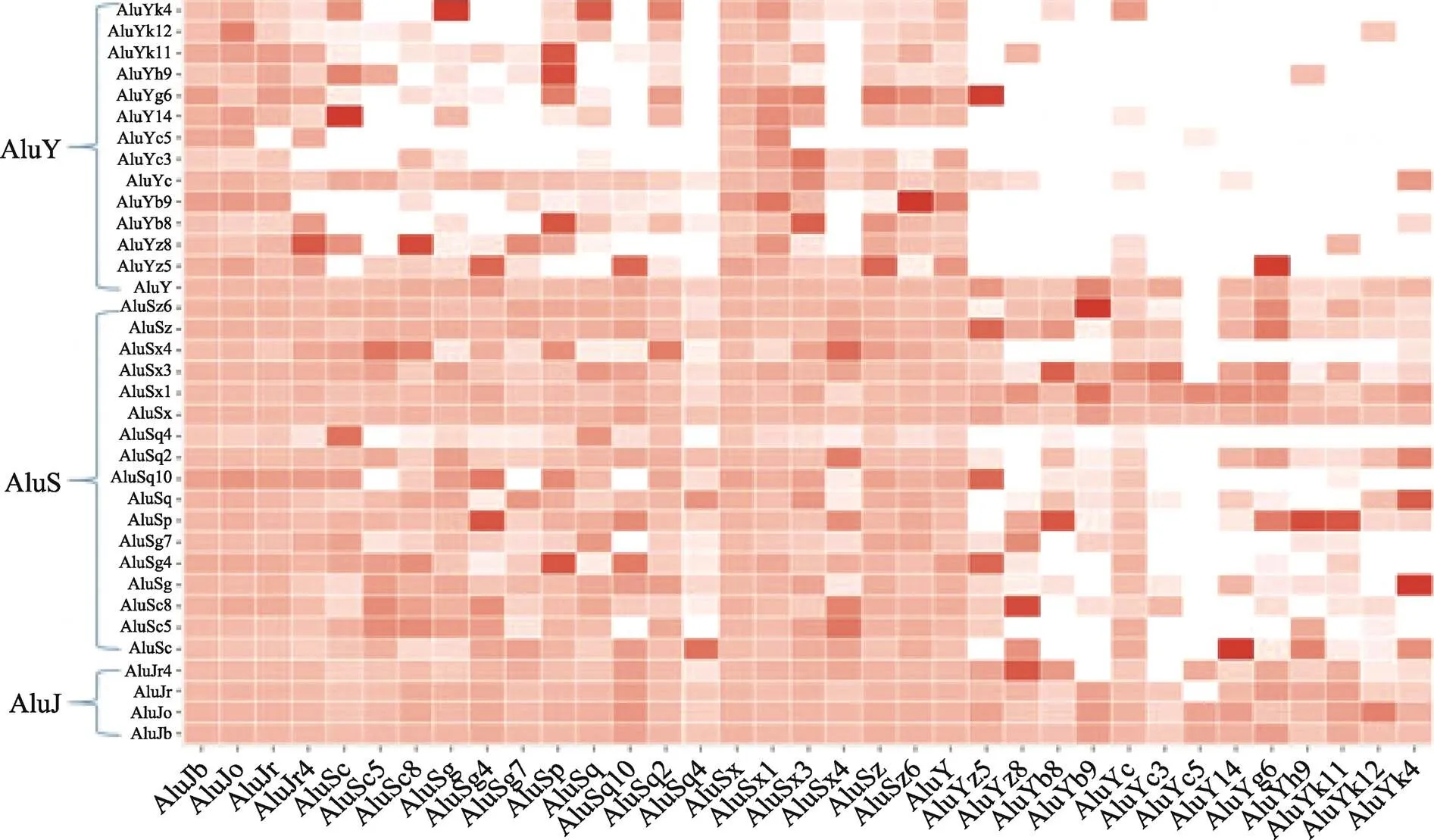
图2 Alu元件子族间相互作用强度分布
横纵坐标代表相应的Alu元件子类。

图3 Alu元件3个子族间相互作用强度分布
基于上述结果,推断Alu元件在染色质相互作用的强度与进化上的关系是存在着一定的正相关性,也就是说一维序列进化距离上比较接近的Alu元件在染色质的三维结构上也会彼此相互靠近。至于是因为它们进化上首先随机跳跃扩展到基因组中的不同区域,但是由于一维序列比较类似因而结合类似的、共同的调控因子从而导致所在区域染色质三维结构相互聚集,还是因为原本在染色质三维构象上,原始的序列位点就比较接近,在进一步的演化过程中,新出现的元件优先扩展到三维距离上更近的区域最终使得所有这些同族的Alu子族在三维分布上也比较接近,还需要后续更多的实验验证。
3 讨论
3.1 Alu元件在染色质三维构象的动态折叠中发挥了重要的作用
细胞核是真核生物独有的一种细胞器,具有高度不均一、严格区室化、亚结构相对独立等特点,可以从外至内分为核被膜、核纤层、核基质、染色质及核仁层结构,其中核纤层、核基质、核仁3者相互连接,构成细胞核内的骨架及支撑结构,染色质围绕这一骨架以复杂而精致的三维形态高度折叠,这一折叠能够维持着相对稳定且高度动态变化的结构,在DNA复制、重排、重组、修复以及基因转录调控等方面发挥重要的功能[16,32,33]。
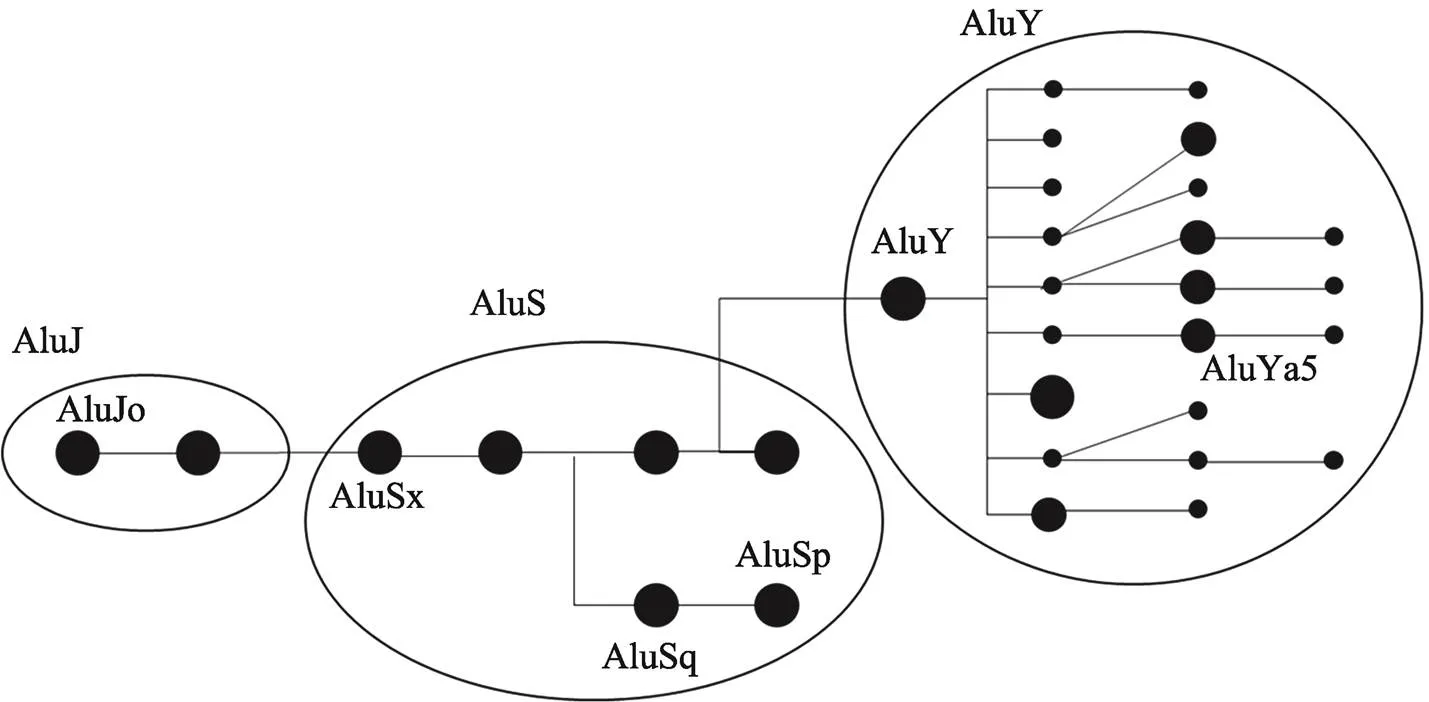
图4 Alu元件进化树
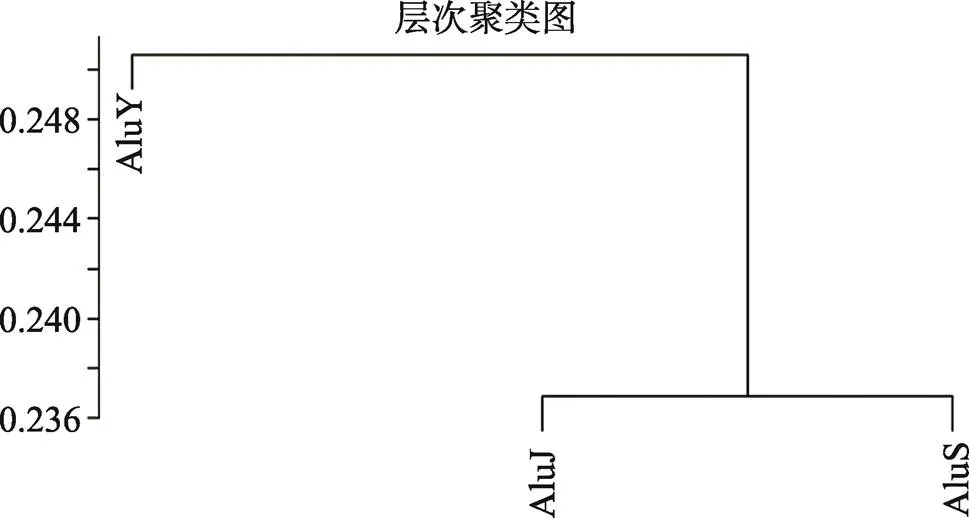
图5 Alu元件相互作用强度层次聚类图
随着技术的发展,人们逐渐发现染色质与蛋白质的多级折叠结构相似,可以分成若干个不同的层级。DNA双螺旋链通过缠绕形成核小体,核小体通过连接DNA形成核小体“串珠”结构,进一步转曲折叠形成螺线管显微结构。共定位、共表达的染色质位点又在染色质内相互靠近,形成染色质或DNA环结构。染色质环又进一步根据相互作用折叠成拓扑结构域(TAD)[28,34]。经研究发现同一TAD内染色质会存在较强的相互作用,TAD间的则相对较弱,且TAD的边界在不同物种、不同类型的细胞系中会相对稳定,因此TAD被认为是染色质三维构象的结构和功能基本单元。近期Bing等[35]整合比较了21种人类不同类型细胞的染色质相互作用,进一步证实TAD边界在不同类型细胞相对保守,且发现了局部相互作用高度富集的染色质活跃区域(frequently interacting regions, FIREs)与超级增强子部分重叠,表明染色质三维折叠与细胞生命活动紧密相关。
在哺乳动物基因组中,一半以上都是各类重复元件序列。重复元件根据结构和复制形式,可分为串联重复序列(tandem repeat)和转座元件(transposable elements)。转座元件又可分为长散在重复元件(long interspersed nuclear elements, LINE)与短散在重复元件(short interspersed nuclear elements, SINE)等[36]。短散在重复元件又根据结构等可分为Alu元件与Mir元件等不同子类。其中Alu元件在哺乳动物尤其是灵长类动物基因组中广泛存在且比例较高,然而对其功能上的研究却一直进展缓慢,早起甚至被认为是垃圾序列[1,37]。最近的一些研究才发现Alu元件在基因组的结构及基因表达调控等方面发挥了一些功能[38,39]。
本研究发现Alu元件在染色质三维构象的动态折叠过程中发挥了重要的作用,在不同种类的细胞系中均参与了较多高强度的染色质间相互作用。且目前多有研究发现,Alu元件与染色质内的增强子有强烈的相互作用。这些都表明Alu元件可能在染色质三维结构的构建、传递和维持中发挥着作用。染色质在细胞核内维持着动态平衡、永不缠绕的结构状态,有着相对稳定的结构,而这样的结构需要一定的网络节点加以固定,因此认为Alu元件在其中可能就发挥着高强度的相互作用网络中心固定节点的作用,从而维持着染色质动态结构的稳定。
3.2 染色质三维构象与Alu元件的演化存在关联
Alu元件在短散在重复序列中广泛分布,且种类繁多[40],目前已有较多根据序列层面分析Alu家族演化过程的研究,但尚未有人从其在染色质三维结构上的分布来分析其演化过程。本研究基于染色质三维结构的数据,对Alu元件的演化历程进行了分析,证明了一维序列演化关系上相对靠近的子类,在染色质三维结构中的相互作用强度也会较高,并用层次聚类的方式得到了基于染色质相互作用强度的进化树。这些工作表明,Alu元件在染色质上相互作用的强度与进化上的关系存在着一定的正相关,可目前的工作尚无法对这一现象的原因进行证实。一方面有可能是因为在进化过程中,Alu元件初代子族随机跳跃扩展到基因组内,某些调控因子对这些初代子族进行调控,使得这些子族所在的染色质位点相互靠拢,对染色质的三维构象进行改变;另一方面有可能在进化过程中,Alu元件的初代子族本身就分布在空间距离比较接近的染色质位点,随着基因组的演化,新产生的Alu元件子族也逐渐跳跃扩展到三维距离相近的染色质位点上,从而使得相同的Alu子族在染色质三维分布上比较靠近。两方面共同作用,从而使得Alu元件的演化与染色质的三维构象紧密相关。
[1] Makalowski W, Genomics. Not junk after all, 2003, 300(5623): 1246–1247.
[2] Wang W, Wang YP. Roles of alu family in human genome., 2007, 29(05): 641–645.王伟, 王亚平. Alu家族在人类基因组中的作用细胞生物学杂志, 2007, 29(05): 641–645.
[3] Lubelsky Y, Ulitsky I. Sequences enriched in alu repeats drive nuclear localization of long rnas in human cells, 2018. 555(7694): 107–111.
[4] Aktaş T, Avşar Ilık I, Maticzka D, Bhardwaj V, Pessoa Rodrigues C, Mittler G, Manke T, Backofen R, Akhtar A. Dhx9 suppresses rna processing defects originating from the alu invasion of the human genome, 2017, 544(7648): 115–119.
[5] Su M, Han D, Boyd-Kirkup J, Yu X, Han JD. Evolution of alu elements toward enhancers, 2014, 7(2): 376–385.
[6] Elbarbary RA, Maquat LE. Distinct mechanisms obviate the potentially toxic effects of Inverted-Repeat alu elements on cellular rna metabolism, 2017, 24(6): 496–498.
[7] Nakama M, Otsuka H, Ago Y, Sasai H, Abdelkreem E, Aoyama Y, Fukao T. Intronic antisense alu elements have a negative splicing effect on the inclusion of adjacent downstream exons, 2018, 664: 84–89.
[8] Dixon JR, Selvaraj S, Yue F, Kim A, Li Y, Shen Y, Hu M, Liu JS, Ren B. Topological domains in mammalian genomes identified by analysis of chromatin interactions, 2012, 485(7398): 376–380.
[9] He C, Li P, Zhang Y, Shi ML, Zhang XY, Xie DJ, Shen WL, Zhao ZH. Bioinformatics analysis of MARs’extensive involvement of forming genome 3D structure., 2017, 33(6): 638–644.何超, 李平, 张彦, 师明磊, 张香媛, 谢德建, 沈文龙, 赵志虎. 核基质附着区广泛参与基因组三维结构折叠的生物信息学分析中国生物化学与分子生物学报, 2017,33(6): 638–644.
[10] Pope BD, Ryba T, Dileep V, Yue F, Wu W, Denas O, Vera DL, Wang Y, Hansen RS, Canfield TK, Thurman RE, Cheng Y, Gülsoy G, Dennis JH, Snyder MP, Stamatoyannopoulos JA, Taylor J, Hardison RC, Kahveci T, Ren B, Gilbert DM. Topologically associating domains are stable units of Replication-Timing regulation, 2014, 515(7527): 402–405.
[11] Dekker J, Rippe K, Dekker M, Kleckner N. Capturing chromosome conformation, 2002, 295(5558): 1306–1311.
[12] Zhao Z, Tavoosidana G, Sjölinder M, Göndör A, Mariano P, Wang S, Kanduri C, Lezcano M, Sandhu KS, Singh U, Pant V, Tiwari V, Kurukuti S, Ohlsson R. Circular chromosome conformation capture (4c) uncovers extensive networks of epigenetically regulated Intra- and interchromosomal interactions, 2006, 38(11): 1341–1347.
[13] Dostie J, Richmond TA, Arnaout RA, Selzer RR, Lee WL, Honan TA, Rubio ED, Krumm A, Lamb J, Nusbaum C, Green RD, Dekker J. Chromosome conformation capture carbon copy (5c): a massively parallel solution for mapping interactions between genomic elements, 2006, 16(10): 1299–1309.
[14] Lieberman-Aiden E, van Berkum NL, Williams L, Imakaev M, Ragoczy T, Telling A, Amit I, Lajoie BR, Sabo PJ, Dorschner MO, Sandstrom R, Bernstein B, Bender MA, Groudine M, Gnirke A, Stamatoyannopoulos J, Mirny LA, Lander ES, Dekker J. Comprehensive mapping of Long-Range interactions reveals folding principles of the human genome, 2009, 326(5950): 289–293.
[15] Zhang XY, He C, Ye BY, Xie DJ, Shi ML, Zhang Y, Shen WL, Li P, Zhao ZH. Optimization and quality control of genome-wide Hi-C library preparation(), 2017, 39(9): 847–855.张香媛, 何超, 叶丙雨, 谢德建, 师明磊, 张彦, 沈文龙, 李平, 赵志虎. 全基因组染色质相互作用Hi-C 文库制备的优化及其质量控制. 遗传, 2017, 39(9): 847–855.
[16] Ma W, Ay F, Lee C, Gulsoy G, Deng X, Cook S, Hesson J, Cavanaugh C, Ware CB, Krumm A, Shendure J, Blau CA, Disteche CM, Noble WS, Duan Z. Fine-Scale chromatin interaction maps reveal the Cis-Regulatory landscape of human lincrna genes, 2015, 12(1): 71–78.
[17] Hsieh TH, Weiner A, Lajoie B, Dekker J, Friedman N, Rando OJ. Mapping nucleosome resolution chromosome folding in yeast by Micro-C, 2015, 162(1): 108–119.
[18] Dryden NH, Broome LR, Dudbridge F, Johnson N, Orr N, Schoenfelder S, Nagano T, Andrews S, Wingett S, Kozarewa I, Assiotis I, Fenwick K, Maguire SL, Campbell J, Natrajan R, Lambros M, Perrakis E, Ashworth A, Fraser P, Fletcher O. Unbiased analysis of potential targets of breast cancer susceptibility loci by capture Hi-C, 2014, 24(11): 1854–1868.
[19] Rao SS, Huntley MH, Durand NC, Stamenova EK, Bochkov ID, Robinson JT, Sanborn AL, Machol I, Omer AD, Lander ES, Aiden EL. A 3d map of the human genome at kilobase resolution reveals principles of chromatin looping, 2014, 159(7): 1665–1680.
[20] Olivares-Chauvet P, Mukamel Z, Lifshitz A, Schwartzman O, Elkayam NO, Lubling Y, Deikus G, Sebra RP, Tanay A. Capturing pairwise and Multi-Way chromosomal conformations using chromosomal walks, 2016, 540(7632): 296–300.
[21] Denker A, de Laat W. The second decade of 3c technologies: detailed insights into nuclear organization, 2016, 30(12): 1357–1382.
[22] Dekker J, Belmont AS, Guttman M, Leshyk VO, Lis JT, Lomvardas S, Mirny LA, O'Shea CC, Park PJ, Ren B, Politz JCR, Shendure J, Zhong S, 4D Nucleome Network. The 4d nucleome project, 2017, 549(7671): 219–226.
[23] Chen N. Using repeatmasker to identify repetitive elements in genomic sequences, 2004, Chapter 4: Unit 4. 10.
[24] Harmston N, Ing-Simmons E, Perry M, Barešić A, Lenhard B. Genomicinteractions: an R/Bioconductor package for manipulating and investigating chromatin interaction data, 2015, 16: 963.
[25] Akdemir KC, Chin L. Hicplotter integrates genomic data with interaction matrices, 2015, 16: 198.
[26] Servant N, Varoquaux N, Lajoie BR, Viara E, Chen CJ, Vert JP, Heard E, Dekker J, Barillot E. Hic-Pro: an optimized and flexible pipeline for Hi-C data processing, 2015, 16: 259.
[27] Lajoie BR, Dekker J, Kaplan N. The hitchhiker's guide to Hi-C analysis: practical guidelines, 2015, 72: 65–75.
[28] Cremer T, Cremer M, Cremer C. The 4d Nucleome: genome compartmentalization in an evolutionary context, 2018, 83(4): 313–325.
[29] Cournac A, Koszul R, Mozziconacci J. The 3d folding of metazoan genomes correlates with the association of sImilarrepetitive elements, 2016, 44(1): 245–255.
[30] Kapitonov V, Jurka J. The age of alu subfamilies, 1996, 42(1): 59–65.
[31] Price AL, Eskin E, Pevzner PA. Whole-Genome analysis of alu repeat elements reveals complex evolutionary history, 2004, 14(11): 2245–2252.
[32] Xie DJ, Shi ML, Zhang Y, Wang TY, Shen WL, Ye BY, Li P, He C, Zhang XY, Zhao ZH. Construction of ctcf degradation cell line by Crispr/Cas9 mediated genome editing, 2016, 38(7): 651–657.谢德建, 师明磊, 张彦, 王天艺, 沈文龙, 叶丙雨, 李平, 何超, 张香媛, 赵志虎. 利用CRISPR/Cas9 技术构建CTCF 蛋白降解细胞系. 遗传, 2016, 38(7): 651–657.
[33] Beagrie RA, Scialdone A, Schueler M, Kraemer DC, Chotalia M, Xie SQ, Barbieri M, de Santiago I, Lavitas LM, Branco MR, Fraser J, Dostie J, Game L, Dillon N, Edwards PA, Nicodemi M, Pombo A. Complex Multi-Enhancer contacts captured by genome architecture mapping, 2017, 543(7646): 519–524.
[34] Marti-Renom MA, Almouzni G, Bickmore WA, Bystricky K, Cavalli G, Fraser P, Gasser SM, Giorgetti L, Heard E, Nicodemi M, Nollmann M, Orozco M, Pombo A, Torres-Padilla ME. Challenges and guidelines toward 4d nucleome data and model standards, 2018, 50(10): 1352–1358.
[35] Schmitt AD, Hu M, Jung I, Xu Z, Qiu Y, Tan CL, Li Y, Lin S, Lin Y, Barr CL, Ren B. A compendium of chromatin contact maps reveals spatially active regions in the human genome, 2016, 17(8): 2042–2059.
[36] Chuong EB, Elde NC, Feschotte C. Regulatory activities of transposable elements: from conflicts to benefits, 2017, 18(2): 71–86.
[37] Ge SX. Exploratory bioinformatics investigation reveals importance of "Junk" DNA in early embryo development, 2017, 18(1): 200.
[38] Padeken J, Zeller P, Gasser SM. Repeat DNA in genome organization and stability, 2015, 31: 12–19.
[39] Gu Z, Jin K, Crabbe MJC, Zhang Y, Liu X, Huang Y, Hua M, Nan P, Zhang Z, Zhong Y. Enrichment analysis of alu elements with different spatial chromatin proximity in the human genome, 2016, 7(4): 250–266.
[40] Tang LM, Pu WY, Xiao L, Li K, Guo ZF, Research progress of a component., 2014, (08): 1–2.汤丽梦, 浦汪洋, 肖莉, 李凯, 郭紫芬. Alu元件研究进展生物技术世界, 2014, (08): 1–2.
Bioinformatics analysis of Alu components at the level of genome 3D structure
Chao He1,2, Wenlong Shen2, Ping Li2, Yan Zhang2, Jing Zeng1, Zuoming Yin1, Zhihu Zhao2
The interphase chromatin is folded in the nucleus in a hierarchical manner, including the nucleosome, the "beads on a string" structure composed of nucleosomes, the solenoid fiber structure, the chromatin/DNA loop structure (chromatin/DNA loop), and the topologically associated domain (TAD). Among them, TAD is considered to be the basic unit of the 3D structure of chromatin because it is relatively stable and conserved in different cell types. Alu elements occupy a large proportion in the mammalian genomes. There are a wide variety of Alu elements, but their functional characterizations are limited to date. This study investigates the role of Alu elements in the assembly of 3D chromatin conformation. The evolutionary process of the Alu subfamily was explored by the distance relationship of the 3D structure of chromatin. We found that the proportion of Alu elements in high-density chromatin interaction increased with higher similarity, indicating that Alu plays an important role in the construction of chromatin 3D structure. There is a certain positive correlation between the strength of the upper interaction and the evolutionary relationship. In sum, the Alu elements with relatively close distances in the 1D sequence will also be close to each other in the 3D structure of chromatin.
genome 3D structure;Alu component;bioinformatics;evolutionary tree
2018-10-30;
2019-01-01
国家自然科学基金项目(编号:31370762,31030026,31272416,81372218)资助[Supported by the National Natural Science Foundation of China (Nos. 31370762, 31030026, 31272416, 81372218)]
何超,硕士,工程师,研究方向:生物信息学。E-mail: hechao2010@tsinghua.org.cn
殷作明,西藏军区总医院院长,研究方向:高原病、战创伤。E-mail: yinzuoming@163.com赵志虎,军事科学院军事医学研究院研究员,研究方向:染色质三维结构。E-mail: zhaozh@bmi.ac.cn
10.16288/j.yczz.18-296
2019/1/14 13:15:30
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20190114.1315.011.html
(责任编委: 吴强)
