枯草芽孢杆菌芽孢皮层裂解酶CwlJ基因的克隆及酶生物信息学分析
2019-03-08马慧娇曾朝玮郭洪伟郭家俊
马慧娇,曾朝玮,郭洪伟,郭家俊,章 中*
(宁夏大学 农学院,宁夏 银川750021)
细菌芽孢对杀菌处理的极端抗性,是造成食品安全问题和食品腐败的重要原因[1]。芽孢极端抗性与其特有结构息息相关,其从外到里有七层结构,分别是孢外壁、芽孢衣、外膜、皮层、细胞壁、内膜、核心[2]。芽孢皮层是维持芽孢休眠状态的关键结构,主要由肽聚糖构成,是芽孢抗压性的一层关键结构。在芽孢萌发而转变成营养体的过程中,皮层裂解酶可将芽孢皮层水解,使得芽孢失去休眠特性[3-6]。皮层裂解酶是芽孢所特有的专一水解皮层肽聚糖的酶,枯草芽孢杆菌(Bacillus subtilis)中有许多皮层裂解酶同系物的存在,CwlJ就是其中之一[7-8]。
高压热杀菌技术(high-pressure thermal sterilization,HPTS)是一种新兴食品加工技术,该技术利用压力和温度的协同效应,可有效杀灭芽孢,并较好地保持食品的营养和感官品质[9-10]。前期研究发现HPTS下芽孢皮层肽聚糖发生了水解,有报道推测HPTS可能激活了皮层裂解酶而导致皮层肽聚糖水解[11-12],那么证实推测并了解其水解机理就成为关键。
本研究对克隆所得的CwlJ基因采用美国国家生物技术信息中心(national center of biotechnology information,NCBI)和ExPASy等网站中的各种信息分析工具,并结合DNAMAN软件对其理化性质、亲/疏水性、二级结构、三级结构、可溶性等进行生物信息预测和分析。该分析结果在揭示皮层裂解酶CwlJ结构与性质的同时,也为后续皮层裂解酶CwlJ的表达、分离纯化以及阐明HPTS下皮层肽聚糖水解机理奠定了理论基础。
1 材料与方法
1.1 材料与试剂
1.1.1 化学试剂
枯草芽孢杆菌(Bacillussubtilis)As1.433:中国普通微生物菌种保藏管理中心(China general microbiological culture collection center,CGMCC);pET-28a(+)、DH5α:生工生物工程(上海)上海股份有限公司;DNA分子质量标准、核酸染料、琼脂糖凝胶回收试剂盒、普通质粒小提试剂盒:天根生化科技(北京)有限公司产品;限制性核酸内切酶、连接酶:英国NEB公司;胰蛋白胨、酵母提取物:英国OXOID公司;琼脂粉、氯化钠:北京化学试剂公司产品;卡那霉素:上海求德生物化工有限公司。实验所用试剂均为分析纯或生化试剂。
1.1.2 培养基
LB液体培养基:胰蛋白胨10 g/L,酵母提取物5 g/L,氯化钠5 g/L,用1 mol/L NaOH溶液调pH值为7.0。LB固体培养基是在LB液体培养基中加入15 g/L的琼脂粉。培养基的灭菌条件均为121 ℃、20 min。
1.2 仪器与设备
AL204型电子天平:梅特勒-托利多仪器(上海)有限公司;DSX-280B型高压灭菌锅:上海申安医疗器械厂;LRH系列生化培养箱:上海一恒科学仪器有限公司;SW-CJ-2FD型超净工作台:苏州洁净技术研究所;DYY-6C型电泳仪、DYCZ-24DN型电泳槽:北京六一仪器厂;T100型梯度聚合酶链式反应(polymerase chain reaction,PCR)仪、Gel doc2000型凝胶成像仪:美国BIO-RAD公司;HH·SY21-Ni恒温水浴器:北京长源实验设备厂。
1.3 方法
1.3.1 皮层裂解酶CwlJ基因的克隆
依据在蛋白质数据库UniProtKB中搜索到的来自Bacillus subtilis-P42249的皮层裂解酶CwlJ的氨基酸序列,通过NCBI序列比对获得CwlJ的基因序列。由基因序列设计全长引物,其上游引物序列为YA0079-1N(5'-GACACCCATGGGCATGGCG-3)';下游引物序列为YA0079-20N(5'-GTGTCCTCGAGAAATGTGTT-3)'(下划线部分分别为限制性核酸内切酶NcoI、XhoI的识别位点),按照95 ℃预变性3 min;95 ℃变性22 s,53 ℃退火20 s,72 ℃延伸30 s,24个循环;72 ℃再延伸5 min的PCR反应体系进行CwlJ全基因合成。合成产物CwlJ进行琼脂糖凝胶电泳检测,并采用回收试剂盒对目的条带进行切胶回收和纯化。对质粒pET-28a(+)和目的基因CwlJ同时使用NcoI和XhoI进行双酶切后用T4DNA连接酶于16 ℃连接过夜,连接产物转化至DH5α感受态细胞,并涂布在含100 g/mL卡那霉素的LB固体培养基上,37 ℃培养过夜,筛选阳性克隆质粒进行双酶切鉴定,并送至昆泰锐武汉生物技术有限责任公司进行菌液测序。
1.3.2 皮层裂解酶CwlJ生物信息学分析
测序结果经translate工具转换得到CwlJ基因编码的蛋白序列,与蛋白数据库中目标序列比对后完全一致。利用ProtParam程序对CwlJ基因编码的蛋白质进行理化性质分析;软件SOPMA对皮层裂解酶CwlJ进行螺旋卷曲分析;SWISS-MODEL对该酶进行三维结构预测;DNAMAN软件对该酶的亲、疏水性和系统进化进行分析;TMHMM工具分析该酶是否有跨膜结构;SignalP软件分析该酶是否为分泌蛋白;TargetP工具对该酶进行亚细胞定位预测[13-14];Harrison统计学模型预测该酶的可溶性。
2 结果与分析
2.1 皮层裂解酶CwlJ基因的克隆
根据已知CwlJ氨基酸序列,合成目的基因CwlJ。合成产物进行1%琼脂糖凝胶电泳,结果见图1。由图1可知,所得产物大小约为450 bp,目的条带上方出现的阴影可能是引物不特异或者退火温度不合理等原因造成的。

图1 CwlJ基因PCR扩增产物电泳图Fig. 1 Electrophoretogram of PCR amplification product of CwlJ gene
目的基因CwlJ克隆至质粒pET-28a(+)后的双酶切验证,结果见图2。由图2可知,显示所切掉的片段和克隆序列大小一致,可确定CwlJ基因已经插入质粒pET-28a(+)中。

图2 双酶切鉴定结果Fig. 2 Results of double enzyme digestion identification
测序结果见图3。由图3可知,该克隆基因全长440 bp,与克隆序列一致,且开放阅读框架正确。由此得出结论pET-28a(+)-CwlJ重组载体构建成功[15]。

图3 克隆基因CwlJ的测序峰图Fig. 3 Sequencing peak map of cloned gene CwlJ
2.2 皮层裂解酶CwlJ生物信息学分析
2.2.1 皮层裂解酶CwlJ一级结构及其理化性质分析

表1 皮层裂解酶CwlJ氨基酸组成Table 1 Amino acid composition of cortex lytic enzyme CwlJ
运用ProtParam程序在线分析芽孢皮层裂解酶CwlJ的理化特性,结果表明皮层裂解酶CwlJ的理论分子质量为16.462 kDa,理论等电点为9.54,原子总数共2 276个,分子式为C731H1116N218O203S8;该酶的亲水性平均系数(grand average of hydropathicity,GRAVY)值为-0.506,GRAVY值通常可以用来表征蛋白质的亲/疏水性,其范围一般在-2~2之间,疏水蛋白的GRAVY 值为正值,亲水蛋白的GRAVY值则为负值,所以皮层裂解酶CwlJ为亲水蛋白;该酶的不稳定系数为42.20,当蛋白质不稳定系数>40时,蛋白不稳定[16],说明CwlJ为不稳定蛋白;CwlJ共编码142个氨基酸(见表1),其中含量最高的是丙氨酸(Ala)和精氨酸(Arg),共占氨基酸总含量的22.6%,含量最低的是色氨酸(Trp)和组氨酸(His),两种氨基酸所占比例相同,各为2.1%,带负电的氨基酸残基总数(Asp+Glu)为13个,带正电的氨基酸残基的总数(Arg+Lys)是20个。
2.2.2 皮层裂解酶CwlJ的二级结构分析
软件SOPMA可以正确预测蛋白二级结构,结果见图4。由图4可知,蛋白二级结构中α-螺旋占26.06%,β-折叠占21.83%,β-转角占4.93%,无规则卷曲占47.18%。

图4 皮层裂解酶CwlJ二级结构分析Fig. 4 Analysis of secondary structure of cortex lytic enzyme CwlJ
2.2.3 皮层裂解酶CwlJ的三级结构预测
SWISS-MODEL建模预测所得皮层裂解酶CwlJ三级结构图见图5。

图5 皮层裂解酶CwlJ三级结构预测Fig. 5 Prediction of tertiary structure of cortex lytic enzyme CwlJ
2.2.4 皮层裂解酶CwlJ的亲/疏水性分析
采用DNAMAN 程序软件分析皮层裂解酶CwlJ的亲、疏水性,结果见图6。由图6可知,皮层裂解酶CwlJ的亲、疏水性分值越高表明疏水性越强,分值越低表明亲水性越强。分析结果显示疏水性最强的区域为第30~35个氨基酸附近,亲水性最强的区域为第95~104个氨基酸附近,经预测该酶的氨基酸大多都是亲水性较强,这也进一步证实了ProtParam程序预测该酶为亲水蛋白的结果。

图6 皮层裂解酶CwlJ的氨基酸疏水性(A)和亲水性(B)分析Fig. 6 Hydrophobicity (A) and hydrophilicity (B) analysis of amino acid of cortex lytic enzyme CwlJ
2.2.5 皮层裂解酶CwlJ的跨膜区分析
借助Expasy中的TMHMM工具对蛋白是否含膜蛋白进行预测。结果见图7。

图7 皮层裂解酶CwlJ的跨膜区预测Fig. 7 Prediction of transmembrane region of cortex lytic enzyme CwlJ
由图7可知,蛋白质序列的长度为142,预测的跨膜螺旋数为0;预期的膜内螺旋氨基酸数量为0.006 44;蛋白质的前60个氨基酸跨膜螺旋中预期的氨基酸数量为0.002 08;蛋白质N端在细胞膜内的可能性为0.340 59。如果outside与inside两条线存在相交,则相交的部分为跨膜区。图中这两条线是平行不相交的,因此不存在跨膜区。通常预期的膜内螺旋氨基酸数量如果数目>18,则很可能该酶是跨膜蛋白,但本研究结果0.006 44明显<18,所以该酶无跨膜区,说明该酶不可能作为膜受体起作用,也不可能是膜锚定蛋白或离子通道蛋白。
2.2.6 皮层裂解酶CwlJ的信号肽分析
应用SignalP预测CwlJ编码的氨基酸序列中是否存在信号肽[17-18]。根据预测的蛋白质来源选择革兰氏阳性菌。结果见图8。

图8 皮层裂解酶CwlJ的信号肽分析Fig. 8 Signal peptide analysis of cortex lytic enzyme CwlJ
由图8可知,S-mean是从N端氨基酸开始到剪切位点处各氨基酸的平均值;D值是S-mean和Y-max的平均值,其对区分是否为分泌蛋白具有重要作用,对于非分泌蛋白,该行输出的分数值非常低(接近目标值0.1)。预测结果中,信号肽(SP)结果为No,且D值非常接近0.1,说明CwlJ没有信号肽,且不是分泌蛋白。
2.2.7 皮层裂解酶CwlJ的亚细胞定位
通过TargetP工具对CwlJ进行亚细胞定位预测。结果见表2。

表2 亚细胞定位预测Table 2 Subcellular location prediction
由表2可知,mTP值越接近1.0,表明蛋白质位于线粒体的可能性越大;SP值越接近1.0,表明有信号肽。其他表明蛋白很有可能位于其他地方。除此之外,其数值范围从1到5,其中RC值越小,说明预测的可靠性越高,本次预测RC值为2,表明可靠性相对较高。结果表明,CwlJ位于线粒体的可能性仅为11.5%,位于其他地方的可能性更大,具体定位地方不确定,且含有信号肽的可能性极低,这也进一步验证了2.2.6的预测结果。
2.2.8 皮层裂解酶CwlJ重组蛋白的可溶性分析
参照WILKINSON D L等[19]两参数预测模型,预测重组蛋白的可溶性。判断的标准值(criterion of values,CV)及概率(probability,P)计算公式如下:

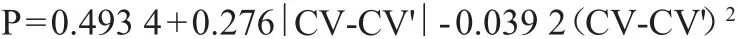
式中:CV 为判断的标准值;CV'为校准值,其为1.71;N、P、G、S、R、K、D、E分别代表天门冬酰胺、脯氨酸、甘氨酸、丝氨酸、精氨酸、赖氨酸、天门冬氨酸、谷氨酸的数量,个;n代表CwlJ序列中全部氨基酸残基的数量,个。
CV-CV'值可用来判断CwlJ基因在大肠杆菌中进行可溶性表达的倾向。如果CV-CV'<0,则该基因有可溶性表达的倾向;如果CV-CV'>0,则该基因在表达时更倾向于形成包涵体;CV-CV'的绝对值越大,倾向性越明显[20]。可溶性(或不可溶性)P则是在体现该可能性的大小。通过计算,该酶中CV-CV'值约为0.76>0,表明CwlJ基因在表达时易形成包涵体,且其概率P为68.11%,预测重组蛋白可溶性可能性较大。
2.2.9 来自不同菌种的皮层裂解酶CwlJ的系统进化分析
用DNAMAN软件对来自不同菌的皮层裂解酶CwlJ构建系统进化树进行同源序列聚类分析,结果见图9。

图9 皮层裂解酶CwlJ系统进化树Fig. 9 Phylogenetic tree of cortex lytic enzyme CwlJ
由图9可知,枯草芽孢杆菌(Bacillus subtilis)皮层裂解酶CwlJ与苏云金芽孢杆菌(Bacillus thuringiensis)CwlJ、苏云金芽孢杆菌(Bacillus thuringiensis)Bt18247 CwlJ处于同一个小分支,表明在进化过程中距离最近,亲缘关系最为密切;与苏云金芽孢杆菌(Bacillus thuringiensis)CwlJ、蜡样芽胞杆菌(Bacillus cereus)CwlJ、炭疽芽孢杆菌(Bacillus anthracis)CwlJ、肺炎链球菌(Streptococcus pneumoniae)CwlJ、类芽孢杆菌(Paenibacillus sp.P22)CwlJ同属一个大分支,表明其具有较高的同源性,反之则亲缘关系较远。
3 结论
通过克隆得到基因CwlJ,其编码142个氨基酸,基因全长为440 bp。而且测序和双酶切结果均表明重组表达载体pET-28(+)-CwlJ构建成功,为后期表达纯化CwlJ奠定了基础。
生物信息学各软件分析结果表明:枯草芽孢杆菌(Bacillus subtilis)皮层裂解酶CwlJ是一个分子量大小为16.462 kDa,亲水性的碱性不稳定蛋白;二级结构分析表明α-螺旋占26.06%,β-延伸链占21.83%,β-转角占4.93%,无规则卷曲占47.18%;DNAMAN 程序分析得该酶多以亲水性氨基酸存在,为亲水蛋白;TMHMM程序分析得CwlJ不是分泌性蛋白;亚细胞定位显示在所预测范围内存在的可能性较小,可能位于其他地方;可溶性预测分析得知表达的酶蛋白倾向于形成包涵体,可能性为68.11%;系统进化分析表明枯草芽孢杆菌(Bacillus subtilis)皮层裂解酶CwlJ与苏云金芽孢杆菌(Bacillus thuringiensis)皮层裂解酶CwlJ、苏云金芽孢杆菌(Bacillus thuringiensis)Bt18247皮层裂解酶CwlJ亲缘关系较近。
克隆表达载体pET-28a(+)-CwlJ构建成功,且通过生物信息学分析,了解了皮层裂解酶CwlJ的组成和结构特性,为后续皮层裂解酶CwlJ大批量异源表达、酶学性质与功能的研究提供了参考信息。
