药用真菌桑黄分子鉴定及遗传多样性分析
2019-03-05王伟科宋吉玲巫优良袁卫东陈观平
王伟科,宋吉玲,巫优良,闫 静,陆 娜,袁卫东,陈观平
(1.杭州市农业科学研究院 蔬菜研究所,浙江 杭州 310024; 2.江山市农业科学研究所,浙江 江山 324104; 3.浙江省中医药研究院,浙江 杭州 310012)
桑黄是一种珍贵的药用真菌,因寄生于桑树树干,子实体呈黄褐色而得名,俗称桑耳、桑臣等,是我国一种传统而名贵的中药材[1]。桑黄真菌种群隶属于担子菌门(Basidiomycotina)、层菌纲(Hymenomycetes)、非褶菌目(Aphyllophorales)、锈革孔菌科(Hymenochaetaceae)[2]。桑黄的功效最早记载在《本草纲目》一书中,《药性论》也有桑黄味微苦、性寒等功效描述[3]。传统中医认为桑黄具利五脏、软坚、排毒、止血和止泻等药效,可用于治疗痢疾、盗汗、血崩、血淋、脱肛和闭经等症[4];现代药理学研究证实,桑黄还具有抗肿瘤、降血糖、护肝、抗炎、免疫调节、抗氧化等药理作用[5-6]。因此,桑黄具有极高的开发利用价值。
当前,桑黄产品的开发主要以桑黄子实体为原料,但由于桑黄野生资源的日益匮乏,加之桑黄子实体生长对环境的依赖性很高,生长周期长,产量不稳定;对桑黄人工栽培等相关技术研究尚处于起步阶段[7],这严重限制了桑黄的进一步开发和利用。而桑黄种质鉴定、新品种选育及高效栽培技术研究等一直是研究者关注的重点。作为珍贵的药用真菌,桑黄真菌大多寄生在桑树、栎树、桦树、杨树等阔叶植物的枯立木及树干上,且该类真菌的子实体均呈蹄形,同时与桑黄真菌在外观形态上相似的大型真菌种类繁多,仅依赖于形态特征的传统鉴定方法很难确定菌株的分类归属[8-10]。在基础研究及开发应用中,往往出现“同种异名”“同名异种”的现象,严重阻碍了桑黄新品种的选育及产业的发展。
rDNA内转录间隔区(ITS)是真菌核糖体RNA基因非转录区的一部分,其在绝大多数的真核生物中表现出了极为广泛的序列多态性,表现为种内菌株间相对保守,种间差异比较明显。这种特点使ITS适合于真菌物种的分子鉴定以及属内物种间或种内差异较明显的菌群间的系统发育关系分析。即使亲缘关系非常接近的2个种都能在ITS序列上表现出差异,显示最近的进化特征。因此,ITS已被广泛应用于微生物菌种分类与鉴定、中药材鉴定和品质评价、植物种质资源鉴定、遗传多样性与亲缘关系、病原诊断、系统发育等不同方面的研究[11-14]。对种属模糊的食用真菌ITS序列进行测序,可通过将测序得到的ITS序列与已知真菌ITS序列比较,从而能获得该菌株的信息及系统的遗传谱系。由于用于真菌鉴定的ITS序列通常包括ITS1、5.8S和ITS2,长度为500~750 bp,故利用高保真PCR技术并结合直接测序法在食用真菌的分类、鉴定中有很大的优越性[15]。
我们在参考真菌rDNA ITS扩增通用引物及GenBank中登录的桑黄真菌属(Phellinusbaumii)rDNA ITS序列基础上,设计并合成了桑黄rDNA ITS通用引物,并利用PCR技术克隆了rDNA ITS基因片段,同时结合直接测序技术,对从杭州千岛湖、桐庐等地区采集到的桑黄真菌进行了分子鉴定及遗传结构分析工作。本文的研究结果可以提供一种准确可靠且简单易行的桑黄真菌分子鉴定技术,有助于明确各桑黄菌株的种属来源及遗传结构,为桑黄真菌的分类及进一步开发应用提供参考依据。
1 材料与方法
1.1 材料
1.1.1 样品采集
测试菌种(株)收集自我国各主要桑黄真菌保藏单位(表1)。此外,检索近期发表的GenBank中登录的其他12种桑黄真菌ITS序列(基因登录号分别为:AF080458.1、AF082102.1、AF200226.1、AF534065.1、AY436626.1、AY513234.1、AY640937.1、DQ103884.1、DQ462333.1、EF506943.1、EF694971.1、FJ940908.1),下载保存,用于序列比较。
1.1.2 仪器与试剂
主要仪器:Veriti型PCR仪(ABI,USA)、DYY-6D电泳仪(北京六一仪器厂,中国)、GelDoc XR凝胶成像系统(Bio-Rad,USA)、3700型DNA测序仪(美国)。
表1桑黄真菌测试菌株来源
Table1The strains used for Sanghuang genetic diversity and molecular identification

序号No.菌株Strain菌株来源Source of strain序号No.菌株Strain菌株来源Source of strain1S1淳安微生物研究所Chunan Institute of Microbiology12S13淳安桑都食用菌专业合作社Chunan “sangdu” Edible Mushroom Professional Cooperative2S2淳安微生物研究所Chunan Institute of Microbiology13S15杭州市食用菌协会Hangzhou Edible Fungi Association3S3四川省农业科学院Sichuan Academy of Agricultural Sciences14S17杭州市食用菌协会Hangzhou Edible Fungi Association4S4四川省农业科学院Sichuan Academy of Agricultural Sciences15S19安徽金寨采集Collection from Jinzhai Anhui5S5浙江桐庐野生采集Wild collection from Tonglu Zhejiang16S21浙江淳安野生采集Wild collection from Chunan Zhejiang6S6浙江桐庐野生采集Wild collection from Tonglu Zhejiang17S22浙江淳安野生采集Wild collection from Chunan Zhejiang7S7浙江临安野生采集Wild collection from Linan Zhejiang 18S23浙江淳安野生采集Wild collection from Chunan Zhejiang8S8浙江淳安野生采集Wild collection from Chunan Zhejiang19S26浙江衢州野生采集Wild collection from Quzhou Zhejiang9S9桐庐桐君堂药业有限公司Tonglu Tong Jun Tang Pharmaceutical Co. Ltd.20S27安徽金寨采集Collection from Jinzhai Anhui10S10浙江桐庐野生采集Wild collection from Tonglu Zhejiang21S28浙江丽水采集Wild collection from Lishui Zhejiang11S11延边朝鲜族自治州农业科学院Yanbian Agricultural Science Institute22SKHCH浙江开化采集Wild collection from Kaihua Zhejiang
主要试剂:Platinum Pfx DNA Polymerase(thermo,USA)、dNTPS(thermo,USA)、agarose(Biowest,上海普飞)、gelred(索莱宝,中国)、marker(天根,中国)。
1.2 方法
1.2.1 菌丝体培养
从培养5~7 d的PDA培养基平板上挑取2 mm×2 mm的菌丝块,将其接种于PDA液体培养基中,25~27 ℃浅层静置培养10~15 d。然后收集菌丝体,用无菌水漂洗2次,灭菌滤纸吸干水分,用于提取基因组DNA。
1.2.2 基因组DNA提取
取0.1 g菌丝体,液氮研磨后加入2%CTAB,用于基因组DNA的提取[16]。以0.8%琼脂糖凝胶电泳检测所提DNA纯度、NanoDrop 2000C超微量分光光度计(Thermo)检测DNA浓度,样品稀释至50 ng·μL-1,用于rDNA ITS扩增。
1.2.3 rDNA ITS扩增
参照White等[17]发表的真菌基因组ITS通用引物及GenBank中公布的桑黄真菌属(Phellinusbaumii)rDNA ITS序列,设计引物:ITS-F(5′-AGTCGTAACAAGGTTTCCGTA-3′);ITS-R(5′-TCCTCCGCTTATTGATATGC-3′)。扩增体系含10×PCR缓冲液5 μL、10 mmol·L-1dNTPs 1 μL、10 μmol·L-1上下游引物各2 μL、100 ng基因组模板DNA及1 UpfuDNA聚合酶,总体积50 μL。PCR扩增程序为:94 ℃预变性3 min;94 ℃变性15 s,57 ℃退火20 s,72 ℃延伸40 s,30个循环;72 ℃延伸7 min。
1.2.4 rDNA ITS扩增产物的检测
取目标产物5 μL,加6×上样缓冲液1 μL(含30 mmol·L-1EDTA,36%丙三醇,0.05%溴酚蓝),混匀后于1.2%琼脂糖凝胶上电泳分离,拍照记录。
1.2.5 rDNA ITS扩增产物的序列测定
将目的条带位置正确的样品送擎科生物测序,以PCR引物为起始引物,双向测通。
1.2.6 序列比较及系统发育分析
用MEGA 7.0.14进行系统发育分析和进化树构建,以最大似然法(construct/test maximum likelihood tree)构建分子进化树,进行1 000次自展抽值(bootstrap sampling)检验分子进化树可靠性。Kimura 2-parameter模型计算遗传距离,所有对位排列结果中的空位(gaps)或缺失数据(missing data)作完全删除(complete deletion)处理。
1.2.7 序列比对分析
用BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)软件进行序列在线比对分析,同时利用clustal W-2.1进行2条序列的比对分析。
2 结果与分析
2.1 桑黄rDNA ITS序列PCR扩增
以桑黄菌丝体基因组DNA为模板,ITS-F/ITS-R为引物,进行PCR扩增。扩增产物经1.2%琼脂糖电泳分析,检测到特异性扩增片段,测试样品扩增产物长度为800 bp左右(图1),符合预期值。
2.2 基于rDNA ITS序列的桑黄真菌系统发育分析
对桑黄rDNA ITS扩增产物进行测序,并依据rDNA ITS序列信息,以最大似然法(construct/test maximum likelihood tree)构建分子进化树,桑黄真菌显著的区分为3个类群(图2)。其中,S3、S28、FJ940908.1、DQ103884.1为一组,S13、S19、S27为一组,其余S1、S2、S4、S5、S6、S7、S8、S9、S10、S11、S15、S17、S21、S22、S23、S26、SKHCH等为一组。
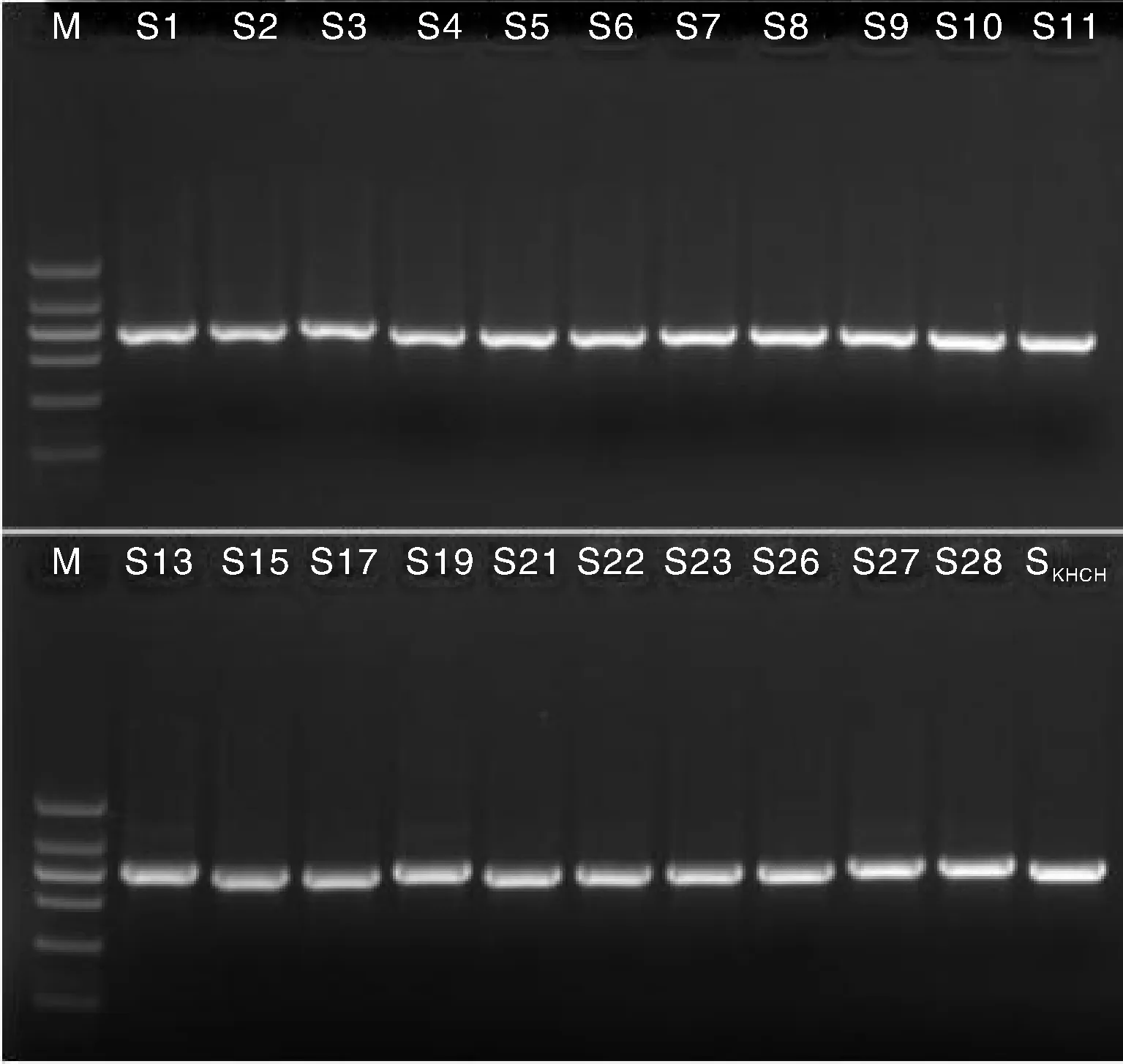
Marker分子量从下到上依次为:200、400、600、800、1 000、1 500 bpThe molecular weight of marker from bottom to top were 200, 400, 600, 800, 1 000 and 1 500 bp.图1 桑黄rDNA ITS序列PCR扩增Fig.1 PCR amplification of rDNA ITS of Phellinus baumii
2.3 基于rDNA ITS序列的桑黄遗传距离分析
对22个桑黄真菌种间及种内的遗传距离进行分析,结果表明,桑黄真菌存在明显的遗传分化,属种间遗传趋异度显著高于种内,S13、S19、S27组存在较高的种内遗传多样性(表2)。其中,S27在ITS区存在较高趋异度(ITS=0.030);其次为S13、S28趋异度(ITS=0.028)。而S1、S2、S4、S5、S6、S7、S8、S9、S10、S11、S15、S17、S21、S22、S23、S26、SKHCH组内基本不存在遗传多样性。
2.4 桑黄真菌rDNA ITS序列分析
对3类桑黄真菌的rDNA ITS序列进行BLAST分析,本文测定的桑黄序列Group Ⅰ(S3、S28)与GenBank中已登录的Fuscoporiagilva(杨树桑黄)真菌rDNA ITS序列(登录号DQ103884.1)显示的序列一致,相似性为100%(图3)。Group Ⅱ(S13、S19、S27)与GenBank中已登录的Inonotusbaumii(丁香桑黄)真菌rDNA ITS序列(登录号FJ940908.1)的同源性较高,相似性为99%(图4)。而Group Ⅲ(S1、S2、S4、S5、S6、S7、S8、S9、S10、S11、S15、S17、S21、S22、S23、S26、SKHCH)与GenBank中已登录的Sanghuangporussanghuang(野生桑树桑黄)真菌rDNA ITS序列(登录号KT693275.1)的同源性较高,相似性为99%(图5)。

图2 基于桑黄真菌rDNA ITS 序列的系统发育树构建Fig.2 Phylogenetic tree based on rDNA ITS sequences of Phellinus baumii
表2桑黄菌各株间的遗传距离分析
Table2Genetic distance among Sanghuang strains

SKHCHS9S8S7S6S5S4S3S28S27S26S23S22S21S2S19S17S15S13S11S10S1SKHCHS90S800S7000S60000S500000S4000000S30.0250.0250.0250.0250.0250.0250.025S280.0250.0250.0250.0250.0250.0250.0250S270.0300.0300.0300.0300.0300.0300.0300.0040.004S2600000000.0250.0250.030S2300000000.0250.0250.0300S2200000000.0250.0250.03000S2100000000.0250.0250.030000S200000000.0250.0250.0300000S190.0280.0280.0280.0280.0280.0280.0280.0030.0030.0030.0280.0280.0280.0280.028S1700000000.0250.0250.030000000.028S1500000000.0250.0250.030000000.0280S130.0280.0280.0280.0280.0280.0280.0280.0030.0030.0030.0280.0280.0280.0280.02800.0280.028S1100000000.0250.0250.030000000.028000.028S1000000000.0250.0250.030000000.028000.0280S100000000.0250.0250.030000000.028000.02800

图3 S3与DQ103884.1(杨树桑黄)rDNA ITS序列比对分析Fig.3 Sequence analysis of rDNA ITS of S3 and Fuscoporia gilva by Clustal W

图4 S13与FJ940908.1(丁香桑黄)rDNA ITS序列比对分析Fig.4 Sequence analysis of rDNA ITS of S13 and Inonotus baumii by Clustal W

图5 S23与KT693275.1(桑树桑黄)rDNA ITS序列比对分析Fig.5 Sequence analysis of rDNA ITS of S23 and Sanghuangporus sanghuang by Clustal W
3 结论与讨论
随着分子生物学技术的发展,位于18S rDNA及28S rDNA间的rDNA内转录间隔区(internal transcribed spacer,ITS)序列分析技术由于进化速率快、稳定性好和测序方便,已广泛运用到多种真菌种属水平的分类鉴定研究中[18]。利用ITS序列分析技术,我们鉴定了杭州、丽水及安徽金寨等地采集到的22个桑黄样品。根据ITS序列聚类分析,将22个桑黄样品明显的区分为3个类群。其中,S3、S28为一组,S13、S19、S27为一组,其余S1、S2、S4、S5、S6、S7、S8、S9、S10、S11、S15、S17、S21、S22、S23、S26、SKHCH为一组。
同时,我们通过BLAST序列比对,分析了以上3类桑黄菌株的rDNA ITS序列,从供试样品中成功鉴定出2份杨树桑黄(Fuscoporiagilva)分别为S3、S28;3份丁香桑黄(Inonotusbaumii)分别是S13、S19、S27;17份桑树桑黄(Sanghuangporussanghuang)分别为S1、S2、S4、S5、S6、S7、S8、S9、S10、S11、S15、S17、S21、S22、S23、S26、SKHCH。
本文22份桑黄样品的rDNA ITS测序结果为Fuscoporiagilva、Inonotusbaumii、Sanghuangporussanghuang的系统发育地位及广泛应用的桑黄品种鉴定提供了可靠的分子证据,同时证明rDNA ITS测序技术能够有效鉴定桑黄种质。利用该技术准确区分了3种桑黄真菌,从分子水平明确提出外观近似的桑黄Fuscoporiagilva、Inonotusbaumii、Sanghuangporussanghuang不是同一个种(图6),而是客观存在且相互独立的生物种。这一结果有助于明确桑黄真菌种类,也能为桑黄种质资源鉴定、菌株培养及有效成分功能鉴定提供参考。
尽管本研究利用rDNA ITS测序技术可将子实体外观形态相似的桑黄种质明确区分为3类,但当前NCBI中GenBank登录的与桑黄相关的序列信息还不够全面;同时桑黄子实体形态也会因所处地理位置及寄主的不同而发生改变;此外,大量的桑黄功效研究所使用的材料也至少包含了桑树桑黄、丁香桑黄、杨树桑黄或火木层孔菌等,究竟哪个种类起到主要作用也不得而知,这些现状均导致学者对桑黄种属的确定始终存在较大争议,因此本研究鉴定区分出的3类桑黄属的明确分类地位还需要进一步的研究。同时,本文虽然成功地将ITS序列作为分子标记对供试样品种类进行了鉴定和区分,但由于ITS在种内间隔区上表现的差异较小,其不适宜属内种的区分,因此,对于本试验中3类桑黄属内种的遗传多样性有待进一步研究。

A,桑树桑黄;B,丁香桑黄;C,杨树桑黄。A, Sanghuangporus sanghuang; B, Inonotus baumii; C, Fuscoporia gilva.图6 野生桑黄子实体Fig.6 Fruit bodies of wild Sanghuang
