基于高通量测序的不同年龄恒河猴肠道菌群结构差异分析
2019-03-01朱华肖冲尚海泉郭亚茜杜晓鹏秦川
朱华,肖冲,尚海泉,郭亚茜,杜晓鹏,秦川
(卫健委人类疾病比较医学重点实验室,中国医学科学院医学实验动物研究所;北京协和医学院比较医学中心,北京 100021)
非人灵长类动物(non-human primate,NHP)具有与人接近的基因背景和表型特征,在脑功能、认知、神经-精神疾病、干细胞研究和传染性疾病机制研究与疫苗研发等领域有着广泛的应用[1]。恒河猴(rhesus macaques)是实验常用的非人灵长类动物,在分类上属于灵长目,猴科,猕猴属,主要分布地包括印度、孟加拉、尼泊尔、越南和我国的西南、华南等地[2-3]。用于构建动物模型的恒河猴需要有清晰的微生物背景。肠道中大多数细菌不能用传统的选择性培养方法进行分离、鉴定,限制了对恒河猴肠道微生物变化规律的研究。高通量测序技术一次可以对几十万、上百万条DNA分子序列进行测定,使研究者可以对一个物种的转录组、基因组、代谢组等进行细致全面的分析,目前已发展到第三代。本研究利用高通量测序技术对不同年龄恒河猴肠道菌群结构和丰度进行分析,为医学研究提供基础数据。
1 材料与方法
1.1 粪便样品的收集与处理
50只恒河猴由中国医学科学院医学实验动物研究所北方资源中心繁殖【SCXK(京)2014-11】,饲养在大群中【SYXY(京)2017-0027】。50只恒河猴随机分为两组,其中,成年组33只(5-10岁)、老年组17只(10岁以上)。每只动物的粪便样品在排泄后20 min内完成收集,所有动物的样品采集在1周内完成。冰盒保存带回实验室后,-80℃冰箱冷冻保存。实验经中国医学科学院医学实验动物研究所伦理委员会批准(ZH17004)。
1.2 PCR扩增、文库构建、测序及生物信息学分析
按参考文献[4]的方法进行基因组DNA提取和PCR扩增文库构建、测序及生物信息学分析。
1.3 统计学方法
所有实验数据采用SPSS 22.0 软件进行差异显著性分析,P< 0.05为差异有统计学意义。
2 结果
2.1 测序结果评估
对收集到的50个样本进行测序共得到4 125 768条原始下机reads,其中4 011 000条可用于OTU的划分。比较成年组、老年组原始下机reads数、过滤后得到的高质量reads数、过滤后得到的高质量序列占raw reads比例(effective%)、Clean reads中测序质量值大于20(错误率小于1%)的碱基数占总碱基(clean reads)数的比例,各组间差异无显著性(表1)。
2.2 肠道菌群多样性分析
根据OTUs代表序列与数据库注释文件对比,在 97%(种)水平上进行OTUs 划分,老年组所获得的OTUs数减少,与成年组比较差异有显著性(P=0.0238)。ACE或Chao1指数数值越大表示菌群丰度越高,老年组这两个指数的数值均降低,与成年组比较差异有显著性(P=0.0121,P=0.0174),说明随年龄增长,恒河猴肠道菌群丰度降低。Shannon指数越大、Simpson指数越小表示肠道菌群的多样性越强,老年组的Shannon指数数值升高(P=0.0132),Simpson指数数值降低(P=0.0258),与成年组比较差异有显著性。提示恒河猴肠道菌群多样性随年龄增长而增高(表2)。

表1 数据过滤统计分析表Table 1 Data filtering statistical analysis

表2 成年与老年恒河猴肠道菌群α-多样性分析指数比较Table 2 Comparison of the results of α-diversity analysis of intestinal flora of the adult and old groups
注:与成年组比较,*P< 0.05(下表同)。
Note. Compared with the adult group,*P< 0.05.(The same in the following tables)
2.3 肠道菌群构成
测序结果显示:恒河猴2个年龄组的绝大多数reads主要分属以下10个门:厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、螺旋体门(Spirochaetes)、软壁体门(Tenericutes)、变形菌门(Proteobacteria)、疣微菌门(Verrucomicrobia)、unidentified_Bacteria、Kiritimatiellaeota、粘胶球形菌门(Lentisphaerae)、纤维杆菌门。老年组与成年组比较,拟杆菌门相对丰度升高(P=0.0013),厚壁菌门、螺旋体门、疣微菌门、粘胶球形菌门相对丰度降低(P=0.0283,P=0.0002,P=0.0482,P=0.0242),差异有显著性(见表3)。
在科水平,老年组与成年组比较,Prevotellaceae相对丰度升高(P=0.0001),Ruminococcaceae、Clostridiales、Spirochaetaceae、Christensenellaceae(P=0.0039,P=0.0080,P=0.0002,P=0.0021)相对丰度降低,差异有显著性(见表4)。
在属水平,老年组unidentified_Prevotellaceae相对丰度升高(P=0.0001),乳杆菌属(Lactobacillus),八迭球菌(Sarcina),unidentified_Spirochaetaceae相对丰度降低(P=0.0114,P=0.0227,P=0.0028),差异有显著性(见表5)。

表3 肠道菌群在门水平上的构成Table 3 Relative abundance of the intestinal flora at the phylum level
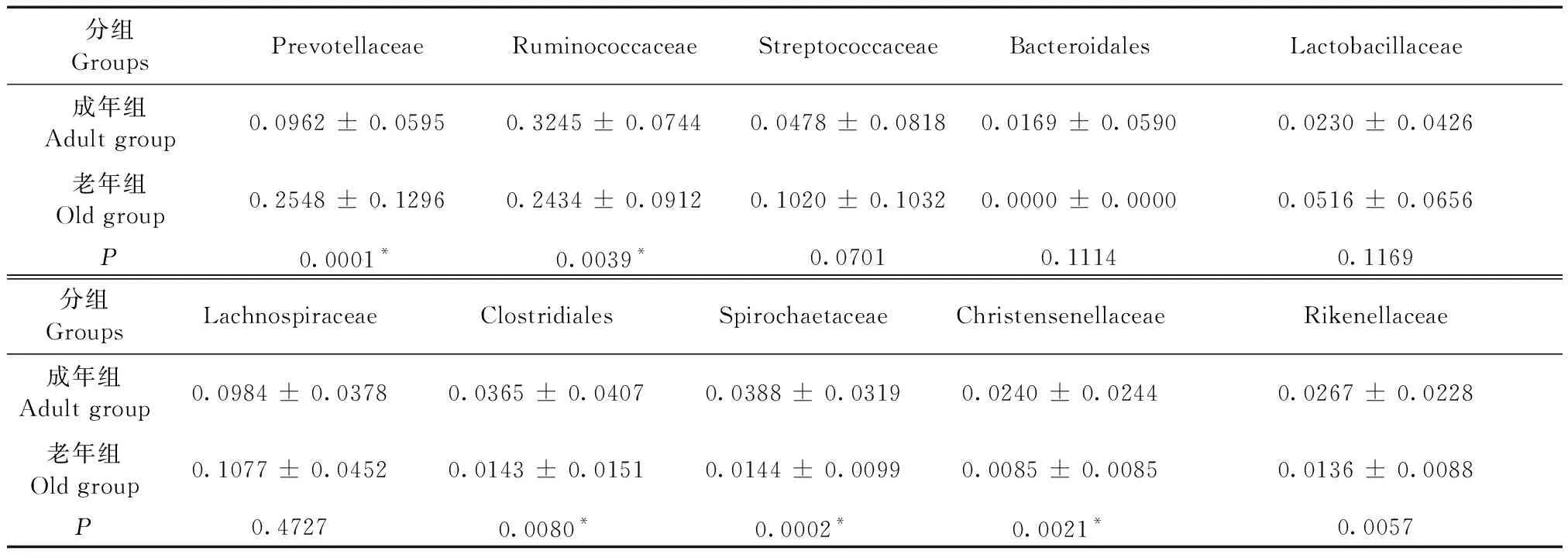
表4 肠道菌群在科水平上的构成Table 4 Relative abundance of the intestinal flora at the family level
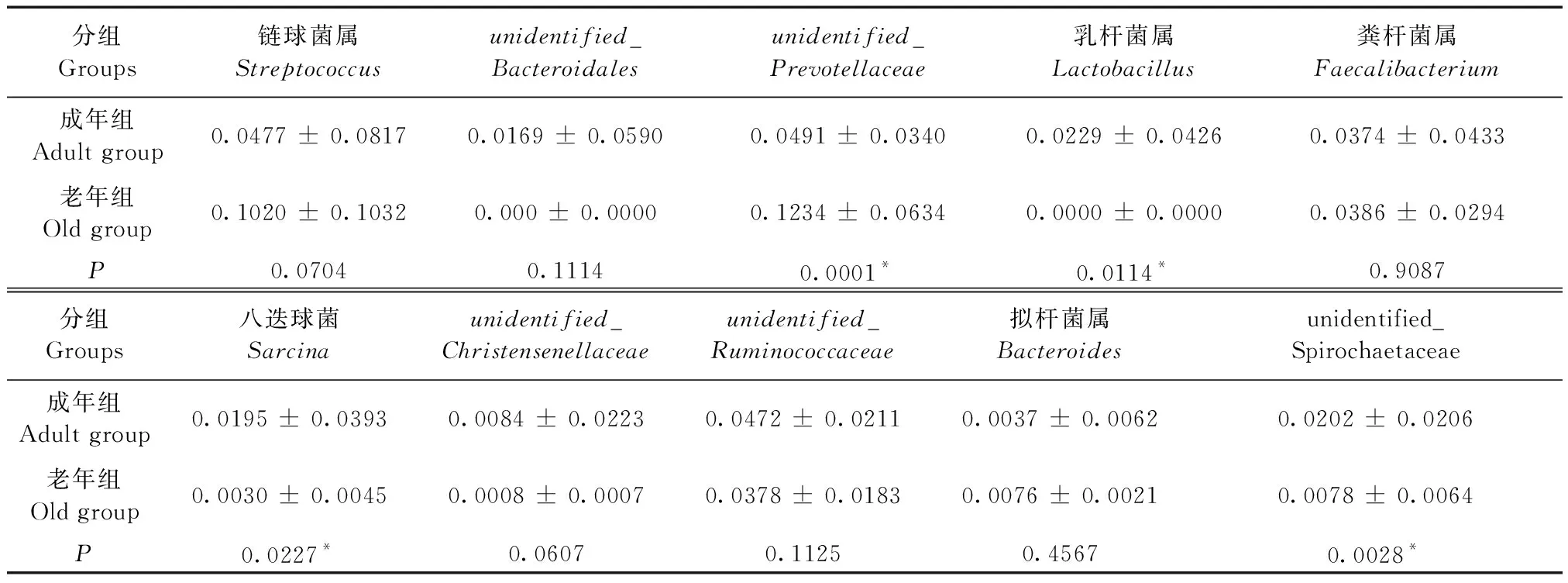
表5 肠道菌群在属水平上的构成Table 5 Gut microbiota distribution at the genus level
2.4 肠道菌群 LEfSe 分析
LEfSe(LDA effect size)分析的意义是在组间寻找具有统计学差异的生物标记物,并强调其统计学意义与生物物种的相关性,用来识别组间不同物种丰度的特征及其关联的类别。 在成年组,链球菌属(Streptococcus)、布赫纳氏菌属(buchnera)、乳杆菌属(lactobacillus)是具有统计学差异的生物标记物(图1)。
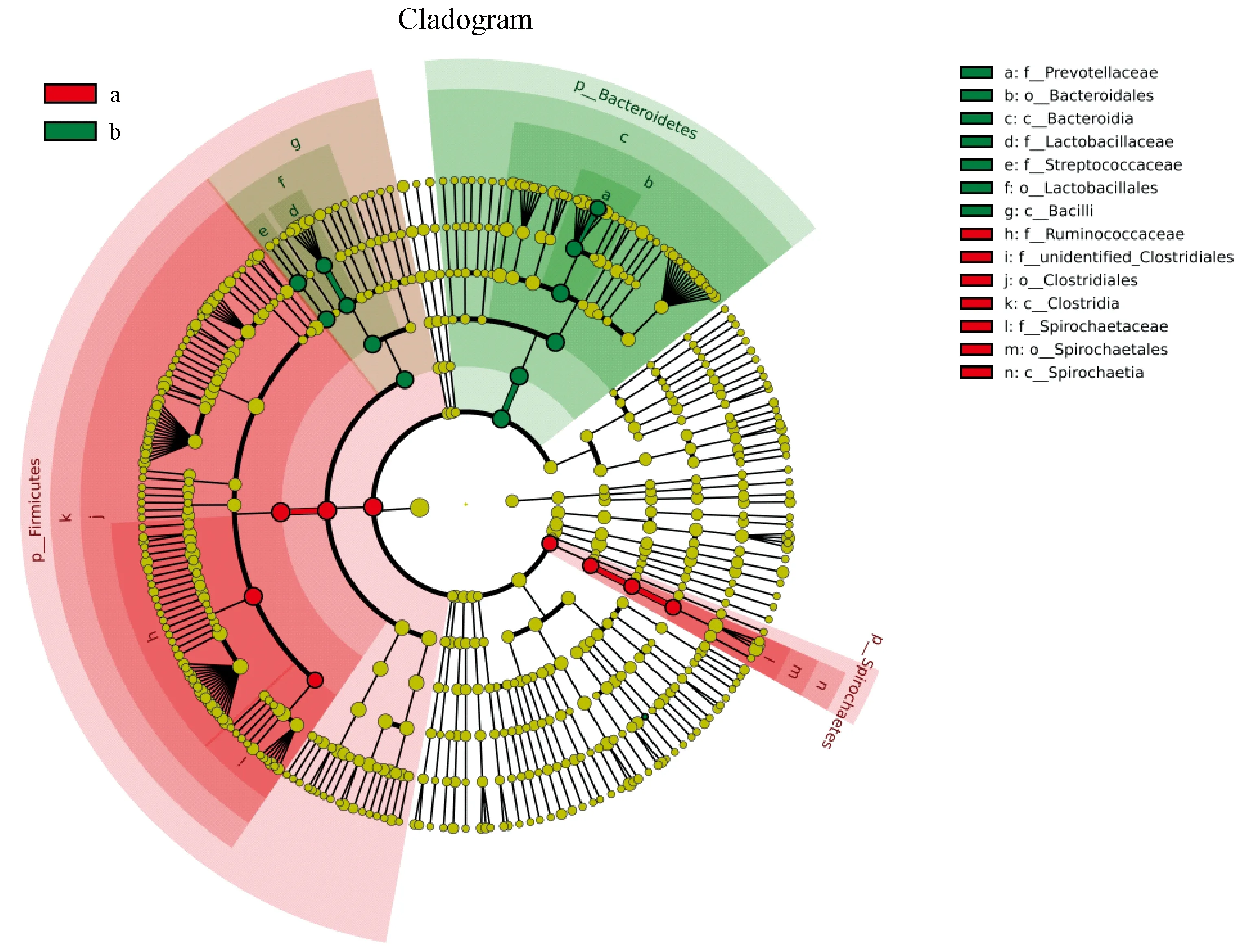
图1 不同年龄组动物LDA 值分布图Figure 1 Cladogram of the linear discriminant analysis (LDA) scores of the adult (a) and old groups (b)
2.5 β-多样性分析
β-多样性研究是使用 Weighted Unifrac 距离来衡量组间相异系数,这个系数越小,表示这两组间在物种多样性方面存在的差异越小。与成年组比较,老年组的 Weighted Unifrac 距离显著升高(P=0.003),表明成年组与老年组在物种多样性方面存在显著性差异,差异有显著性(图2A)。β-多样性分析结果显示,老年组恒河猴肠道菌群与成年组分布在不同区域(图2B)。
2.6 菌群功能预测分析
通过KEGG基因功能富集分析,结果显示青年组与老年组优势基因分区明显(图3A),青年组优势基因集中在与代谢相关的酵解酶、转运体、外泌体等(图3B)。
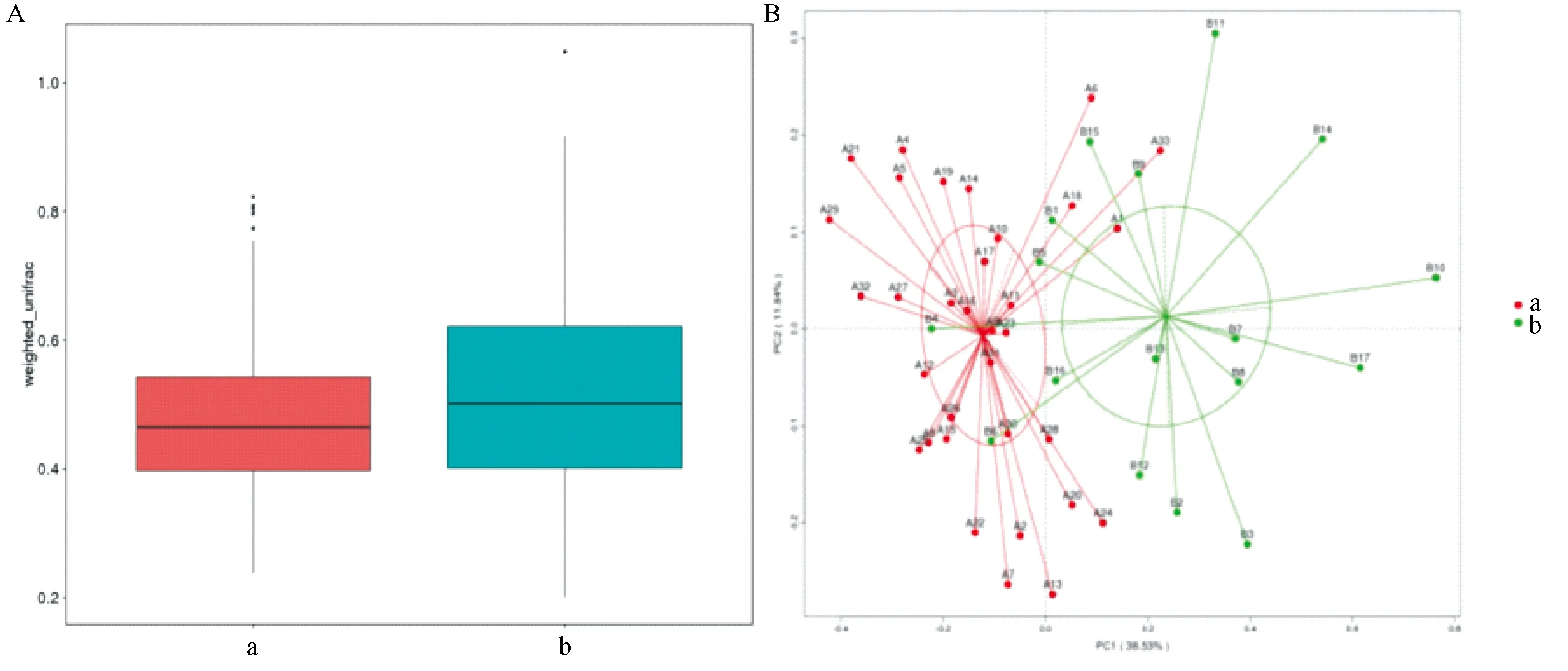
图2 成年组(a)与老年组(b)β-多样性分析Figure 2 β-diversity of the adult (a) and old groups (b)
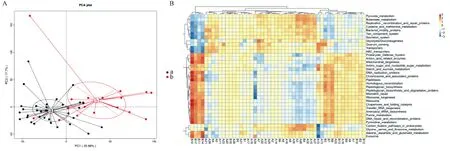
图3 成年组(a)与老年组(b)功能基因预测Figure 3 Gene function prediction of the adult (a) and old groups(b)
3 讨论
实验动物是医学研究的必要工具,美国、欧盟和中国的食品药品监督管理机构都规定新药研发临床前必须经过动物实验评价其安全性和有效性。高通量测序技术被广泛应用以来,更多的证据显示肠道菌群可通过很多方式影响人体的健康[5-6]。但如何影响?怎样通过合理利用动物模型进行相关的实验研究?研究者只有在充分了解实验动物与人体肠道菌群的差异后,才有可能进行更深入的探讨。高通量测序技术被广泛应用以来,对于人体菌群的研究计划很多:如由美国NIH发起的“人类微生物组计划”,欧盟发起的“人类肠道宏基因组计划”等。这些研究投入巨大,成果显著。相比之下,关于常用实验动物特别是非人灵长类肠道菌群的研究报道却很少。
由于宿主自身和外界环境等因素的影响,非人灵长类动物肠道菌群的结构和丰度存在着种间和种内的差异。影响因素主要包括以下4点:宿主种类及其系统发育关系[7-9]、食性[10-11]、年龄与性别[12-13]、社会互动[14-15]。本实验的研究对象均为恒河猴,饲喂统一的市售颗粒饲料,生活在同样的社会自然环境下,排除了种属、饮食、环境等因素的干扰,主要探讨年龄因素对宿主肠道菌群的影响。
据文献报道,人体肠道菌群在门水平主要由厚壁菌门、拟杆菌门、放线菌门和变形菌门构成,但其结构与相对丰度会随年龄增长而改变,老年人肠道菌群多样性程度比成年人高,未知菌种所占比例也比较高[16]。2006年,Mueller等[17]学者发表了一篇代表性研究成果:通过对法国、德国、意大利、瑞典四个欧洲国家230位健康志愿者的粪便样品进行分析,发现这四个国家所有参与分析的老年人肠道中肠杆菌含量普遍高,最后结论是这种菌群结构特征与志愿者所处地理位置无关,而主要与年龄相关。我们的测序结果显示,成年组、老年组恒河猴α-多样性指数存在显著性差异,随年龄增长,恒河猴肠道菌群丰度降低,多样性增加。β-多样性分析,老年组恒河猴肠道菌群与成年组分布在不同区域,老年组的 Weighted Unifrac 距离显著升高(P=0.003),表明成年组与老年组在物种多样性方面存在显著性差异。在丰度排名前10位的菌属中,未知菌属占一半,与人的结果一致。但特征性优势菌属与人不同。
非人灵长类肠道菌群主要由来自12个门的细菌组成的包括拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、 疣微菌门(Verrucomicrobia)、软壁菌门(Tenericutes)、放线菌门(Actinobacteria)、螺旋体门(Spirochaetes)、粘胶球形菌门(Lentisphaerae)、纤维杆菌门(Fibrobacteres)、 酸杆菌门(Acidobacteria)、梭杆菌门(Fusobacteria)和浮霉菌门(Planctomycetes)[2]。其中,拟杆菌门和厚壁菌门在所有非人灵长类肠道菌群中所占比例都很高,并且在多数非人灵长类中是比例最高的两个菌门。在我们的结果中,成年组、老年组恒河猴肠道菌群这两个门的比例分别为87.09%和91.94%,与报道相符[18]。
厚壁菌门/拟杆菌门比值是微生物发酵的能量产出指标之一[19],成年组、老年组恒河猴的这个比值分别为3.3654和1.8994(P=0.012),差异明显。在成年组的优势菌群中,值得关注度是乳杆菌属。乳杆菌属于厚壁菌门,在人体肠道中也普遍存在,是一种厌氧、无芽孢、革兰阳性菌,其发酵碳水化合物的终产物主要是乳酸。乳杆菌可刺激机体产生免疫球蛋白,增强机体的后天获得性免疫力,常被用于益生菌菌种[20]。乳杆菌还可以通过酸化肠道微生态环境阻止某些有害菌与肠粘膜上皮细胞的黏附,阻碍有害菌在肠道内定植,减少疾病的发生[21]。代谢产物乳酸是可以帮助机体消化吸收的短链脂肪酸。厚壁菌门/拟杆菌门比值、优势菌属的结果都与恒河猴成年个体比老年个体有更高的营养和能量需求的生理特点相符。功能基因的预测结果也验证了这个结果。
本实验利用高通量测序技术,从比较微生态学角度,对不同年龄恒河猴肠道菌群的结构与丰度进行分析,期望能够为肠道微生态与疾病关系研究用模型动物的选择与应用提供参考。
参考文献
[1] 胡新天, 仇子龙, 顾勇,等. 非人灵长类模型 [J]. 中国科学院院刊, 2016, 31(7): 773-782.
Hu XT, Qiu ZL, Gu Y, et al.Non-human primate brain model [J]. Clin Acad Sci J, 2016, 31(7):773-782.
[2] 伊丽娜,刘学聪,蒋志刚.非人灵长类肠道菌群组成及影响因素 [J]. 动物学杂志,2018, 53(3): 479-494.
Yi LN, Liu XC, Jiang ZG. Gut bacterial composition and its influencing factors of non-human primates [J]. Chin J Zool, 2018, 53(3): 479-494.
[3] Roger SJ, Gibbs RA. Comparative primate genomics: emerging patterns of genome content and dynamics [J]. Nat Rev Genet, 2014, 15(5): 347-359.
[4] 朱华,郭亚茜,杜晓鹏,等. 链脲佐菌素诱导糖尿病大鼠模型肠道菌群变化 [J]. 中国实验动物学报, 2018, 26(3): 349-356.
Zhu H, Guo YX, Du XP, et al. Analysis of gut microbiota in SD rat model of diabetes mellitus induced by streptozotocin [J]. Acta Lab Anim Sci Sin, 2018, 26(3): 349-356.
[5] Goodman AL, Mcnulty NP, Zhao Y, et al. Identifying genetic determinants needed to establish a human gut symbiont in its habitat [J]. Cell Host Microbe, 2009, 6(3): 279-289.
[6] Lee YK, Mazmanian SK. Has the microbiota played a critical role in the evolution of the adaptive immune system? [J]. Science, 2010, 330(6012): 1768-1773.
[7] Ochman H, Worobey M, Kuo CH, et al. Evolutionary relationships of wild hominids recapitulated by gut microbial communities [J]. PLoS Biol, 2010, 8(11): e1000546.
[8] Yildirim S, Yeoman CJ, Sipos M, et al. Characterization of the fecal microbiome from non-human wild primates reveals species specific microbial communities [J]. PLoS One, 2010, 5(11):e13963.
[9] Amato KR, Martinez-Mota R, Righini N, et al. Phylogenetic and ecological factors impact the gut microbiota of two Neotropical primate species [J]. Oecologia, 2016, 180(3): 717-733.
[10] McKenney EA, Ashwell M, Lambert JE, et al. Fecal microbial diversity and putative function in captive western lowland gorillas (Gorillagorillagorilla), common chimpanzees (Pantroglodytes), hamadryas baboons (Papiohamadryas) and binturongs (Arctictisbinturong) [J]. Integr Zool, 2014, 9(5): 557-569.
[11] Sun B, Wang X, Bernstein S, et al. Marked variation between winter and spring gut microbiota in free-ranging Tibetan macaques (Macacathibetana) [J]. Sci Rep, 2016, 6: 26035.
[12] Degnan PH, Pusey AE, Lonsdorf EV, et al. Factors associated with the diversification of the gut microbial communities within chimpanzees from Gombe National Park [J]. Proc Natl Acad Sci U S A, 2012, 109(32): 13034-13039.
[13] Amato KR, Leigh SR, Kent A, et al. The role of gut microbes in satisfying the nutritional demands of adult and juvenile wild, black howler monkeys (Alouattapigra) [J]. Am J Phys Anthropol, 2014, 155(4): 652-664.
[14] Moeller AH, Peeters M, Ndjango J, et al. Sympatric chimpanzees and gorillas harbor convergent gut microbial communities [J]. Genome Res, 2013, 23(10): 1715-1720.
[15] Tung J, Barreiro LB, Burns MB, et al. Social networks predict gut microbiome composition in wild baboons [J]. Elife, 2015, 4.e05224
[16] Arumugam M, Raes J, Pelletier E, et al. Enterotypes of the human gut microbiome [J]. Nature, 2011, 473: 174-180.
[17] Mueller S, Saunier K, Hanisch C, et al. Differences in fecal microbiota in different European study populations in relation to age, gender, and country: a cross-sectional study [J]. Appl Environ Microbiol, 2006, 72 (2): 1027-1033.
[18] Ley RE, Hamady M, Lozupone C, et al. Evolution of mammals and their gut microbes [J]. Science, 2008, 320(5883): 1647-1651.
[19] Turnbaugh PJ, Ley RE, Mahowald MA, et al. An obesity-associated gut microbiome with increased capacity for energy harvest [J]. Nature, 2006, 444(7122): 1027-1031.
[21] Ljungh A, Wadström T. Lactic acid bacteria as probiotics [J]. Curr Issues Intest Microbiol, 2006,7 (2): 73-89.
