云南新现蝙蝠SARS样冠状病毒密码子偏性及其聚类分析
2019-01-21,,,,,
,, ,,,
严重急性呼吸道综合症(Severe acute respiratory syndrome coronavirus, SARS)是由SARS冠状病毒(SARS-CoV)引起的一种严重的呼吸道疾病[1],是21世纪第1个在全球暴发流行的传染病。该病以其起病急、发展迅猛、死亡率高的特点,引起了全世界民众的恐慌[2]。SARS-CoV初步认为起源于蝙蝠SARS样冠状病毒(SARSr-CoV)[3],其中从菊头蝠体内分离出的冠状病毒基因组结构与SARS-CoV相似,且核苷酸一致性在88%~92%之间[4]。虽然到目前为止SARSr-CoV还未出现暴发流行,但其核苷酸与SARS-CoV的高度一致性,仍应引起相关部门的高度重视。
密码子(codon)是生物体内遗传信息传递不可缺少的物质,在编码氨基酸过程中,多种密码子可以编码同一种氨基酸,即同义密码子[5]。在不同的生物体内,甚至同一种生物不同的蛋白质基因对密码子的使用频率不尽相同,具有一定的偏性,即同义密码子使用的偏性[6]。密码子偏性在单细胞物种和多细胞物种的基因组和基因形成中起着重要作用[7-8]。同时,病毒密码子偏性的使用情况也可揭示不同毒株间的进化关系[5]。
2017年12月,中科院武汉病毒研究所石正丽课题组再次报道了11株云南新现蝙蝠SARSr-CoV,并与SARS-CoV的基因序列具有高度相似性,且其S蛋白上具有SARS-CoV的受体结合区(RBD)[3]。本研究以这11株云南新现的蝙蝠SARSr-CoV为主要对象,分析了它们与SARS-CoV密码子偏性的异同,并以密码子偏性为基础进行聚类分析,探索不同时期和地点发现的SARSr-CoV与SARS-CoV密码子偏性之间的关系。
1 材料与方法
1.1 材 料
1.1.1目的序列来源 本研究涉及的冠状病毒基因编码序列(CDS)均来自NCBI数据库(https://www.ncbi.nlm.nih.gov/)。
1.1.2使用软件 本研究使用EMBOSS(http://emboss.toulouse.inra.fr/)子程序CUSP计算密码子Frequency值[9],CodonW(https://sourceforge.net/projects/codonw/)计算密码子ENC、GC、GC3S、RSCU值[10],Lasergene子程序EditSeq和MegAlign用于比对和截取蛋白编码序列。使用Sigmaplot绘图、SPSS22.0进行聚类分析。
1.2 方 法
1.2.1有效密码子数 有效密码子数(Effective number of codon, ENC)是由 Wright[11]于1990 年提出的一种描述密码子使用偏离随机选择程度的方法。它是一个基因的密码子使用频率与同义密码子平均使用频率的量化值。同时该值可单独由密码子使用数据计算得出,与基因的长度及氨基酸组成无关[12],因此可以对基因的密码子偏性程度提供一个比较客观的标准。本研究采用CodonW计算出特定序列中有效密码子的ENC值。其取值范围为20(每个氨基酸只使用一个密码子的极端情况)~61(各个密码子均被平均使用)。因此,当ENC值越接近20时其偏性越强,而ENC值越接近61,其偏性就越弱甚至没有偏性[6]。
1.2.2相对密码子使用度 相对密码子使用度(Relative Synonymous Codon Usage,RSCU)是指对于某一特定的密码子在编码对应氨基酸的同义密码子间的相对频率,它去除了氨基酸组成对密码子使用的影响[13],当密码子不存在偏好时,该密码子的RSCU值等于1。当某一密码子的RSCU值大于1时,代表该密码子为使用相对较多的密码子,即偏性密码子,反之亦然[14]。
1.2.3ENC-Plot分析 ENC-Plot分析是用于确定密码子使用偏爱性的影响因素(尤其是突变偏倚/突变压力)。GC3S值代表同义密码子的第3个密码子位置中的鸟嘌呤或胞嘧啶的频率,不包括Met、Trp和终止密码子。ENC-Plot分析方法是用GC3S为横坐标,ENC为纵坐标,来预测ENC和GC3S之间的功能关系。即针对GC3S值绘制ENC值[11]。其中,标准曲线是指在不存在选择压力,密码子的偏性完全取决于突变压力的情况[15]。故当基点位于标准曲线上或散布在标准曲线附近时,说明密码子的偏爱性受突变影响较大。反之,说明密码子使用模式受到自然选择的影响比较大。
1.2.4中性分析 中性分析(Neutrality Plot)是另外一种确定密码子使用偏性的影响因素(尤其是自然选择偏倚/自然选择压力),是以每一个编码基因的密码子的GC1、GC2平均值为纵坐标,GC3为横坐标绘制的二维散点图。如果散点的趋势线分布于对角线(斜率=1),则表明基因仅受突变的影响。反之,斜率为0说明该基因在进化过程中受到选择压力的影响[14]。
1.2.5奇偶规则分析 奇偶规则分析(Parity Rule 2 plot analyses)也被应用于密码子使用偏性的分析。此规则是一种核苷酸内链规则,如果在2个互补链DNA之间不存在任何突变或选择效应上的偏倚,则预测A=T和G=C[16]。如果由4个密码子编码的氨基酸的第3位密码子中的奇偶规则存在显著偏倚,则在进化过程中则以自然选择压力为主[17]。因此规则规定仅选择由4个密码子编码的氨基酸。以G3/(G3+C3)|4为横坐标,以A3/(A3+T3)|4为纵坐标绘制二维散点图[15]。图的中心处遵循PR2原则,A=T且G=C,即横纵坐标都为0.5。从这个中心出发的矢量表示了PR2偏倚的程度和方向。
1.2.6基于密码子偏性的聚类分析 本研究采用CondonW计算出所需的SARS-CoV及SARSr-CoV基因序列的密码子使用频数,使用SPSS 22.0进行聚类分析。
2 结 果
2.1有效密码子数目分析 SARSr-CoV的全基因组由29 727个核苷酸组成,由11个开放阅读框(ORF)。共编码S、E、M和N共4种结构蛋白和ORF1a、ORF1b、ORF3a、ORF3b、ORF6、ORF7a、ORF7b、ORF8等多种功能未知的蛋白[18]。研究表明,新发现的11株云南蝙蝠SARSr-CoV各蛋白有效密码子数目(ENC)值均接近61(如表1所示),初步表明密码子偏性总体较低。并且,云南新现蝙蝠SARSr-CoV与SARS-CoV相同蛋白的ENC值非常接近。
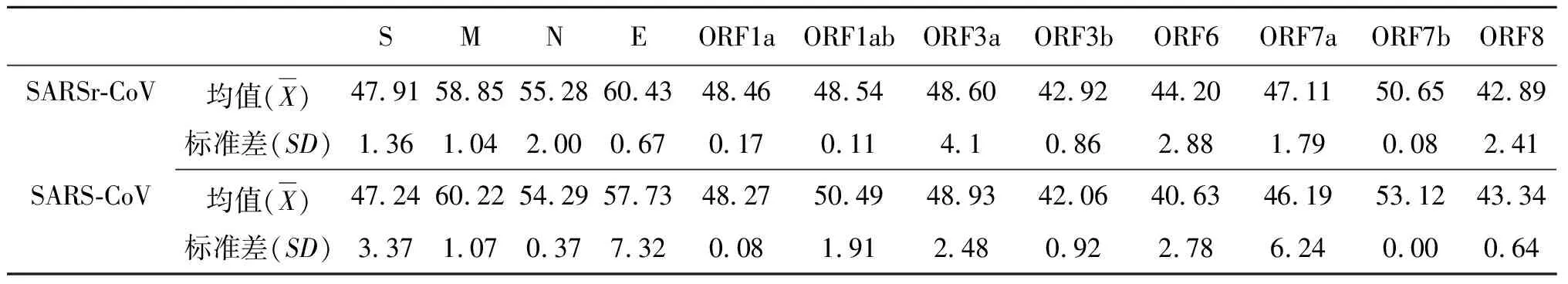
表1 云南新现蝙蝠SARSr-CoV与SARS-CoV的ENC值(均值)Tab.1 ENC value of emerging bat SARSr-CoV in Yunnan province and SARS-CoV (Average)
2.2相对密码子使用度 运用CodonW软件分别计算新现11株云南蝙蝠SARSr-CoV与SARS-CoV(SZ3、GZ02、Tor2、BJ01、PC4-227)12条蛋白各同义密码子的RSCU平均值(结果见表2)。结果显示,S、M、N、E、ORF1a、ORF1ab、ORF3a、ORF3b、ORF6、ORF7a、ORF7b、ORF8分别具有27、25、30、28、26、27、25、27、25、27、18、30个偏性密码子(RSCU>1),并以A、U结尾的密码子为主;其中,ACU为新现云南蝙蝠SARSr-CoV的12种蛋白共有的偏爱密码子,而GGG、CGG、AGC、UGG、UAG、AUC、AUG为非偏性密码子。

表2 云南新现蝙蝠SARSr-CoV的同义密码子平均使用度(RSCU)Tab.2 Relative synonymous codon usage (RSCU) of emerging bat SARSr-CoV in Yunnan province

AAC0.60 1.10 0.79 0.80 0.83 0.75 1.05 2.00 0.00 0.84 1.00 0.61 LysAAA1.22 1.33 1.37 2.00 0.99 1.02 1.28 0.91 0.30 1.33 2.00 1.45 AAG0.78 0.67 0.63 0.00 1.01 0.98 0.72 1.09 1.70 0.67 0.00 0.55 AspGAU1.18 0.38 0.96 2.00 1.23 1.25 0.82 0.00 1.53 0.97 1.00 0.81 GAC0.82 1.62 1.04 0.00 0.77 0.75 0.99 0.00 0.47 1.03 1.00 1.19 GluGAA1.07 1.04 1.01 2.00 1.11 1.10 0.86 0.00 0.80 0.79 1.50 1.84 GAG0.93 0.96 0.99 0.00 0.89 0.90 1.14 2.00 1.20 1.21 0.50 0.16 CysUGU1.18 1.27 0.00 0.67 1.29 1.28 0.61 0.09 0.00 0.68 1.00 1.48 UGC0.82 0.73 0.00 1.33 0.71 0.72 1.39 1.91 0.00 1.32 1.00 0.52 TERUGA0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.55 2.77 0.00 0.00 TrpUGG1.00 1.00 1.00 0.00 1.00 1.00 0.91 0.09 1.00 0.09 1.00 1.00 ArgCGU1.29 1.42 0.97 3.00 2.11 1.85 0.79 0.26 0.00 0.90 0.00 2.04 CGC0.56 1.32 1.55 0.00 0.64 0.76 0.00 0.00 0.00 0.08 0.00 2.18 CGA0.36 0.90 1.18 3.00 0.30 0.21 0.00 2.68 0.00 0.18 0.00 0.14 表2(续)AACondonSMNEORF1aORF1abORF3aORF3bORF6ORF7aORF7bORF8CGG0.02 0.45 0.00 0.00 0.08 0.10 0.00 0.08 0.00 0.06 0.00 0.00 SerAGU0.87 0.47 1.23 0.87 1.25 1.27 1.02 0.51 2.09 0.11 0.00 1.65 AGC0.37 0.94 0.90 0.87 0.50 0.49 0.83 0.99 0.00 0.78 0.00 0.27 ArgAGA2.06 1.04 1.83 0.00 2.09 2.25 4.42 1.62 2.00 4.72 0.00 1.37 AGG1.70 0.87 0.48 0.00 0.78 0.82 0.79 1.36 4.00 0.06 0.00 0.27 GlyGGU1.67 1.31 0.90 2.00 2.30 2.17 1.35 0.00 0.00 1.82 0.00 1.15 GGC1.19 0.90 1.41 0.00 0.80 0.83 1.85 4.00 0.00 0.91 0.00 0.55 GGA1.00 0.80 1.33 1.64 0.75 0.86 0.81 0.00 0.00 1.15 0.00 2.31 GGG0.14 1.00 0.36 0.36 0.16 0.14 0.00 0.00 0.00 0.12 0.00 0.00
终止密码子及Trp和Met(RSCU值为1)未列入表中;AA表示氨基酸
2.3ENC-Plot关联分析 本研究进一步绘制了SARS-CoV与新现的11株云南蝙蝠SARSr-CoV各蛋白的ENC和GC3S的散点图(图1),可以看出两者几乎落在了同一区域,这进一步说明两者的密码子偏好受到了相似因素的影响。其中大部分点落在了远离标准曲线的位置,这说明云南新现蝙蝠SARSr-CoV与SARS-CoV受自身突变和自然选择的双重影响,但以自然选择为主。
2.4中性分析 为进一步分析11株云南新现蝙蝠SARSr-CoV与SARS-CoV密码子偏爱性的影响因素,我们以S蛋白、ORF1a两蛋白作为结构蛋白与非结构蛋白的代表,分析突变及自然选择压力对两者密码子偏性的影响(图2)。
如图2所示,云南新现蝙蝠SARSr-CoV与SARS-CoV的S、ORF1a两蛋白的斜率(b)分别是0.208 4和0.257 9,更接近0,表明两者主要受到自然选择的影响。

图1 云南新现蝙蝠SARSr-CoV与SARS-CoV相关蛋白编码基因的ENC-Plot分析Fig.1 ENC-Plot analysis about the protein coding sequences of emerging bat SARSr-CoV in Yunnan province and SARS-CoV

图2 云南新现蝙蝠SARSr-CoV及SARS-CoV相关蛋白编码基因的中性绘图分析Fig.2 Neutrality Plot analysis of the protein coding gene of emerging bat SARSr-CoV in Yunnan province and SARS-CoV
2.5奇偶规则分析 为进一步明确新现的11株云南蝙蝠SARSr-CoV与SARS-CoV密码子偏性形成中自然选择压力的作用,我们分析了由4个密码子编码的氨基酸的第3位密码子中的奇偶规则。如图3所示,两者大部分基因落在了PR2图的左下方,这意味着在密码子第3位C和T(嘧啶)的使用频率高于G和A(嘌呤)。即其频率存在显著偏倚,提示两者在进化过程中主要受自然选择压力为主[17]。
2.6基于密码子偏性的聚类分析 我们进一步从密码子偏爱性角度分析了新发现的11株蝙蝠SARSr-CoV毒株与以往发现的SARSr-CoV毒株以及SARS-CoV毒株之间的进化关系。为了能够和石正丽课题组基于基因序列构建系统进化树的结果进行比较,我们同样选择了包膜蛋白S和非结构蛋白基因ORF1a 2个蛋白的密码子使用频率进行聚类分析,如图4。

图3 云南新现蝙蝠SARSr-CoV与 SARS-CoV相关蛋白编码基因的奇偶规则分析(PR2)Fig.3 Parity Rule Analysis (PR2) of the protein coding gene of emerging bat SARSr-CoV in Yunnan province and SARS-CoV

SARS-CoV为紫色字体;云南新现的蝙蝠SARSr-CoV为红色加粗字体;其余在云南省发现的SARSr-CoV为黑色加粗字体。图4 基于S蛋白、ORF1a蛋白密码子偏爱性的聚类分析Fig.4 Cluster analysis of S and ORF1a proteins based on codon bias
对S蛋白的聚类分析发现,云南新报道的11株蝙蝠SARSr-CoV分散聚类于先前报道的SARSr-CoV中。其中,新报道的Rs7327、Rs9401、Rs4084、Rs4231、Rs4874这5株云南蝙蝠SARSr-CoV与既往报道的3株云南SARSr-CoV (Rs/YN2013、Rs/WIVI1、Rs/WIV16)一起与SARS-CoV聚为一类。其中, Rs/YN2013、Rs/WIVI1、Rs/WIV16与SARS-CoV进化关系最为密切。新报道的6株云南蝙蝠SARSr-CoV(Rs4081、Rs4255、As6526、Rs4237、Rs4247、Rf4092)则与香港(HKU3-1~ HKU3-13)、广西(Rs/GX2013)发现的SARSr-CoV聚为一类。既往报道的云南株YNLF_31C、YNLF_34C、RS3367与贵州(Rs/Rs672)、广西(Rs/Rp3)、湖北(Rf1、Rm1、HuB2013)、河北(HeB2013)、吉林(RfJL2012)发现的病毒株聚在了一起;此外,山西毒株Rf/SX2013和陕西毒株Rp/SAX2011则单独聚为一类。
而非结构蛋白ORF1a的聚类分析发现,云南蝙蝠SARSr-CoV(包括11株新报道SARSr-CoV及以往报道的YNLF_31C和YNLF_34 C)均与SARS-CoV聚在了一起,提示这些毒株与SARS-CoV的密码子偏爱性相似度较高,该分支同时包括了来自于贵州(Rs/Rs672)、广西(Rs/Rp3、Rs/GX2013)、湖北(Rf1、Rm1)的毒株。而香港报道的SARSr-CoV毒株与SARS-CoV差异较大,与一株来自湖北的毒株(HuB2013)共同聚为一类。山西毒株Rf/SX2013、陕西毒株Rp/SAX2011、河北毒株HeB2013、湖北毒株Rf1、吉林毒株RfJL2012等SARSr-CoVs则共同聚为一类。
值得注意的是,与SARS-CoV密码子偏爱性最为密切的SARSr-CoV毒株均来自云南。其余省份检测到的SARSr-CoVs与SARS-CoV的距离较远。
3 讨 论
密码子是蛋白质编码基因中的基本单位和进化单位,生物信息学的发展使得分析与计算大量基因序列数据成为可能。本研究中,我们使用一些常用的生物信息软件围绕密码子使用模式,对新报道的11株云南蝙蝠SARSr-CoVs密码子偏性及其与SARS-CoVs以及以往报道的SARSr-CoVs的进化关系进行分析。
本研究发现,这11株SARSr-CoVs各蛋白间有效密码子数目(ENC)值均接近61,密码子偏性总体较低。此外云南蝙蝠SARSr-CoV与SARS-CoV进化过程中,在偏爱密码子的选择上非常接近。通过RSCU分析我们发现云南蝙蝠SARSr-CoV各蛋白偏爱性密码子以A、U结尾的密码子居多,这与中东呼吸综合征冠状病毒(MERS-CoV)偏爱以A、U结尾的密码子的结果相似[19]。ENC-plot、中性绘图分析、PR2分析均显示,云南蝙蝠SARSr-CoV与SARS-CoV的密码子偏性主要受到自然选择等其他因素的影响,该结果也和MERS-CoV一致[19]。
通过基于密码子偏性的聚类分析发现,无论是S蛋白还是ORF1a蛋白,与SARS-CoV密码子偏性相似度较高的SARSr-CoV毒株均来自云南。另发现这11株新报道的云南蝙蝠SARSr-CoV在非结构蛋白基因ORF1a上均彼此接近,且与SARS-CoV紧密地聚在了一起。这些结果提示,不论是结构蛋白和非结构蛋白,云南蝙蝠SARSr-CoV的密码子偏性与SARS-CoV具有较高的相似性,该结果也从密码子偏性的角度,佐证了云南蝙蝠SARSr-CoV可能是SARS-CoV进化起源的观点[3]。
此外,不同地区来源的SARSr-CoV其密码子偏爱性存在差异,并与地理位置之间可能存在一定的关联。该发现也与石正丽课题组在ORF1蛋白系统进化的结果一致[3]。这可能与邻近地区之间蝙蝠的SARSr-CoV相互传播过程有关。此外,中国其他省份发现的SARSr-CoV几乎均可在云南找到相似密码子偏性的毒株,这进一步验证了石正丽课题组关于云南蝙蝠SARSr-CoV可能是SARS-CoV、及其他地区SARSr-CoV重要的天然基因库的观点[3]。
