百合CBL1与CBL3基因的克隆与表达分析
2019-01-14张秀省高祥斌义鸣放吕福堂
曹 兴,张秀省,高祥斌,义鸣放,吕福堂*
(1.聊城大学农学院,山东聊城252059;2.中国农业大学园艺学院/花卉发育与品质调控北京市重点实验室,北京100193)
百合(Liliumspp.)为百合科百合属球根花卉,具有重要的观赏、食药用价值。百合多数喜微酸性土壤,土壤高盐分会抑制百合的生长发育,降低切花的产量和质量,并造成种球退化。因此,解析百合的盐胁迫响应机制,鉴定调控耐盐性的关键基因,对于采用基因工程技术来提高百合的耐盐性具有重要的理论和实践意义。
作为植物体中普遍存在的第二信使,Ca2+参与调控植物的生长发育和多种逆境胁迫响应。植物感受外界刺激后,细胞中的Ca2+浓度在时空上出现特异性的变化,这种变化会被相应的Ca2+感受器所感知,进一步将信号传递给下游的靶蛋白,从而引发细胞内一系列的生理生化反应来响应逆境胁迫。目前,植物中发现的Ca2+受体蛋白主要分为三类:钙调蛋白(calmodulin,CaM) 、类钙调磷酸酶B亚基蛋白(calcineurin B-like proteins,CBL)和钙依赖蛋白激酶(calcium-dependent protein kinase,CDPK)[1]。其中,CBL仅存在于高等植物中,与其下游靶蛋白CIPK(CBL-interacting protein kinase) 组成CBL-CIPK 信号转导途径,在植物响应逆境胁迫过程中发挥重要作用。到目前为止拟南芥中鉴定了10个CBL基因和25个CIPK基因,CBL1、CBL4、CBL10都能与CIPK24互作提高拟南芥的耐盐性[2-4]。
本研究以麝香百合杂种系(L.longiflorumHybrids)品种‘白天堂’(‘White Heaven’)为试验材料,从中克隆了CBL基因,分析了百合CBL蛋白特性及CBL基因在盐胁迫下的表达模式,以期为进一步研究CBL基因在百合耐盐性调节方面的功能及作用机制提供理论参考。
1 材料与方法
1.1 试验材料
试验材料为麝香百合杂种系品种‘白天堂’(L.longiflorumcv. ‘White Heaven’)组培苗,培养条件为22 ℃,光照时间为16 h/d。亚细胞定位使用的材料为紫皮洋葱(Alliumcepa)。引物由生工生物工程(上海)股份有限公司合成,试验所用引物见表1。DNA测序由北京六合华大基因科技有限公司完成。
1.2 试验方法
1.2.1 百合CBL基因的克隆 取百合叶片用液氮研磨,根据天根公司多糖多酚植物总RNA提取试剂盒说明书提取RNA。cDNA的合成根据Takara公司的Reverse Transcriptase M-MLV(RNase H)说明书进行,反转录接头引物为AP。
(1)LlCBL1基因的克隆。基因5′端序列的克隆:按照TaKaRa公司5′-Full RACE kit说明书进行。根据实验室之前获得的百合CBL1基因片段设计两条下游特异引物SPR1和SPR2,与试剂盒自带的两条5′端接头引物进行巢式PCR扩增。第一轮引物为5′RACE Outer primer和SPR1,反应程序为:94 ℃ 5 min;94 ℃ 30 s,62 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 10 min。取第一轮产物1 μL进行第二轮反应,引物为5′ RACE Inner primer和SPR2,反应程序为:94 ℃ 5 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 1 min,33个循环;72 ℃ 10 min。
开放阅读框(Open Reading Frame,ORF)序列的校正:将已经获得的目的基因片段用DNAMAN 5软件拼接,并在NCBI(http://www.ncbi.nlm.nih.gov/)上预测其ORF,根据ORF两端序列设计上游特异引物SPF1和下游特异引物SPR3,用保真性较高的PrimeSTAR®HS DNA Polymerase扩增目的基因的ORF序列。反应程序为:98 ℃ 5 min;98 ℃ 10 s,60 ℃ 15 s,72 ℃ 50 s,33个循环;72 ℃ 10 min。
PCR产物用10 g/L的琼脂糖凝胶电泳检测后切胶回收,连接到pUM-T载体并转化至大肠杆菌DH5α感受态中,Amp抗性培养基过夜培养后,挑取单克隆进行菌落PCR检测,将含有目的基因片段的阳性克隆送公司测序。
(2)LlCBL3基因的克隆。根据NCBI上已报道的拟南芥(Arabidopsisthaliana)AtCBL3的mRNA序列,利用BLAST程序搜索与其高度相似的百合表达序列标签(Expressed Sequence Tag,EST)。然后将该序列在NCBI上进行同源比对,用ORF Finder程序预测其ORF。在起始密码子上游设计特异引物SPF2(上游),在终止密码子下游设计特异引物SPR4(下游),使用PrimeSTAR®HS DNA聚合酶进行ORF全长校正,PCR反应条件:98 ℃ 5 min,98 ℃ 10 s,60 ℃ 15 s,72 ℃ 1 min,33个循环;72 ℃ 10 min。
1.2.2 LlCBL1与LlCBL3的生物信息学分析 用Primer Premier 5软件将目的基因的ORF序列翻译成氨基酸序列;用EXPASY (https://www.expasy.org/tools/)中的ProtParam、SMART、SignalP、TMHMM、PSORT、NetPhos,PSMP(http://plantsp.genomics.purdue.edu/myrist.html)等在线工具对蛋白质进行生物信息学分析;用DNAMAN 5对氨基酸序列进行多重比对分析;用MEGA 5软件构建系统进化树。
1.2.3 LlCBL1与LlCBL3的亚细胞定位 利用DNAMAN 5分析LlCBL1 ORF序列的酶切位点,结合pSAT6-GFP载体,选用HindⅢ和PstI作为融合表达载体构建的酶切位点,并设计上游特异引物SPF3和下游特异引物SPR5进行PCR扩增。双酶切pSAT6-GFP空载质粒和PCR扩增产物,纯化酶切产物并用T4连接酶连接,获得pSAT6-LlCBL1-GFP重组表达载体,双酶切和测序验证连接是否成功。用同样的方法构建pSAT6-LlCBL3-GFP重组表达载体,选用的限制性内切酶为HindⅢ和PstI,设计的特异引物为SPF4(上游)和SPR6(下游)。用基因枪轰击法[5]将构建好的重组表达载体轰击到在MS培养基上预培养24 h的洋葱表皮细胞中,暗培养24 h后用激光共聚焦显微镜(Nikon Eclipse TE2000-E)观察和拍照,并用EZ-C1软件对照片进行分析和处理。
1.2.4LlCBL1与LlCBL3的表达分析 选择生长良好、长势一致的‘白天堂’组培苗作为基因表达分析的材料。
LlCBL1、LlCBL3在不同组织中的表达:分别取常温(22 ℃)培养的百合组培苗的根、鳞茎和叶片。
LlCBL1、LlCBL3在盐胁迫下的表达:常温(22 ℃)下,用20 mL 100 mmol/L NaCl溶液浸没百合组培苗根系,分别在处理0、1、3、6和12 h取叶片。
所取组织用液氮速冻并保存于-80 ℃的超低温冰箱中备用提取RNA。采用实时荧光定量PCR(quantitative real-time PCR,qPCR)的方法检测基因的表达,LlCBL1的扩增引物为qF1和qR1,LlCBL3的扩增引物为qF2和qR2,内参基因18S rRNA的扩增引物为18SqF和18SqR。实验参照Takara公司的SYBR Premix Ex TaqTMkit荧光定量试剂盒说明书进行,仪器为Applied Biosystems Prism 7300 PCR仪。采用两步法进行PCR扩增,反应程序为:95 ℃ 10 s,95 ℃ 5 s,60 ℃ 31 s,40个循环;95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s,1个循环。每个样品重复3次。采用2-ΔΔCT方法[6]分析基因的相对表达量,用Excel软件分析数据。

表1 PCR引物
表中下划线表示酶切位点
Underline represents enzyme cleavage site
2 结果与分析
2.1 百合LlCBL1与LlCBL3基因的克隆
将获得的目的基因在NCBI上进行BLAST比对,分别定名为LlCBL1(GenBank登录号JQ911694)和LlCBL3(GenBank登录号JQ713001)。其中,LlCBL1基因ORF区序列长为642 bp,编码213个氨基酸(图1,a);LlCBL3基因ORF区序列长为672 bp,编码223个氨基酸(图1,b)。
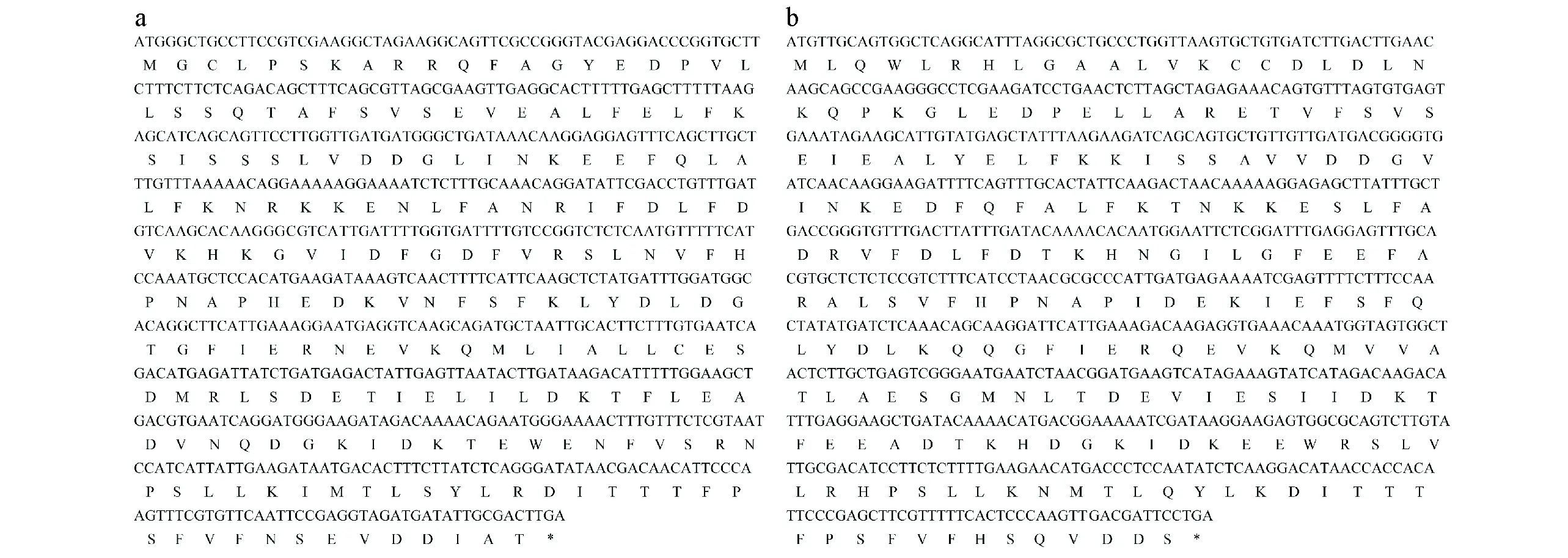
a.LlCBL1开放阅读框及编码的氨基酸序列;b.LlCBL3开放阅读框及编码的氨基酸序列a.The open reading frame of LlCBL1 and the predicted amino acid sequences;b.The open reading frame of LlCBL3 and the predicted amino acid sequences图1 百合CBL基因开放阅读框及编码的氨基酸序列Fig.1 The open reading frame of lily CBL genes and the predicted amino acid sequences
2.2 百合LlCBL1与LlCBL3的生物信息学分析
ProtParam在线工具预测百合LlCBL1蛋白包含213个氨基酸残基,分子式为C1102H1709N279O334S6,分子量为24.41 ku,属于稳定蛋白、亲水性蛋白和酸性蛋白。LlCBL3蛋白包含223个氨基酸残基,分子式为C1162H1806N294O349S6,分子量为25.67 ku,属于不稳定蛋白、亲水性蛋白和酸性蛋白。蛋白质信号肽预测程序SignalP预测LlCBL1和LlCBL3均不含信号肽,蛋白质跨膜区域预测软件TMHMM未发现LlCBL1和LlCBL3有明显的跨膜区域,蛋白质亚细胞定位工具PSORT预测LlCBL1主要定位于细胞质、细胞核内,LlCBL3主要定位于细胞质、线粒体内。
蛋白质磷酸化位点预测工具NetPhos预测LlCBL1含有13个丝氨酸(Ser)、5个苏氨酸(Thr)及1个酪氨酸(Tyr)潜在磷酸化位点,LlCBL3含有12个丝氨酸(Ser)、7个苏氨酸(Thr)潜在磷酸化位点,这些潜在的磷酸化位点可能是CBL活性和功能调控所必需的。蛋白质结构域预测工具SMART预测LlCBL1和LlCBL3均含有4个EF手型基序(EF-hand motif)(图2)。EF手型结构域是CBL的特征结构,该结构能够结合Ca2+,使得CBL能够解码环境Ca2+信号。与百合钙调蛋白LlCaM3[7]的典型EF手(canonical EF-hand)相比,LlCBL1的EF1和EF2的关键氨基酸位点发生了取代,导致Ca2+结合活性降低甚至不能结合Ca2+,这些称为非典型EF手(noncanonical EF-hand),因此LlCBL1含有2个典型EF手型基序和2个非典型EF手型基序。与LlCaM1相比,LlCBL3的4个EF手均为非典型EF手型基序。LlCBL1和LlCBL3的C端含有1个保守的PFPF基序(PFPF motif),在该结构域中被苯丙氨酸-脯氨酸残基(Phe-Pro)和Phe残基包围的Ser残基是CIPK磷酸化作用的靶点(图2)。与LlCBL3相比,蛋白质豆蔻酰化预测工具PSMP在LlCBL1的N端发现了1个豆蔻酰化基序(N-Myristoylation motif)(图2),该基序可能在LlCBL1的蛋白互作和膜锚定过程中发挥重要作用。
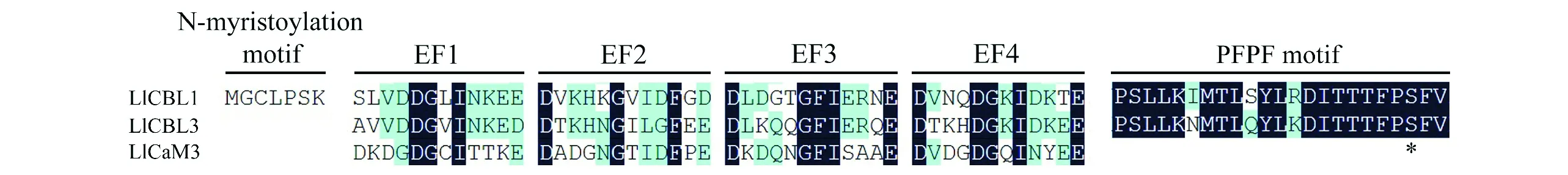
Ll:百合Lilium longiflorum;LlCBL1:AFU36090;LlCBL3:AFI41211;LlCaM3:AFA89863图2 百合CBL与CaM氨基酸序列比对Fig.2 Alignment of the amino acid sequences of LlCBL1,LlCBL3 and LlCaM3

图中各节点处数字代表可信度。Eg:油棕;Pd:海枣;Ao:石刁柏;Ll:百合;Os:水稻;Ta:小麦;Vv:葡萄;At:拟南芥The numbers at the node of the figure is credibility.Eg:Elaeis guineensis;Pd:Phoenix dactylifera;Ao:Asparagus officinalis;Ll:Lilium longiflorum;Os:Oryza sativa;Ta:Triticum aestivum;Vv:Vitis vinifera;At:Arabidopsis thaliana图3 百合LlCBL1、LlCBL3与其他植物CBL家族系统进化树Fig.3 Phylogenetic relationship of LlCBL1,LlCBL3 with other plant CBLs
将百合LlCBL1及与其同源性较高的其他植物的CBL1、LlCBL3及与其同源性较高的其他植物的CBL3、拟南芥AtCBL1-AtCBL10的氨基酸序列,用MEGA 5构建系统进化树,结果显示CBL家族可分为两个亚族:第Ⅰ亚族包括CBL1、CBL4、CBL5、CBL8、CBL9;CBL2、CBL3、CBL6、CBL7、CBL10归属第Ⅱ亚族。百合LlCBL1与同为单子叶植物的油棕(Elaeisguineensis)EgCBL1、海枣(Phoenixdactylifera)PdCBL1的亲缘关系最近(图3),相似度达92%。百合LlCBL3与同为百合科的石刁柏(Asparagusofficinalis)AoCBL3的亲缘关系最近(图3),相似度达92%。特征结构域和系统进化分析的结果进一步说明本研究分离得到的LlCBL1和LlCBL3基因分别属于植物CBL1和CBL3的同源基因。
2.3 百合LlCBL1与LlCBL3亚细胞定位
利用基因枪将构建好的瞬时表达载体pSAT6-LlCBL1-GFP和pSAT6-LlCBL3-GFP轰击到经过24 h预培养的洋葱表皮细胞中,暗培养24 h后放置于激光共聚焦显微镜下观察。结果表明,空载对照pSAT6-GFP的荧光信号分布在整个细胞中,包括细胞膜、细胞质和细胞核,在细胞膜、细胞质和细胞核中也检测到了pSAT6-LlCBL3-GFP的荧光信号,而pSAT6-LlCBL1-GFP的荧光信号仅在细胞膜中检测到(图4)。表明LlCBL3在细胞膜、细胞质和细胞核中表达,LlCBL1在细胞膜中表达。
2.4 百合LlCBL1与LlCBL3的表达分析
利用qPCR检测了LlCBL1与LlCBL3的时空表达。结果表明,常温(22 ℃)下,LlCBL1、LlCBL3在百合根、鳞茎和叶中均有表达,LlCBL1在根中的表达量最高,鳞茎中次之,叶中最低;LlCBL3在叶中的表达量最高,根中次之,鳞茎中最低(图5,a)。在盐胁迫处理1~12 h内,百合叶片中LlCBL1的相对表达量呈现出先上升后下降的趋势,但始终高于对照,在处理6 h时达到峰值,约为对照的6.33倍(P<0.05);百合叶片中LlCBL3的相对表达量也呈现出先上升后下降的趋势,但仅在1~3 h内的相对表达量显著高于对照(P<0.05),盐胁迫处理3 h的相对表达量是对照的3.10倍(图5,b)。

图4 LlCBL1与LlCBL3在洋葱表皮中的亚细胞定位Fig.4 Subcellular localization of LlCBL1 and LlCBL3 in onion epidermal cells

a.LlCBL1与LlCBL3在根、鳞茎、叶中的表达;b.叶片LlCBL1与LlCBL3在不同NaCl处理时间下的表达,*表示各点与0 h的表达量在P<0.05水平差异显著a.The expression of LlCBL1 and LlCBL3 in root,bulb and leaf;b.The expression of LlCBL1 and LlCBL3 in leaf treated with 100 mM NaCl at different time,*represent significant correlation at 0.05 level图5 LlCBL1与LlCBL3时空表达模式Fig.5 Expression pattern of LlCBL1 and LlCBL3 in lily
3 讨论
百合属于盐敏感性植物,我国北方地区土壤的盐渍化阻碍了百合的种植及发展,因此培育耐盐品种是百合育种的重要内容。目前,有关百合的盐胁迫研究主要集中在以下几个方面:(1)百合对盐胁迫的形态及生理响应[8-9]。(2)百合种质耐盐性比较[10-11]。(3)利用化学、微生物或杂交育种的方法提高百合的耐盐性[12-15]。除常规杂交育种外,百合耐盐转基因育种也取得了初步进展。盐胁迫条件下,转JERF3基因的百合小鳞茎的发芽率显著高于野生型,死亡率显著低于野生型,表明转基因百合的耐盐性能明显提高[16]。将6-磷酸山梨醇脱氢酶(S6PDH)基因导入麝香百合,盐胁迫下转化植株的电导率和MDA含量均小于对照,可溶性蛋白含量高于对照,且具有比对照更高的酶活性,说明转化苗的抗盐性较强[17]。
对百合耐盐功能基因的研究,除了上述的S6PDH、JERF3外,还包括APX[18]、LLA23[19]等,但对于在植物盐胁迫响应中起关键作用的CBL基因的研究尚未见报道。CBL通过与CIPK互作解码特异钙信号所组成的Ca2+-CBL-CIPK信号系统是植物逆境信号传导的关键途径,在植物的耐盐性调控中发挥重要作用[20]。CBL为多基因家族,现已发现拟南芥、水稻、玉米和杨树CBL家族均含有10个成员,本研究从百合中分离鉴定了LlCBL1和LlCBL3基因,还分离鉴定了LlCBL2与LlCBL4(未发表),百合的基因组较大,推测百合中还存在其他CBL基因。CBL蛋白主要由N端和C端两个球状结构域组成,每个结构域均含有一对EF手型基序,每个EF手型基序由12个氨基酸残基组成。与CaM的典型EF手型基序的氨基酸序列(DXD(N)XD(N)(S)GXI(V)D(N)(S)XXE)相比,CBL的EF手型结构存在不同程度的变异,如拟南芥CBL家族的10个成员中,AtCBL1和AtCBL9有2个典型EF手型基序,AtCBL6、AtCBL7、AtCBL8和AtCBL10只含有一个典型EF手型基序,而AtCBL2、AtCBL3、AtCBL4和AtCBL5则没有典型EF手型基序[1]。百合LlCBL1和LlCBL3的EF手型基序的部分关键位点也发生了氨基酸取代,LlCBL1含有2个典型EF手型基序和2个非典型EF手型基序,LlCBL3的4个EF手型基序均为非典型EF手型基序。EF手型基序中氨基酸序列的变化导致CBL具有不同的Ca2+结合活性,可能解码来自不同环境胁迫的Ca2+信号。在LlCBL1和LlCBL3的C端含有保守的PFPF基序,其中P、M、L、F、P和F氨基酸残基完全保守,位于P与F之间的S残基是CIPK磷酸化作用的靶点,这一磷酸化机制能够促进CBL与CIPK互作,并参与对下游靶蛋白活性的调控,是CBL-CIPK信号途径的重要调控机制之一[21]。根据N端的结构,CBL可以分为两个亚族,第Ⅰ亚族的N端具有高度保守的豆蔻酰化模序MGCXXSK/T,豆蔻酰化是蛋白质翻译后修饰的一种方式,在蛋白互作和膜定位方面发挥作用。在MGCXXSK/T模序中,豆蔻酰化作用发生在蛋氨酸后的甘氨酸残基,发生豆蔻酰化作用的CBL蛋白具有较低的膜定位活性,但当甘氨酸后面的半胱氨酸残基发生棕榈酰化后,就能将蛋白定位在细胞膜上[22]。在LlCBL1的N端发现了1个豆蔻酰化基序,亚细胞定位的结果证实了LlCBL1主要在细胞膜上表达,这与对同属第Ⅰ亚族AtCBL1和AtCBL9的研究结果类似[20]。第Ⅱ亚族的N端较长,缺少豆蔻酰化、棕榈酰化等脂质修饰位点。LlCBL3归属于第Ⅱ亚族,亚细胞定位的结果表明LlCBL3在细胞膜、细胞质和细胞核中均有表达,这与同属第Ⅱ亚族的AtCBL3在细胞膜和液泡膜上表达的结果不同[20]。同属第Ⅱ亚族的AtCBL2和AtCBL6定位于液泡膜上,AtCBL10定位在细胞膜和液泡膜上,AtCBL7在细胞膜、细胞质和细胞核中均有表达[20]。这些多样而特异的亚细胞定位暗示CBL作为钙感受器能够快速传递局部钙释放事件。
在植物进化过程中,CBLs和CIPKs基因数量的增加很可能与CBL-CIPK信号系统的功能进化相关,这有利于时空特异性Ca2+信号的识别与解码,暗示CBL和CIPK进化的复杂性与植物的环境适应性相一致[23]。拟南芥可以通过CBL4-CIPK24-SOS1通路[3]或CBL10-CIPK24-NHX通路[4]来提高植株对盐胁迫的耐受性。过表达玉米ZmCBL9、烟草NsylCBL10或杨树PeCBL6提高了转基因拟南芥的耐盐性[24-26]。AtCBL1通过互作将AtCIPK24带至细胞膜参与盐胁迫反应,AtCBL1还可以与AtCIPK23互作调控AKT1介导的根细胞K+的吸收与运输,AtCBL1还参与了干旱胁迫和冷胁迫应答等[23]。沙冬青AmCBL1受盐、干旱和CaCl2诱导表达,在烟草中过表达AmCBL1提高了植株的耐盐性和耐旱性[27]。杜梨PbCBL1的表达受盐胁迫诱导上调,PbCBL1的转入可以提高大肠杆菌对盐胁迫的耐受能力[28]。盐胁迫显著促进了水稻OsCBL1、OsCBL3和杨树PeCBL1、PeCBL3的表达[29-30]。本研究中,LlCBL1和LlCBL3的表达受盐胁迫诱导,但二者的表达模式存在差异。LlCBL3在早期(1~3 h)响应盐胁迫,LlCBL1在1~12 h内持续响应盐胁迫,且LlCBL1的相对表达量上调倍数显著高于LlCBL3。
多基因家族、多样而特异的表达模式和亚细胞定位表明了CBL/CIPK信号转导的特异性与功能的多样性。课题组从百合中分离了两个CBL家族基因LlCBL1和LlCBL3,研究了LlCBL1和LlCBL3的亚细胞定位和基因表达模式,结果表明LlCBL1可能与百合的盐胁迫响应密切相关,但是还需要进一步在模式植物和百合上分析基因功能。本研究为探讨百合CBL基因在百合耐盐性调节方面的功能及作用机制奠定了理论基础。
