花生PDAT基因家族的全基因组生物信息学分析
2019-01-14田海莹李新国张智猛孟静静万书波彭振英
田海莹,单 雷,李新国,郭 峰,张智猛,孟静静,万书波,彭振英*
(1.山东省农业科学院生物技术研究中心/山东省作物遗传改良与生态生理重点实验室,山东 济南 250100;2.山东省花生研究所,山东 青岛 266100; 3.山东省农业科学院,山东 济南 250100)
花生是我国重要的油料作物之一,在国民食用油供给中具有举足轻重的地位[1-2]。植物体以甘油三磷酸为骨架、以脂肪酸为底物通过不同的代谢途径合成三酰甘油(triacylgycerol,TAG),最终以油体形式贮藏在种子中,成为植物油的主要成分。过去研究认为,TAG主要是由二酰甘油酰基转移酶(diacylglycerol acyltransferase, DGAT)催化完成的,二酰甘油 (diacylglycerol, DAG) 和脂酰 CoA在DGAT的催化下合成TAG,DGAT是TAG合成的限速酶,这一途径被称为Kennedy途径[3-4],该途径依赖脂酰-CoA。后来研究发现植物从DAG到TAG的合成还有另外的途径,这是一种不依赖脂酰-CoA的合成途径,即PDAT(phospholipids:diacylglycerol acyltransferase,PDAT)途径,PDAT催化DAG和磷脂合成TAG与溶血磷脂[5]。
PDAT活性最早是由Dahlqvist等人在向日葵(Helianthusannuus)、蓖麻(Ricinuscommunis)、还阳参属植物(Crepispalaestina)的微粒体中发现的[6]。随后他们在酿酒酵母中也发现了PDAT活性,并鉴定了第一条PDAT基因,该基因与人类卵磷脂胆固醇酰基转移酶(lecithin-cholesterol acyltransferase,LCAT,EC 2.3.1.43)具有较高的相似性,并证实其在酵母TAG合成中具有重要作用[5, 7]。Fan等人认为PDAT和DGAT在TAG合成中功能存在部分冗余,且对于种子和花粉发育是必需的。同时,他们用拟南芥tgd1-1突变体证明PDAT1在营养组织TAG生物合成中是必需的,在此背景下破坏PDAT导致叶片发育严重迟缓,配子体缺陷甚至细胞过早死亡[8]。PDAT对底物的特异性在不同物种是存在差异的,对拟南芥AtPDAT1功能研究显示,该基因可以催化不同链长(C10-C22)以及不同饱和程度的酰基供体合成TAG,尤其是对含有多双键、羟化酰基和环氧化的酰基供体具有较高的偏好性[9]。在蓖麻中,Kim等发现RcoPDAT1-2具有与类似FAH12的功能,参与蓖麻油酸向TAG转化,被认为是蓖麻特异性基因。将该基因转化拟南芥显著提高了拟南芥种子中羟基脂肪酸的积累量,高达25%[10]。
在真核生物中,可变剪切(alternative splicing,AS)是一种重要的转录后调控机制[11],随着新一代测序技术以及多种组学的交叉应用,越来越多的AS基因被发现[12-14]。在拟南芥中60%多外显子基因具有AS现象;在番茄幼苗、花和发育幼果等组织中59.3%的基因发生了AS[15],水稻33%的基因具有这种情况[16-17]。虽然在不同物种中发生AS的基因数量与剪接类型各不相同,但它们在植物生长发育以及应对各种逆境胁迫中都具有重要作用。Zheng等(2017)研究了AhDGAT1基因7种可变剪接体,并分别在缺陷酵母H1426中验证,发现除AhDGAT1.2和AhDGAT1.4的C-末端截短没有DGAT酶活性外,其余五个较长的AhDGAT1变体表现出很高的酰基转移酶活性并且能够互补该菌株表型[18]。
到目前为止,在花生中对该基因的研究还未见相关文献报道。本研究利用生物信息学方法在全基因组水平对AhPDAT基因家族的进化关系、表达模式和可变剪接等方面进行了系统分析,为进一步深入研究该家族的功能奠定了理论基础。
1 材料与方法
1.1 基因检索
从TAIR数据库中下载2个已知的拟南芥PDAT基因(AT5G13640,AT3G44830)为探针,通过 BLAST 搜索花生、大豆、苜蓿等10个物种的PDAT同源基因。从花生数据库Peanutbase[1](https://www.peanutbase.org/)中下载基因组与蛋白数据,利用SeqHunter1.0,E-value<1.0e-15本地Blast,搜索AhPDAT基因。文中涉及的大豆、苜蓿其他植物的PDAT基因的相关序列来自Phytozome(http://phytozome.jgi.doe.gov/)数据库。同时,将序列利用在线网站Pfam Search(http://pfam.xfam.org/search)进行家族鉴定分析,预测的蛋白包含卵磷脂胆固醇酰基转移酶功能结构域(PF02450)即为PDAT蛋白。
1.2 AhPDATs基因结构分析
将AhPDATs的基因组序列和CDS利用在线网站Gene Structure Display Server(GSDS, http://gsds.cbi.pku.edu.cn/)绘制外显子-内含子结构图[19]。
1.3 AhPDATs序列特征与蛋白特性
运用BioXM2.6分析AhPDATs蛋白的理化性质;运用TMHMM(www.cbs.dtu.dk/services/TMHMM/)[20]在线分析分析跨膜结构域;运用CELLO v.2.5(http://cello.life.nctu.edu.tw/)在线软件预测亚细胞定位情况;运用SignalP4.1Server (http://www.cbs.dtu.dk /services/SignalP)在线预测信号肽。
1.4 AhPDATs系统进化与保守基序分析
为了研究花生与其他物种PDATs的进化关系,以酿酒酵母PDAT蛋白(NM_001183185)为外类群,选取1.1中获得的10个物种(蓖麻、菜豆、大豆、可可、棉花、苜蓿、拟南芥、亚麻、杨树、花生)中氨基酸数目大于500的37个PDATs蛋白序列,利用Clustal X进行多重比对,并运用 MEGA6.0软件中的邻接法(neighbor-joining,NJ)对构建进化树,设定泊松校正法计算进化距离,Bootstrap值设为1000[21]。同时,运用MEME[22](http://meme.nbcr.net/meme/cgi-bin/meme.cgi)在线分析PDAT 蛋白的保守基序信息,最大基序检索值设为10,并利用TBtools工具导出图片。
1.5 AhPDATs表达模式分析
为研究AhPDATs基因的时空表达模式,根据本实验室转录组测序数据(NCBI: PRJNA354652)中AhPDATs的FPKM值,利用HemI软件绘制其在根、叶、种子组织的表达模式图。转录组所测组织为花生的根(Root)、叶(Leaf)、果针入土30 d(Seed1)和50 d(Seed2)。
1.6 可变剪接分析
结合花生基因组(https://www.peanutbase.org/)和转录组数据(NCBI: PRJNA354652),用ASTALAVISTA program (http://genome.crg.es/astalavista/)分析其可变剪接情况。本研究涉及到5种AS事件,分别为:转录起始位置可变剪切(transcription start site,TSS)、转录终止位置可变剪接(transcription terminal site,TTS)、外显子跳跃(exon skipping,ES)、内含子滞留(intron retention,IR)、可变外显子 5'或3'端剪切(alternative exon 5' or 3'ends,AE)[23]。
2 结果与分析
2.1 AhPDATs基因家族的鉴定
通过Blast和Pfam鉴定,最终从花生数据库共检索到17个AhPDATs基因(表1),它们不均匀地分布在9条染色体上,其中A基因组中10条,B基因组中7条。Aradu.S9XBY和Araip.I19GZ、Aradu.AQ5JR和Araip.HH1X5、Araip.18K24和Aradu.4X75W、Aradu.UA9D8和Araip.WVH6X为4对同源基因,位于3对染色体上,其中Aradu.AQ5JR和Aradu.4X75W、Araip.18K24和Araip.HH1X5分别位于Aradu.A03和Araip.B03染色体上。
AhPDATs编码蛋白的氨基酸个数介于88~742之间,其中Araip.8M2BC编码最短,仅88个氨基酸,分子量为9.72 kD,序列不完整;Aradu.S9XBY编码最长,共742个氨基酸,分子量为83.76 kD。等电点的变化幅度比较大(表1),其中8个成员为酸性蛋白,9个为碱性蛋白,最小的是Araip.8M2BC,为4.37;最大的为Aradu.AQ5JR,达到9.6。
2.2 AhPDATs跨膜结构和亚细胞定位分析
利用TMHMM在线工具对17个AhPDATs进行跨膜结构分析,结果表明,仅有Araip.I19GZ、Araip.18K24、Aradu.S9XBY三个蛋白具有跨膜结构(见表1),其N端位于膜内,C端位于膜外。亚细胞定位分析发现,Aradu.UA9D8、Araip.WVH6X、Araip.8M2BC定位于叶绿体;Aradu.S9XBY、Araip.I19GZ、Aradu.ELK98定位在线粒体;Aradu.43KMV和Aradu.AQ5JR定位于质膜;Aradu.EJC3Z、Aradu.8Y8QZ、Araip.WT4S8定位于细胞外;其余均定位于细胞质中。此外,SignalP 在线分析显示,仅Aradu.4X75W存在信号肽(见表1)。
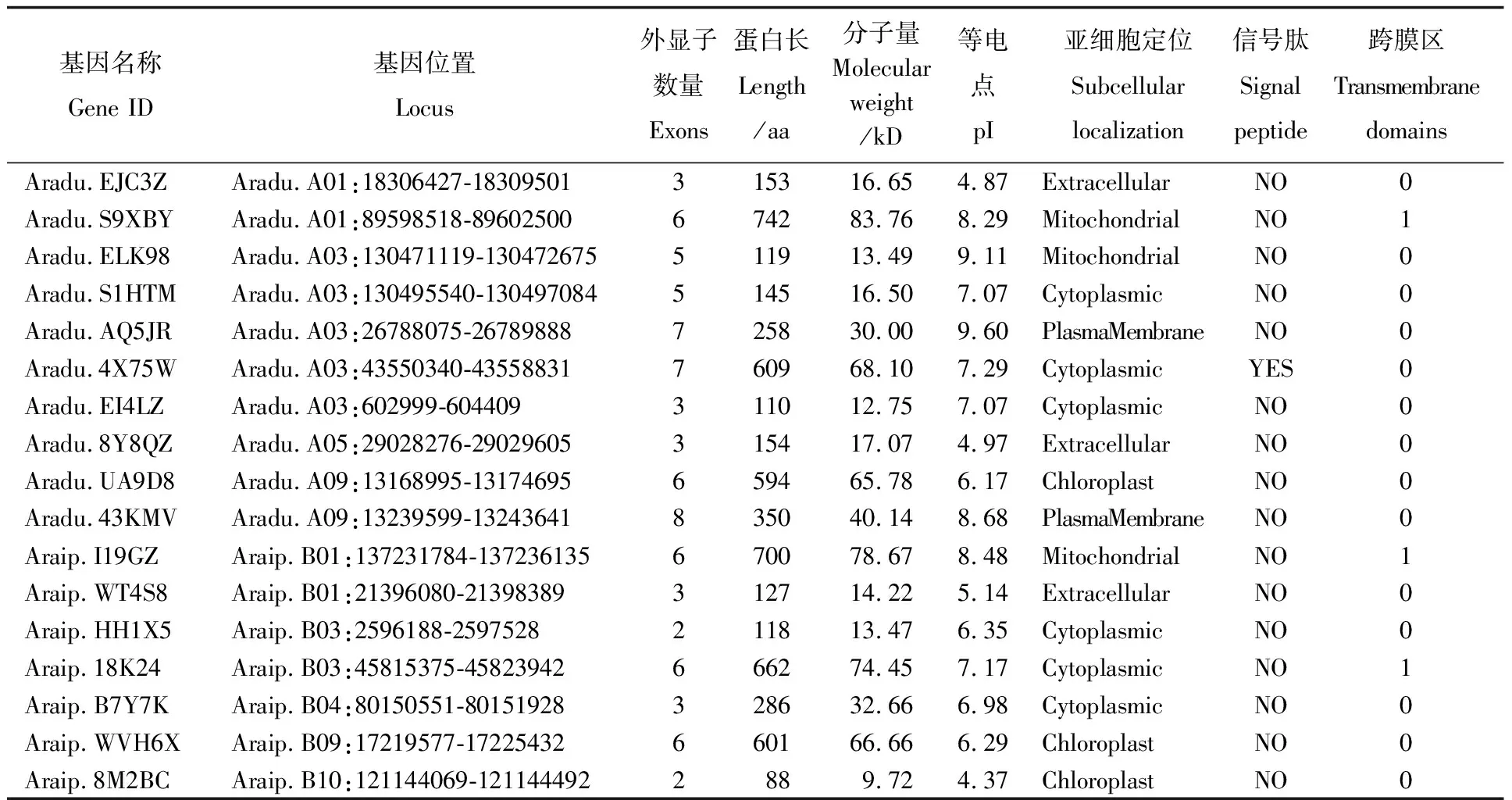
表1 AhPDATs家族基因的基本信息
2.3 AhPDATs的基因结构分析
外显子—内含子结构分析,显示AhPDAT家族基因的外显子数目变化幅度较大(见图1),从2至8不等,其中Araip.8M2BC和Araip.HH1X5的外显子个数最少,仅有2个外显子;Aradu.43KMV比较特殊,全长仅有350 aa,但外显子个数最多,共8个。同源基因之间基因结构以及内含子相位高度保守,外显子个数介于6~8之间,其中Aradu.S9XBY与Araip.I19GZ外显子个数基本一致,内含子相位差异较大;Aradu.4X75W较Araip.18K24多了一个外显子和一个内含子,其余部分无差异。此外,编码蛋白序列较短(< 500 aa)的AhPDAT基因内含子长度差异较大,保守性低,具体原因有待深入研究。
2.4 AhPDATs系统进化与保守基序分析
在17个AhPDAT中,仅有Aradu.S9XBY、Araip.I19GZ、Araip.18K24、Aradu.4X75W、Aradu.UA9D8和Araip.WVH6X可编码完整的蛋白,因此选用此6个蛋白用于系统进化分析。

图1 AhPDATs的基因结构分析 Fig.1 Gene structure analysis of AhPDAT genes
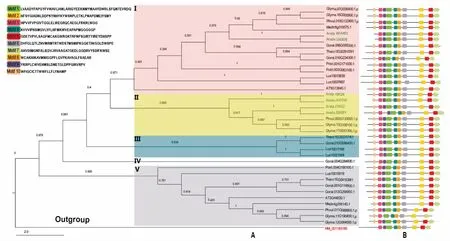
图2 花生及其他植物PDAT的系统进化与保守基序分析Fig.2 Phylogenetic relationships and conserved motifs analysis of PDAT from peanut and other plants 注: A:系统进化树(Phylogenetic tree); B:保守基序(Conserved motifs)。
以酿酒酵母PDAT为外类群,利用邻近法初步分析AhPDAT与拟南芥、大豆、蒺藜苜蓿等双子叶植物31个PDATs蛋白的进化关系(图2)。结果表明,除酿酒酵母(红色字体标注)外,双子叶植物进化成了I、II、III、IV、V五条独立分支,在进化树末端共形成9对旁系同源基因,其中6个AhPDAT(绿色字体标注)分布在I、II亚组,形成3对旁系同源基因。等位基因Aradu.UA9D8和Araip.WVH6X位于I亚组,与蒺藜苜蓿PDAT进化关系较近;Araip.18K24和Aradu.4X75W、Aradu.S9XBY和Araip.I19GZ被分在II亚组,与菜豆(Phvul.003G133000.1.p)、大豆(Glyma.13G108100.1.p和Glyma.17G051300.1.p)关系密切。III、IV、V亚组成员数分别为4、1、10,其中Gorai.004G284600.1单独组成IV亚组。另外,保守基序分析结果显示,各亚组之间基序组成与分布高度保守,除酿酒酵母NM_001183185、Aradu.UA9D8和Araip.WVH6X没有Motif10外,其余均含有10个保守基序。
2.5 AhPDATs表达模式分析
根据转录组数据对AhPDATs进行表达模式分析,结果显示Aradu.S9XBY、Araip.I19GZ、Araip.WVH6X、Aradu.UA9D8和Aradu.AQ5JR在所有组织均有表达,其中Araip.WVH6X在叶中表达量最高,而Aradu.AQ5JR在叶中表达最低;Araip.WVH6X、Aradu.S9XBY、Araip.I19GZ和Aradu.UA9D8在根与叶中的表达量大于种子,其中Araip.WVH6X在叶中表达量最高;其余12个基因在四个组织中表达量都较低。这些基因在花生不同组织以及种子不同发育阶段呈现不同的表达模式,预示它们功能也随之有所差异。整体而言,AhPDATs在根和叶中的表达量远远高于种子中,显示出它们发挥作用的地方主要是在根和叶中,而对于种子中TAG的合成所起作用相对较小。

图3 AhPDATs的表达模式分析 Fig.3 Expression pattern analysis of AhPDATs
2.6 AhPDATs可变剪接分析
基于RNA-Seq数据,统计并分析AhPDATs的AS情况。结果发现17个AhPDAT基因中有5个具有可变剪接体,约占29.41%,其中AE是最多的AS形式,共18个,其次为TSS,有17个。Aradu.S9XBY、Aradu.AQ5JR在四个组织中均发现了不同的AS形式,而且Aradu.S9XBY的可变剪接体最多,高达12个,其中最多的剪接形式为AE,其次为TTS。根据各组织中AS的分布,发现种子中AS的数目以及剪接形式最多,其次为叶,根中最少,仅Aradu.S9XBY在根中存在可变剪接体,主要形式为AE(表2)。AhPDAT基因家族在不同组织以及不同发育阶段产生了不同的可变剪接体,预示AS对其功能具有重要的调节作用。

表2 AhPDATs的可变剪接分析
3 讨 论
PDAT的功能同DGAT一样,都是催化TAG合成的最后一步反应,虽然目前对于PGAT的研究远远滞后于DGAT,但也取得了一些进展。研究发现,PDAT不仅具有TAG合成的功能[24-25],还在非生物胁迫中也具有重要作用[26-27]。Fan 等研究发现PDAT1在介导TAG合成中起关键作用,进而保护植物快速生长组织中FFA诱导的细胞死亡[8]。在拟南芥中PDAT1介导的TAG积累增加了植株耐热性,与野生型幼苗相比,pdat1突变体幼苗对严重热胁迫更敏感,同时幼苗存活率显著降低。在绿藻中研究表明,在缺氮胁迫下有助于MiPDAT将膜脂向TAG转化。以上研究说明PDAT在植物生长发育以及应对外界环境变化中具有重要作用[24,27]。通过对17个AhPDATs基因的表达模式分析表明,AhPDATs主要在根和叶中表达,可见它在种子TAG合成中所起的作用相对较小,花生种子中TAG的合成主要是由AhDGAT催化完成的,而AhPDAT在根和叶发育及其应对环境变化中具有重要作用。这与Fan等人报道是一致的,Fan等人认为PDAT对叶中TAG的合成具有重要作用,过表达PADT可以显著提高叶片中脂肪酸和TAG的含量[8]。
研究表明大部分植物PDAT具有6个外显子[28-29],而莱茵衣藻PDAT具有15个外显子[30],微胞藻PDAT仅有1个外显子[29]。本实验对AhPDAT的基因结构进行了初步分析,AhPDATs外显子数目(2~8个不等)以及内含子相位存在一定差异,但是有三对等位基因能够编码完整蛋白,它们多数有6个外显子。Aradu.43KMV比较特殊,有8个外显子。将Aradu.43KMV在NCBI中nr数据库中进行blast比对,结果表明其编码蛋白C-端完整,含有PDAT家族的保守结构域,与其他完整PDAT序列相比中间有一些片段缺失,可能是由于预测基因序列不准确所致,因此有关该基因真实的基因结构需要进一步用实验证实。其余一些外显子数目较少的AhPDAT基因大多是由于序列不完整所致。植物PDAT的保守结构域位于序列的中后部,其N-端序列保守性较差,因此预测其N-端序列是很困难的,需要进一步用RACE方法进行验证。
研究证明,植物PDAT通常位于内质网和质体中发挥作用[8],蓖麻PDAT位于内质网上[31],缺刻缘绿藻PDAT则位于质膜上[27]。本研究分析结果显示,AhPDATs不仅可以定位于叶绿体和质膜上,还可以定位于线粒体和细胞质上,甚至作为分泌蛋白定位于细胞外。这种复杂的定位形式反应了AhPDAT功能的多样性。Simpsona等人报道,蜡果杨梅的果实表面覆盖一层厚厚的腊质,其中富含TAG,而且这些TAG是在细胞外合成的,因此推测催化TAG合成的酰基转移酶应该是位于细胞外的;Simpsona又对其进行了转录谱分析,结果表明DGAT2和DGAT3、PDAT1、GPAT以及LPAAT3均具有很高的表达水平,显示出这些基因在蜡果杨梅果实蜡质层TAG合成中的重要作用[32]。部分AhPDAT具有信号肽定位于细胞外,也许和花生表皮腊质形成有关。
进化分析显示,AhPDATs与苜蓿、菜豆聚为一枝,亲缘关系较近。同时,在进化树末端发现形成3对花生特异性旁系同源基因,胡利宗等在分析大豆PDAT基因的进化关系时也发现了这种现象[28],说明这些旁系同源基因是特定物种分化后形成的基因重复事件。保守基序结果显示,只有Aradu.UA9D8和Araip.WVH6X和酿酒酵母(NM_001183185)一样,不含有Motif10,推测可能是由于花生基因组注释不完善,没有正确预测这两个蛋白的C-端序列。
利用二代测序数据对AhPDAT家族基因进行AS分析,发现约29.41%的基因存在AS现象,这与水稻中具有AS基因的比例基本一致,而拟南芥有61%的基因具有AS情况,这说明虽然可变剪接现象是普遍存在的,但不同物种中,以至于同一物种的不同基因家族之间的比例还是有很大差异的,原因可能是不同物种间基因结构以及外界环境存在差异造成的[15-16,33]。AhPDAT不同剪接异构体组织表达差异明显,表明植物AS现象会因组织差异和外界环境不同而变化。例如水稻中的OsIM基因在正常条件下可以形成OsIM1和OsIM2两个可变剪接体,在盐胁迫下,耐盐品种中OsIM1表达水平提升,而OsIM2的含量显著下降,这些研究为我们今后进一步探究AhPDAT不同剪接异构体的功能分工提供了很好的思路[16]。
