数据挖掘分析线粒体转录延伸因子在胰腺癌中的表达及意义
2018-12-28孙美涛王唯斯李素芬自加吉张晓娟
孙美涛,梅 雯,王唯斯,李素芬,自加吉,张晓娟,熊 伟
数据挖掘分析线粒体转录延伸因子在胰腺癌中的表达及意义
孙美涛1,2,梅雯1,王唯斯1,李素芬1,自加吉1,张晓娟3,*熊 伟1
(1. 大理大学基础医学院,云南,大理 671000;2. 杭州医学院基础医学与法医学院,浙江,杭州 311300;3. 大理市第一人民医院呼吸内科,云南,大理 671000)
线粒体转录延伸因子(mitochondrial transcription elongation factor, TEFM)在线粒体基因转录调控中发挥重要作用,但其在胰腺癌中的表达及意义未见系统研究。本研究利用各种肿瘤生物信息学数据库进行数据挖掘分析TEFM基因在胰腺癌及正常胰腺组织中的表达情况,并进一步探讨TEFM基因对胰腺癌患者预后的影响。利用Oncomine数据库分析TEFM基因在正常胰腺组织和胰腺癌组织中mRNA水平的表达变化。通过基因表达谱动态分析(Gene Expression Profiling Interactive Analysis, GEPIA)人体正常组织和其相应的肿瘤组织中TEFM基因的表达差异。经MethHC分析胰腺癌和正常胰腺组织中TEFM基因DNA启动子区甲基化水平的差异。利用OncoLnc对TEFM基因的表达水平与胰腺癌患者生存率作Kaplan-Meier生存分析和Log-rank检验。在String-DB数据库中探索TEFM基因在线粒体基因转录调控中相互作用的基因。结果显示:与正常胰腺组织相比,胰腺癌组织中TEFM基因在mRNA水平呈高表达,且TEFM基因DNA启动子区甲基化水平升高。TEFM基因的表达水平与胰腺癌患者的总体生存时间无显著相关性。线粒体RNA聚合酶(mitochondrial RNA polymerase, POLRMT)与TEFM蛋白有明显的相互作用。大样本数据挖掘能迅速地获取胰腺癌组织中TEFM基因表达的相关信息,为深入研究TEFM基因在胰腺癌发生发展中的作用机制奠定理论基础。
胰腺癌;线粒体转录延伸因子;数据挖掘
胰腺癌是严重危害人类健康的消化道恶性肿瘤[1]。其早期症状隐匿,临床确诊率较低,并且容易发生局部侵袭和淋巴结转移,使得临床手术、放化疗的治疗效果较差,严重威胁患者的生命[2]。对于胰腺癌患者的治疗,除了进行外科手术以外,以放化疗为主的综合治疗倍受重视和关注,因此找到更多有效的治疗方法以改善胰腺癌患者的预后是亟待解决的问题[3]。
线粒体基因组复制和转录两个过程的协调进行对肿瘤细胞的能量代谢起着重要的调控作用。恶性肿瘤的发生不仅与核内遗传物质有关,而且与核外的线粒体DNA(mitochondrial DNA, mtDNA)也有一定的关系[4]。人类的线粒体转录延伸因子(mitochondrial transcription elongation factor, TEFM)又称C17orf42,是线粒体基因转录调控中的重要蛋白因子[5]。TEFM基因定位于17q11.2,含有4个外显子,其mRNA转录本(NM_024683.3)的全长为1357 bp,编码一个由360个氨基酸残基组成的蛋白质,N端的1~35个氨基酸残基为其前导肽序列[6]。研究显示,TEFM是人类mtDNA复制和转录转换的一个关键的分子开关[7]。TEFM蛋白能够与线粒体RNA聚合酶特异性结合,并在体内和体外都能增强mtDNA的转录活性[8-9]。目前,关于TEFM在胰腺癌中的研究还未见相关报道。本研究旨在利用现有的各种肿瘤生物信息数据库,通过数据挖掘分析TEFM基因在胰腺癌中的表达及其预后意义。
1 材料与方法
1.1 Oncomine数据库提取数据
本研究设定的筛选条件为:①“Gene: c17orf42”;②“Analysis Type: Cancer vs. Normal Analysis”;③“Cancer Type: Pancreatic Cancer”;④“Date Type: mRNA”;⑤“Sample Type: Clinical Specimen”;⑥临界值设定条件(value<1E-4, fold change 2, gene rank = top 10%)。
1.2 Oncomine数据库分析TEFM基因在胰腺癌中的表达
在Oncomine数据库(http://www.oncomine.org)所提取出的数据集里,选择样本数量最大的Badea Pancreas(78个样本)的基因芯片研究分析TEFM基因在胰腺癌组织中的表达。
1.3 GEPIA数据库分析TEFM基因在各肿瘤组织中的表达
通过基因表达谱动态分析(Gene Expression Profiling Interactive Analysis, GEPIA) (http://gepia.cancer-pku.cn)TEFM基因在胰腺癌和正常胰腺组织中的表达差异,同时分析人体其它肿瘤组织和其相应的正常组织中TEFM基因的表达差异。
1.4 MethHC数据库分析TEFM基因在胰腺癌中的甲基化水平
通过MethHC数据库(http://MethHC.mbc.nctu. edu.tw)分析TEFM基因在胰腺癌和正常胰腺组织中的甲基化水平。
1.5 OncoLnc数据库分析TEFM基因的表达与胰腺癌患者预后的关系
从OncoLnc数据库(https://www.oncolnc.org)选择TCGA数据集中的PAAD项目,输入TEFM基因,高表达组与低表达组都设置为33%,最终得到57例TEFM基因高表达和57例TEFM基因低表达样本进行Kaplan-Meier分析。
1.6 String-DB数据库分析与TEFM蛋白相互作用的蛋白质
利用String-DB数据库(https://string-db.org)分析TEFM蛋白在线粒体基因转录调控中相互作用的蛋白质。
1.7 统计学分析
正常胰腺组织和对应的胰腺癌组织之间TEFM表达的差异采用检验。多组样本均数采用单因素方差分析(One-Way ANOVA),均数间的两两比较采用LSD法。TEFM表达与胰腺癌预后的关系采用Kaplan-Meier模型分析和Log-rank检验。所用统计学分析采用Graphpad 5.0进行,以<0.05为差异有统计学意义,<0.005为差异极显著。
2 结果
2.1 转录水平上TEFM基因与胰腺癌的相关性
Oncomine数据库显示,有关胰腺癌的基因芯片研究中共有8项涉及TEFM基因,相关文献分别发表于Hepatogastroenterology[10]、Oncogene[11]、Neoplasia[12]、Am J Pathol[13]、 Cancer Sci[14]、Cancer Cell[15]、Clin Cancer Res[16]。荟萃分析8项研究的结果发现,与正常胰腺组织(对照组)相比,TEFMmRNA在胰腺癌组织中呈高表达,中位表达数值为4217.0,= 0.105(图1)。

图1 8项研究显示TEFM基因在正常胰腺组织(对照组)与胰腺癌组织中的表达差异
2.2 TEFM在胰腺癌组织及正常胰腺组织的表达差异
在Oncomine数据库中,对样本数量最多的Badea Pancreas子数据集的分析结果显示,与正常胰腺组织相比,TEFM基因mRNA在胰腺导管腺癌组织中的表达量升高,上调了1.542倍,且组间差异极显著(<0.005)(图2)。

0: 正常胰腺组织(n = 39);1: 胰腺导管腺癌(n = 39);
2.3 人体各种肿瘤组织及其相应正常组织中TEFM的表达差异
通过GEPIA分析TCGA数据库中胰腺癌数据集,结果同样显示,与正常胰腺组织相比,TEFM基因在胰腺癌组织中呈高表达,且差异具有统计学意义(< 0.05)(图3)。GEPIA分析TCGA数据库中TEFM基因在人体31种肿瘤组织及其相应的正常组织中的表达差异,结果显示,TEFM基因在大多数人体肿瘤组织中均呈高表达(图4)。

图3 TCGA数据集中胰腺癌组织和正常胰腺组织中TEFM mRNA表达差异(*P<0.05)
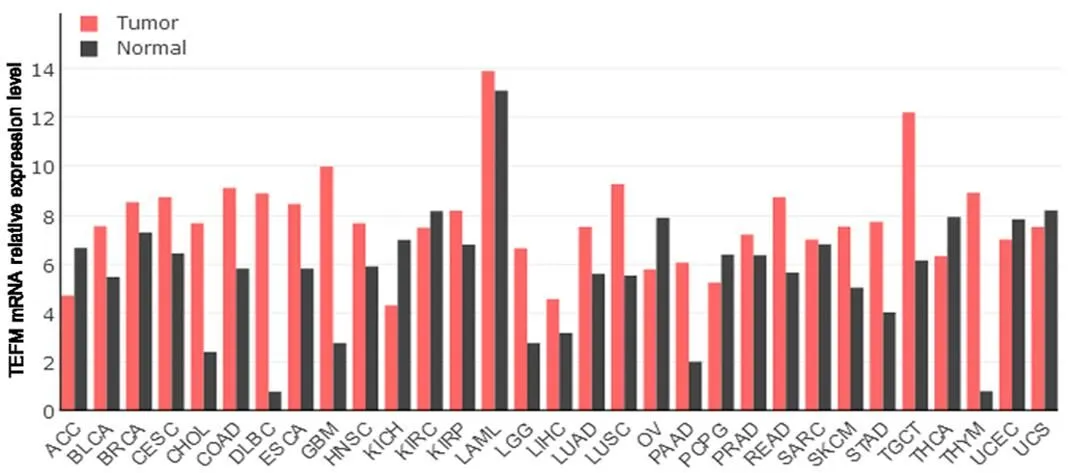
图4 人体各种肿瘤组织及其相应的正常组织中TEFM基因的表达差异
2.4 胰腺癌中TEFM基因DNA启动子区甲基化水平分析
在MethHC中,TEFM基因的转录模板NM_024683,与NCBI数据库中报道的一致。研究发现,胰腺癌组织与正常胰腺组织中TEFM基因DNA启动子区的甲基化水平具有显著差异性,与正常胰腺组织相比,胰腺癌组织中NM_024683表达水平高,TEFM基因启动子区甲基化水平升高(< 0.05)(图5)。结果提示,DNA甲基化水平并非影响TEFM基因表达水平的唯一因素,可能还有其它的表观遗传学因素影响TEFM基因在胰腺癌中的表达水平。
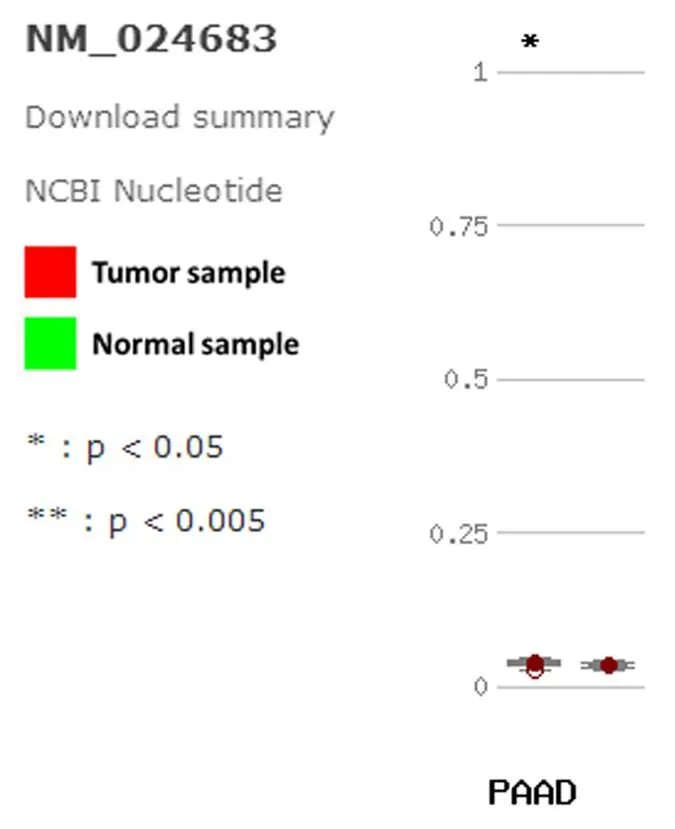
图5 MethHC数据库分析胰腺癌组织和正常胰腺组织中TEFM基因甲基化水平
2.5 胰腺癌数据集的在线生存分析
OncoLnc数据库检索结果显示:TEFM基因的表达水平对胰腺癌患者的总生存时间无显著影响。与TEFM基因高表达组相比,TEFM基因低表达的胰腺癌患者总体生存率无明显变化(Logrank= 0.44)(图6)。

图6 OncoLnc数据库胰腺癌数据集中TEFM表达量与患者预后的Kaplan-Meier生存分析
2.6 与TEFM蛋白相互作用的蛋白质
通过String-DB分析发现,TEFM在线粒体基因转录过程中发挥重要作用,PPI富集= 0.5,节点数为1个(图7)。与TEFM蛋白有相互作用的蛋白质为线粒体RNA聚合酶(mitochondrial RNA polymerase, POLRMT)。这与文献报道的结果完全一致[7-9]。
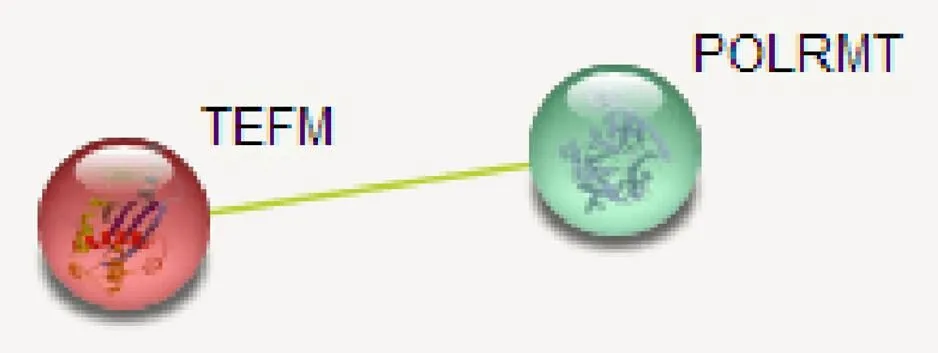
图7 线粒体转录过程中与TEFM相互作用的蛋白质
3 讨论
Oncomine数据库是目前世界上最大的癌基因芯片数据库和整合数据挖掘平台,旨在挖掘癌症基因信息,可用于比较主要癌症类型和各自正常组织的差异表达分析,也可用于探索各种癌症亚型以及基于临床和病理学的分析[17]。基因表达谱动态分析(GEPIA)是由北京大学研制开发可用于分析基因在癌症和正常组织的差异表达的在线应用,可对TCGA数据库进行可视化分析[18]。MethHC 数据库包含18种人类肿瘤组织及其对应正常组织中的DNA甲基化和mRNA/miRNA 表达谱,可用于证明正常组织和肿瘤组织中的甲基化模式,并分析甲基化和表达之间的相关性,还可以用于基因组分析揭示差异甲基化基因和基因簇[19]。OncoLnc 数据库可对癌症数据集进行在线Kaplan-Meier生存分析[20]。String-DB数据库是分析基因或蛋白质相互作用的检索工具,包含已证实的和可以预测的蛋白质-蛋白质相互作用的生物数据库和网络资源[21]。
胰腺癌起病隐匿,其恶性程度高,进展迅速,患者预后极差[22-24]。随着基因治疗技术的不断发展与进步,分子靶向治疗成为临床治疗胰腺癌研究的热点[25]。由于临床上缺乏胰腺癌特异性的分子靶标,不利于胰腺癌的早期诊断、治疗方法的选择及治疗效果[26-28]。因此,深入研究胰腺癌发生发展的分子机制同时探索有助于胰腺癌早期诊断的分子标志物是至关重要的[22]。TEFM是线粒体基因转录调控中的重要蛋白质因子,但目前关于TEFM在胰腺癌中的表达及其预后意义尚未见报道。
本研究中我们利用Oncomine数据库分析TEFM基因在胰腺癌组织及正常胰腺组织中的表达差异。通过GEPIA数据库分析发现TEFM基因在各肿瘤组织中的表达情况。然后,通过MethHC数据库分析了在胰腺癌和正常胰腺组织中TEFM基因DNA启动子区的甲基化水平的变化,MethHC分析结果显示:与正常胰腺组织相比,胰腺癌组织中TEFM基因在DNA启动子区的甲基化水平升高。结果提示除了DNA甲基化水平外,可能还有其它的表观遗传学因素(如组蛋白修饰,乙酰基化改变,miRNAs等)影响TEFM基因在胰腺癌组织中的表达水平,这有待于进一步实验研究和探讨。
最后,利用OncoLnc数据库分析TEFM基因的表达水平对胰腺癌患者的总生存时间的影响,OncoLnc数据结果显示:与TEFM基因高表达组相比,TEFM基因低表达的胰腺癌患者总体生存时间无明显变化(Logrank= 0.44)。另外,我们对TEFM基因在线粒体基因转录调控中的作用进行了初步探索,通过String-DB数据库分析发现:线粒体RNA聚合酶(POLRMT)与TEFM有明显的相互作用。
通过对各种肿瘤生物信息学数据库的数据挖掘和分析发现,在mRNA转录水平上,TEFM基因在胰腺癌中较正常胰腺组织高表达;相比正常胰腺组织,胰腺癌组织中TEFM基因在DNA启动子区的甲基化水平显著升高;与TEFM基因高表达组相比,TEFM基因低表达的胰腺癌患者总体生存时间无明显差异。这些结果为后续的实验研究提供了理论基础,也为进一步探究TEFM在肿瘤发生发展中的分子机制和抗肿瘤靶向治疗提供了理论依据。
[1] Baron T H, Kozarek R A. Preoperative biliary stents in pancreatic cancer-proceed with caution[J]. N Engl J Med, 2010, 362(2): 170-172.
[2] Keleg S, Büchler P, Ludwig R,et al. Invasion and metastasis in pancreatic cancer[J]. Mol Cancer, 2003, 2:14.
[3] Hidalgo M. Pancreatic cancer[J]. N Engl J Med, 2010, 362(17): 1605-1617.
[4] 刘启梁. 线粒体DNA异常与肿瘤[J]. 生命的化学, 2016, 36(6): 862-867.
[5] Hillen H S, Parshin A V, Agaronyan K, et al.Mechanism of transcription anti-termination in human mitochondria[J]. Cell,2017,171(5):1082-1093.
[6] 杨勇琴,张晓娟,孙美涛,等. 人线粒体转录延伸因子蛋白结构与功能的生物信息学分析[J]. 生物技术,2015,25(5): 475-480.
[7] Agaronyan K,Morozov Y I,Anikin M,et al. Mitochondrial biology.Replication-transcription switch in human mitochondria[J]. Science, 2015, 347(6221): 548-551.
[8] Posse V, Shahzad S, Falkenberg M,et al. TEFM is a potent stimulator of mitochondrial transcription elongation factor in vitro[J]Nucleic Acid Res, 2015, 43(5): 2615-2624.
[9] Minczuk M, He J, Duch A M, et al. TEFM (c17ofr42) is necessary for transcription of human mtDNA[J]. Nucleic Acid Res, 2011, 39(10): 4284-4299.
[10] Badea L,Herlea V,Dima S O,et al. Combinedgeneexpressionanalysisofwhole-tissueandmicrodissectedpancreaticductaladenocarcinomaidentifiesgenesspecificallyoverexpressedintumorepithelia[J]. Hepatogastroenterology, 2008, 55(88): 2016-2027.
[11] Buchholz M,Braun M,Heidenblut A,et al.Transcriptome analysis of microdissected pancreatic intraepithelial neoplastic lesions[J]. Oncogene, 2005, 24(44): 6626-6636.
[12] Grutzmann R, Pilarskty C, Ammerpohl O, et al. Gene expression profiling of microdissected pancreatic ductal carcinomas using high-density DNA microarrays[J]. Neoplasia, 2004, 6(5): 611-622.
[13] Iacobuzio-Donabue C A,Maitra A,Olsen M,et al. Exploration of global gene expression patterns in pancreatic adenocarcinoma using cDNA microarrays[J]. Am J Pathol, 2003, 162(4):1151-1162.
[14] Ishikawa M, Yoshida K, Yamashita Y, et al. Experimental trial for diagnosis of pancreatic ductal carcinoma based on gene expression profiles of pancreatic ductal cells[J]. Cancer Sci, 2005, 96(7):387-393.
[15] Pei H, Li L, Fridley BL, et al. FKBP51 affects cancer cell response to chemotherapy by negatively regulating Akt[J]. Cancer Cell, 2009, 16(3): 259-266.
[16] Segara D, Biankin A V, Kench J D, et al. Expression of HOXB2, a retinoic acid signalingtarget in pancreatic cancer and pancreatic intraepithelial neoplasia[J]. Clin Cancer Res, 2005, 11(9): 3587-3596.
[17] Rhodes D R, Kalyana-Sundaram S, Mahavisno V, et al. Oncomine 3.0: genes, pathways, and networks in a collection of 18,000 cancer gene expression profiles[J]. Neoplasia (New York), 2007, 9(2): 166-180.
[18] Tang Z, Li C, Kang B, et al. GEPIA: a web server for cancer and normal gene expression profiling and interactive analyses[J]. Nucleic Acids Research, 2017, 45(Web Server issue): W98-W102.
[19] Huang W Y,Hsu S D,Huang H Y,et al.MethHC: A database of DNA methylation and gene expression in human cancer[J]. Nucleic Acids Research, 2015, 43(Database issue):856-861.
[20] 齐闯,赵虎,陈展,等. 基于数据挖掘分析酪氨酸蛋白激酶受体TYRO3在膀胱癌中的表达及临床意义[J]. 现代肿瘤医学, 2018, 26(1) : 57-61.
[21] Szklarczyk D,Morris J H,Cook H,et al.The STRING database in 2017: Quality- controlled protein-protein association networks, made broadly accessible[J]. Nucleic Acids Research, 2017, 45 (Database issue): D362-D368.
[22] Garrido-Laguna I, Hidalgo M. Pancreatic cancer: from state-of-the-art treatments to promising novel therapies[J]. Nat Rev Clin Oncol, 2015, 12(6): 319-334.
[23] 倪泉兴,虞先濬,刘亮. 中国胰腺癌临床诊断标准的探讨[J]. 中国癌症杂志,2012, 22(2): 81-87.
[24] Saif M W. Advancements in the management of pancreatic cancer: 2013[J]. JOP, 2013, 14(2): 112-118.
[25] Chiaravalli M, Reni M, O’Reilly E M. Pancreatic ductal adenocarcinoma: State-of-the-art 2017 and new therapeutic strategies[J]. Cancer Treat Rev, 2017, 60: 32-43.
[26] Fleming A K, Storz P. Protein kinase c isoforms in the normal pancreas and in pancreatic disease[J]. Cell Signal, 2017, 40: 1-9.
[27] Neault M, Mallette F A, Richard S. miR-137 Modulates a tumor suppressor network-inducing senescence in pancreatic cancer cells[J].Cell Rep,2016,14(8):1966-1978.
[28] Hu B, Shi C, Jiang H X, et al. Identification of novel therapeutic genes and pathway in pancreatic cancer by integrative analysis[J]. Medicine (Baltimore), 2017, 96(42): e8261.
Expression and significance of TEFM in Pancreatic cancer: Evidence from data mining
SUN Mei-tao1,2, MEI Wen1, WANG Wei-si1, LI Su-fen1, ZI Jia-ji1, ZHANG Xiao-juan3,*XIONG Wei1
(1. College of Basic Medical Sciences, Dali University, Dali, Yunnan 671000, China; 2. Department of Basic Medical Sciences and Forensic Medicine, Hangzhou Medical College, Hangzhou, Zhejiang 311300, China; 3. Department of Respiratory Medicine, First People’s Hospital of Dali City, Dali, Yunnan 671000, China)
Mitochondrial transcription elongation factor is an important protein factor in the regulation of mitochondrial gene transcription, but there is no systematic study of TEFM expression in Pancreatic cancer. Based on data mining, the expression of TEFM in Pancreatic cancer and normal tissues and its impact on the prognosis of Pancreatic cancer were analyzed. The mRNA level of TEFM gene in Pancreatic cancer was determined by Oncomine database. The differences in expression of TEFM gene in normal tissues and their corresponding tumor tissues were analyzed by GEPIA. The differences of TEFM gene DNA promoter methylation level in Pancreatic cancer and normal pancreas tissue were analyzed by MethHC analysis. The correlation between the expression level of TEFM gene and the survival time of patients with Pancreatic cancer was analyzed by OncoLnc. The location of the TEFM gene in the cell signal transduction pathway and the upstream and downstream genes associated with it was explored in the String-DB database. Compared with normal pancreatic tissue, the expression level of TEFM gene in pancreatic cancer tissue is higher in mRNA level, the methylation level of TEFM gene DNA promoter region is higher. The expression level of TEFM gene is not significantly correlated with the overall survival time of pancreatic cancer patients. POLRMT protein have obvious interaction with TEFM. Large sample data mining can quickly obtain information about the expression of TEFM in pancreatic cancer, which lays a foundation for further study on the mechanism of TEFM gene in the occurrence and development of pancreatic cancer.
pancreatic cancer; TEFM; data mining
1674-8085(2018)05-0038-06
R735.9
A
10.3969/j.issn.1674-8085.2018.05.008
2018-06-06;
2018-08-02
国家自然科学基金项目(81560458,31601155,31760331);大理大学大学生创新创业计划项目(S-CXCY-2017-8);云南省教育厅科学研究基金项目(2016ZDX01,2016ZDX05);云南省中青年学术和技术带头人后备人才项目(2017HB077)
孙美涛(1990-),女,山东日照人,助理实验师,硕士,主要从事分子病理学研究(E-mail: 1103293583@qq.com);
梅 雯(1988-),女,云南曲靖人,硕士生,主要从事细胞分子生物学研究(E-mail: 1457789546@qq.com);
王唯斯(1991-),男,内蒙古呼伦贝尔人,硕士生,主要从事细胞分子生物学研究(E-mail: 313731142@qq.com);
李素芬(1993-),女,山东济宁人,硕士生,主要从事细胞分子生物学研究(E-mail: 1446198172@qq.com);
自加吉(1980-),男,云南大理人,讲师,硕士,主要从事分子病理学研究(E-mail: dldxjcyxyzjj@163.com);
张晓娟(1984-),女,云南大理人,护师,主要从事内科护理学研究(E-mail: 791372366@163.com);
*熊 伟(1982-),男,湖南株洲人,教授,博士,硕士生导师,主要从事肿瘤细胞分子生物学和生物信息学研究(E-mail: xwailp@163.com).
