新一代DNA测序技术的应用与研究进展
2018-12-21徐疏梅
徐疏梅
(上海海洋大学 水产与生命学院,上海 201306)
1953年,Watson 和Crick 发现DNA 双螺旋结构[1],之后人们认识到生物的遗传信息是由DNA 序列决定的, 即A、T、C、G 这4种碱基的排列方式决定了生物的形态、生长发育以及疾病等特征.那么,如何破解“生命密码”,探究物种的DNA 序列及其完整性,则成为生命科学研究领域的热门话题.随着科学技术的进步与发展,DNA 测序技术(又称基因测试技术)在业界人士的关注、研究与促进下,有了迅猛的发展,不仅在传统生物学、医学研究等领域开辟了新的视角,并推动生物信息学、系统生物学、分子遗传学、基因组学、精准医学等学科的进一步发展,而这些学科的发展又促进生命科学研究的进步.1977年,被奉为标志性测序技术的Sanger链终止测序法诞生,自此第一代DNA测序方法正式浮出水面,并因其高准确性一直沿用至今.21世纪以来,第二代高通量测序技术取得了快速发展及广泛的应用,而第三代单分子测序技术逐渐走向成熟和多元化,受到广大科研人员的关注与欢迎.这些测序技术的进步与发展,加快了对人类信息探索的发展,如2008年1月,“国际千人基因组计划”启动,它由中国深圳华大基因研究院、美国国立人类基因组研究所、英国桑格研究所承担,绘制了有史以来最有医学应用价值、最为详尽的人类基因组遗传多态性图谱;2012年11月,该计划研究人员首次对比分析了千人规模以上的基因组,发布了1 000余人的基因数据,在生命科学研究中取得了划时代的进展[2].DNA测序技术的发展路径如图1[3]所示.本文阐释了DNA测序技术的应用与研究进展,旨在为生命科学研究人员提供研究依据与方向.
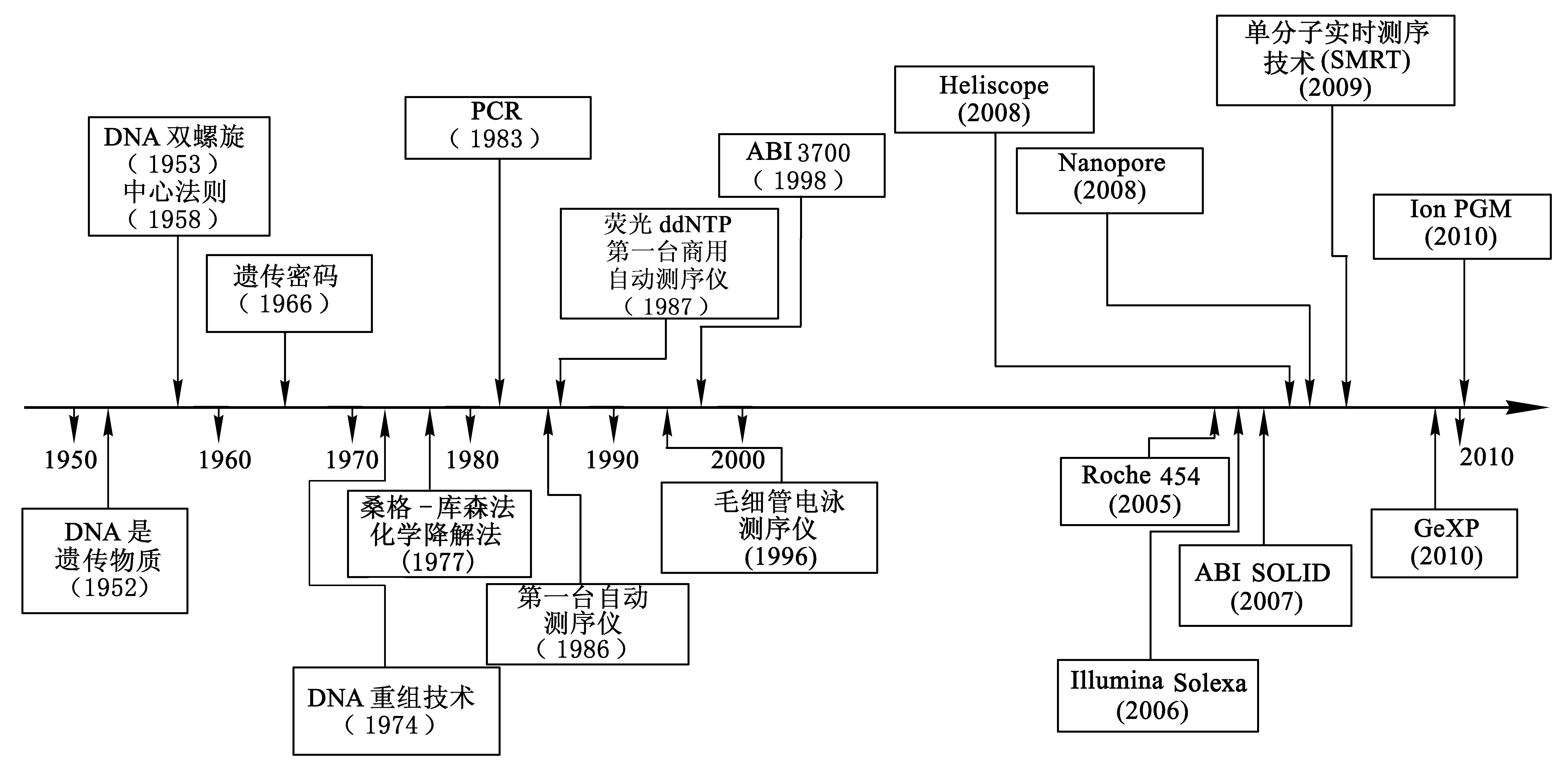
图1 DNA测序技术发展历程
1 第一代DNA测序技术
DNA双螺旋结构被发现之后,在1954年,Whitfeld等[4]利用磷酸单酯酶的脱磷酸作用、高碘酸盐的氧化作用,将含脱氧核糖的寡核昔酸从链末端逐一分离,并测定其种类,即为测量多聚核糖核苷酸的化学降解法.然而,这种方法的操作极其复杂,无法被广泛应用,直到1977年,英国生物化学家Sanger等提出的双脱氧核苷酸末端终止测序法,以及Maxam等提出的类似的化学降解法,标志着DNA测序技术的正式诞生.
1.1 桑格-库森法(Sanger-Coulson method)
1977年,Sanger等[5]提出了双脱氧核苷酸末端终止测序法,发明了第一代测序技术.我国科学技术名词审定委员会审定公布的《遗传学名词》( 2007 年)、《生物化学与分子生物学名词》(2008 年)、《细胞生物学名词》(2009 年)中,均将其命名为桑格-库森法(Sanger-Coulson method),我国很多网站和学术论文将其简称为Sanger测序法.桑格-库森法的定义为以2,3-双脱氧核苷三磷酸为底物,快速测定DNA 中核苷酸序列的方法[6].该方法的核心原理是因为双脱氧核苷酸(ddNTP)的2′和3′上都不含羟基,使得DNA的合成过程中无法形成磷酸二酯键,因而将其用以中断DNA合成反应.具体而言就是在4个DNA合成反应体系中,在DNA 模板链上分别加入一定比例的互补参入却不能延伸的4种双脱氧核苷三磷酸ddNTP(ddATP,ddCTP,ddGTP,ddTTP),而它们都带有放射性同位素标记,然后与正常的4种脱氧核苷三磷酸(dNTP)竞争,再通过凝胶电泳和放射自显影的方法,根据4条泳道上的条带顺序来确定待测分子的DNA序列.
1.2 化学降解法(Maxam-Gilbert method)
1977年,Maxam等[7]发明了DNA片段序列的测定方法,即化学降解法.化学降解法与桑格-库森法类似,其核心原理是首先将1个DNA片段的5′端磷酸基作32P 放射性标记,然后利用特殊试剂降解,即采用不同的化学方法,修饰、裂解特定碱基,从而产生一系列长度不一且5′端被标记的DNA片段,再将这些以特定碱基结尾的片段群,通过聚丙烯酰胺凝胶电泳进行片段分离,并采用放射性自显影技术,判断各片段末端碱基,读出目的DNA序列[8-9].
1.3 第一代测序技术的应用与发展
迄今为止,人类获得的绝大部分DNA序列都是基于桑格-库森法获得的[10],它与化学降解法的应用,带来了DNA测序技术的快速发展:1986年,美国应用生物系统公司(Applied Biosystems Inc,ABI)推出的第一代商用ABI 370A 测序仪可在双脱氧核苷酸上直接标记不同颜色荧光基团;1998年,ABI采用其开发的毛细管凝胶电泳技术,推出的ABI Prism 3700毛细管测序仪可同时进行96 个并行测序反应,真正实现了测序规模化[11-12];ABI Prism 3730是ABI Prism 3700基础上发展而来的,至今仍是第一代测序的主力机型,也常用于验证其他新测序设备的准确性.在这一时期,还出现了诸如焦磷酸测序法、链接酶法等其他测序技术.
毛细管电泳测序方法的出现与应用,促进了人类基因组计划的完成.1985年,美国科学家提出人类基因组计划,于1990年正式启动,测定了人类染色体的30亿个碱基对组成的核苷酸序列,绘制了人类基因组图谱,并且识别其载有的基因及其序列信息,达到破译人类基因遗传密码的最终目的[13].截止到2005年,人体全序列的基因测定工作已经完成[14-15].整个计划历时15 a,其中中国、美国、英国、法国、德国和日本6个国家的科学家共同参与了这一预算达30亿美元的人类基因组计划,而人类基因序列图则己成为全人类共同的财富[16].该项目的完成标志着分子医学时代的到来,也开启了人类基因组测序的新时代.
2 第二代DNA测序技术
尽管第一代DNA测序技术以其可达1 000 bp的测序读长、99.999%的高准确性帮助人们完成了大量的测序工作,但其测试速度慢、成本高、通量低等方面的不足,也致使其不能得到大众化的应用.随着科学技术的进步以及科研人员对测序技术的努力开发,2005年Roche公司发布的454测序系统标志着测序技术跨入高通量并行测序的时代.第二代DNA测序技术又称次世代测序技术(next generation sequencing,NGS)、大量并行测序技术(massive parallel sequencing,MPS)、高通量测序技术(high-throughput sequencing,HTS),以低成本、99%以上的准确度,1次可对几百、几千个样本的几十万至几百万条DNA分子同时进行快速测序分析.这一时期的代表技术有Roche公司的454、Illumina公司的Solexa、ABI公司的SOLID,由于该时期的测序技术十分前沿,因而市场主要被这3家公司所垄断.
2.1 Roche 454测序技术
2005年,美国的罗氏(Roche)公司利用焦磷酸测序法研发出不同于第一代的测序技术,即454测序技术.这项技术的核心原理是依靠生物发光技术对DNA 序列进行分析,在DNA 聚合酶、ATP 硫酸化酶、荧光素酶和双磷酸酶的协同作用下,将引物上的dNTP 聚合与1次荧光信号释放偶联起来,通过检测荧光信号的强度,达到实时测定DNA 序列的目的.其测序步骤是利用喷雾法把待测的DNA样本主要打断成300~800 bp长的序列片段后构建测序文库、微乳液PCR的扩增及测序分析[17].454测序系统是第一个被商业化的平台,但随着测序技术的进步,罗氏公司已宣布454焦磷酸测序仪退出市场.
2.2 Illumina Solexa 测序技术
2006年, Solexa 公司发明了Solexa 测序技术,后被美国的Illumina公司收购.Solexa测序技术基于高密度的单分子阵列进行序列测定,其步骤是:1)首先用超声或氮气等方法将DNA 随机打断成100~200 bp 片段,再在两端加上通用接头,然后进行PCR扩增,构建ssDNA文库;2)将接好接头的待测DNA片段放入含有基片的流通池内,片段的另一端因随机与附近的另一接头序列互补而被固定,使得打断的DNA 片段两端固定在基片上,形成桥状结构,即桥式扩增;3)在8个微流道的测序芯片上进行测序,可以重复几十个循环[18-19].
2.3 ABI SOLID测序技术
2007年,美国应用生物系统公司基于连接酶法开发了SOLID测序技术,并将其用于商业测序.SOLID测序技术以连接酶的连接反应取代聚合酶的聚合反应,并采用双碱基编码的方式获取DNA序列信息.其测序步骤是:1)将基因组DNA 打断成100~200 bp 片段,在片段两端加上测序接头,构建单链DNA文库;2)微乳液PCR的扩增,其过程与454测序方法类似,只是测试模板微球更小,仅1 μm;3)加入连接酶进行连接测序[20-21].
2.4 三种测序技术的比较
以上三种测序技术的特征、成本及特点见表1.

表1 第二代DNA典型测序技术的特征、成本及特点
2.5 第二代测序技术的应用与发展
继三种典型测序技术之后,有了新的发展,先后出现了Ion Torrent、Heliscope、Nanopore、SMRT、Ion PGM、GeXp等第二代、第三代测序技术.这些技术的开发与应用极大地加速了基因组重测序、宏基因组测序、DNA甲基化测序、转录组测序、目标基因组区域再测序、基因的表观遗传修饰检测、微生物检测等领域的研究工作,解决了第一代测序技术无法大规模进行试验的现实问题.
3 第三代DNA测序技术
近年来,为了更加精确与高效地挖掘DNA序列信息,科研人员研究、开发出第三代测序技术,即单分子测序( single molecule sequencing)技术.这项技术与前两代技术不同的是测序时不需要进行PCR扩增,而是基于单分子水平的边合成边测序思想,实现了对每一条DNA分子的单独测序.目前其测序技术原理主要分为两大类:1)单分子荧光测序[22],以Helisope Bioscience公司的SMS 技术、Pacfic Bioscience 公司的SMRT 技术为代表,用荧光标记脱氧核苷酸进行探测,用显微镜观测、记录荧光强度的实时变化;2)纳米孔测序[23],以英国牛津纳米孔公司为代表,利用直径非常细小的纳米孔,根据不同碱基产生的电信号的差异进行测序.
第三代DNA测序技术相较于前两代测序技术,具有超长读长、运行快、无需模板扩增、直接检测表观修饰位点等特点,主要用于基因组测序、甲基化研究、突变鉴定(SNP检测)等方面. 第三代测序技术的优点是巨大而不可比拟的,但该代技术尚处于发展阶段,还未成熟和多元化,测序精度还低于前两代技术,用于商业化的测序仪相对较少.
4 结语
第一代DNA测序技术的产生宣示了划时代的生命科学研究的到来,其高准确性的特点使其至今仍然被应用;第二代DNA测序技术具有速度快、成本低、通量高等优点,是目前最为成熟、应用最为广泛的技术,其发展给生命科学领域的研究带来了空前的进步,但也存在所测DNA长度较短的问题,需要第一代测序技术的佐证;第三代技术大幅降低了测序费用,是未来的发展方向.从三代DNA测序技术的发展与应用可知,DNA测序技术的产生与快速发展为生命科学研究带来了革命性的改变,可以一次性对某个物种的DNA到RNA的遗传信息进行全貌解析,使得人类知晓并掌握自身的全基因组序列,以及水稻、小麦、家蚕等其他物种的基因序列,甚至包括细菌的基因序列.同时,SNP的大量存在,也使人们认识到人类基因组图谱并不是独一无二的,每个人的独特图谱是实际存在的.因而,如何快速、经济、高通量、高精确性地探明一段DNA序列所代表的生物学意义,使人类对自然和自身的认知进入到新的科学层面,则成为科研人员的孜孜不倦的追求目标.
