细粒棘球绦虫原头蚴microRNA表达谱分析
2018-11-30王正荣薄新文张艳艳卢平萍徐梦飞孟季蒙
王正荣,薄新文*,张艳艳,马 勋,卢平萍,,徐梦飞,,孟季蒙
(1. 新疆农垦科学院畜牧兽医研究所,省部共建绵羊遗传改良与健康养殖国家重点实验室, 石河子 832000; 2.石河子大学动物科技学院,石河子 832000)
MicroRNA(miRNA)是一类非编码的内源性小RNA分子,其长度在22个核苷酸左右。它们在动植物体内参与转录后基因的表达调控,并且通过部分或者全部序列互补的形式结合到靶标基因的转录子上,直接导致其降解或翻译受阻[1-6]。截至目前,在miRBase (http://www. Mirbase. org/ release 22.0)数据库中注册提交的miRNA序列已经达到271个物种的48 885条成熟序列。已提交的棘球属绦虫的miRNA 成熟序列147条,其中细粒棘球绦虫的miRNA序列为133条。
包虫病是由棘球属幼虫——棘球蚴寄生于中间宿主牛、羊等动物以及人类,引起严重的脏器损伤,是一种危害性极其严重的人畜共患寄生虫病[7-8]。该病是导致我国西部农牧区牧民因病致贫、因病返贫的主要原因之一。目前,该病呈世界性分布,我国20省份有该病发生,主要流行于西部一些牧区,包括青海、西藏、新疆等地。据估计国内包虫病的患者人数35万~130万[7]。另据文献报道,我国包虫病的受威胁人口近5 000万,患者数量庞大,与现有的全球包虫病流行数据相比较,显示我国包虫病受威胁人口数和患者数仍居全球首位[7, 9]。该病严重危害着疫区群众的生命财产安全,同时也严重影响着我国的国际声誉。
细粒棘球绦虫是包虫病的主要病原之一,其原头蚴具有双向发育的特性,即在中间宿主体内原头蚴可经无性繁殖发育为包囊,在终末宿主体内,原头蚴可经有性繁殖发育为成虫。这一发育现象必然受到众多因素的调控,已有研究报道在原头蚴发育为成虫的过程中,胆酸盐信号通路发挥着重要的功能[10]。但有没有其他的调控因子(如非编码RNA等)不得而知,为了进一步揭示miRNA在原头蚴发育中调控机制,本研究对细粒棘球绦虫原头蚴表达的miRNA进行了测序研究,结果发现了大量的新miRNA分子,这些发现一方面丰富了细粒棘球绦虫miRNA数据,另一方面为揭示miRNA在细粒棘球绦虫原头蚴生长发育中的调控机制提供了新的视角。
1 材料与方法
1.1 实验材料
在新疆石河子市牛、羊定点屠宰场采集细粒棘球蚴感染的绵羊肝,低温保存带回实验室,先用高压灭菌的生理盐水将脏器表面清洗干净,然后用酒精棉球对脏器表面做消毒处理,无菌注射器吸取囊液,于无菌条件下剪开包囊的顶部取出内囊,置于一无菌平皿中,用加双抗的无菌PBS液反复吹打冲洗3~5次,收集虫体沉淀,备用。
1.2 总RNA的提取、miRNA库的建立以及测序
按照Trizol(Invitrogen,Gaithersburg,MD,USA)法提取约100 mg的细粒棘球绦虫原头节总RNA,柱纯化后用Nanodrop ND-1000(Nanodrop Nechnologies,Wilmington,DE)分析浓度,用Agilent BioAnalyzer 2100(Agilent Technologies,Palo Alto,CA)进行质量检测。总RNA于-80 ℃保存备用。
首先,采用15%变性聚丙烯酰胺凝胶电泳技术(PAGE)从总RNA中分离小片段15~30 nt RNA,纯化后对小片段RNA连接3′和5′接头。然后运用SuperScriptⅡ反转录酶将连接有5′和3′接头的RNA进行反转录,合成cDNA,并使用下列程序进行PCR扩增:98 ℃预变性30 s;98 ℃变性10 s、60 ℃ 退火30 s、72 ℃延伸15 s,14个循环;72 ℃延伸10 min;4 ℃保存。扩增后的cDNA经纯化检测合格后,送北京诺禾致源科技股份有限公司进行Illumina HiSeqTM2500测序。
1.3 保守与新miRNA的鉴定
对序列数据进行去除低质量、污染、接头的处理,然后统计序列总数、种数和长度,最后获得全部有效序列用作后续分析。首先将有效序列比对到GenBank、Repeat sequence、Rfam数据库,去除rRNA、tRNA、snoRNA、snRNA、repeat等非编码小RNAs;然后将剩余序列比对到miRBase 21.0中已知miRNA的成熟序列或前体序列(默认G-U配对,允许1~2个碱基错配),从而得到在细粒棘球绦虫已知的miRNA;最后将剩余序列比对到细粒棘球绦虫EST数据库,通过截取与小RNA匹配的50~70 bp 的EST序列,利用Mireap软件预测细粒棘球绦虫特有的miRNA。
1.4 miRNA靶标基因的预测
利用Miranda算法[11-12]预测miRNA靶基因,该算法采用动态规划算法搜索miRNA与 3′ UTR互补同时稳定形成双链的区域,预测miRNA靶基因过程中所使用到的阈值参数为:S≥150,Δ G≤-126 kJ·mol-1,同时必须满足5′端种子序列的配对。
1.5 wnt/β-catenin信号通路相关miRNA及其潜在靶标基因在细粒棘球绦虫原头蚴体外培养不同时间的mRNA转录水平分析
1.5.1 细粒棘球绦虫原头蚴的体外培养及总RNA的提取cDNA合成 将无菌采集的细粒棘球绦虫原头蚴按2 000枚·mL-1的培养密度接种于含10%胎牛血清的RPMI-1640为主的培养液中,于37 ℃,5% CO2的恒温培养箱中培养,每3 d更换一次培养液,分别在培养0、4、10 d采集样品,保存于液氮中,备用。总RNA提取采用Invitrogen Trizol Reagent试剂,提取方法参照试剂说明书步骤进行。mRNA和miRNA反转录cDNA合成分别采用康为世纪公司10 μL反转录体系的CW2582和CW2141S试剂盒。
1.5.2 miRNA荧光定量 采用康为世纪公司CW2142S试剂盒推荐的20 μL荧光定量反应体系。加入2× miRNA qPCR Mixture 10 μL,10 μmol·L-1Forward primer 0.4 μL,10 μmol·L-1Reverse primer 0.4 μL,模板cDNA 1 μL,ddH2O 8.2 μL。反应条件依据引物退火温度而略有不同,PCR程序:95 ℃预变性10 min,95 ℃变性10 s,58 ℃退火30 s,72 ℃延伸 30 s,共进行40个循环。
1.5.3 mRNA的荧光定量 提取体外培养0、4和10 d的原头蚴组织样品总RNA,反转录成cDNA,以上述cDNA为模板,以efa为内参基因,对miRNA靶标基因进行qRT-PCR扩增,反应体系:SYBR PremixExTaqMix(2×) 10 μL,上游引物(10 mmol·L-1)0.4 μL,下游引物(10 mmol·L-1)0.4 μL,cDNA 1 μL,加ddH2O 至20 μL。反应条件:95 ℃预变性30 s;95 ℃ 5 s,60 ℃ 20 s,共40个循环。每个基因做三个重复,采用相对比较△Ct(Qr=2-△△Ct)[13]法计算目的基因的相对表达量。mRNA的定量PCR采用的内参基因是efa,miRNA 定量PCR采用的内参基因是5.8S,引物序列参见表1。
表1定量PCR检测引物
Table1Primersusedinthisstudy

基因Genes引物序列(5′→3′)Primers sequencesaxinForwardTGATCACTTACCTCCGCCTCReverseGAGTGGCATTTCTCTCAGCGdisForwardAGCTCCACTTCAACCTCCTCReverseCGGTTGAGACGTAGAATGCGβ-cateninForwardTGTCACGCAAAGGAGGAGATReverseGGGATTATGTGGCGGTTGACefaForwardATGAAGCCGGGTATGATCGTReverseTAACTTGGGCGGTGAACTCT36258ForwardTAGACGCCGCAAGCGCGTA36364ForwardGCTGCAGGCCGGAAATT25846ForwardACGCGAGATTTGGTTTGTCCTCReverseOffered by miRNA q-PCR assay kit (CW2142s; Beijing ComWin Biotech Co., Ltd)5.8SForwardTCGATGAAGAGTGCAGCCGACReverseATGCGTTCGAAGTGTCGATGTT
1.6 wnt/β-catenin信号通路相关miRNA潜在靶标基因在细粒棘球绦虫原头蚴的组织分布
根据miRNA靶基因的序列设计荧光原位杂交探针,探针由广州外显子生物技术有限公司合成。全量组织荧光原位杂交的操作流程主要参照Olson实验室网站(http://www.olson lab.com/)公开的操作方法以及参考文献所述方法进行[14],主要包括样品的固定、蛋白酶处理、预杂交、杂交、复染等过程。
2 结 果
2.1 Small RNA测序数据预处理
笔者总共从细粒棘球绦虫原头节得到22 752 526 条reads序列,在去除低质量的reads序列和接头序列后,最终还剩21 708 040条洁净序列,其中细粒棘球绦虫特有序列1 172 539条。这些洁净reads序列的长度大多集中在21~23 nt之间(图1),此结果提示本研究建立的小RNA文库中含有丰富的miRNA序列。除miRNA外,在文库中还鉴定到其他一些小RNA的序列,如rRNAs、tRNAs、snRNAs和snoRNAs,并且rRNAs和tRNAs在文库中占有较高的比例。
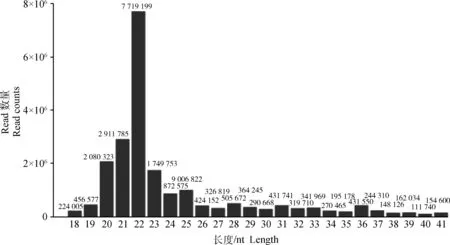
图1 原头蚴small RNA reads长度的分布
Fig.1 The length distribution of reads for small RNA in protoscoleces
2.2 序列比对注释
2.2.1 参考基因组比对 将洁净的reads序列首先与细粒棘球绦虫参考基因组进行比对,统计结果显示,比对到参考基因组上的reads序列为16 828 531 条,占全部reads总数的77.52%,其中细粒棘球绦虫原头蚴特异的reads数为529 664个,占总数的45.17%。
2.2.2 miRNA数据库(miRNABase数据库)比对注释 采用Bowtie软件将序列与miRNABase中该miRNA成熟体序列进行无错配比对,比对上的序列认为是已知的miRNA。结果显示有7 505 269个reads序列可以比对到miRNABase数据库,占总数的34.57%,其中细粒棘球绦虫特异性序列650条,占总数的0.05%。
和细粒棘球绦虫现已公布的227个已知miRNA 分子序列对比发现,在细粒棘球绦虫原头蚴中,存在121个已知的miRNA序列,但经过比对后有109个miRNA表达。其中相对表达量处于前10位的miRNA分子如下:egr-miR-10、egr-let-7、egr-bantam、egr-miR-71、egr-miR-2b、egr-miR-4989、egr-miR-61、egr-miR-125、egr-miR-3479和egr-miR-2a,见表2。
表2细粒棘球绦虫原头蚴中表达量前10的已知的miRNA分子
Table2TherelativeabundanceofthetoptenhighestexpressedknownmaturemiRNAsinPSCofE.granulosus

miRNA名称Name of miRNA表达量Expression levelegr-miR-103 439 042egr-let-72 085 925egr-bantam982 374egr-miR-71273 283egr-miR-2b103 300egr-miR-498983 696egr-miR-6182 295egr-miR-12551 371egr-miR-347951 269egr-miR-2a43 385
2.3 新miRNA预测
2.3.1 新miRNA的预测 采用miRDeep2软件结合该物种同源的miRNA 序列、RNAfold等RNA二级结构预测软件预测识别新miRNA的成熟体(star miRNA与mature miRNA)和前体序列。针对每个新预测的miRNA,miRDeep2软件采用各种评价方法评估其预测准确度,结果显示共预测出189条新的miRNA发夹前体,其中含有189个mature 序列和189个star序列,但经过比对后只有260个miRNA有表达。其中相对表达量处于前10位 的novel miRNA分别为HG328392.1_6805、HG328414.1_36428、HG328424.1_38644*、HG328465.1_42151、HG328457.1_41721*、HG328407.1_34056、HG328397.1_22187、HG328392.1_10888*、HG328393.1_11978和HG328391.1_3385*,见表3。
表3细粒棘球绦虫原头蚴中表达量前10的新的miRNA分子
Table3TherelativeabundanceofthetoptenhighestexpressednovelmiRNAsinPSCofE.granulosus

miRNA名称Name of miRNA表达量Expression levelHG328392.1_680516 122HG328414.1_3642810 952HG328424.1_38644∗10 134HG328465.1_421517 269HG328457.1_41721∗2 234HG328407.1_340561 077HG328397.1_22187893HG328392.1_10888∗548HG328393.1_11978520HG328391.1_3385∗385
2.3.2 新的miRNA二级结构预测 通过RNAfold等RNA二级结构预测软件预测识别新miRNA的成熟体(star miRNA与mature miRNA)和前体序列。结果显示本研究测序获得的novel miRNA都具有典型的hairpin发卡结构(如图2所示),本研究预测了wnt/β-catenin信号通路相关的3个miRNA,即HG328399.1_25846、HG328414.1_36364*和HG328413.1_36258的二级结构,见图2A~C。
2.4 miRNA靶标基因的预测
根据miranda软件预算法则,本研究对测序获得的细粒棘球绦虫原头蚴miRNA潜在的靶标基因进行了预测,结果显示已知的109个miRNA共预测得到132个潜在的靶标基因。对260个有表达的新鉴定得到的miRNA的预测结果表明,有3 152个潜在的靶标基因。同时本研究还对机体生长发育相关wnt以及胆酸盐信号通路相关的miRNA进行了分析,结果显示与上述信号通路潜在互作的miRNA分子分别为30个和13个,其具体的互作关系如图3、图4所示。
2.5 wnt/β-catenin信号通路相关miRNA的筛选及qPCR验证
采用qPCR方法对软件预测的wnt/β-catenin信号通路相关miRNA以及靶标基因在原头蚴体外培养0、4、10 d的相对转录量进行了分析,结果显示,β-catenin基因在原头蚴体外培养0、4、10 d的过程中其相对转录量呈逐渐下降的趋势,其对应的miRNAHG328413.1_36258的相对转录量呈现递增趋势,dis以及axin基因在原头蚴体外培养0、4、10 d的过程中其相对转录量呈逐渐升高的趋势, 而其对应的miRNAHG328414.1_36364* 和HG328399.1_25846的相对转录量则呈现逐渐下降的趋势,见图5。
2.6 wnt/β-catenin信号通路相关miRNA潜在靶标基因在细粒棘球绦虫原头蚴的组织分布
对细粒棘球绦虫原头蚴wnt/β-catenin信号通路相关miRNA潜在靶标基因的全量组织原位杂交分析结果表明,β-catenin、axin以及dis基因在原头蚴顶突的口钩部位均有分布,除此之外,β-catenin和axin基因在虫体原头蚴散在的细胞中也有表达,见图6。
3 讨 论
2011年,Cucher等[15]首次在细粒棘球绦虫中鉴定得到了38种microRNA,其中16种和其他后生动物同源,18种和更低等的生物同源,还有4种可能是棘球属绦虫特有的。同时研究还发现,这些microRNA在虫体不同发育阶段的表达存在差异,提示其功能可能与发育相关。近年,Bai等[16]对细粒棘球绦虫3个不同发育阶段(即成虫、幼虫以及包囊)的microRNA进行了检测,最终发现了94个前体序列,编码91个成熟的miRNA和39个miRNA stars。

图2 HG328399.1_25846(A)、HG328414.1_36364*(B)和HG328413.1_36258(C)发卡结构预测
Fig.2 Hairpin structure prediction of HG328399.1_25846(A)、HG328414.1_36364*(B) and HG328413.1_36258(C)

图3 细粒棘球绦虫原头蚴中wnt信号通路相关miRNA-mRNA的互作网络
Fig.3 The miRNA-mRNA networks related wnt signaling pathway of protoscoleces in E. granulosus

图4 细粒棘球绦虫原头蚴中胆酸盐信号通路相关miRNA-mRNA的互作网络
Fig.4 The miRNA-mRNA networks related bile-salt signaling pathway of protoscoleces in Echinococcus granulosus
研究发现有些miRNA是组织特异性表达的,提示其可能与虫体特有组织的发育相关。其中还有42个miRNA在不同发育阶段差异表达,推测其可能与虫体双向发育、营养代谢以及神经系统发育相关。进一步的研究发现在细粒棘球绦虫中,30个 保守的miRNAs家族中的很多成员都存在缺失,这可能与虫体较低的复杂性以及较高的适应性相关[17]。
已有的研究表明,一些棘球属的miRNAs在脊椎动物宿主中是缺失的,这些miRNAs可能是棘球绦虫特有的,提示这些分子也许可以作为疾病诊断的潜在靶标。起初有研究者在宿主血清中检测到曼氏血吸虫的miRNAs分子,包括miR-277 和bantam,由于这些miRNAs分子在未感染的宿主中并不存在,因此可以作为血吸虫病检测的靶标基因[18]。无独有偶,之后又有研究者分析发现了一些棘球属绦虫特有,而宿主缺失的miRNAs,包括miR-71、miR-4989 (miR-277家族) 和bantam,或者是一些由宿主同源的分子分化而来的分子,诸如let-7等,也可作为包虫病诊断的潜在靶标基因[19]。
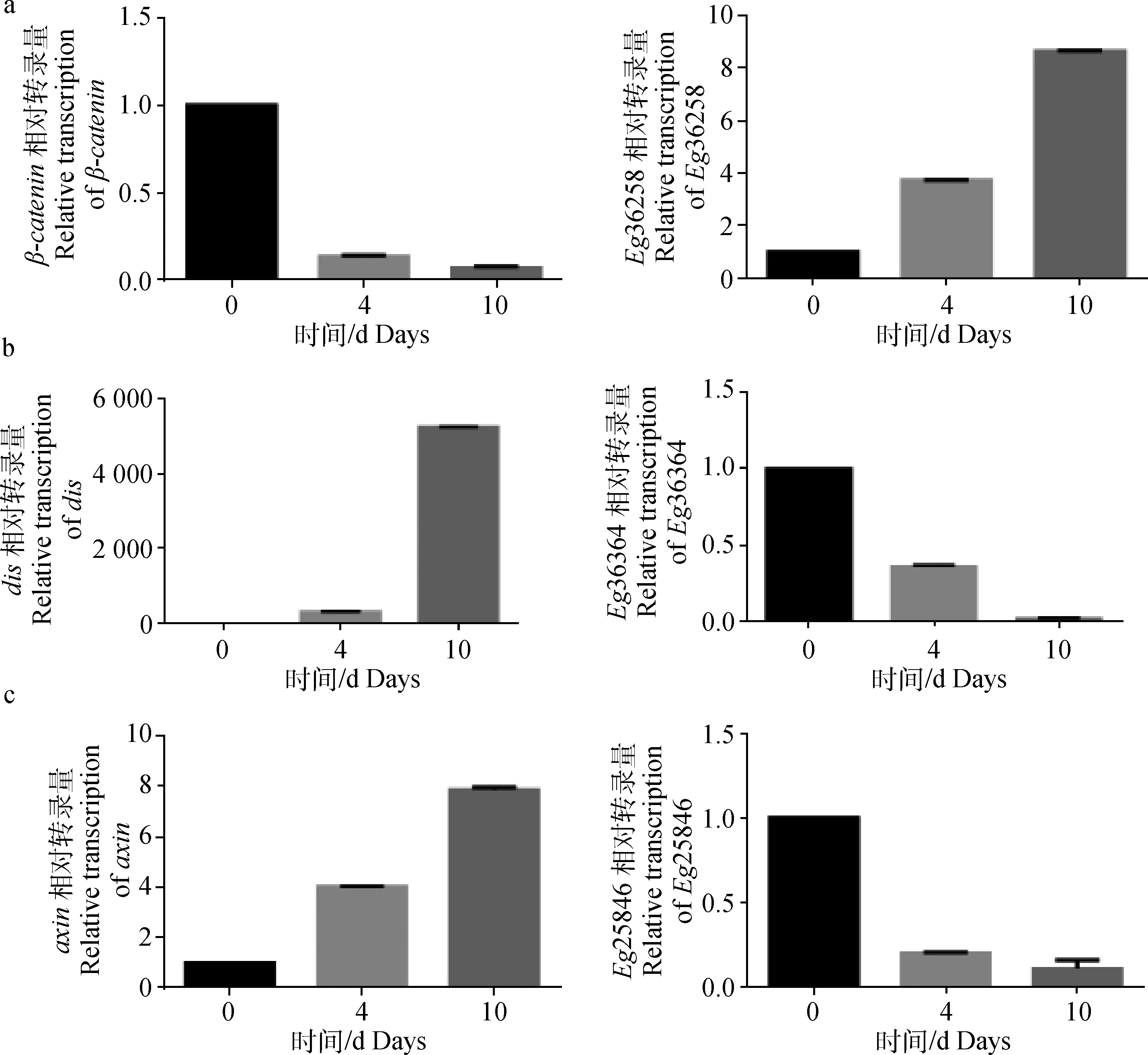
a. HG328413.1_36258 vs β-catenin; b. HG328414.1_36364* vs dis; c. HG328399.1_25846 vs axin
图5 miRNA和候选靶基因的相对转录水平分析
Fig.5 The relative transcription of miRNA and their candidate targets genes

H.口钩;C.散在的细胞。阳性杂交结果呈绿色,细胞核呈红色
H. Hooks; C. Disperse cells. The positive results of Whole-mount in situ hybridization signal is shown in green and nuclear PI staining in red
图6 β-catenin、dis和axin基因在细粒棘球绦虫原头蚴的原位杂交分析
Fig.6 The results of β-catenin,dis and axin in E. granulosus protoscoleces in situ hybridization
这一研究结论在后期的研究中又得到了进一步的佐证,有学者在感染多房棘球绦虫的小鼠血清中检测到2种虫体特异性的miRNAs,包括miR-10和miR-227[20]。
本研究发现,在细粒棘球绦虫原头蚴中,存在109个已知的miRNA序列,其中表达量相对较高的有egr-bantam、egr-let-7、egr-miR-10、egr-miR-125、egr-miR-190、egr-miR-2162、egr-miR-2a、egr-miR-2b、egr-miR-3479、egr-miR-4989、egr-miR-4989*、egr-miR-61和egr-miR-71。可以看出前面研究报道的棘球属特有的miRNA分子诸如miR-71、miR-4989、miR-277和bantam等同样存在于细粒棘球绦虫原头蚴,且表达量较高。除此之外,本研究从细粒棘球绦虫原头蚴中分离得到189个新的成熟序列和189个star序列,后经比对后发现,只有260个miRNA有表达。为了更好地理解miRNA在细粒棘球绦虫原头蚴的调控作用,本研究分析了与动物机体5大信号通路(即wnt、cAMP、Hedgehog、NF-κB 和Notch信号通路)相关的miRNA分子,结果发现,与上述信号通路潜在互作的miRNA分子分别为30、34、14、6和13个,其中已知的miRNA分子有egr-miR-4990、egr-new-125和egr-miR-125,其余均为本研究新发现的miRNA分子。由于细粒棘球绦虫原头蚴具有双向发育性,并且已有的研究已证实,在原头蚴发育为成虫的过程中受到胆酸盐信号通路的调节。因此,本研究同样分析了与胆酸盐信号通路存在潜在互作的miRNA分子,发现有13个miRNA分子可能参与调控此信号通路,且均为新发现的miRNA分子。
经典的wnt信号通路,即wnt/β-catenin为动物体轴发育关键的信号通路[21-22]。前期的研究表明,细粒棘球绦虫具有完整的wnt信号通路主要成员[23]。本研究采用miranda软件预测了与wnt/β-catenin信号通路主要基因互作的miRNA分子,结果显示有12个miRNA分子,文库编号为HG328403.1_31513*、HG328429.1_39208*、HG328392.1_10158、HG328392.1_6661、HG328399.1_25562、HG328396.1_20495、HG328408.1_34466*、HG328395.1_18164*、HG328399.1_25846、HG328413.1_36258、HG328414.1_36364*和HG328392.1_7008,其潜在的靶基因分别为wnt2、frizzled5、LRP、β-catenin、GSK-3β、dis和axin,后经qPCR验证表明,仅有HG328413.1_36258和β-catenin、HG328414.1_36364*和dis以及HG328399.1_25846和axin其表达趋势相反,存在潜在的调控关系。之后笔者又采用全量组织原位杂交技术分析了这三个潜在靶标基因的组织分布规律,结果发现其主要分布于细粒棘球绦虫原头蚴顶突的口钩、内部散在的细胞以及表皮部位。
4 结 论
对细粒棘球绦虫原头蚴表达的miRNA进行了测序研究。结果发现了大量的新miRNA分子,通过对这些miRNAs靶基因预测以及初步验证发现,这些非编码的小分子可能广泛参与细粒棘球绦虫原头蚴的生长发育过程。这些数据为进一步揭示miRNA在细粒棘球绦虫原头蚴生长发育中的调控机制提供了新的视角。
