RACE法克隆巴什拜羊SP-A基因及序列分析
2018-11-30王继雪孙延鸣
王继雪,孙延鸣
(石河子大学动物科技学院,新疆 石河子 832003)
【研究意义】肺表面活性物质相关蛋白A(Pulmonary surfactant-associated protein A, SP-A)是C-型凝素家族的成员之一,具亲水性的一种糖蛋白,作为肺部主要的防御和免疫分子,在肺中不仅在天然免疫反应发挥着作用,而且在局部防御中也起着不可取代的作用[1]。【前人研究进展】周佳磊[2]等通过研究具有肺炎的普通患儿与重症患儿血清中的SP-A表达水平,发现其血清中SP-A表达水平重症患儿要明显高于普通患儿。此外,许明峰[3]等应用基因测序技术对SP-A基因多态性与儿童支原体肺炎易感的相关性进行了研究,结果发现SP-A基因第186位碱基以及第655位碱基具有多态性且儿童易患支原体肺炎可能与该多态性相关,并且分析,可能正是由于个体的SP-A基因突变,进而影响了该个体对肺炎支原体的先天性免疫和抗病作用。黄文川[4]等研究了SP-A对大鼠肺组织的作用,结果发现当大鼠肺组织灌洗液中SP-A浓度下降时,肺组织损伤加重,进而得出SP-A对肺组织有明显的保护作用。Virginia L[5]等通过对宿主防御肺部病原体的研究表明SP-A在宿主吸入病原体的初始防御中起着关键作用。【本研究切入点】巴什拜羊作为新疆地方的优良品种,具有耐粗饲、生长发育快、抗病力强、早熟性好等优点[6]。剡文亮等[7]对新疆地区绵羊肺炎支原体的染病率做了调查,结果表明不同品种的绵羊对肺炎支原体的感染与发病有明显的差异,巴什拜羊在临床上很少发生绵羊支原体肺炎。目前对SP-A基因的研究在小鼠、家猪和人上比较集中,而对于巴什拜羊SP-A基因的研究报道在国内外还尚未出现。【拟解决的关键问题】把新疆巴什拜羊作为本研究的研究对象,克隆巴什拜羊SP-A基因的cDNA全长并对其序列进行分析,为巴什拜羊SP-A基因的载体的构建、蛋白的表达以及生物学功能的研究奠定了基础。
1 材料与方法
1.1 试验动物
巴什拜羊5只,均在塔城地区巴什拜羊繁殖基地购买。
1.2 主要试剂
Trizol (Invitrogen)、LATaq酶(TaKaRa)、TAKARA SMARTer® RACE 5’/3’Kit Protocol-At-A-Glance试剂盒及pMD18-T载体(TaKaRa)均购自宝生物工程(大连)有限公司。Trans2K DNA Marker K189购自北京全式金生物技术(TransGen Biotech)有限公司, 薄型琼脂糖凝胶DNA回收试剂盒购自中科瑞泰(北京)生物科技有限公司。大肠杆菌DH5α由石河子大学动物科技学院生物化学实验室保存。
1.3 引物设计
参考GenBank中绵羊的SP-A基因序列,又因不确定开放阅读框以外序列,便在开放阅读框内用Primer5软件设计特异性引物,5’RACE引物为GSP1:5’-GTTGTGCTTCTTCACGATGCTGGTAATGGCCTCATTCTCCT-3’;3’RACE引物为GSP2:5’-TGCTGTGCTCTTTGACCCTTAT-3’。引物由北京六合华大基因科技股份有限公司合成。
1.4 总RNA提取
通过颈动脉放血屠宰巴什拜羊,无菌手术打开羊胸腔,取出肺组织并剪成小块放入1 mL无菌无酶液氮冻存管,随后立即放入液氮罐中保存,用液氮研磨法将肺组织研成粉末,称重并按每100 mg组织加入1 mL Trizol,按说明书操作提取总RNA,微量紫外检测仪测定RNA的浓度,后用2%的琼脂糖凝胶对RNA进行电泳检测,选用电泳条带清晰完整且OD260/OD280为1.9~2.0的RNA用SMARTer®RACE 5’/3’ Kit Protocol-At-A-Glance反转录试剂盒分别制备5’和3’RACE ReadycDNA。
1.5 巴什拜羊SP-A基因cDNA序列5’端的RACE扩增
把用RACE试剂盒反转的5’RACE Ready cDNA作为模板,GSP1为下游引物,试剂盒提供的UPM(Long primer=5’-TAATACGACTCACTATAGGGCAAGCAGTGGTATCAACGCAGAGT-3′;Short primer=5’-CTAATACGACTCACTATAGGGC-3’)为上游引物,进行5’端RACE扩增。扩增体系为:PCR-Grade H2O 15.5μl,2X SeqAmpTMBuffer 25.0 μl,SeqAmp DNA Polymerase 1.0 μl,5’-RACE-Ready cDNA 2.5μl,10X UPM 5.0 μl, GSP1 1.0 μl,Total Volume 50.0 μl。扩增程序为:94 ℃预变性5 min,94 ℃变性35 s,72 ℃延伸3 min,5个循环;94 ℃变性35 s,70 ℃退火30 s,72 ℃延伸3 min,25个循环;94 ℃变性35 s,69 ℃退火30 s,72 ℃延伸3 min,5个循环,4 ℃ Store。用1 %琼脂糖凝胶对扩增产物进行电泳检测,后用胶回收试剂盒将目的片段进行胶回收。把胶回收产物与PMD18-T载体相连接并转化到大肠杆菌DH5α感受态细胞中,PCR筛选阳性克隆并选取5个阳性克隆送北京六合华大基因科技有限公司测序。
1.6 巴什拜羊SP-A基因cDNA序列3’端的RACE扩增
3’端的 RACE 扩增体系与 5’端相同,3’-RACE Ready cDNA 为模板,GSP2 和UPM(与5’扩增所用UPM引物序列一致)为引物。扩增程序为94 ℃预变性5 min,94 ℃变性40 s,70 ℃退火35 s,72 ℃延伸3 min,5个循环;94 ℃变性40 s,65 ℃退火35 s,72 ℃延伸3 min,5个循环;94 ℃变性40 s,60 ℃退火35 s,72 ℃延伸3 min,5个循环,94 ℃变性40 s,58 ℃退火35 s,72 ℃延伸3 min,4 ℃ Store。用1 %琼脂糖凝胶对扩增产物进行电泳检测,后用胶回收试剂盒将目的片段进行胶回收。把胶回收产物与PMD18-T载体相连接并转化到大肠杆菌DH5α感受态细胞中,PCR筛选阳性克隆并挑选5个阳性克隆送北京六合华大基因科技有限公司测序。
1.7 巴什拜羊SP-A基因全长cDNA序列的获得及序列分析
用DNAMAN软件将5’RACE和3’RACE扩增片段测序结果拼接得到SP-A基因cDNA全长,用DNAstar软件进行理化分析,用MEGA7软件构建氨基酸进化树。
2 结果与分析
2.1 巴什拜羊肺组织总RNA的提取
由图1显示,用Trizol法提取肺组织的总RNA,经1 %琼脂糖凝胶电泳并用凝胶成像系统检测后发现28S、18S和5S RNA条带很清晰,且28S是18S的1倍以上,证明RNA完整性较好,可用于后续试验。

图1 巴什拜羊肺组织RNA电泳图Fig.1 RNA electrophoresis of lung in Bashibay sheep
2.2 巴什拜羊SP-A基因5’端的RACE扩增
由图2可以看出,以5’RACE Ready cDNA为模板,应用GSP1为下游引物,UPM为上游引物,开展5’端RACE,扩增出了657 bp条带。
2.3 巴什拜羊SP-A基因3’端的RACE扩增
由图3可以看出,以3’RACE Ready cDNA为模板,应用GSP2为上游引物,UPM为下游引物,开展3’端RACE,扩增出了1847 bp条带。
2.4 巴什拜羊SP-A基因分析
如图4所示,将SP-A基因5’端与3’端用DNAMAN软件拼接后最终得到巴什拜羊SP-A基因的cDNA全长序列,该序列总长度为1962 bp,通过DNASTAR软件分析,巴什拜羊SP-A基因开放阅读框789 bp(70~858 bp),5’非编码区长69 bp,3’非编码区长1104 bp且具有典型的真核生物polyA加尾信号AATAAA及polyA结构。编码氨基酸序列如图4灰色部分,共编码262个氨基酸。其中有22个强碱性氨基酸(K、R),32个强酸性氨基酸(D、E),53个极性氨基酸(Y、N、C、S、T、Q),72个疏水氨基酸(V、A、F、W、I、L)。碱基使用情况为A=22.81 %[180],T=20.28 %[160],C=25.35 %[200],G=31.56 %[249],不确定碱基=0.00 %[0],(A+T)=43.09 %[340],(C+G)=56.91 %[449]。预测蛋白质分子量为27.53kDa,等电点4.949。

图2 SP-A基因5’端的RACE电泳图Fig.2 5 ′RACE electrophoresis of SP-A gene

图3 SP-A基因3’端的RACE电泳图Fig.3 3 ′RACE electrophoresis of SP-A gene
2.5 系统进化分析
如表1所示,将克隆得到的巴什拜羊SP-A基因序列在NCBI网站进行Blast比对,结果显示巴什拜羊SP-A基因与绵羊、山羊、藏羚羊、家牛、空齿鹿、小须鲸、羊驼、犀牛、马、中华菊头蝠、东北虎的同源性比较高,尤其与绵羊同核苷酸源性最高。
如表2所示,将巴什拜羊SP-A基因编码氨基酸序列在NCBI网站进行Blast比对,结果显示巴什拜羊SP-A基因编码氨基酸序列与绵羊、山羊、藏羚羊、家牛、骆驼、羊驼、野猪、家鼠、东非狒狒的同源性较高,尤其与绵羊氨基酸同源性最高。
如图5所示,将巴什拜羊、绵羊、山羊、藏羚羊、家牛等动物的SP-A基因编码氨基酸序列调入MEGA7软件,进行进化树分析,可清楚地看到进化树形成了2个团簇,在巴什拜羊、绵羊、山羊、藏羚羊、家牛这个团簇中巴什拜羊与绵羊又形成对称的合枝,表明它们在进化关系上最近,进化树上还有独立分离的树枝野猪、家鼠、东非狒狒,表明巴什拜羊与它们的进化关系最远。
3 讨 论
杜智慧等[8]对巴什拜羊与其杂交羊分别人工感染MO,通过观察所感染的羊的临床症状及剖解后的肺组织学病理变化,采集并检测羊血清中的IFN-γ、IL-1β、IL-6及IL-9浓度,综合分析后最终得出巴氏拜羊可抗MO病原体感染。许明峰[3]等发现SP-A基因第186位碱基以及第655位碱基具有多态性,且儿童易患支原体肺炎与该基因位碱基多态性具有相关性,由此表明SP-A基因是具有多态性的。但巴什拜羊具有抗MO感染的特性是否也与SP-A基因相关位点基因的多态性有关,本次实验通过RACE技术扩增巴什拜羊SP-A基因全长,为我们进一步研究巴什拜羊抗MO感染的特性与SP-A基因多态性是否具有相关性奠定了基础。

图4 巴什拜羊SP-A基因全长及编码蛋白的氨基酸序列Fig.4 Full-length amino acid sequence and encoding protein of SP-A gene in Bashibay sheep
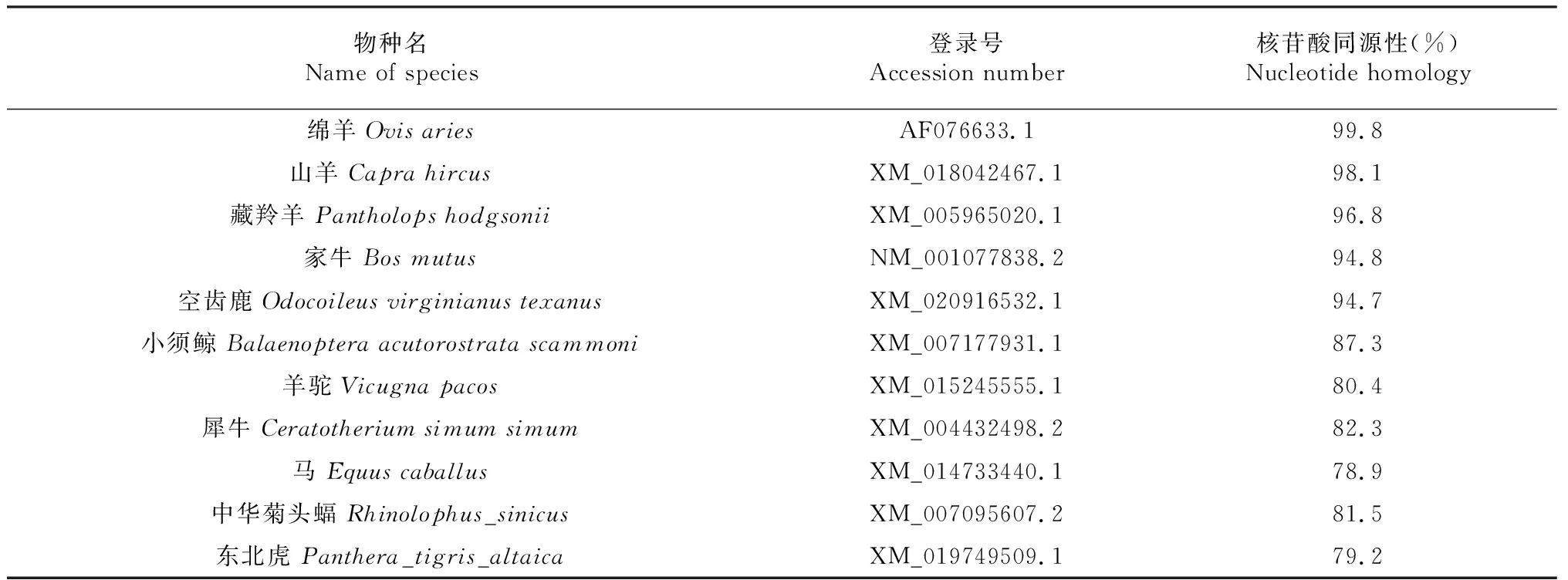
物种名Name of species 登录号Accession number核苷酸同源性(%)Nucleotide homology绵羊 Ovis ariesAF076633.199.8山羊 Capra hircusXM_018042467.198.1藏羚羊 Pantholops hodgsoniiXM_005965020.196.8家牛 Bos mutusNM_001077838.294.8空齿鹿 Odocoileus virginianus texanusXM_020916532.194.7小须鲸 Balaenoptera acutorostrata scammoniXM_007177931.187.3羊驼 Vicugna pacosXM_015245555.180.4犀牛 Ceratotherium simum simumXM_004432498.282.3马 Equus caballusXM_014733440.178.9中华菊头蝠 Rhinolophus_sinicusXM_007095607.281.5东北虎 Panthera_tigris_altaicaXM_019749509.179.2
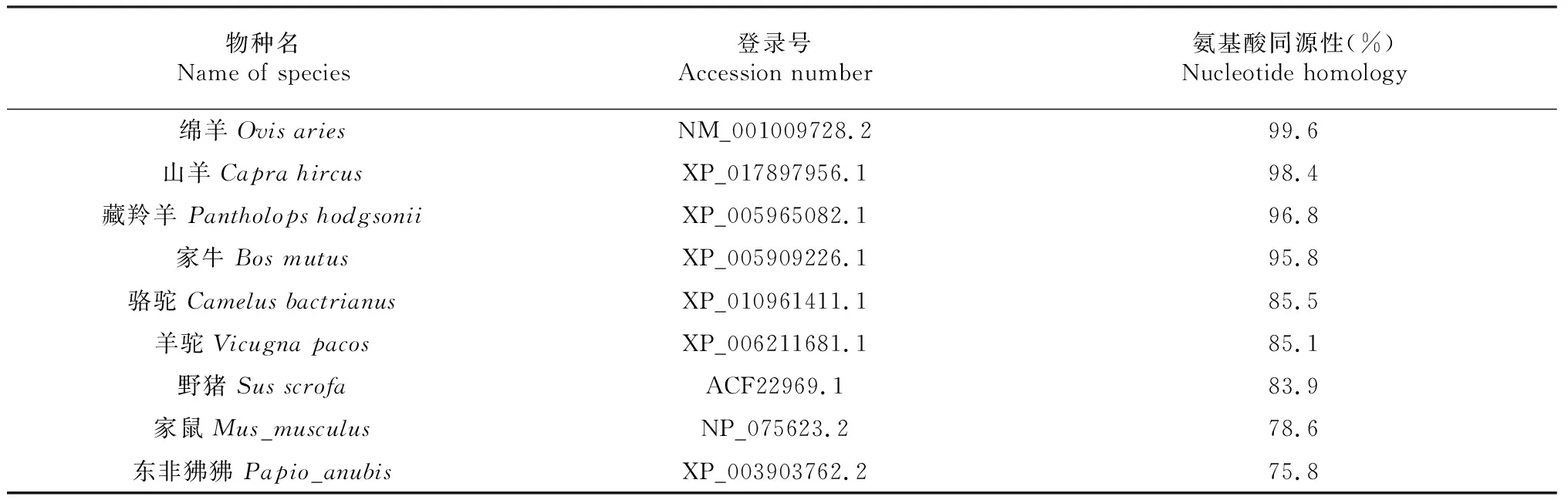
表2 巴什拜羊SP-A基因编码氨基酸与不同动物的同源性
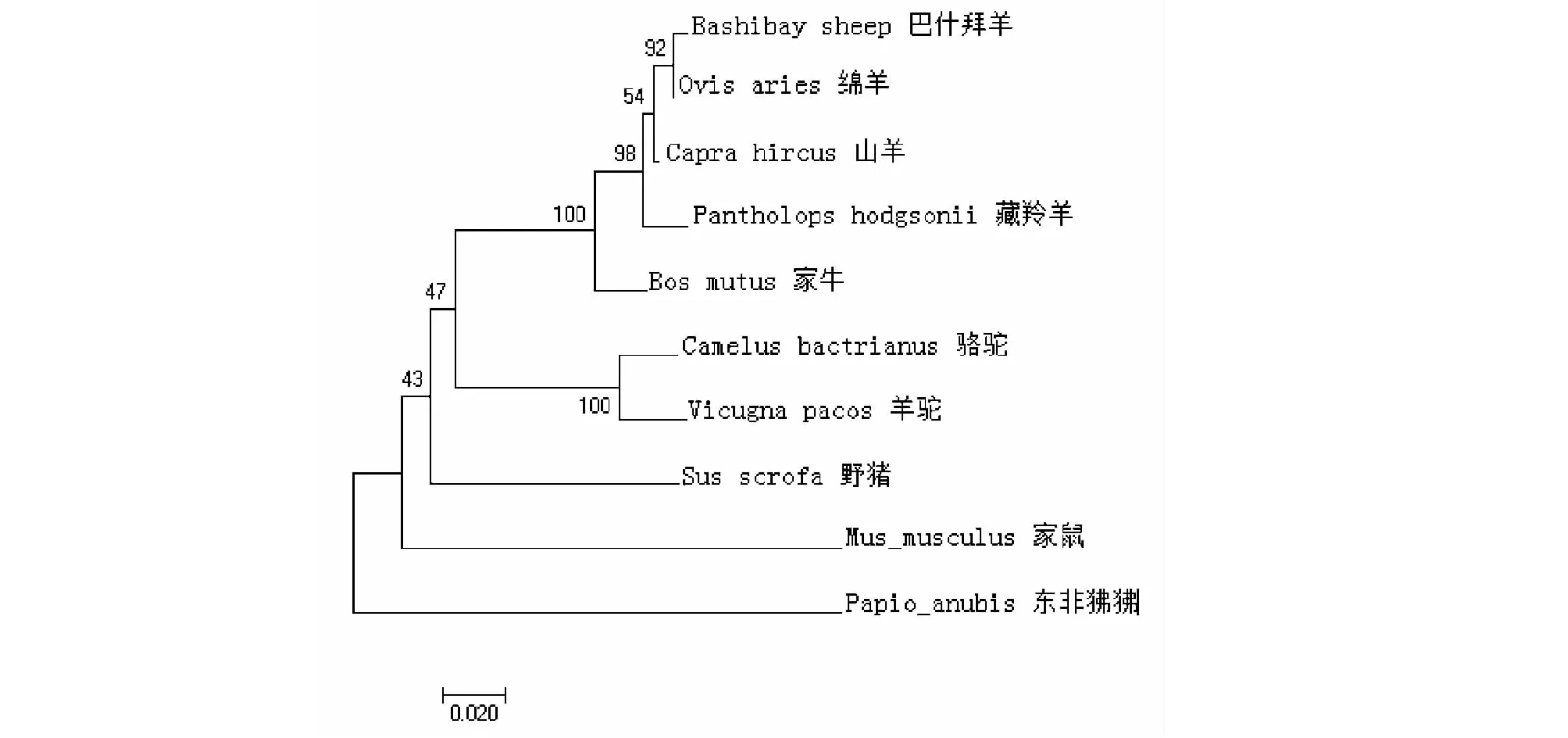
图5 巴什拜羊SP-A基因编码氨基酸进化分析Fig.5 Evolutionary analysis of amino acids encoded by SP-A gene in Bashibay sheep
应用RACE技术可利用低丰度转录本分别扩增出cDNA的3′末端及5′末端,短时间内便可获得目的mRNA的全长序列,又由于该技术简单、廉价、快速,现已成为一种被广泛应用的分子生物学技术[9]。近年来,越来越多的全长RNA被扩增出来,这都得益于RACE技术的应用[10]。本研究从绵羊SP-A基因的开放阅读框内设计特异引物,又采用灵敏高效的TAKARA SMARTer® RACE 5’/3’ Kit Protocol-At-A-Glance试剂盒,这样便可以保证RACE方法可扩增出巴什拜羊SP-A基因,将扩增出的DNA片段测序并与SP-A基因的开放阅读框比对可确定所扩增出的DNA片段是否为目的片段。按照RACE试剂盒说明书扩增出巴什拜羊SP-A基因的5’端和3’端后,经测序以及DNAMAN软件比对发现SP-A基因的5’端和3’端序列重合部位发生在GenBank上查到的绵羊SP-A基因开放阅读框序列,这与当初设计引物所要达到的效果一致。
在NCBI上将巴什拜羊SP-A基因全长进行BLAST比对发现巴什拜羊SP-A基因与绵羊(GenBank登录号:NM_001009728.2)同源性最高,可达99.8 %。与绵羊相比巴什拜羊SP-A基因开放阅读框也是789 bp,也编码262个氨基酸,不同的是5’端比绵羊短11 bp,3’端比绵羊长26 bp,进一步分析差异发现开放阅读框内有2处核苷酸差异,即第198位碱基由G变成A,密码子由ATG变成ATA,导致氨基酸由M(蛋氨酸)变成I(异亮氨酸);第703位碱基由A变成G,密码子由AAT变成GAT,导致氨基酸由N(天冬酰胺)变成D(天冬氨酸)。
氨基酸的突变往往会使相关蛋白的结构及其功能的发生改变[11],由此可推想巴什拜羊SP-A基因编码氨基酸的突变有没有引起其相关蛋白结构或功能的变化,要证实这一点还需进一步的研究。
进化树又称之为系统树,它是把图表形象的画成树的形状,把各类物种安放在树的节点上,如果某些物种在进化关系上比较密切则对应的进化树上的两个分支节点的距离也会比较贴近。每个物种在进化树上的分枝都有一定的长度,它是对生物遗传距离和进化时间的一个度量[12]。以往对于进化的研究其主导思想是比较各物种之间的特征差异,特征越是相近的物种则认为它们在遗传进化上越是贴近,但这样得到的信息往往不可靠[13],因此本次研究选择使用进化树,以SP-A基因为研究对象,通过MEGA7软件将巴什拜羊SP-A基因编码蛋白序列与其它同源性较高的各类动物SP-A基因编码蛋白序列进行对比,然后构建出系统进化树,这样有了理论依据,而不是单纯的凭外貌特征来判断各物种间的进化关系,因而可以得出更科学可靠的结果。从进化树上可以清楚的看到巴什拜羊与绵羊、山羊、藏羚羊、家牛亲缘关系比较近,尤其是与绵羊亲缘关系最近,与骆驼、羊驼、野猪、家鼠、东非狒狒亲缘关系比较远,尤其与野猪、家鼠、东非狒狒亲缘关系最远。
4 结 论
本研究使用RACE法首次获得了新疆巴什拜羊SP-A基因cDNA全长序列,通过对其进行序列分析及简单的信息学分析,发现巴什拜羊及与之同源性最高的绵羊SP-A基因5’端和3’端长度有所差异,进一步分析差异性在开放阅读框内发现2处碱基差异且两处碱基差异均使得编码的氨基酸发生改变。这使得进一步认识了新疆巴什拜羊的SP-A基因,也为后续研究中SP-A基因载体的构建、蛋白的表达、蛋白的结构功能研究以及其他相关功能的研究奠定了基础。
