Trichoderma lentiforme ACCC30425脂肪酶基因的原核表达及其酶学性质的初步研究
2018-11-28王宇洲陈敬师李世贵龚明波顾金刚
王宇洲, 马 锐, 向 杰, 陈敬师, 李世贵, 龚明波, 顾金刚*
1.中国农业科学院农业资源与农业区划研究所, 农业农村部农业微生物资源收集保藏重点实验室, 北京 100081;2.中国农业科学院饲料研究所, 农业农村部饲料生物技术重点实验室, 北京 100081
脂肪酶(EC 3.1.1.3)被认为是最重要的商业酶之一,可在油水界面上催化甘油三酯水解释放甘油二酯、长链脂肪酸(>8碳)和甘油[1]。在水解过程中,脂肪酶与甘油酰基结合形成脂肪酶酰基复合物,然后将酰基转移到水的-OH上;而在非水性催化条件下,脂肪酶将羧酸酰基转移至亲核化合物上[2]。根据蛋白质结构的相似性,脂肪酶属于α/β水解酶家族,其中催化三联体(通常为丝氨酸、组氨酸、谷氨酸或天冬氨酸)和氧离子空穴是催化反应的关键[3],此外,盖子结构、底物亲和性也与活性位点的结合能力有着紧密关系。
脂肪酶广泛存在于微生物、植物和动物中,其中,细菌脂肪酶、真菌脂肪酶因具有产量高、易操作等特点,一直是研究和应用的重点。研究表明,细菌和酵母来源的脂肪酶可在高温下保持较高的酶活力,如来源于Pseudomonasaeruginosa、嗜热芽孢杆菌(Bacillusthermophilus)和酵母(Kurtzmanomycessp.)的脂肪酶,最适温度在60~75℃[4,5]。
木霉(Trichoderma)具有显著的木质纤维素降解能力,能产生多种水解酶、裂解酶和辅助酶,包括纤维素酶、木聚糖酶、几丁质酶、漆酶、脂肪酶等[6],而且木霉是酶制剂产业的重要表达系统之一[7]。目前,已完成了13株木霉菌株的基因组测序[8,9],其中绿色木霉[10]、里氏木霉[11]、哈茨木霉[12]和T.gamsii[13]的基因组序列分析结果表明,木霉普遍具有多个脂肪酶基因[14]。本课题组在前期研究中筛选获得了1株脂肪酶酶活力较高的木霉菌株T.lentiformeACCC30425[15],本研究依据该菌株的全基因组测序结果,注释了15个脂肪酶基因,以此进行基因合成和原核异源表达,并对脂肪酶的酶学性质进行初步研究,以期为耐热脂肪酶的开发利用提供参考。
1 材料与方法
1.1 菌株与质粒
大肠杆菌感受态细胞EscherichiacoliTransI和BL21(DE3)均购自北京全式金生物技术有限公司;质粒pET-30a(+)载体购于美国Invitrogen公司。
1.2 溶剂和底物配制
异丙基硫代β-D-半乳糖苷(isopropyl-β-D-thiogalactopyranoside,IPTG)、硫酸卡那霉素、LB培养基等配制方法均参考孙乔乔等[16]的配方。
底物配制:A液:取固体对硝基苯酚棕榈酸酯(p-nitrophenylpalmitate,pNPP)8 mmol溶解于10 mL异丙醇中,温热溶解,于4℃保存,有效期为1周;B液:用蒸馏水配制20 mmol/L Tris,加0.22 g阿拉伯胶,用盐酸调至pH 8(视具体情况而定),于4℃保存,有效期为1周。按照1∶9的比例混合A液和B液,制成底物溶液。
1.3 脂肪酶基因分析
利用KOG/KEGG/GO数据库对全基因组测序结果进行注释,获得脂肪酶基因。再利用Vector NTI 10软件(美国Invitrogen公司)对cDNA序列进行翻译并获得推导氨基酸序列,分析等电点和蛋白质分子量,然后利用SignalP(http://www.cbs.dtu.dk/services/SignalP)线上软件预测信号肽长度,最后利用BlastP(http://www.ncbi.nlm.nih.gov/BLAST/)分析序列相似性。同时,利用Swiss-model(https://swissmodel.expasy.org/)完成预测脂肪酶的同源建模以及与PDB数据库(https://www.rcsb.org/)中序列的比对,并采用MEGA 6和ClustalX 1.81构建系统发育树,进行蛋白质聚类分析。
1.4 脂肪酶基因合成与原核表达
根据ACCC30425全基因组序列,经数据库注释获得了15个脂肪酶基因cDNA,以此为基础,手动删除信号肽,选择合适酶切位点,删除原有终止密码子,将所需合成的成熟肽链的DNA序列信息交至上海捷瑞生物工程有限公司,以合成密码子优化的适合原核表达的核苷酸序列。对合成产物以及pET30a(+)质粒进行EcoRⅠ与XhoⅠ双酶切后,使用T4 DNA连接酶进行连接(20℃过夜)。将连接产物pET30a(+)-lip热激转化至E.coliBL21(DE3)感受态细胞中,获得pET30a(+)-lip-BL21,挑单菌落验证阳性克隆。再利用IPTG诱导菌株表达,并通过超声波破碎获得脂肪酶粗酶液[16]。
1.5 重组脂肪酶的酶学性质分析
1.5.1pNPP法测定脂肪酶酶活力 按照pNPP法测定脂肪酶酶活力,于37℃反应15 min后,加入2 mL无水乙醇终止反应,随后12 000 r/min离心2 min取上清液,于410 nm处测定吸光度(OD值),进而计算重组脂肪酶的活性[15]。1个脂肪酶活性单位(U)定义为在一定的反应条件下,每分钟分解pNPP生成1 μmoL对硝基酚(p-Nitrophenol,pNP)所需的脂肪酶酶量[16]。
1.5.2酶学性质分析 酶活粗筛:按照1.5.1分别测定重组酶酶活力。
最适反应pH:保持A液不变,用不同pH(7.0、8.0、8.5、9.0、9.5和10.0)的20 mmol/L Tris-HCl缓冲液配制B液。其他步骤同1.5.1。
最适反应温度:在酶的最适pH条件下,于不同温度(40℃、50℃、60℃、65℃、70℃、80℃、90℃和95℃)中测定酶活力。其他步骤同1.5.1。
2 结果与分析
2.1 脂肪酶基因分析
经T.lentiformeACCC30425基因组序列分析,共注释获得15个脂肪酶编码基因(表1)。Vector NTI 10软件分析结果表明菌株ACCC30425的脂肪酶分子量在55~70 kDa之间,其中ACCC30425_8519基因编码的脂肪酶分子量最大,高达68.8 kDa,而ACCC30425_5744编码的脂肪酶分子量最小。脂肪酶的等电点处于4.0~6.0之间。SignalP线上软件确定所有脂肪酶均为胞外分泌。
基于Swissprot数据库的Blast比对,有11个脂肪酶与已知序列的相似性低于50%,其中有5个低于40%;而于PDB数据库中序列比对,有10个脂肪酶与已知序列的相似性低于40%,其中有3个低于30%。结合2个数据库的比对结果,有5个脂肪酶基因(表1中下划线标注)在酶学性质和结构比对方面都具有一定的新颖性。
2.2 T. lentiforme ACCC30425脂肪酶的聚类分析
将T.lentiformeACCC30425的15个脂肪酶基因与相似序列以最大相似法构建聚类关系树(图1)。结果表明,菌株ACCC30425的脂肪酶与哈茨木霉(T.harzianum)和贵州木霉(T.guizhouense)来源的脂肪酶的同源性更高,其中脂肪酶ACCC30425_2714更接近于T.lixii来源的脂肪酶。
2.3 T. lentiforme ACCC30425重组脂肪酶的酶学性质分析
T.lentiformeACCC30425的15个脂肪酶基因经密码子优化后分别与载体pET30a(+)连接以构建重组质粒,并在E.coliBL21(DE3)感受态细胞中表达。
2.3.1脂肪酶酶活力筛选 按照1.5.1测定重组脂肪酶酶活力,结果表明,共有11个阳性克隆产脂肪酶,分别为LipA、LipB、LipC、LipD、LipH、LipI、LipK、LipL、LipM、LipP、LipQ,具体酶活力见表2。其中,LipP、LipK、LipH、LipM、LipQ、LipD的酶活力均高于0.1 U/mL,LipB的酶活最低,而其他4个克隆未产脂肪酶。
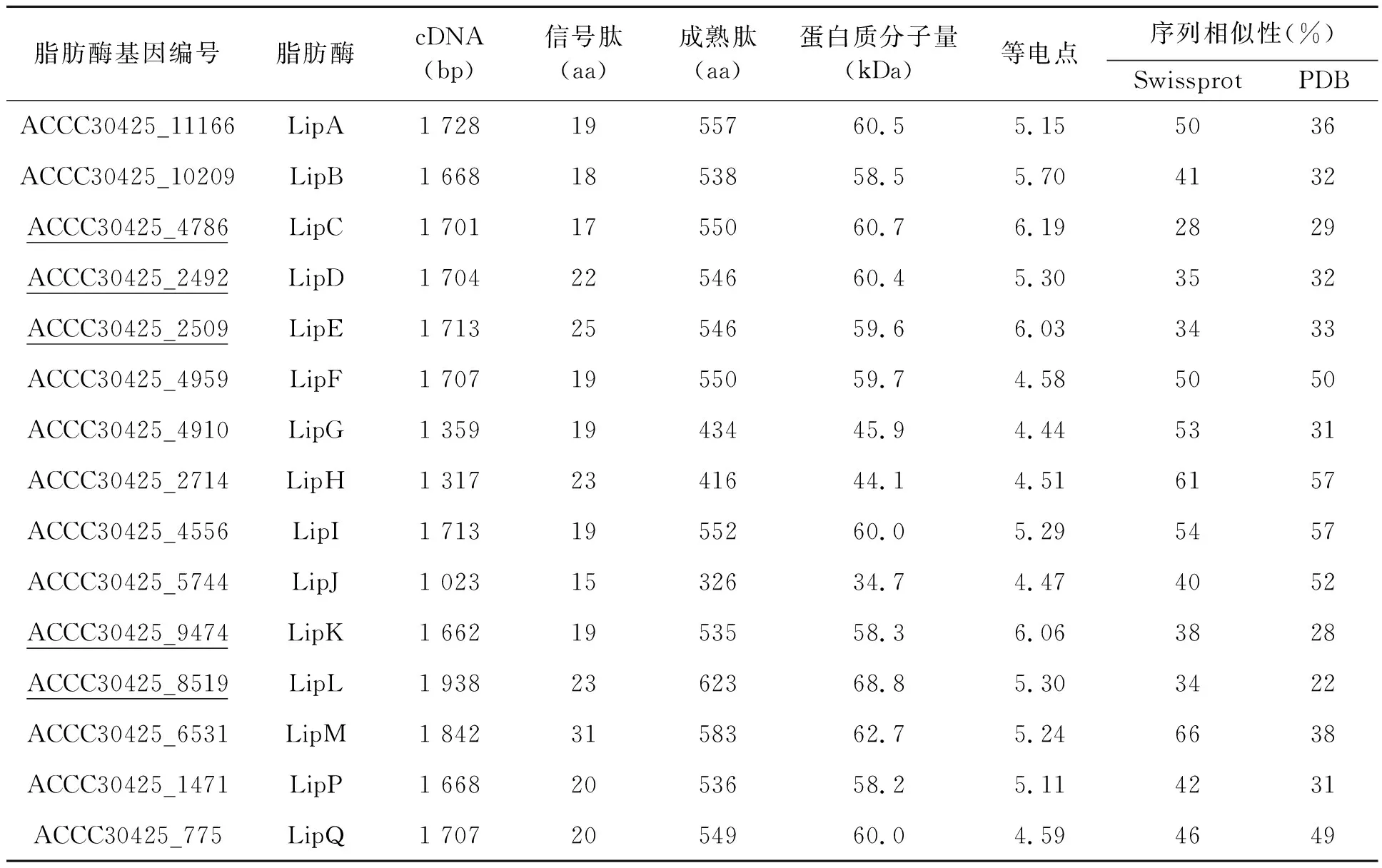
表1 Trichoderma lentiforme ACCC30425的脂肪酶基因分析Table 1 Analysis of the lipase genes from Trichoderma lentiforme ACCC30425.
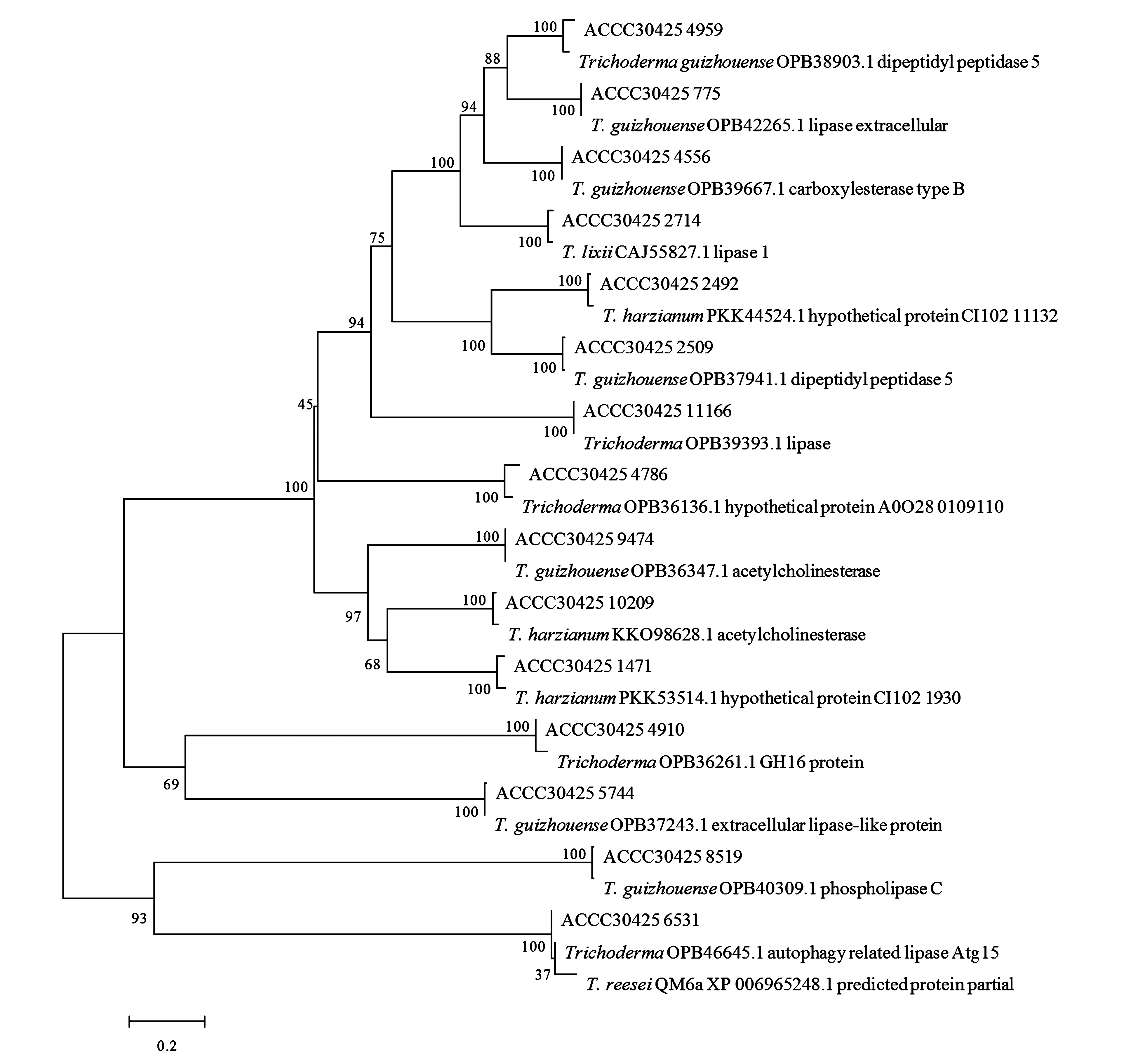
图1 T. lentiforme ACCC30425脂肪酶的聚类分析Fig.1 Cluster analysis of the lipases from T. lentiforme ACCC30425.
2.3.2最适反应pH 在37℃、不同pH条件下测定T.lentiformeACCC30425重组脂肪酶酶活力。如图2所示,LipB最适pH为8.5,LipA、LipH最适pH为9.0,而LipC、LipD、LipI、LipK、LipL、LipM、LipP和LipQ的最适pH均为9.5。当pH<8.5时,LipL失去活性;当pH>10.0时,LipA和LipB的相对活性低于60%;而其他脂肪酶均保持着60%以上的相对活性。因此,T.lentiformeACCC30425脂肪酶普遍具有耐碱性,即在碱性条件下可保持较高的酶活力。

表2 重组脂肪酶活性Table 2 Activities of recombinant lipases.

图2 pH对Trichoderma lentiforme ACCC30425重组脂肪酶酶活力的影响Fig.2 Effect of pH on the activities of recombinant lipases from Trichoderma lentiforme ACCC30425.A:具有特性的酶LipA、LipB、LipH;B:性质相近的酶LipC、LipI、LipM、LipP;C:性质相近的酶LipD、LipK、LipL、LipQ。
2.3.3最适反应温度 在最适pH、不同温度的条件下测定脂肪酶酶活力。结果如图3所示,LipQ为中温酶,最适温度为50℃,当温度高于60℃时,其酶活力急剧下降。其他脂肪酶均属高温脂肪酶,最适温度在70~90℃。其中,LipP的最适温度为90℃,且在中温环境也保持50%以上的相对活性;LipA、LipL的最适温度为70℃;而其他脂肪酶的最适温度均为80℃。
2.3.4最适条件下测定酶活力 如图4所示,重组脂肪酶在最适条件下的酶活力,相较于标准条件均有明显提高。如在标准条件下酶活力最低的LipB,在最适条件下其酶活力提高了87倍;LipP酶活力仍为最高,在最适条件下其酶活力提高了8倍,高达1.5 U/mL。最适温度为80℃的脂肪酶在最适条件下的酶活力普遍在0.8~1.2 U/mL范围内。
3 讨论
筛选具有特殊性质的脂肪酶一直备受研究领域和工业应用领域的关注,微生物来源的脂肪酶最适温度普遍在30~50℃之间,温度过高会导致脂肪酶结构变性至失活,国内单志文等[17]在2001年首次筛选到1个来源于Pseudomonasfragi的脂肪酶,其最适温度为65~70℃,具有耐热性。
在实际应用中,脂肪酶参与的工业催化反应过程大多需要较高的反应温度,尤其是替代化石能源的生物柴油的生产工艺的研究。近年来,人们一直在挖掘耐热以及超耐热酯酶/脂肪酶,通常是先在热泉和笼屉等高温环境下筛选嗜热菌株,再对嗜热微生物中的脂肪酶基因进行克隆、异源表达,亦或构建突变体,从而改良酶学性质,如来自嗜热细菌[18]、短梗霉[19]、假单胞菌[20]、芽孢杆菌[21,22]等嗜热微生物[23]的耐热酯酶/脂肪酶基因重组表达后的最适反应温度往往在60~75℃,此外,来自嗜热古细菌的酯酶[24]的最适反应温度为90℃,此为迄今国内发现的最适反应温度最高的酯酶,然而超耐热脂肪酶的研究近来鲜有报道。
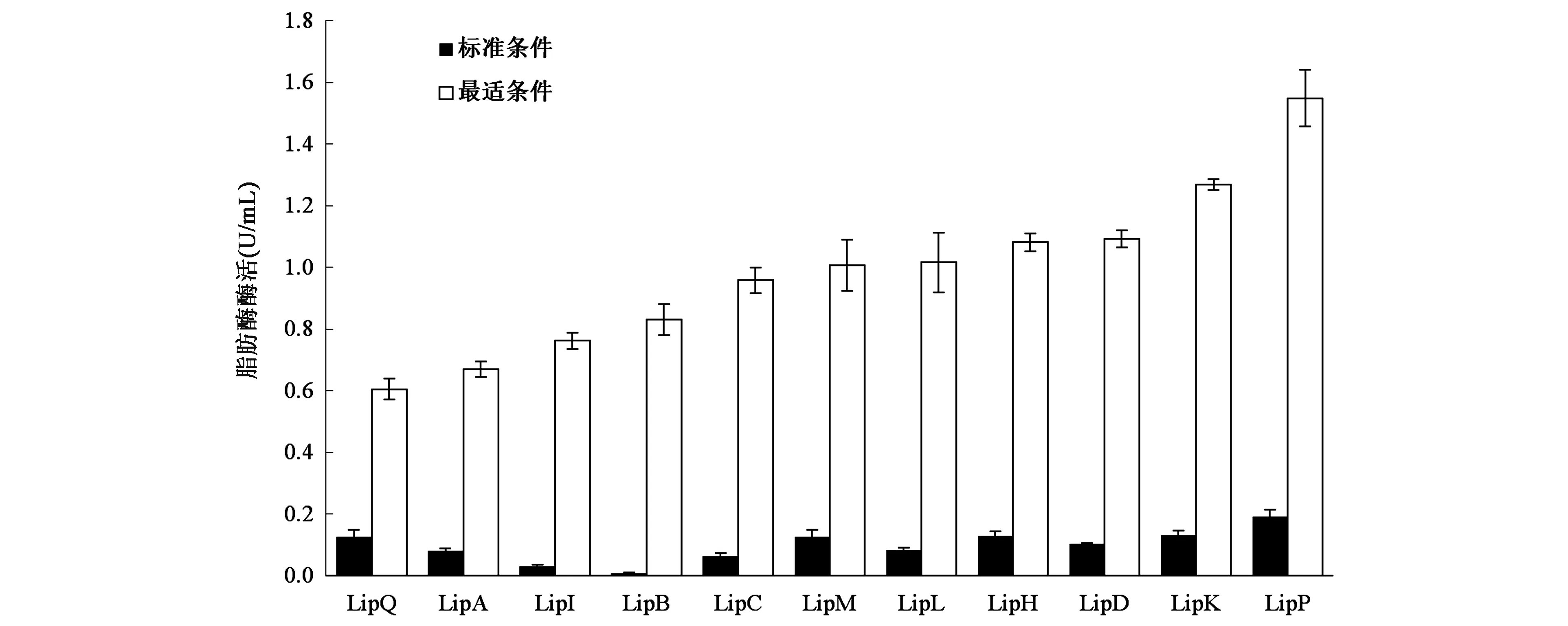
图4 脂肪酶在标准条件酶活和最适反应条件下的酶活力Fig.4 The activities of lipases under standard and optimal reaction conditions.
本研究首次报道了基于基因组序列分析的木霉Trichodermalentiforme脂肪酶的系统研究,来源于T.lentiformeACCC30425的脂肪酶LipP最适温度为90℃,是目前已知的最适反应温度最高的脂肪酶。此外,本研究还发现了多个具有耐碱性(pH 8.5~9.5)、耐热性(70~90℃)的脂肪酶,为脂肪酶的基础研究和潜在应用提供了宝贵的材料。这些性质各异的木霉脂肪酶具有很好的应用前景和开发潜能,需要通过更加高效的表达系统进行表达和纯化,以便在酶学的结构功能上进行更深一步的研究。
