甘薯TPS基因家族分析及IbTPS7a基因克隆与功能研究
2018-11-28邵欢欢郑海燕廖若星费雪婷马沁沁袁向华王晓燕
邵欢欢, 郑海燕, 廖若星, 徐 攀, 费雪婷,雍 彬*, 马沁沁, 袁向华, 王晓燕
(1.四川师范大学 生命科学学院,四川 成都 610101;2.四川师范大学 附属中学,四川 成都 610101)
关键字:甘薯;海藻糖;抗逆;数字表达谱
农作物在生长过程中容易受到干旱、极端温度、高盐及病虫害等胁迫,使其生长发育受到限制,进而影响产量。为了应对来自环境中的非生物或生物胁迫,经过长期的进化,植物形成了复杂的响应机制,例如植物可以改变其形态和生理生化状态来适应环境的变化[1]。海藻糖(Trehalose)是一种非还原性二糖,由两分子葡萄糖通过半缩醛羟基缩合反应形成的α,α-1,1糖苷键连接而成,在细菌、真菌、昆虫、无脊椎动物和植物中广泛存在[2]。海藻糖在酸、碱、高温等条件下具有很强的稳定性和强吸水性,可以有效地保护细胞并使其具有抗脱水作用。海藻糖分子中具有较多的羟基可以与多种生物大分子结合形成氢键,对生物大分子能起到保护作用并降低失活风险,有助于维持细胞或生物体的正常生命过程,进而提升生物体的抗逆能力[3]。自然界中已经发现了5个海藻糖合成通路,分别是TPS-TPP途径、TreP途径、TreS途径、TreY-TreZ途径和TreT途径,其中TPS-TPP途径是高等植物唯一的海藻糖合成途径[4-6]。TPS-TPP途径是由两个关键酶经过两步反应形成,首先在TPS催化下尿苷二磷酸葡萄糖(UDPG)和 6-磷酸-葡萄糖(G6P)反应生成 6-磷酸海藻糖(T6P),T6P在磷酸海藻糖磷酸酯酶(TPP)催化下最终形成海藻糖[7]。
甘 薯 (Ipomoea batatas (L.) Lam.), 旋 花 科(Convolvulaceae)番薯属(Ipomoea)一年生草本植物,淀粉含量高且营养丰富,是一种重要的粮食作物。中国甘薯资源十分丰富,种植面积和产量均居世界第一[8]。甘薯多被种植在不发达地区的边缘土地中,产量很高但并不占用良田,对环境要求较低,容易受到干旱、盐碱、极端温度等非生物胁迫,因此其抗胁迫能力也较强。
在高等植物中已经发现了较多的TPS基因,有些植物中具有由一系列TPS基因组成的TPS基因家族,例如拟南芥基因组中具有11个TPS基因(AtTPS1-11)组成的AtTPS基因家族[9],根据拟南芥TPS基因家族序列特点在杨树和水稻等植物中也发现了类似的TPS基因家族,其中杨树TPS基因家族有12个PtTPS基因,水稻TPS基因家族有11个OsTPS基因[10-11]。目前还没有关于甘薯TPS基因家族及功能研究的报道,作者根据NCBI数据库中拟南芥和杨树的TPS基因家族序列,利用本地Blast(BlastN和BlastP)在甘薯转录组数据库[12]中共发现了7个TPS基因,成功克隆得到了IbTPS7a基因,分析了IbTPS7a基因在甘薯不同组织中的表达量,在大肠杆菌和酿酒酵母对IbTPS7a基因的抗逆功能进行了验证,有助于了解甘薯的TPS基因抗逆功能和作用机制,为未来甘薯的育种及基因工程改造研究打下了基础。
1 材料与方法
1.1 材料
1.1.1 薯材、菌株、质粒 甘薯品种为徐薯18,大肠杆菌克隆及表达菌株分别为Escherichia coli DH5α和 E.coli BL21 (DE3), 酵 母 表 达 菌 株 为Saccharomyces cerevisiae INVSc1,大肠杆菌表达载体为 pET-32a(+),酵母表达载体为 pYES2。
1.1.2 主要试剂、工具酶和分析软件 RNA提取试剂 盒 MiniBEST Plant RNA Extraction Kit: 购 自TaKaRa公司;PCR高保真酶PrimeSTAR®HS(Premix):购自TaKaRa公司;酵母基因组提取试剂盒:购自北京天根生物科技有限公司;胶回收试剂盒E.Z.N.A.TMPlasmid Mini Kit I:购自Omega Bio-tek 公司;M-MLV逆转录酶: 购自 Invitrogen;RNase inhibitor及各种常规限制内切酶:购自Thermo Scientific公司;引物合成及测序:成都擎科梓熙生物技术有限公司合成。
1.2 方法
1.2.1 甘薯TPS基因家族分析及进化树构建 利用已知的单子叶植物水稻和双子叶植物拟南芥中TPS基因家族核酸序列和蛋白序列,用BlastParser软件对甘薯转录组数据库分别进行本地BlastN和BlastP(e-value为1e-10)比对,获得相关同源序列,结合两种比对结果分析甘薯转录组数据库中可能存在的TPS基因家族。将获得的甘薯TPS基因家族序列与拟南芥的相关序列使用MEGA 5.0软件采用Neighbor-joining法制作系统进化树。
1.2.2 总RNA提取及反转录 根据TaKaRa公司植物总RNA提取试剂盒的推荐方法对分别对徐薯18的叶片、块根和茎提取总RNA,将去除基因组DNA后无降解的总RNA使用Thermo公司的Nanodrop 2000进行RNA浓度测定,并根据浓度按照比例将3个组织样品的总RNA进行等量混合,以M-MLV反转录酶反转录得到cDNA第一链,分装后保存在-20℃冰箱中备用。
1.2.3 IbTPS7a基因克隆及表达载体构建 以甘薯转录组测序数据及前期经比对得到的甘薯TPS基因家族中的IbTPS7a序列为模板,使用SLIC(sequence and ligation independent cloning) 法设计引 物 , 引 物 序 列 所 下 所 示 :TPS-32sF:5’ -GAAAACCTGTACTTCCAGGGTATGATGTCCAAATC GTATACCAACTT-3’ ;TPS-32sR:5’-GTTTAGAGG CCCCAAGGGGTTATCAATCCGAGCTTCCGGTGG-3’ ;PET_TPS-F:5’-CCACCGGAAGCTCGGATTGA TAACCCCTTGGGGCCTCTAAAC-3’ ;PET_TPS-R:5’-ACCCTGGAAGTACAGGTTTTCACCAGAAGAAT GATGATGATGATGG-3’其中下划线部分为pET32a(+)载体骨架部分。以反转录得到的cDNA和pET32a(+)质粒为模板,分别以 TPS-32sF和TPS-32sR以及PET_TPS-F和PET_TPS-R为引物,使用PrimeSTAR®HS(Premix)分别进行 PCR扩增IbTPS7a基因和线性 pET32a(+)载体,PCR产物经电泳后胶回收,回收产物经T4 DNA Polymerase处理、退火,构建得到重组质粒pET32-IbTPS。把得到的pET32-IbTPS质粒经PCR验证后送至成都擎科梓熙生物技术有限公司进行测序,以测序正确的质粒为模板使用引物TPS-yF(5’-CGGGATCCCGATG ATGTCCAAATCGTATACCAACTT-3’) 和 TPS-yR(5’ -GCTCTAGAGCTCAATCCGAGCTTCCGGTGG-3’)进行PCR,将纯化后的PCR产物和pYES2载体使用BamH I和Xba I进行双酶切,胶回收使用T4 DNA ligase于18℃过夜连接后转化大肠杆菌DH5α,获得的阳性克隆进行培养后提取质粒,将质粒酶切和PCR验证后送至公司进行测序。
1.2.4 生物信息学分析 根据测序得到IbTPS7a基因 序 列 , 用 BLAST(http://blast.ncbi.nlm.nih.gov/Blast.cgi)和DNAMAN进行基因及蛋白同源序列比对;使用 ProtParam(http://web.expasy.org/protparam/)对IbTPS7a蛋白质理化性质进行预测;进化树使用MEGA 5.0软件的Neighbor-Joining法构建,蛋白质氨基酸进化距离计算采用Poisson correction模型,bootstrap自检重复1 000次;信号肽使用Signal IP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)进行预测;跨膜结构分析使用TMpred(http://www.ch.embnet.org/software/TMPRED_form.html); 亚细胞定位 使 用 Wolf psort (http://www.genscript.com/wolfpsort.html)。
1.2.5 原核表达及非生物胁迫耐受性分析 将成功构建的pET32-IbTPS质粒转化E.coli BL21(DE3)菌株,使用含有氨苄青霉素(Ampicillin,50 μg/mL)抗性平板筛选,挑取单菌落至2 mL的液体LB中过夜培养,取500 μL菌液加入50 mL液体LB中,37℃ 培养至 A600nm为 0.4~0.6时加入 IPTG(Isopropyl β-D-Thiogalactoside,终浓度为 1 mmol/L),将50 mL菌液放置于18℃培养箱中诱导表达16 h,SDS-PAGE电泳检测IbTPS7a蛋白表达效果。
挑取含有pET32-IbTPS质粒的阳性克隆菌落于含50 μg/mL Amp的 2 μL LB液体培养基中 37℃震荡过夜培养,取100 μL将其接入10 mL液体LB(含 50 μg/mL Amp)中当 A600nm为 0.6 时,加入IPTG至终浓度为1 mmol/L,18℃诱导培养4 h。按1%比例,取500 μL此培养物分别移入50 mL含0.6 mol/L NaCl、0.8 mol/L NaCl、 质 量 分 数 20%PEG6000和30%PEG6000的液体LB培养基(IPTG终浓度为1 mmol/L)中,在37℃培养箱中持续震荡培养3 d,每间隔4 h取样并测定其A600nm值,绘制生长曲线,实验均重复3次,含空载体的对照菌株按照相同的方法绘制生长曲线。
1.2.6 酿酒酵母转化及非生物胁迫耐受性分析利用醋酸锂法将pYES2-IbTPS和pYES2质粒分别转化S.cerevisiae INVSc1,将两种菌落接种至液体培养基中,使用酵母基因组提取试剂盒提取含有质粒的酿酒酵母总DNA,用引物TPS-yF和TPS-yR以提取得到的DNA为模板进行PCR扩增,检测成功转化质粒的菌株,鉴定正确的菌液划线固体平板保存至4℃冰箱中短暂保存。
分别挑取含有pYES2空载体和pYES2-IbTPS的单菌落接种于10 mL SD-URA液体培养基中,30℃振荡培养24 h,测定A600nm值,将菌液转接至含有半乳糖(20 g/L)的诱导培养基中,使初始A600nm值均为0.4,30℃诱导表达30 h,将最终菌液A600nm值调为一致。取1 mL诱导的酵母菌液,离心后去上清液,菌体分别悬浮于1 mL的5 mol/L NaCl、4 mol/L山梨醇和PBS溶液(pH 7.4),置于4℃胁迫处理24 h,稀释1 000倍,取50 μL涂布于不含半乳糖的SD-URA固体平板上,放至30℃培养箱中培养,通过平板计数计算酿酒酵母细胞的生存率。
1.2.7 数字表达谱分析 利用Illumina Pipeline软件获取甘薯的嫩叶、成熟叶片、茎、须根、初始块根、膨大块根和收获块根等7个不同组织原始的21 bp的DGE标签,Bowtie定位有关IbTPS7a基因完整CDS的clean tag(仅允许一个碱基错配)。使用edgeR package程序对RNA的组成和不同的测序库规模进行误差评估,对获得的基因的表达量进行标准化,从而获得准确的IbTPS7a基因的表达量[13]。

表1 甘薯的TPS基因家族Table 1 TPS family genes in Ipomoea batatas
2 结 果
2.1 甘薯TPS基因家族挖掘及分析
为了挖掘甘薯的TPS基因家族,作者根据拟南芥和水稻的TPS家族核酸及蛋白序列对甘薯转录组数据库分别进行BlastN和BlastP,将比对得到的contig序列翻译得到的TPS蛋白进行序列和功能单元分析,共筛选得到7个全长IbTPS基因(如表1所示),其中有两个IbTPS蛋白序列经比对发现其同源性达到75%,且这两个IbTPS蛋白与其它物种的TPS7蛋白序列同源性最高,因此分别被命名为IbTPS7a和IbTPS7b。生物信息学分析发现甘薯TPS蛋白都具有TPS蛋白所含有的N端GT1_TPS保守区域及C端Trehalose_PPase保守区域。信号肽分析结果表明7个甘薯TPS蛋白都没有信号肽,亚细胞定位分析发现IbTPS1和IbTPS7a最有可能位于叶绿体 中 ,IbTPS5、IbTPS6、IbTPS7b 和 IbTPS11 可能位于细胞质中,而IbTPS9可能位于细胞核中,这表明甘薯不同的IbTPS蛋白在胞内分布区域及功能可能有所不同。利用已知的拟南芥11个TPS蛋白与甘薯7个TPS蛋白进行进化树分析,结果发现与拟南芥TPS蛋白家族类似,甘薯的TPS蛋白也可以分为两个亚家族,其中IbTPS1属于I类家族,其余IbTPS蛋白属于II类家族(如图1所示)。
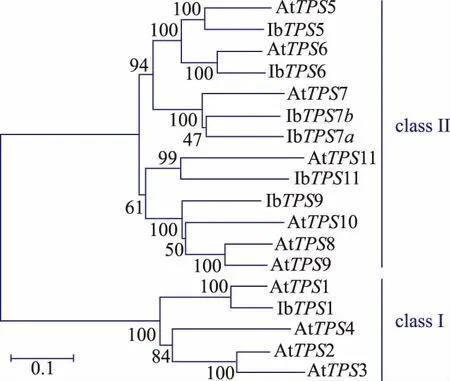
图1 甘薯和拟南芥中TPS蛋白家族进化树分析Fig.1 Phylogeneticanalysisofthe TPS family in Arabidopsis and Ipomoea batatas
2.2 IbTPS7a基因克隆及表达载体构建
以提取得到的徐薯18总RNA为模板,使用通用引物oligo(dT)15进行反转录得到cDNA,再以cDNA为模板使用TPS-32sF和TPS-32sR进行PCR,以 pET32a(+)质粒为模板使用 PET_TPS-F和PET_TPS-R进行扩增,电泳检测在约2 600 bp左右和5 900 bp左右各有一明亮条带(图2),与之前预期的扩增的IbTPS7a基因大小和pET32a(+)质粒大小一致;使用SLIC法构建pET32-TPS质粒,以测序正确的pET32-TPS质粒为模板,使用TPS-yF和TPS-yR进行PCR扩增,在2 600 bp左右有一明亮条带,将PCR片段与pYES2载体分别双酶切后连接转化,得到pYES2-IbTPS质粒。

图2 IbTPS7a基因PCR产物及载体构建Fig.2 Agarose electrophoresis analysis and expression vector construction of IbTPS7a
2.3 IbTPS7a生物信息学分析
生物信息学分析结果显示,IbTPS7a基因长度为2 568 bp,其翻译的蛋白长度为855 aa,IbTPS7a蛋白分子式为C4310H6710N1158O1277S40,蛋白相对分子质量为 94.46×103,理论等电点 5.58,脂肪系数为87.22,亲水性值为-0.230,因此IbTPS7a是一个亲水性蛋白;信号肽预测分析发现IbTPS7a没有明显的信号肽,可见IbTPS7a是一个胞内蛋白,主要在细胞内发生作用,跨膜结构分析发现IbTPS7a具有两个明显的由外到内跨膜结构区域:474-491和790-812;通过亚细胞定位软件Wolf psort对IbTPS7a蛋白在甘薯细胞内的位置进行了预测,结果发现K最近邻(k-Nearest Neighbor,KNN)值为 14,其中 7 个位于叶绿体中,5个位于细胞核中,2个位于液泡中,即IbTPS7a定位可能性为:叶绿体>细胞核>液泡。
2.4 IbTPS7a蛋白进化树分析
使用DNAMAN软件将10种其它植物的TPS蛋白与甘薯IbTPS7a蛋白进行序列比对,发现它们同源性都在82%以上,其中IbTPS7a与Nicotiana sylvestris和Solanum tuberosum蛋白同源性最高为86.7%,与Eucalyptus grandis的TPS7蛋白同源性最低为82%(如图3所示)。将这11条蛋白序列首先进行MEGA 5.0软件进行Clustal W比对,然后将比对后的蛋白序列采用Neighbor-joining法构建系统进化树,Bootstrap自检1 000次,甘薯IbTPS7a蛋白与Nicotiana sylvestris和Solanum tuberosum的TPS7蛋白亲缘关系最近,该结果与同源比对结果一致。

图3 IbTPS7a蛋白进化树分析Fig.3 Phylogenetic tree of IbTPS7a protein
2.5 含IbTPS7a基因的大肠杆菌BL21重组子抗逆境胁迫分析
为了验证IbTPS7a基因在原核细胞中的功能,将 pET32-IbTPS质粒转化 E.coli BL21(DE3)并使用IPTG进行诱导表达,分别使用0.6、0.8 mol/L NaCl、质量分数20%PEG6000和30%PEG6000对IbTPS7a基因过表达菌株进行高盐及干旱模拟胁迫,并对生长曲线进行测定。如图4显示,在无胁迫的LB培养基中含有空载体的对照菌株和转IbTPS7a基因菌株的生长曲线几乎没有差异(图4);在0.6 mol/L NaCl胁迫处理下,转IbTPS7a基因菌株(0 h)相比对照菌株(4 h)更早进入指数生长期,但是两者进入稳定期的时间差异不大,转基因菌株生长效果较好于对照菌株;在0.8 mol/L NaCl胁迫处理下,两种菌株的生长都受到抑制,但转基因菌株的生长明显优于对照菌株;在20%PEG6000胁迫处理下,两种菌株的生长效果差异并不明显;在30%PEG6000胁迫处理下,对照菌株生长困难,转基因菌株明显优于对照菌株,但是相比20%PEG6000胁迫转基因菌株进入指数生长期的时间也明显延长。

图4 重组大肠杆菌BL21/IbTPS7a在盐胁迫及干旱胁迫培养基中的生长曲线Fig.4 Growth curves of IbTPS7a in E.coli BL21 under salt and drought stress
2.6 转基因酵母抗逆境胁迫分析
如图5所示,在无胁迫压力下,含有pYES2空载体的酵母菌株和转IbTPS7a的菌株涂平板后的菌落数基本一致,这表明IbTPS7a基因的过表达对酿酒酵母的正常生长没有明显的影响;两种菌株分别在5 mol/L NaCl和4 mol/L山梨醇进行胁迫处理后,转IbTPS7a基因的酿酒酵母菌株的存活率明显高于对照菌株,因此实验结果表明IbTPS7a基因的过表达可以有效提升酿酒酵母的生存率,降低外界环境改变对细胞的伤害。
2.7 IbTPS7a基因数字表达谱分析
利用数字表达谱对甘薯IbTPS7a基因的组织特异性表达进行分析,结果(如图6所示)发现,甘薯在多个组织中的表达量相对较低,其中在茎(ST)中的表达量最高为15.35 TPM,其次是嫩叶 (YL)为12.23 TPM,在根部组织的表达量都比较低,其中在须根(FR)中的表达量最低为0.84 TPM。
3 讨论
TPS基因可通过多种信号传导途径参与植物的代谢、生长发育及抗逆作用,不同物种的TPS基因对胁迫条件的反应机制存在差异,TPS基因在不同的组织中的表达量也不尽相同[14-15]。甘薯是一种重要的粮食作物,其栽培品种是六倍体,基因组庞大且结构复杂,由于甘薯基因组测序尚未完成,现在对于甘薯遗传资源的挖掘多通过转录组数据来实现[16-17]。在已完成基因组测序的高等植物中都发现有TPS基因家族,而目前尚没有对甘薯TPS基因家族研究的报道。

图5 重组酿酒酵母菌在盐胁迫及干旱胁迫条件下的生长Fig.5 Growth of IbTPS7a in yeast under salt and drought stresses

图6 IbTPS7a基因在甘薯不同组织样品中的表达量Fig.6 Expression level of IbTPS7a in different tissues of Ipomoea batatas by digital gene expression profiling(TPM means the transcript per million)
4 结语
作者使用生物信息学方法对甘薯转录组数据库进行比对分析,首次鉴定得到7个具有完整ORF结构的甘薯TPS基因家族成员,其中有两个TPS基因经鉴定为TPS7基因的两个亚型即IbTPS7a和IbTPS7b,本研究对于TPS基因家族的挖掘基于转录组数据,没有找到全部的甘薯TPS基因,显示了数据不足的缺憾,但是也为未来甘薯TPS基因的研究提供了重要的数据。将甘薯的TPS基因家族与其拟南芥的TPS基因进行了进化树分析,结果发现与拟南芥类似,甘薯TPS基因家族也可以被分为两个亚家族,其中IbTPS1归属于class I,其余的IbTPS基因属于class II。拟南芥的TPS1被证明具有TPS活力,但class II的TPS尚未被证明具有活力[18],为了研究甘薯class II中TPS基因的功能,作者利用RT-PCR方法成功克隆得到class II的IbTPS7a基因,并对其在大肠杆菌和酿酒酵母中的抗逆作用进行的研究,结果发现转IbTPS7a基因后诱导过表达的大肠杆菌菌株相比对照菌株在高盐和干旱胁迫下生长能力明显提升,在酿酒酵母中过表达IbTPS7a基因也可以有效增强真核细胞在高盐和干旱胁迫下生存率也显著提高,这说明过表达甘薯IbTPS7a有助于提升细胞的耐盐和耐干旱作用,增强细胞适应外界环境变化及抵御胁迫的能力。细菌中海藻糖合成除了TreS和TreY/Z途径外还可以通过TPP/TPS途径,酵母中也具有TPP/TPS途径,研究发现外源TPS基因的导入可以很好的修复相关缺陷菌株的海藻糖合成能力,因此甘薯TPS基因的过表达可能有助于促进大肠杆菌和酵母胞内海藻糖的合成,从而保护细胞免受环境胁迫造成的伤害[19-20]。大豆和拟南芥的TPS基因在根和根瘤等组织中的表达量较高[21],而作者发现IbTPS7a基因在茎和嫩叶中的表达量较高,但是在多个根部组织中的表达量较低,这可能是因为甘薯IbTPS7a基因与其它植物中的TPS基因在功能上有所不同。由于海藻糖对生物体优良的保护性能,其在食品、保健、医疗等行业应用越来越普遍[22],因此本研究不仅为进一步研究甘薯TPS基因家族及IbTPS7a的功能打下了基础,也为拓展甘薯海藻糖合成酶在未来的应用提供了理论依据。
