几种植物淀粉合成酶的生物信息学分析
2018-11-06田亚珍武国凡秦绪军牛世全孔维宝
田亚珍, 武国凡, 秦绪军, 牛世全, 孔维宝*
(1.西北师范大学 生命科学学院,甘肃 兰州,730070;2.第四军医大学 预防医学院,陕西 西安,710032)
淀粉是高等植物合成的具有重要功能的碳水化合物之一,并且是一种廉价易得的可再生性资源,已成为很多生产领域的重要原料[1]。淀粉作为植物中能量储存分子以及许多动物基本能量的来源[2],其合成主要发生在两个阶段,一是在形成临时淀粉的光合作用阶段,另一个则是在成为贮藏淀粉的营养积累阶段[3]。淀粉的生物合成包括两种形式:在光合组织叶绿体中进行的瞬时淀粉合成和在非光合组织造粉体中完成的贮藏淀粉合成,淀粉合成酶是淀粉生物合成途径中关键的酶[4]。淀粉合成酶(Starch Synthase,SS,EC 2.4.1.21 )是一个葡萄糖转移酶,主要负责延伸直链淀粉和直链淀粉的葡萄糖链,通过转移ADP葡萄糖的糖基到α-1,4葡萄糖的非还原性末端来延长α-1,4葡萄糖多聚体[5],多聚体又作为淀粉分支酶的底物合成支链淀粉[6]。生物信息学是在生命科学的研究中形成的建立在分子生物学基础上的以计算机为工具对生物信息进行储存、检索和分析的新兴科学,它以数学、信息学、计算机科学为主要手段,对原始序列数据进行储存、管理、注释、加工,通过查询、搜索、比对分析,从而预测其分子的结构与功能及其两者间的相互作用关系[7]。
作者用生物信息学的方法对马铃薯、红薯等6种重要农作物的SS基因及相应的氨基酸序列进行比对分析,进而对其理化性质、二级结构、结构域等进行预测分析,以期为今后开展SS的深入研究和利用提供一定的理论依据。
1 材料与方法
1.1 实验材料
马铃薯 (Solanum tuberosum)、 红薯(Impomoea batatas)、 小麦 (Triticum aestivum)、 高粱(Sorghum bicolor)、 南瓜 (Cucurbita moschata) 、 水稻(Oryza sativa)SS mRNA序列和氨基酸序列来源于NCBI的基因数据库,网址见表1[8]。登录号分别为:X52417.1、U44126.1、D10657.1、U41446.1、JN828808.1和FJ750946.1。

表1 生物信息学分析数据库和软件的相关网址Table 1 Websites of bioinformatics database and software
1.2 实验方法
通过NCBI网站检索合适的DNA序列,在选择基因序列的时候选择完整的线性序列。利用CpG岛分析6种物种的甲基化位点;通过DNAstar程序中的EditSeq确定其完整编码框并预测蛋白质理化性质;通过ProtScale程序分析蛋白质的亲水性/疏水性;通过在线工具TMHMM 2.0 Server分析蛋白质跨膜结构域;通过PSIPRED网站的在线分析功能完成蛋白质二级结构的预测;利用Smart分析物种的结构域;利用Blast完成核酸及氨基酸序列的同源性比对;利用Mega 6.0构建分子进化树。
2 结果与分析
2.1 6种植物SS甲基化位点分析
DNA甲基化是最早发现的表观遗传修饰途径之一,是指在DNA甲基化转移酶的作用下,将S-腺苷甲硫氨酸提供的甲基基团共价到CpG二核苷酸的胞嘧啶5’碳位上的过程[9]。基因启动子及其附近区域内CpG甲基化是众多基因实现去表达 (沉默)和基因印记的重要途径,通过测定启动子CpG岛甲基化状态了解基因是否去表达,为研究基因表达提供了DNA水平进行的途径[10]。DNA相关区域的每个CpG位点有特异性的甲基化修饰,导致复杂的信息类型,形成特异性CpG甲基化谱[11]。CpG岛的甲基化是表观遗传中基因表达调控的重要机制。通过CpG岛分析结果发现[12],红薯、小麦、高粱和水稻都含有甲基化位点,马铃薯和南瓜不含甲基化位点。红薯含有3个甲基化位点,小麦含有701个甲基化位点,高粱含有154个甲基化位点,水稻含有1 799个甲基化位点。
2.2 SS一级结构分析
2.2.1 核苷酸及其对应的氨基酸序列的组成成分和理化性质分析用 DNAstar,ORF Finder和ProtParam分析马铃薯、红薯、小麦、高粱、南瓜、水稻6种植物相关基因序列,对基因的核苷酸及其对应的氨基酸序列的组成成分和理化性质进行分析,结果见表2。淀粉合成酶核苷酸序列的全长平均为1 847 bp,开放阅读框的长度约为877 bp,起始密码子为ATG,终止密码子有TAA、TGA、GAC。开放阅读框所编码的氨基酸残基平均数286;平均相对分子质量为31 915.88;平均等电点为 8.264 5;pH为7的中性溶液中平均带电荷为-0.598 1;平均亲水氨基酸 63个,平均疏水氨基酸 102个,预测SS为疏水性蛋白。
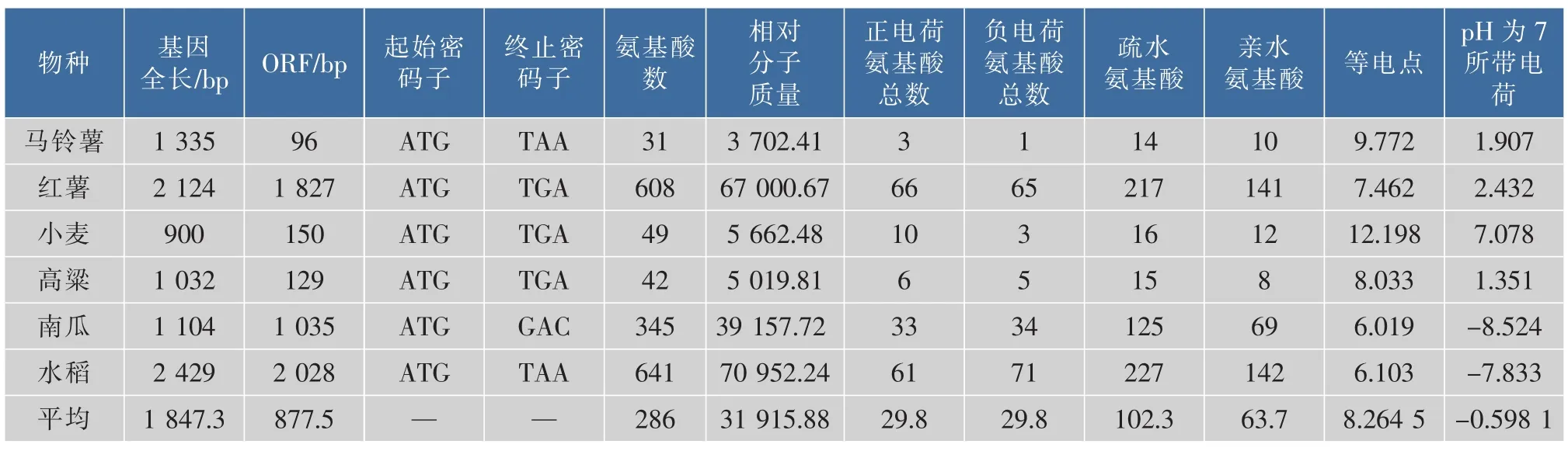
表2 不同植物中SS组成成分及理化性质分析Table 2 Composition analysis and physicochemical characteristics of SS in different plants
2.2.2 SS氨基酸疏水性/亲水性分析蛋白质疏水性/亲水性的组成是蛋白质折叠的主要驱动力。蛋白质折叠会形成亲水内核和亲水表面,同时在潜在跨膜区形成高疏水值区域,据此可以推测跨膜二级螺旋等二级结构和蛋白质表面氨基酸分布。分析正值越大表示越疏水,负值越大表示越亲水,而介于-0.5~+0.5之间的主要为两性氨基酸[13]。用ProtScale对马铃薯等6种植物淀粉合成酶氨基酸序列进行疏水性/亲水性预测,结果见图1。预测结果表明,马铃薯SS多肽链中的氨基酸(异亮氨酸,ILE)具有最高的分值(2.111)位于第130、131位,表明该位点的氨基酸疏水性最强,而氨基酸(精氨酸,Arg)的分值最低(-0.667)位于第 1145、1146位,表明该位点的氨基酸亲水性最强。而就整体来看,疏水性氨基酸多于亲水性氨基酸。因此,整个多肽链表现为疏水性,可认为马铃薯SS是疏水性蛋白。另外对其它5种物种的SS氨基酸序列进行疏水性/亲水性预测,结果表明其均为疏水性蛋白。SS整条链表现为疏水性,这与前面的预测结果一致,即表明SS为疏水性蛋白。
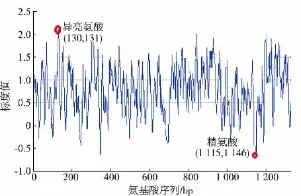
图1 马铃薯SS疏水性/亲水性预测分析Fig.1 Predictive analysis of hydrophobic/hydrophili of Ipomoea batatas SS
2.2.3 跨膜结构域的预测和分析跨膜结构域是膜内在蛋白和膜脂相结合的主要部位,它可能作为膜受体起作用,也可能定位于膜的锚定蛋白或者离子通道蛋白等[14]。因而,预测和分析跨膜结构域对于了解蛋白质的结构和功能以及在细胞中的作用部位具有十分重要的意义。使用TMHMM 2.0 Server对马铃薯等6种植物的SS氨基酸序列的跨膜结构域进行预测和分析,判定其跨膜结构域,红薯和水稻的预测结果见图2。红薯和水稻这两种SS序列都含有结构域,马铃薯、小麦、高粱、南瓜4种均没有结构域。
2.3 淀粉合成酶二级结构分析
2.3.1 蛋白质二级结构元件组成蛋白质的二级结构指它的多肽主链中有规则重复的构象,蛋白质分子的多肽链通常折叠和盘曲成比较稳定的空间结构,以形成特有的生物学活性和理化性质[15]。因此,预测和分析蛋白质的二级结构对了解其功能和空间结构有重要意义。蛋白质的二级结构预测是蛋白质结构组学研究的重点问题之一[16]。
目前预测二级结构的方法有很多,但是准确率却不是很理想。目前的预测方法中,PSlPRED方法的预测结果比较理想,因此采用此方案。常见的二级结构元件主要有α-螺旋、β-转角、β-折叠片、无规则卷曲和延伸链等[17]。表3为PSlPRED法预测6种植物SS氨基酸的二级结构原件比例,结果显示其二级结构以无规则卷曲和延伸链为主要构件。图3高粱SS氨基酸的二级结构为:α-螺旋含有22个,分别在53~64、97~106等区域其所占蛋白质比例为13.33%。 β-折叠片含有 29 个,分别在 5~15、19~23、86~90、116~121、161~162 等区域,所占蛋白质比例17.58%。无规则卷曲和延伸链含有114个,分别在1 ~4、16 ~18、24 ~52、65 ~85、91 ~96、107 ~115、122 ~160、163~165等区域,所占蛋白质比例为69.09%。
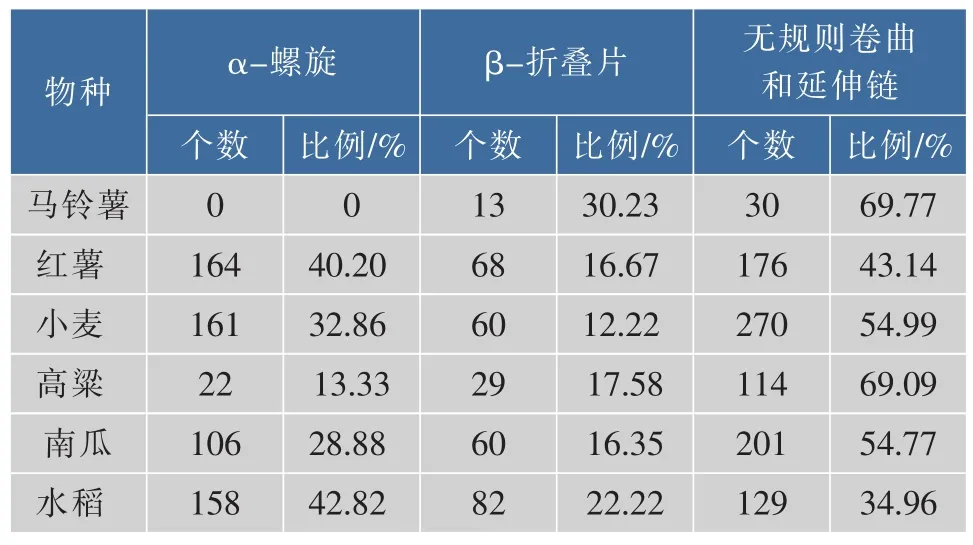
表3 SS蛋白二级结构分析Table 3 Analysis of SS protein secondary stucture
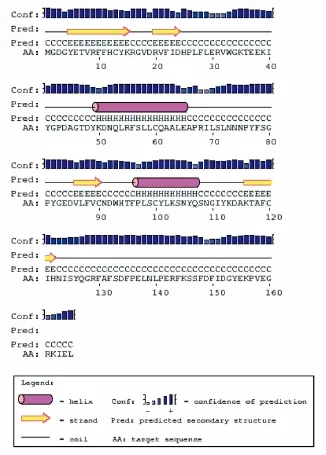
图3 高粱SS二级结构预测Fig.3 Prediction of secondary structure of Sorghum Bicolor SS
2.3.2 结构域的分析结构域是一种相对独立的区域性的结构,是介于二级和三级结构之间的另一种层次结构,是蛋白质亚基中的紧密球状结构区域,在蛋白质中起着独立的结构单位、功能单位与折叠单位的功能。一个蛋白质可以包含一个结构域也可以由几个结构域构成功能单位,通过Smart对6种植物的SS结构域进行预测。对红薯和水稻SS结构域的预测结果见图4,其中红色小方块表示低复杂度区域。红薯SS预测结果表明,其低度复杂区位于98~115处,长度为17 bp。对水稻SS结构域的预测结果表明其含有两个低复杂度区域,其低复杂度区域位于2~14处和99~108处,总长度为21 bp。其余4种植物SS均不含有结构域。
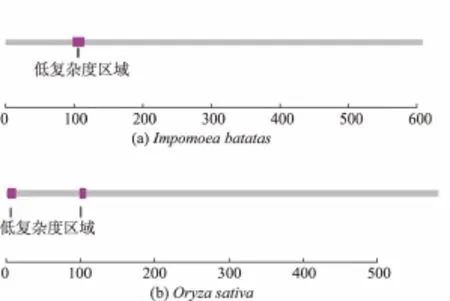
图4 红薯和水稻SS的结构域预测Fig.4 Prediction of structural domain of SS in Impomoea batata and Oryza sativa
2.4 核酸及蛋白质序列的比对分析
序列的相似性与序列的同源性有一定关系,一般来说,序列间的相似性越高,它们是同源序列的可能性就越高,所以通常通过序列的相似性推测序列是否同源。用Blast程序分别对6种物种的SS核酸(Blastn)及蛋白质序列(Blastp)进行同源性比对,结果见表4。表4数据显示,南瓜与高粱的核酸序列同源度分别为74%,其他物种均无同源序列。
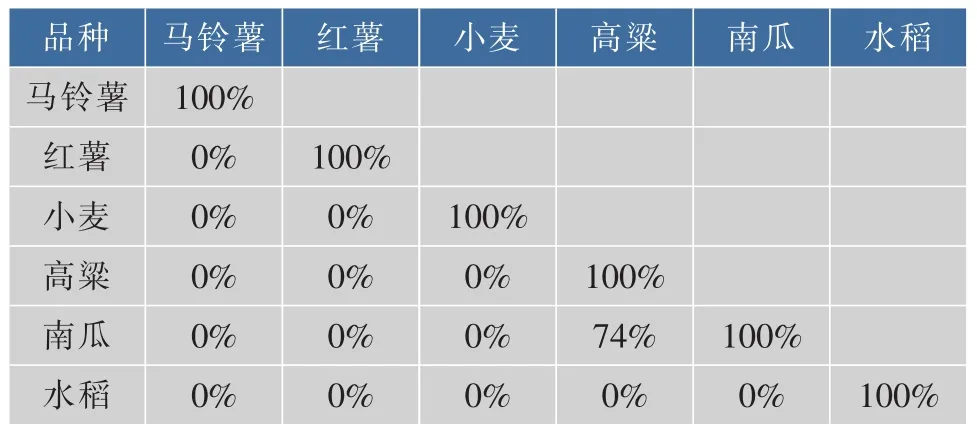
表4 6种植物间核酸序列的相关性比对Table 4 Correlation of nucleotides sequences between six different plants
Blastp比对5种植物蛋白质序列的同源性,结果见表5。水稻与小麦、高粱的同源度分别为86%、87%。南瓜与高粱的同源度分别为82%,高粱与小麦的同源度为64%。

表5 5种植物间蛋白质序列的相关性比对Table 5 Correlation between protein sequences of five different plants
Blastn与Blastp的结果对比发现,核酸的同源度远远低于蛋白质的同源度,这是因为密码子的简并性即由一种以上的密码子编码同一个氨基酸的现象,对应于同一氨基酸的密码子又称为同义密码子[18]。Blastp比对结果显示:红薯与欧洲山杨(Populustremula,CAI77773.1)、 抗 寒 性 烟 草(Nicotiana sylvestris,XP009777240.1)、 柑橘(Citrus clementinal,XP 006441435.1)、 葡 萄 (Vitis vinifera,CAN63617.1)、 可 可 (Theobromacacao,XP 0070029360.1)的同源度分别为 37%、33%、36%、32%、32%。 小 麦 与 高 粱 (Sorghum bicolor,KXG39419.1)、 节 节 麦 (Aeqilopstauschii,EMT23342.1)、 大 麦 (Hordeumvulqare,AAA32972.1)、 籼 稻 (Oryza sativaindica group,EEC82681.1)、小米(Setaria italica,XP004958680.1)同源度分别为 64%、92%、71%、64%、87%。高粱与玉米 (Zea mays,NP 001149266.1)、 籼 稻 (Oryza sativaindica group,EAZ09149.1)、 小 麦 (Triticum monococcum,AHJ14569.1)、大叶藻(Zostera marina,KMZ64620.1)、葡 萄 (Vitis vinifera,CAN76946.1)的同源度分别 89%、85%、71%、88%、57%。南瓜与西瓜 (Citrullus lanatus,AEV46188.1)、 草莓(Fragaria ananassa,AAT40976.1)、 山 荆 子 (Malus baccata,AED99204.1)、刺梨(Rosa roxburghii,AAT28434.1)、山葡萄(Vitis amurensis,AAR08831.1)的同源度分别为 83%、46%、45%、48%、45%。 水稻与小米(Setaria italica,XP 004977081.1)、 玉 米 (Zea mays,NP 001105759.1)、 小 麦 (Triticumaestivum,CBG91898.1) 、 高 粱 (Sorghumbicolor,XP 002455594.1)、 毛 果 杨 (Populus trichocarpa,XP 002320314.1)的同源度分别为 90%、86%、86%、87%、77%。
2.5 进化树构建与分析
用MEGA 6.0[19]软件分析淀粉合成酶的6个物种之间的亲缘关系并构建进化树,结果见图5。结果显示,参与分析的6个物种在进化树上呈现出一个大的分支:小麦和高梁SS进化程度相似,和水稻进化程度接近,和南瓜的进化相距较远。其中南瓜和马铃薯、红薯进化程度相近。6个物种之间呈现明显的亲缘关系。

图5 淀粉合成酶进化树分析Fig.5 Phylogenetic tree analysis of SS
3 结语
红薯、小麦、高粱和水稻都含有甲基化位点,马铃薯和南瓜不含甲基化位点。红薯含有3个甲基化位点,小麦含有701个甲基化位点,高粱含有154个甲基化位点,水稻含有1 799个甲基化位点。
淀粉合成酶核苷酸序列的全长平均为1 847 bp,开放阅读框的长度约为877 bp,起始密码子为ATG,终止密码子有TAA、TGA、GAC。开放阅读框所编码的氨基酸残基平均数286,平均相对分子质量为31 915.88;平均等电点为 8.264 5,pH为7的中性溶液中平均带电荷为-0.598 1。平均亲水氨基酸63个,平均疏水氨基酸 102个,预测SS为疏水性蛋白。
淀粉合成酶为疏水性蛋白,小麦和水稻分别存在一个跨膜结构。6种植物淀粉合成酶的二级结构表明,其二级结构以无规则卷曲和延伸链为主要构件。Blastn比对6种物种的同源性,结果表明,南瓜与高粱的同源度分别为74%,其他物种均无同源序列。Blastp比对5种植物蛋白质的同源性表明,水稻与小麦、高粱的同源度分别为86%、87%。南瓜与高粱的同源度分别为82%,高粱与小麦的同源度为64%。通过进化树分析淀粉合成酶显示,小麦和高梁进化程度相似,和南瓜的进化相距较远。这可能与淀粉合成酶在不同物种上进化程度有关。
